KR20170040249A - 항-cdh6 항체 약물 접합체 - Google Patents
항-cdh6 항체 약물 접합체 Download PDFInfo
- Publication number
- KR20170040249A KR20170040249A KR1020177003507A KR20177003507A KR20170040249A KR 20170040249 A KR20170040249 A KR 20170040249A KR 1020177003507 A KR1020177003507 A KR 1020177003507A KR 20177003507 A KR20177003507 A KR 20177003507A KR 20170040249 A KR20170040249 A KR 20170040249A
- Authority
- KR
- South Korea
- Prior art keywords
- seq
- variable region
- chain variable
- light chain
- heavy chain
- Prior art date
Links
- 229940049595 antibody-drug conjugate Drugs 0.000 title claims abstract description 191
- 239000000611 antibody drug conjugate Substances 0.000 title claims abstract description 152
- 206010028980 Neoplasm Diseases 0.000 claims abstract description 95
- 102000008394 Immunoglobulin Fragments Human genes 0.000 claims abstract description 66
- 108010021625 Immunoglobulin Fragments Proteins 0.000 claims abstract description 66
- 201000011510 cancer Diseases 0.000 claims abstract description 56
- 238000011282 treatment Methods 0.000 claims abstract description 26
- 210000004027 cell Anatomy 0.000 claims description 270
- 238000009739 binding Methods 0.000 claims description 229
- 230000027455 binding Effects 0.000 claims description 228
- 239000000427 antigen Substances 0.000 claims description 172
- 108091007433 antigens Proteins 0.000 claims description 170
- 102000036639 antigens Human genes 0.000 claims description 170
- 239000003814 drug Substances 0.000 claims description 153
- 239000012634 fragment Substances 0.000 claims description 153
- 238000000034 method Methods 0.000 claims description 106
- 229940079593 drug Drugs 0.000 claims description 103
- 239000000203 mixture Substances 0.000 claims description 73
- 241000282414 Homo sapiens Species 0.000 claims description 61
- 150000001413 amino acids Chemical group 0.000 claims description 61
- 239000003112 inhibitor Substances 0.000 claims description 59
- -1 apoptosis promoters Substances 0.000 claims description 56
- 239000000562 conjugate Substances 0.000 claims description 50
- 150000007523 nucleic acids Chemical class 0.000 claims description 47
- 229940124597 therapeutic agent Drugs 0.000 claims description 43
- JSHOVKSMJRQOGY-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 4-(pyridin-2-yldisulfanyl)butanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCCSSC1=CC=CC=N1 JSHOVKSMJRQOGY-UHFFFAOYSA-N 0.000 claims description 37
- 102000039446 nucleic acids Human genes 0.000 claims description 32
- 108020004707 nucleic acids Proteins 0.000 claims description 32
- 239000003153 chemical reaction reagent Substances 0.000 claims description 31
- 108020004414 DNA Proteins 0.000 claims description 29
- 206010033128 Ovarian cancer Diseases 0.000 claims description 27
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 26
- 239000008194 pharmaceutical composition Substances 0.000 claims description 23
- 239000013598 vector Substances 0.000 claims description 23
- 108010056045 K cadherin Proteins 0.000 claims description 22
- 229940123711 Bcl2 inhibitor Drugs 0.000 claims description 21
- 239000003795 chemical substances by application Substances 0.000 claims description 18
- 229940122035 Bcl-XL inhibitor Drugs 0.000 claims description 16
- 239000012664 BCL-2-inhibitor Substances 0.000 claims description 13
- 230000002285 radioactive effect Effects 0.000 claims description 11
- VRDGQQTWSGDXCU-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 2-iodoacetate Chemical compound ICC(=O)ON1C(=O)CCC1=O VRDGQQTWSGDXCU-UHFFFAOYSA-N 0.000 claims description 10
- BQWBEDSJTMWJAE-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 4-[(2-iodoacetyl)amino]benzoate Chemical compound C1=CC(NC(=O)CI)=CC=C1C(=O)ON1C(=O)CCC1=O BQWBEDSJTMWJAE-UHFFFAOYSA-N 0.000 claims description 10
- 239000002829 mitogen activated protein kinase inhibitor Substances 0.000 claims description 10
- FUHCFUVCWLZEDQ-UHFFFAOYSA-N 1-(2,5-dioxopyrrolidin-1-yl)oxy-1-oxo-4-(pyridin-2-yldisulfanyl)butane-2-sulfonic acid Chemical compound O=C1CCC(=O)N1OC(=O)C(S(=O)(=O)O)CCSSC1=CC=CC=N1 FUHCFUVCWLZEDQ-UHFFFAOYSA-N 0.000 claims description 9
- 102100021569 Apoptosis regulator Bcl-2 Human genes 0.000 claims description 9
- 108091012583 BCL2 Proteins 0.000 claims description 9
- 229940124647 MEK inhibitor Drugs 0.000 claims description 8
- 208000006990 cholangiocarcinoma Diseases 0.000 claims description 8
- 230000002519 immonomodulatory effect Effects 0.000 claims description 8
- GTBCXYYVWHFQRS-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 4-(pyridin-2-yldisulfanyl)pentanoate Chemical compound C=1C=CC=NC=1SSC(C)CCC(=O)ON1C(=O)CCC1=O GTBCXYYVWHFQRS-UHFFFAOYSA-N 0.000 claims description 7
- 230000006907 apoptotic process Effects 0.000 claims description 7
- 150000003839 salts Chemical class 0.000 claims description 7
- 102000029749 Microtubule Human genes 0.000 claims description 6
- 108091022875 Microtubule Proteins 0.000 claims description 6
- 206010038389 Renal cancer Diseases 0.000 claims description 6
- 208000024770 Thyroid neoplasm Diseases 0.000 claims description 6
- 238000004113 cell culture Methods 0.000 claims description 6
- 230000008878 coupling Effects 0.000 claims description 6
- 238000010168 coupling process Methods 0.000 claims description 6
- 238000005859 coupling reaction Methods 0.000 claims description 6
- 229940124302 mTOR inhibitor Drugs 0.000 claims description 6
- 239000003628 mammalian target of rapamycin inhibitor Substances 0.000 claims description 6
- 210000004688 microtubule Anatomy 0.000 claims description 6
- 201000002510 thyroid cancer Diseases 0.000 claims description 6
- 239000012624 DNA alkylating agent Substances 0.000 claims description 5
- 208000008839 Kidney Neoplasms Diseases 0.000 claims description 5
- 101710181812 Methionine aminopeptidase Proteins 0.000 claims description 5
- 208000032383 Soft tissue cancer Diseases 0.000 claims description 5
- 201000007455 central nervous system cancer Diseases 0.000 claims description 5
- 201000010982 kidney cancer Diseases 0.000 claims description 5
- 201000007270 liver cancer Diseases 0.000 claims description 5
- 208000014018 liver neoplasm Diseases 0.000 claims description 5
- 239000003381 stabilizer Substances 0.000 claims description 5
- 101100004408 Arabidopsis thaliana BIG gene Proteins 0.000 claims description 4
- 102100024457 Cyclin-dependent kinase 9 Human genes 0.000 claims description 4
- 239000012623 DNA damaging agent Substances 0.000 claims description 4
- 102100024746 Dihydrofolate reductase Human genes 0.000 claims description 4
- 102100025012 Dipeptidyl peptidase 4 Human genes 0.000 claims description 4
- 101100485279 Drosophila melanogaster emb gene Proteins 0.000 claims description 4
- 102100029095 Exportin-1 Human genes 0.000 claims description 4
- 101000980930 Homo sapiens Cyclin-dependent kinase 9 Proteins 0.000 claims description 4
- 101001056180 Homo sapiens Induced myeloid leukemia cell differentiation protein Mcl-1 Proteins 0.000 claims description 4
- 101000684208 Homo sapiens Prolyl endopeptidase FAP Proteins 0.000 claims description 4
- 102100026539 Induced myeloid leukemia cell differentiation protein Mcl-1 Human genes 0.000 claims description 4
- PEEHTFAAVSWFBL-UHFFFAOYSA-N Maleimide Chemical compound O=C1NC(=O)C=C1 PEEHTFAAVSWFBL-UHFFFAOYSA-N 0.000 claims description 4
- OFOBLEOULBTSOW-UHFFFAOYSA-N Malonic acid Chemical compound OC(=O)CC(O)=O OFOBLEOULBTSOW-UHFFFAOYSA-N 0.000 claims description 4
- 229940079156 Proteasome inhibitor Drugs 0.000 claims description 4
- 229940123573 Protein synthesis inhibitor Drugs 0.000 claims description 4
- 101100485284 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) CRM1 gene Proteins 0.000 claims description 4
- 101150094313 XPO1 gene Proteins 0.000 claims description 4
- 239000000362 adenosine triphosphatase inhibitor Substances 0.000 claims description 4
- 108010044540 auristatin Proteins 0.000 claims description 4
- 108020001096 dihydrofolate reductase Proteins 0.000 claims description 4
- 108700002148 exportin 1 Proteins 0.000 claims description 4
- 239000003481 heat shock protein 90 inhibitor Substances 0.000 claims description 4
- 239000003276 histone deacetylase inhibitor Substances 0.000 claims description 4
- 229940043355 kinase inhibitor Drugs 0.000 claims description 4
- 229910021645 metal ion Inorganic materials 0.000 claims description 4
- 210000003470 mitochondria Anatomy 0.000 claims description 4
- 239000003757 phosphotransferase inhibitor Substances 0.000 claims description 4
- 239000003207 proteasome inhibitor Substances 0.000 claims description 4
- 239000000007 protein synthesis inhibitor Substances 0.000 claims description 4
- 239000012625 DNA intercalator Substances 0.000 claims description 3
- 102000003867 Phospholipid Transfer Proteins Human genes 0.000 claims description 3
- 108090000216 Phospholipid Transfer Proteins Proteins 0.000 claims description 3
- 230000001268 conjugating effect Effects 0.000 claims description 3
- 230000000368 destabilizing effect Effects 0.000 claims description 3
- AMRJKAQTDDKMCE-UHFFFAOYSA-N dolastatin Chemical compound CC(C)C(N(C)C)C(=O)NC(C(C)C)C(=O)N(C)C(C(C)C)C(OC)CC(=O)N1CCCC1C(OC)C(C)C(=O)NC(C=1SC=CN=1)CC1=CC=CC=C1 AMRJKAQTDDKMCE-UHFFFAOYSA-N 0.000 claims description 3
- 229930188854 dolastatin Natural products 0.000 claims description 3
- 239000003937 drug carrier Substances 0.000 claims description 3
- 238000006276 transfer reaction Methods 0.000 claims description 3
- 102000000578 Cyclin-Dependent Kinase Inhibitor p21 Human genes 0.000 claims description 2
- 108010016788 Cyclin-Dependent Kinase Inhibitor p21 Proteins 0.000 claims description 2
- 229940126161 DNA alkylating agent Drugs 0.000 claims description 2
- 229940124179 Kinesin inhibitor Drugs 0.000 claims description 2
- 238000012258 culturing Methods 0.000 claims description 2
- 229940121372 histone deacetylase inhibitor Drugs 0.000 claims description 2
- 239000012216 imaging agent Substances 0.000 claims description 2
- 102100027670 Islet amyloid polypeptide Human genes 0.000 claims 5
- 102100029756 Cadherin-6 Human genes 0.000 claims 3
- 101000794604 Homo sapiens Cadherin-6 Proteins 0.000 claims 3
- NZNMSOFKMUBTKW-UHFFFAOYSA-M cyclohexanecarboxylate Chemical compound [O-]C(=O)C1CCCCC1 NZNMSOFKMUBTKW-UHFFFAOYSA-M 0.000 claims 1
- 125000005647 linker group Chemical group 0.000 description 92
- 108090000623 proteins and genes Proteins 0.000 description 85
- 206010057190 Respiratory tract infections Diseases 0.000 description 75
- 230000014509 gene expression Effects 0.000 description 75
- 102000004169 proteins and genes Human genes 0.000 description 69
- 235000018102 proteins Nutrition 0.000 description 65
- 125000003275 alpha amino acid group Chemical group 0.000 description 56
- 235000001014 amino acid Nutrition 0.000 description 56
- 229940024606 amino acid Drugs 0.000 description 55
- 108090000765 processed proteins & peptides Proteins 0.000 description 50
- 108010047041 Complementarity Determining Regions Proteins 0.000 description 48
- 229910052805 deuterium Inorganic materials 0.000 description 47
- 230000000694 effects Effects 0.000 description 45
- YZCKVEUIGOORGS-OUBTZVSYSA-N Deuterium Chemical compound [2H] YZCKVEUIGOORGS-OUBTZVSYSA-N 0.000 description 43
- 229940127121 immunoconjugate Drugs 0.000 description 41
- 239000000872 buffer Substances 0.000 description 39
- 102000004196 processed proteins & peptides Human genes 0.000 description 39
- 239000000243 solution Substances 0.000 description 37
- 238000000746 purification Methods 0.000 description 36
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 34
- 239000003431 cross linking reagent Substances 0.000 description 33
- 238000004091 panning Methods 0.000 description 33
- 239000008055 phosphate buffer solution Substances 0.000 description 33
- 238000006243 chemical reaction Methods 0.000 description 32
- 239000002609 medium Substances 0.000 description 32
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 31
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 31
- 229920001184 polypeptide Polymers 0.000 description 30
- 230000008569 process Effects 0.000 description 27
- 238000012360 testing method Methods 0.000 description 26
- FPKVOQKZMBDBKP-UHFFFAOYSA-N 1-[4-[(2,5-dioxopyrrol-1-yl)methyl]cyclohexanecarbonyl]oxy-2,5-dioxopyrrolidine-3-sulfonic acid Chemical compound O=C1C(S(=O)(=O)O)CC(=O)N1OC(=O)C1CCC(CN2C(C=CC2=O)=O)CC1 FPKVOQKZMBDBKP-UHFFFAOYSA-N 0.000 description 25
- 108091033319 polynucleotide Proteins 0.000 description 25
- 102000040430 polynucleotide Human genes 0.000 description 25
- 239000002157 polynucleotide Substances 0.000 description 25
- 239000000523 sample Substances 0.000 description 24
- 239000011347 resin Substances 0.000 description 23
- 229920005989 resin Polymers 0.000 description 23
- DHMQDGOQFOQNFH-UHFFFAOYSA-N Glycine Chemical compound NCC(O)=O DHMQDGOQFOQNFH-UHFFFAOYSA-N 0.000 description 21
- 238000012216 screening Methods 0.000 description 21
- 238000002965 ELISA Methods 0.000 description 19
- 150000001875 compounds Chemical class 0.000 description 19
- 239000013604 expression vector Substances 0.000 description 19
- 238000010791 quenching Methods 0.000 description 19
- 210000001519 tissue Anatomy 0.000 description 19
- 238000003556 assay Methods 0.000 description 18
- 239000013078 crystal Substances 0.000 description 18
- 238000002474 experimental method Methods 0.000 description 18
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 17
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 17
- 238000002347 injection Methods 0.000 description 17
- 239000007924 injection Substances 0.000 description 17
- 239000011780 sodium chloride Substances 0.000 description 17
- 238000002560 therapeutic procedure Methods 0.000 description 17
- 241000699666 Mus <mouse, genus> Species 0.000 description 16
- 241000700159 Rattus Species 0.000 description 16
- 208000035475 disorder Diseases 0.000 description 16
- 230000000069 prophylactic effect Effects 0.000 description 16
- 230000000171 quenching effect Effects 0.000 description 16
- 239000000126 substance Substances 0.000 description 16
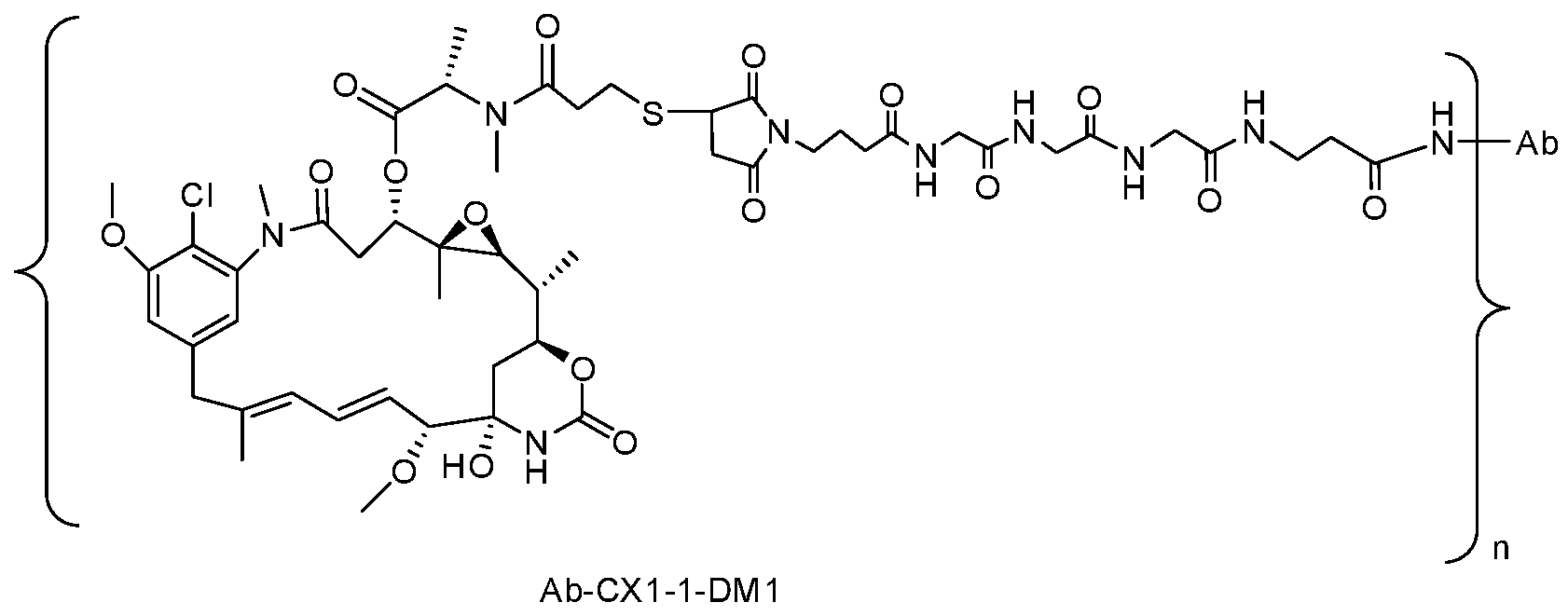
- IADUWZMNTKHTIN-MLSWMBHTSA-N (2,5-dioxopyrrolidin-1-yl) 4-[[3-[3-[[(2S)-1-[[(1S,2R,3S,5S,6S,16E,18E,20R,21S)-11-chloro-21-hydroxy-12,20-dimethoxy-2,5,9,16-tetramethyl-8,23-dioxo-4,24-dioxa-9,22-diazatetracyclo[19.3.1.110,14.03,5]hexacosa-10,12,14(26),16,18-pentaen-6-yl]oxy]-1-oxopropan-2-yl]-methylamino]-3-oxopropyl]sulfanyl-2,5-dioxopyrrolidin-1-yl]methyl]cyclohexane-1-carboxylate Chemical compound CO[C@@H]1\C=C\C=C(C)\Cc2cc(OC)c(Cl)c(c2)N(C)C(=O)C[C@H](OC(=O)[C@H](C)N(C)C(=O)CCSC2CC(=O)N(CC3CCC(CC3)C(=O)ON3C(=O)CCC3=O)C2=O)[C@]2(C)O[C@H]2[C@H](C)[C@@H]2C[C@@]1(O)NC(=O)O2 IADUWZMNTKHTIN-MLSWMBHTSA-N 0.000 description 15
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 15
- 241000699670 Mus sp. Species 0.000 description 15
- 229940127089 cytotoxic agent Drugs 0.000 description 15
- 201000010099 disease Diseases 0.000 description 15
- 238000000338 in vitro Methods 0.000 description 15
- 230000003993 interaction Effects 0.000 description 15
- 125000003729 nucleotide group Chemical group 0.000 description 15
- 125000000539 amino acid group Chemical group 0.000 description 14
- 230000001413 cellular effect Effects 0.000 description 14
- 231100000599 cytotoxic agent Toxicity 0.000 description 14
- 239000002773 nucleotide Substances 0.000 description 14
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 13
- 229960005091 chloramphenicol Drugs 0.000 description 13
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 13
- 230000013595 glycosylation Effects 0.000 description 13
- 238000006206 glycosylation reaction Methods 0.000 description 13
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 12
- 238000013459 approach Methods 0.000 description 12
- 239000011230 binding agent Substances 0.000 description 12
- 238000004422 calculation algorithm Methods 0.000 description 12
- 238000003776 cleavage reaction Methods 0.000 description 12
- RAXXELZNTBOGNW-UHFFFAOYSA-N imidazole Natural products C1=CNC=N1 RAXXELZNTBOGNW-UHFFFAOYSA-N 0.000 description 12
- 238000001727 in vivo Methods 0.000 description 12
- 230000009467 reduction Effects 0.000 description 12
- 230000007017 scission Effects 0.000 description 12
- 230000001225 therapeutic effect Effects 0.000 description 12
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Chemical compound O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 12
- 238000012447 xenograft mouse model Methods 0.000 description 12
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 11
- 108020004705 Codon Proteins 0.000 description 11
- 102100024319 Intestinal-type alkaline phosphatase Human genes 0.000 description 11
- 229920001213 Polysorbate 20 Polymers 0.000 description 11
- 239000002246 antineoplastic agent Substances 0.000 description 11
- 238000002648 combination therapy Methods 0.000 description 11
- 238000010494 dissociation reaction Methods 0.000 description 11
- 230000005593 dissociations Effects 0.000 description 11
- 239000006166 lysate Substances 0.000 description 11
- 125000002496 methyl group Chemical group [H]C([H])([H])* 0.000 description 11
- 230000035772 mutation Effects 0.000 description 11
- 239000013612 plasmid Substances 0.000 description 11
- 239000000256 polyoxyethylene sorbitan monolaurate Substances 0.000 description 11
- 235000010486 polyoxyethylene sorbitan monolaurate Nutrition 0.000 description 11
- 238000005406 washing Methods 0.000 description 11
- 108091028043 Nucleic acid sequence Proteins 0.000 description 10
- 239000007983 Tris buffer Substances 0.000 description 10
- 230000001580 bacterial effect Effects 0.000 description 10
- 239000011324 bead Substances 0.000 description 10
- 238000009295 crossflow filtration Methods 0.000 description 10
- 229910052739 hydrogen Inorganic materials 0.000 description 10
- 238000004519 manufacturing process Methods 0.000 description 10
- 239000011159 matrix material Substances 0.000 description 10
- 238000001890 transfection Methods 0.000 description 10
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 9
- 102000035195 Peptidases Human genes 0.000 description 9
- 108091005804 Peptidases Proteins 0.000 description 9
- 229920002684 Sepharose Polymers 0.000 description 9
- 239000006180 TBST buffer Substances 0.000 description 9
- 239000004480 active ingredient Substances 0.000 description 9
- 230000000259 anti-tumor effect Effects 0.000 description 9
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 description 9
- 230000037396 body weight Effects 0.000 description 9
- 239000002254 cytotoxic agent Substances 0.000 description 9
- 230000001687 destabilization Effects 0.000 description 9
- 238000005516 engineering process Methods 0.000 description 9
- 238000009472 formulation Methods 0.000 description 9
- 230000012010 growth Effects 0.000 description 9
- 239000001257 hydrogen Substances 0.000 description 9
- 230000001965 increasing effect Effects 0.000 description 9
- 239000000843 powder Substances 0.000 description 9
- 238000002360 preparation method Methods 0.000 description 9
- 241000894007 species Species 0.000 description 9
- 238000006467 substitution reaction Methods 0.000 description 9
- 239000006228 supernatant Substances 0.000 description 9
- FALRKNHUBBKYCC-UHFFFAOYSA-N 2-(chloromethyl)pyridine-3-carbonitrile Chemical compound ClCC1=NC=CC=C1C#N FALRKNHUBBKYCC-UHFFFAOYSA-N 0.000 description 8
- 101100112922 Candida albicans CDR3 gene Proteins 0.000 description 8
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 8
- RJKFOVLPORLFTN-LEKSSAKUSA-N Progesterone Chemical compound C1CC2=CC(=O)CC[C@]2(C)[C@@H]2[C@@H]1[C@@H]1CC[C@H](C(=O)C)[C@@]1(C)CC2 RJKFOVLPORLFTN-LEKSSAKUSA-N 0.000 description 8
- 238000004458 analytical method Methods 0.000 description 8
- 230000000295 complement effect Effects 0.000 description 8
- 238000001514 detection method Methods 0.000 description 8
- 108020001507 fusion proteins Proteins 0.000 description 8
- 102000037865 fusion proteins Human genes 0.000 description 8
- 239000008103 glucose Substances 0.000 description 8
- 238000013507 mapping Methods 0.000 description 8
- 239000000463 material Substances 0.000 description 8
- BASFCYQUMIYNBI-UHFFFAOYSA-N platinum Chemical compound [Pt] BASFCYQUMIYNBI-UHFFFAOYSA-N 0.000 description 8
- 229940014800 succinic anhydride Drugs 0.000 description 8
- 239000000725 suspension Substances 0.000 description 8
- 238000013268 sustained release Methods 0.000 description 8
- 239000012730 sustained-release form Substances 0.000 description 8
- 102100036364 Cadherin-2 Human genes 0.000 description 7
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 7
- 108090000371 Esterases Proteins 0.000 description 7
- OWXMKDGYPWMGEB-UHFFFAOYSA-N HEPPS Chemical compound OCCN1CCN(CCCS(O)(=O)=O)CC1 OWXMKDGYPWMGEB-UHFFFAOYSA-N 0.000 description 7
- 101000714537 Homo sapiens Cadherin-2 Proteins 0.000 description 7
- 241001465754 Metazoa Species 0.000 description 7
- 238000007792 addition Methods 0.000 description 7
- 238000005377 adsorption chromatography Methods 0.000 description 7
- 229910002091 carbon monoxide Inorganic materials 0.000 description 7
- 239000011248 coating agent Substances 0.000 description 7
- 238000000576 coating method Methods 0.000 description 7
- 239000002299 complementary DNA Substances 0.000 description 7
- 238000013270 controlled release Methods 0.000 description 7
- 239000002552 dosage form Substances 0.000 description 7
- 239000003623 enhancer Substances 0.000 description 7
- 150000002148 esters Chemical class 0.000 description 7
- 238000003018 immunoassay Methods 0.000 description 7
- 238000012423 maintenance Methods 0.000 description 7
- 230000004048 modification Effects 0.000 description 7
- 238000012986 modification Methods 0.000 description 7
- 239000002245 particle Substances 0.000 description 7
- 239000008188 pellet Substances 0.000 description 7
- 239000012071 phase Substances 0.000 description 7
- 229920000642 polymer Polymers 0.000 description 7
- 239000011550 stock solution Substances 0.000 description 7
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 6
- QTBSBXVTEAMEQO-UHFFFAOYSA-N Acetic acid Chemical compound CC(O)=O QTBSBXVTEAMEQO-UHFFFAOYSA-N 0.000 description 6
- 241000699802 Cricetulus griseus Species 0.000 description 6
- 239000004471 Glycine Substances 0.000 description 6
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 6
- 239000004472 Lysine Substances 0.000 description 6
- 239000004698 Polyethylene Substances 0.000 description 6
- 238000012300 Sequence Analysis Methods 0.000 description 6
- 229930006000 Sucrose Natural products 0.000 description 6
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 6
- 239000002253 acid Substances 0.000 description 6
- RJURFGZVJUQBHK-UHFFFAOYSA-N actinomycin D Natural products CC1OC(=O)C(C(C)C)N(C)C(=O)CN(C)C(=O)C2CCCN2C(=O)C(C(C)C)NC(=O)C1NC(=O)C1=C(N)C(=O)C(C)=C2OC(C(C)=CC=C3C(=O)NC4C(=O)NC(C(N5CCCC5C(=O)N(C)CC(=O)N(C)C(C(C)C)C(=O)OC4C)=O)C(C)C)=C3N=C21 RJURFGZVJUQBHK-UHFFFAOYSA-N 0.000 description 6
- 125000000217 alkyl group Chemical group 0.000 description 6
- 229960002685 biotin Drugs 0.000 description 6
- 235000020958 biotin Nutrition 0.000 description 6
- 239000011616 biotin Substances 0.000 description 6
- 210000004899 c-terminal region Anatomy 0.000 description 6
- 238000012512 characterization method Methods 0.000 description 6
- 238000002050 diffraction method Methods 0.000 description 6
- 239000001963 growth medium Substances 0.000 description 6
- 238000004949 mass spectrometry Methods 0.000 description 6
- 230000002611 ovarian Effects 0.000 description 6
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 6
- 235000019833 protease Nutrition 0.000 description 6
- 239000007787 solid Substances 0.000 description 6
- 239000002904 solvent Substances 0.000 description 6
- 239000005720 sucrose Substances 0.000 description 6
- 239000003053 toxin Substances 0.000 description 6
- 231100000765 toxin Toxicity 0.000 description 6
- 108700012359 toxins Proteins 0.000 description 6
- LENZDBCJOHFCAS-UHFFFAOYSA-N tris Chemical compound OCC(N)(CO)CO LENZDBCJOHFCAS-UHFFFAOYSA-N 0.000 description 6
- 241000283707 Capra Species 0.000 description 5
- DLGOEMSEDOSKAD-UHFFFAOYSA-N Carmustine Chemical compound ClCCNC(=O)N(N=O)CCCl DLGOEMSEDOSKAD-UHFFFAOYSA-N 0.000 description 5
- UHDGCWIWMRVCDJ-CCXZUQQUSA-N Cytarabine Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@@H](O)[C@H](O)[C@@H](CO)O1 UHDGCWIWMRVCDJ-CCXZUQQUSA-N 0.000 description 5
- 108090000695 Cytokines Proteins 0.000 description 5
- 102000004127 Cytokines Human genes 0.000 description 5
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 5
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 5
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 5
- KDXKERNSBIXSRK-YFKPBYRVSA-N L-lysine Chemical compound NCCCC[C@H](N)C(O)=O KDXKERNSBIXSRK-YFKPBYRVSA-N 0.000 description 5
- 239000002033 PVDF binder Substances 0.000 description 5
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 5
- 239000012505 Superdex™ Substances 0.000 description 5
- 238000010521 absorption reaction Methods 0.000 description 5
- 239000000443 aerosol Substances 0.000 description 5
- 239000000556 agonist Substances 0.000 description 5
- 210000003323 beak Anatomy 0.000 description 5
- 239000012472 biological sample Substances 0.000 description 5
- 230000000903 blocking effect Effects 0.000 description 5
- 238000005119 centrifugation Methods 0.000 description 5
- 238000010367 cloning Methods 0.000 description 5
- 230000021615 conjugation Effects 0.000 description 5
- 230000001186 cumulative effect Effects 0.000 description 5
- 239000002619 cytotoxin Substances 0.000 description 5
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 5
- 230000029087 digestion Effects 0.000 description 5
- 238000010790 dilution Methods 0.000 description 5
- 239000012895 dilution Substances 0.000 description 5
- 125000005179 haloacetyl group Chemical group 0.000 description 5
- 230000001939 inductive effect Effects 0.000 description 5
- 238000001802 infusion Methods 0.000 description 5
- 230000005764 inhibitory process Effects 0.000 description 5
- 238000001990 intravenous administration Methods 0.000 description 5
- 239000003446 ligand Substances 0.000 description 5
- 239000002502 liposome Substances 0.000 description 5
- 239000007788 liquid Substances 0.000 description 5
- 210000004962 mammalian cell Anatomy 0.000 description 5
- 238000005259 measurement Methods 0.000 description 5
- 239000012528 membrane Substances 0.000 description 5
- 210000004379 membrane Anatomy 0.000 description 5
- GLVAUDGFNGKCSF-UHFFFAOYSA-N mercaptopurine Chemical compound S=C1NC=NC2=C1NC=N2 GLVAUDGFNGKCSF-UHFFFAOYSA-N 0.000 description 5
- 239000000546 pharmaceutical excipient Substances 0.000 description 5
- 125000001997 phenyl group Chemical group [H]C1=C([H])C([H])=C(*)C([H])=C1[H] 0.000 description 5
- 229920002981 polyvinylidene fluoride Polymers 0.000 description 5
- 102000005962 receptors Human genes 0.000 description 5
- 108020003175 receptors Proteins 0.000 description 5
- 230000002829 reductive effect Effects 0.000 description 5
- 238000013207 serial dilution Methods 0.000 description 5
- 238000002741 site-directed mutagenesis Methods 0.000 description 5
- 230000004083 survival effect Effects 0.000 description 5
- 230000002195 synergetic effect Effects 0.000 description 5
- 239000003643 water by type Substances 0.000 description 5
- JWDFQMWEFLOOED-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 3-(pyridin-2-yldisulfanyl)propanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCSSC1=CC=CC=N1 JWDFQMWEFLOOED-UHFFFAOYSA-N 0.000 description 4
- 206010063836 Atrioventricular septal defect Diseases 0.000 description 4
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 4
- 108010092160 Dactinomycin Proteins 0.000 description 4
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 4
- 241000588724 Escherichia coli Species 0.000 description 4
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 4
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 4
- 239000007995 HEPES buffer Substances 0.000 description 4
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 4
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 4
- 239000002136 L01XE07 - Lapatinib Substances 0.000 description 4
- 241000124008 Mammalia Species 0.000 description 4
- 229930126263 Maytansine Natural products 0.000 description 4
- 101100438153 Mus musculus Cdh6 gene Proteins 0.000 description 4
- 102000057297 Pepsin A Human genes 0.000 description 4
- 108090000284 Pepsin A Proteins 0.000 description 4
- 101100438155 Rattus norvegicus Cdh6 gene Proteins 0.000 description 4
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 4
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 4
- 210000001015 abdomen Anatomy 0.000 description 4
- 239000013543 active substance Substances 0.000 description 4
- 235000004279 alanine Nutrition 0.000 description 4
- 125000004429 atom Chemical group 0.000 description 4
- 230000015572 biosynthetic process Effects 0.000 description 4
- 230000006287 biotinylation Effects 0.000 description 4
- 229930189065 blasticidin Natural products 0.000 description 4
- 210000004369 blood Anatomy 0.000 description 4
- 239000008280 blood Substances 0.000 description 4
- 239000006227 byproduct Substances 0.000 description 4
- 150000001720 carbohydrates Chemical class 0.000 description 4
- 229910052799 carbon Inorganic materials 0.000 description 4
- 239000006285 cell suspension Substances 0.000 description 4
- 239000012539 chromatography resin Substances 0.000 description 4
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 4
- 239000012228 culture supernatant Substances 0.000 description 4
- 238000011161 development Methods 0.000 description 4
- 230000018109 developmental process Effects 0.000 description 4
- 231100000673 dose–response relationship Toxicity 0.000 description 4
- 239000012636 effector Substances 0.000 description 4
- 238000001211 electron capture detection Methods 0.000 description 4
- GTTBEUCJPZQMDZ-UHFFFAOYSA-N erlotinib hydrochloride Chemical compound [H+].[Cl-].C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 GTTBEUCJPZQMDZ-UHFFFAOYSA-N 0.000 description 4
- 125000001495 ethyl group Chemical group [H]C([H])([H])C([H])([H])* 0.000 description 4
- 238000011156 evaluation Methods 0.000 description 4
- BRZYSWJRSDMWLG-CAXSIQPQSA-N geneticin Chemical compound O1C[C@@](O)(C)[C@H](NC)[C@@H](O)[C@H]1O[C@@H]1[C@@H](O)[C@H](O[C@@H]2[C@@H]([C@@H](O)[C@H](O)[C@@H](C(C)O)O2)N)[C@@H](N)C[C@H]1N BRZYSWJRSDMWLG-CAXSIQPQSA-N 0.000 description 4
- 238000010438 heat treatment Methods 0.000 description 4
- 210000004408 hybridoma Anatomy 0.000 description 4
- 238000011065 in-situ storage Methods 0.000 description 4
- 238000011534 incubation Methods 0.000 description 4
- BPHPUYQFMNQIOC-NXRLNHOXSA-N isopropyl beta-D-thiogalactopyranoside Chemical compound CC(C)S[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O BPHPUYQFMNQIOC-NXRLNHOXSA-N 0.000 description 4
- BCFGMOOMADDAQU-UHFFFAOYSA-N lapatinib Chemical compound O1C(CNCCS(=O)(=O)C)=CC=C1C1=CC=C(N=CN=C2NC=3C=C(Cl)C(OCC=4C=C(F)C=CC=4)=CC=3)C2=C1 BCFGMOOMADDAQU-UHFFFAOYSA-N 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 108010082117 matrigel Proteins 0.000 description 4
- WKPWGQKGSOKKOO-RSFHAFMBSA-N maytansine Chemical compound CO[C@@H]([C@@]1(O)C[C@](OC(=O)N1)([C@H]([C@@H]1O[C@@]1(C)[C@@H](OC(=O)[C@H](C)N(C)C(C)=O)CC(=O)N1C)C)[H])\C=C\C=C(C)\CC2=CC(OC)=C(Cl)C1=C2 WKPWGQKGSOKKOO-RSFHAFMBSA-N 0.000 description 4
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 4
- 229930182817 methionine Natural products 0.000 description 4
- 125000000325 methylidene group Chemical group [H]C([H])=* 0.000 description 4
- 229940111202 pepsin Drugs 0.000 description 4
- 229910052697 platinum Inorganic materials 0.000 description 4
- 229920001223 polyethylene glycol Polymers 0.000 description 4
- 239000011148 porous material Substances 0.000 description 4
- 229960003387 progesterone Drugs 0.000 description 4
- 239000000186 progesterone Substances 0.000 description 4
- 230000035755 proliferation Effects 0.000 description 4
- 238000000159 protein binding assay Methods 0.000 description 4
- 235000020183 skimmed milk Nutrition 0.000 description 4
- 239000007790 solid phase Substances 0.000 description 4
- 230000009870 specific binding Effects 0.000 description 4
- 238000011105 stabilization Methods 0.000 description 4
- 238000003756 stirring Methods 0.000 description 4
- 239000000758 substrate Substances 0.000 description 4
- 125000000472 sulfonyl group Chemical group *S(*)(=O)=O 0.000 description 4
- SUVMJBTUFCVSAD-UHFFFAOYSA-N sulforaphane Chemical compound CS(=O)CCCCN=C=S SUVMJBTUFCVSAD-UHFFFAOYSA-N 0.000 description 4
- 229910052717 sulfur Inorganic materials 0.000 description 4
- 208000024891 symptom Diseases 0.000 description 4
- HVXKQKFEHMGHSL-QKDCVEJESA-N tesevatinib Chemical compound N1=CN=C2C=C(OC[C@@H]3C[C@@H]4CN(C)C[C@@H]4C3)C(OC)=CC2=C1NC1=CC=C(Cl)C(Cl)=C1F HVXKQKFEHMGHSL-QKDCVEJESA-N 0.000 description 4
- WYWHKKSPHMUBEB-UHFFFAOYSA-N tioguanine Chemical compound N1C(N)=NC(=S)C2=C1N=CN2 WYWHKKSPHMUBEB-UHFFFAOYSA-N 0.000 description 4
- 230000009261 transgenic effect Effects 0.000 description 4
- 230000014616 translation Effects 0.000 description 4
- 238000002054 transplantation Methods 0.000 description 4
- 230000004614 tumor growth Effects 0.000 description 4
- JXLYSJRDGCGARV-CFWMRBGOSA-N vinblastine Chemical compound C([C@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-CFWMRBGOSA-N 0.000 description 4
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 4
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 4
- 229960004528 vincristine Drugs 0.000 description 4
- 238000002424 x-ray crystallography Methods 0.000 description 4
- QWPXBEHQFHACTK-KZVYIGENSA-N (10e,12e)-86-chloro-12,14,4-trihydroxy-85,14-dimethoxy-33,2,7,10-tetramethyl-15,16-dihydro-14h-7-aza-1(6,4)-oxazina-3(2,3)-oxirana-8(1,3)-benzenacyclotetradecaphane-10,12-dien-6-one Chemical compound CN1C(=O)CC(O)C2(C)OC2C(C)C(OC(=O)N2)CC2(O)C(OC)\C=C\C=C(C)\CC2=CC(OC)=C(Cl)C1=C2 QWPXBEHQFHACTK-KZVYIGENSA-N 0.000 description 3
- QDITZBLZQQZVEE-YBEGLDIGSA-N (5z)-5-[(4-pyridin-4-ylquinolin-6-yl)methylidene]-1,3-thiazolidine-2,4-dione Chemical compound S1C(=O)NC(=O)\C1=C\C1=CC=C(N=CC=C2C=3C=CN=CC=3)C2=C1 QDITZBLZQQZVEE-YBEGLDIGSA-N 0.000 description 3
- IAYGCINLNONXHY-LBPRGKRZSA-N 3-(carbamoylamino)-5-(3-fluorophenyl)-N-[(3S)-3-piperidinyl]-2-thiophenecarboxamide Chemical compound NC(=O)NC=1C=C(C=2C=C(F)C=CC=2)SC=1C(=O)N[C@H]1CCCNC1 IAYGCINLNONXHY-LBPRGKRZSA-N 0.000 description 3
- WEVYAHXRMPXWCK-UHFFFAOYSA-N Acetonitrile Chemical compound CC#N WEVYAHXRMPXWCK-UHFFFAOYSA-N 0.000 description 3
- 206010069754 Acquired gene mutation Diseases 0.000 description 3
- ULXXDDBFHOBEHA-ONEGZZNKSA-N Afatinib Chemical compound N1=CN=C2C=C(OC3COCC3)C(NC(=O)/C=C/CN(C)C)=CC2=C1NC1=CC=C(F)C(Cl)=C1 ULXXDDBFHOBEHA-ONEGZZNKSA-N 0.000 description 3
- QADPYRIHXKWUSV-UHFFFAOYSA-N BGJ-398 Chemical compound C1CN(CC)CCN1C(C=C1)=CC=C1NC1=CC(N(C)C(=O)NC=2C(=C(OC)C=C(OC)C=2Cl)Cl)=NC=N1 QADPYRIHXKWUSV-UHFFFAOYSA-N 0.000 description 3
- CWHUFRVAEUJCEF-UHFFFAOYSA-N BKM120 Chemical compound C1=NC(N)=CC(C(F)(F)F)=C1C1=CC(N2CCOCC2)=NC(N2CCOCC2)=N1 CWHUFRVAEUJCEF-UHFFFAOYSA-N 0.000 description 3
- KXDAEFPNCMNJSK-UHFFFAOYSA-N Benzamide Chemical compound NC(=O)C1=CC=CC=C1 KXDAEFPNCMNJSK-UHFFFAOYSA-N 0.000 description 3
- 108010006654 Bleomycin Proteins 0.000 description 3
- COVZYZSDYWQREU-UHFFFAOYSA-N Busulfan Chemical compound CS(=O)(=O)OCCCCOS(C)(=O)=O COVZYZSDYWQREU-UHFFFAOYSA-N 0.000 description 3
- 102100021935 C-C motif chemokine 26 Human genes 0.000 description 3
- 108091026890 Coding region Proteins 0.000 description 3
- 102000001301 EGF receptor Human genes 0.000 description 3
- 108060006698 EGF receptor Proteins 0.000 description 3
- 102000004190 Enzymes Human genes 0.000 description 3
- 108090000790 Enzymes Proteins 0.000 description 3
- 102000009109 Fc receptors Human genes 0.000 description 3
- 108010087819 Fc receptors Proteins 0.000 description 3
- 101000897493 Homo sapiens C-C motif chemokine 26 Proteins 0.000 description 3
- 101001012157 Homo sapiens Receptor tyrosine-protein kinase erbB-2 Proteins 0.000 description 3
- 108060003951 Immunoglobulin Proteins 0.000 description 3
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 3
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 3
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 3
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 3
- 239000005517 L01XE01 - Imatinib Substances 0.000 description 3
- CZQHHVNHHHRRDU-UHFFFAOYSA-N LY294002 Chemical compound C1=CC=C2C(=O)C=C(N3CCOCC3)OC2=C1C1=CC=CC=C1 CZQHHVNHHHRRDU-UHFFFAOYSA-N 0.000 description 3
- QWPXBEHQFHACTK-UHFFFAOYSA-N Maytansinol Natural products CN1C(=O)CC(O)C2(C)OC2C(C)C(OC(=O)N2)CC2(O)C(OC)C=CC=C(C)CC2=CC(OC)=C(Cl)C1=C2 QWPXBEHQFHACTK-UHFFFAOYSA-N 0.000 description 3
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 3
- 102000016943 Muramidase Human genes 0.000 description 3
- 108010014251 Muramidase Proteins 0.000 description 3
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 3
- 108010062010 N-Acetylmuramoyl-L-alanine Amidase Proteins 0.000 description 3
- CXQHYVUVSFXTMY-UHFFFAOYSA-N N1'-[3-fluoro-4-[[6-methoxy-7-[3-(4-morpholinyl)propoxy]-4-quinolinyl]oxy]phenyl]-N1-(4-fluorophenyl)cyclopropane-1,1-dicarboxamide Chemical compound C1=CN=C2C=C(OCCCN3CCOCC3)C(OC)=CC2=C1OC(C(=C1)F)=CC=C1NC(=O)C1(C(=O)NC=2C=CC(F)=CC=2)CC1 CXQHYVUVSFXTMY-UHFFFAOYSA-N 0.000 description 3
- 108091007960 PI3Ks Proteins 0.000 description 3
- KDLHZDBZIXYQEI-UHFFFAOYSA-N Palladium Chemical compound [Pd] KDLHZDBZIXYQEI-UHFFFAOYSA-N 0.000 description 3
- 102000003993 Phosphatidylinositol 3-kinases Human genes 0.000 description 3
- 108090000430 Phosphatidylinositol 3-kinases Proteins 0.000 description 3
- 241000404883 Pisa Species 0.000 description 3
- 206010035226 Plasma cell myeloma Diseases 0.000 description 3
- 229920002873 Polyethylenimine Polymers 0.000 description 3
- 239000004365 Protease Substances 0.000 description 3
- 108010076504 Protein Sorting Signals Proteins 0.000 description 3
- 241000589516 Pseudomonas Species 0.000 description 3
- 102100030086 Receptor tyrosine-protein kinase erbB-2 Human genes 0.000 description 3
- 208000006265 Renal cell carcinoma Diseases 0.000 description 3
- 229920005654 Sephadex Polymers 0.000 description 3
- 239000012507 Sephadex™ Substances 0.000 description 3
- 108010088160 Staphylococcal Protein A Proteins 0.000 description 3
- 108010090804 Streptavidin Proteins 0.000 description 3
- 239000004098 Tetracycline Substances 0.000 description 3
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 3
- LUJZZYWHBDHDQX-QFIPXVFZSA-N [(3s)-morpholin-3-yl]methyl n-[4-[[1-[(3-fluorophenyl)methyl]indazol-5-yl]amino]-5-methylpyrrolo[2,1-f][1,2,4]triazin-6-yl]carbamate Chemical compound C=1N2N=CN=C(NC=3C=C4C=NN(CC=5C=C(F)C=CC=5)C4=CC=3)C2=C(C)C=1NC(=O)OC[C@@H]1COCCN1 LUJZZYWHBDHDQX-QFIPXVFZSA-N 0.000 description 3
- RJURFGZVJUQBHK-IIXSONLDSA-N actinomycin D Chemical compound C[C@H]1OC(=O)[C@H](C(C)C)N(C)C(=O)CN(C)C(=O)[C@@H]2CCCN2C(=O)[C@@H](C(C)C)NC(=O)[C@H]1NC(=O)C1=C(N)C(=O)C(C)=C2OC(C(C)=CC=C3C(=O)N[C@@H]4C(=O)N[C@@H](C(N5CCC[C@H]5C(=O)N(C)CC(=O)N(C)[C@@H](C(C)C)C(=O)O[C@@H]4C)=O)C(C)C)=C3N=C21 RJURFGZVJUQBHK-IIXSONLDSA-N 0.000 description 3
- 238000001042 affinity chromatography Methods 0.000 description 3
- 150000001412 amines Chemical class 0.000 description 3
- 125000003277 amino group Chemical group 0.000 description 3
- 239000003242 anti bacterial agent Substances 0.000 description 3
- 230000004071 biological effect Effects 0.000 description 3
- 238000007413 biotinylation Methods 0.000 description 3
- 230000008499 blood brain barrier function Effects 0.000 description 3
- 210000001218 blood-brain barrier Anatomy 0.000 description 3
- 239000002775 capsule Substances 0.000 description 3
- 235000014633 carbohydrates Nutrition 0.000 description 3
- 229960005243 carmustine Drugs 0.000 description 3
- 230000010261 cell growth Effects 0.000 description 3
- 238000004587 chromatography analysis Methods 0.000 description 3
- 238000011260 co-administration Methods 0.000 description 3
- KTEIFNKAUNYNJU-GFCCVEGCSA-N crizotinib Chemical compound O([C@H](C)C=1C(=C(F)C=CC=1Cl)Cl)C(C(=NC=1)N)=CC=1C(=C1)C=NN1C1CCNCC1 KTEIFNKAUNYNJU-GFCCVEGCSA-N 0.000 description 3
- 230000009260 cross reactivity Effects 0.000 description 3
- 238000002425 crystallisation Methods 0.000 description 3
- 230000008025 crystallization Effects 0.000 description 3
- 229960000684 cytarabine Drugs 0.000 description 3
- 229960000640 dactinomycin Drugs 0.000 description 3
- 239000000032 diagnostic agent Substances 0.000 description 3
- 229940039227 diagnostic agent Drugs 0.000 description 3
- 238000000502 dialysis Methods 0.000 description 3
- 230000006334 disulfide bridging Effects 0.000 description 3
- 239000006196 drop Substances 0.000 description 3
- 229940000406 drug candidate Drugs 0.000 description 3
- 238000004520 electroporation Methods 0.000 description 3
- 229940088598 enzyme Drugs 0.000 description 3
- 238000001914 filtration Methods 0.000 description 3
- 230000004927 fusion Effects 0.000 description 3
- QBKSWRVVCFFDOT-UHFFFAOYSA-N gossypol Chemical compound CC(C)C1=C(O)C(O)=C(C=O)C2=C(O)C(C=3C(O)=C4C(C=O)=C(O)C(O)=C(C4=CC=3C)C(C)C)=C(C)C=C21 QBKSWRVVCFFDOT-UHFFFAOYSA-N 0.000 description 3
- 230000036541 health Effects 0.000 description 3
- 238000004191 hydrophobic interaction chromatography Methods 0.000 description 3
- 229910052588 hydroxylapatite Inorganic materials 0.000 description 3
- KTUFNOKKBVMGRW-UHFFFAOYSA-N imatinib Chemical compound C1CN(C)CCN1CC1=CC=C(C(=O)NC=2C=C(NC=3N=C(C=CN=3)C=3C=NC=CC=3)C(C)=CC=2)C=C1 KTUFNOKKBVMGRW-UHFFFAOYSA-N 0.000 description 3
- 230000005847 immunogenicity Effects 0.000 description 3
- 102000018358 immunoglobulin Human genes 0.000 description 3
- 238000003364 immunohistochemistry Methods 0.000 description 3
- 230000002401 inhibitory effect Effects 0.000 description 3
- 238000007918 intramuscular administration Methods 0.000 description 3
- 238000007912 intraperitoneal administration Methods 0.000 description 3
- 238000002372 labelling Methods 0.000 description 3
- 238000004895 liquid chromatography mass spectrometry Methods 0.000 description 3
- 229960000274 lysozyme Drugs 0.000 description 3
- 239000004325 lysozyme Substances 0.000 description 3
- 235000010335 lysozyme Nutrition 0.000 description 3
- 230000014759 maintenance of location Effects 0.000 description 3
- SGDBTWWWUNNDEQ-LBPRGKRZSA-N melphalan Chemical compound OC(=O)[C@@H](N)CC1=CC=C(N(CCCl)CCCl)C=C1 SGDBTWWWUNNDEQ-LBPRGKRZSA-N 0.000 description 3
- MYWUZJCMWCOHBA-VIFPVBQESA-N methamphetamine Chemical compound CN[C@@H](C)CC1=CC=CC=C1 MYWUZJCMWCOHBA-VIFPVBQESA-N 0.000 description 3
- 125000000956 methoxy group Chemical group [H]C([H])([H])O* 0.000 description 3
- 238000002156 mixing Methods 0.000 description 3
- 201000000050 myeloid neoplasm Diseases 0.000 description 3
- JLYAXFNOILIKPP-KXQOOQHDSA-N navitoclax Chemical compound C([C@@H](NC1=CC=C(C=C1S(=O)(=O)C(F)(F)F)S(=O)(=O)NC(=O)C1=CC=C(C=C1)N1CCN(CC1)CC1=C(CCC(C1)(C)C)C=1C=CC(Cl)=CC=1)CSC=1C=CC=CC=1)CN1CCOCC1 JLYAXFNOILIKPP-KXQOOQHDSA-N 0.000 description 3
- 230000007935 neutral effect Effects 0.000 description 3
- 230000030147 nuclear export Effects 0.000 description 3
- 239000012038 nucleophile Substances 0.000 description 3
- 239000002674 ointment Substances 0.000 description 3
- XYJRXVWERLGGKC-UHFFFAOYSA-D pentacalcium;hydroxide;triphosphate Chemical compound [OH-].[Ca+2].[Ca+2].[Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O XYJRXVWERLGGKC-UHFFFAOYSA-D 0.000 description 3
- 229940068977 polysorbate 20 Drugs 0.000 description 3
- 125000001436 propyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])[H] 0.000 description 3
- 235000019419 proteases Nutrition 0.000 description 3
- 238000001959 radiotherapy Methods 0.000 description 3
- 239000011541 reaction mixture Substances 0.000 description 3
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 3
- 230000001105 regulatory effect Effects 0.000 description 3
- 238000011160 research Methods 0.000 description 3
- 210000002966 serum Anatomy 0.000 description 3
- 229960002930 sirolimus Drugs 0.000 description 3
- QFJCIRLUMZQUOT-HPLJOQBZSA-N sirolimus Chemical compound C1C[C@@H](O)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 QFJCIRLUMZQUOT-HPLJOQBZSA-N 0.000 description 3
- 238000002415 sodium dodecyl sulfate polyacrylamide gel electrophoresis Methods 0.000 description 3
- 229940074404 sodium succinate Drugs 0.000 description 3
- ZDQYSKICYIVCPN-UHFFFAOYSA-L sodium succinate (anhydrous) Chemical compound [Na+].[Na+].[O-]C(=O)CCC([O-])=O ZDQYSKICYIVCPN-UHFFFAOYSA-L 0.000 description 3
- 230000037439 somatic mutation Effects 0.000 description 3
- 238000001179 sorption measurement Methods 0.000 description 3
- 239000007921 spray Substances 0.000 description 3
- 230000006641 stabilisation Effects 0.000 description 3
- 239000008362 succinate buffer Substances 0.000 description 3
- 125000004434 sulfur atom Chemical group 0.000 description 3
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 3
- 238000003786 synthesis reaction Methods 0.000 description 3
- 229930101283 tetracycline Natural products 0.000 description 3
- 229960002180 tetracycline Drugs 0.000 description 3
- 235000019364 tetracycline Nutrition 0.000 description 3
- 150000003522 tetracyclines Chemical class 0.000 description 3
- 230000000699 topical effect Effects 0.000 description 3
- 238000013518 transcription Methods 0.000 description 3
- 230000035897 transcription Effects 0.000 description 3
- 210000004881 tumor cell Anatomy 0.000 description 3
- 229940121358 tyrosine kinase inhibitor Drugs 0.000 description 3
- 239000005483 tyrosine kinase inhibitor Substances 0.000 description 3
- 238000000870 ultraviolet spectroscopy Methods 0.000 description 3
- 229960003048 vinblastine Drugs 0.000 description 3
- 239000013603 viral vector Substances 0.000 description 3
- 230000003612 virological effect Effects 0.000 description 3
- LLXVXPPXELIDGQ-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 3-(2,5-dioxopyrrol-1-yl)benzoate Chemical compound C=1C=CC(N2C(C=CC2=O)=O)=CC=1C(=O)ON1C(=O)CCC1=O LLXVXPPXELIDGQ-UHFFFAOYSA-N 0.000 description 2
- WGMMKWFUXPMTRW-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 3-[(2-bromoacetyl)amino]propanoate Chemical compound BrCC(=O)NCCC(=O)ON1C(=O)CCC1=O WGMMKWFUXPMTRW-UHFFFAOYSA-N 0.000 description 2
- RKGUGSULMVQEIX-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 3-[2-[2-[6-(2,5-dioxopyrrol-1-yl)hexanoylamino]ethoxy]ethoxy]propanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCOCCOCCNC(=O)CCCCCN1C(=O)C=CC1=O RKGUGSULMVQEIX-UHFFFAOYSA-N 0.000 description 2
- PVGATNRYUYNBHO-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 4-(2,5-dioxopyrrol-1-yl)butanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCCN1C(=O)C=CC1=O PVGATNRYUYNBHO-UHFFFAOYSA-N 0.000 description 2
- PMJWDPGOWBRILU-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 4-[4-(2,5-dioxopyrrol-1-yl)phenyl]butanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCCC(C=C1)=CC=C1N1C(=O)C=CC1=O PMJWDPGOWBRILU-UHFFFAOYSA-N 0.000 description 2
- WCMOHMXWOOBVMZ-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 6-[3-(2,5-dioxopyrrol-1-yl)propanoylamino]hexanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCCCCNC(=O)CCN1C(=O)C=CC1=O WCMOHMXWOOBVMZ-UHFFFAOYSA-N 0.000 description 2
- DIGQNXIGRZPYDK-WKSCXVIASA-N (2R)-6-amino-2-[[2-[[(2S)-2-[[2-[[(2R)-2-[[(2S)-2-[[(2R,3S)-2-[[2-[[(2S)-2-[[2-[[(2S)-2-[[(2S)-2-[[(2R)-2-[[(2S,3S)-2-[[(2R)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[2-[[(2S)-2-[[(2R)-2-[[2-[[2-[[2-[(2-amino-1-hydroxyethylidene)amino]-3-carboxy-1-hydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1,5-dihydroxy-5-iminopentylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]hexanoic acid Chemical compound C[C@@H]([C@@H](C(=N[C@@H](CS)C(=N[C@@H](C)C(=N[C@@H](CO)C(=NCC(=N[C@@H](CCC(=N)O)C(=NC(CS)C(=N[C@H]([C@H](C)O)C(=N[C@H](CS)C(=N[C@H](CO)C(=NCC(=N[C@H](CS)C(=NCC(=N[C@H](CCCCN)C(=O)O)O)O)O)O)O)O)O)O)O)O)O)O)O)N=C([C@H](CS)N=C([C@H](CO)N=C([C@H](CO)N=C([C@H](C)N=C(CN=C([C@H](CO)N=C([C@H](CS)N=C(CN=C(C(CS)N=C(C(CC(=O)O)N=C(CN)O)O)O)O)O)O)O)O)O)O)O)O DIGQNXIGRZPYDK-WKSCXVIASA-N 0.000 description 2
- SVNJBEMPMKWDCO-KCHLEUMXSA-N (2s)-2-[[(2s)-3-carboxy-2-[[2-[[(2s)-5-(diaminomethylideneamino)-2-[[4-oxo-4-[[4-(4-oxo-8-phenylchromen-2-yl)morpholin-4-ium-4-yl]methoxy]butanoyl]amino]pentanoyl]amino]acetyl]amino]propanoyl]amino]-3-hydroxypropanoate Chemical compound C=1C(=O)C2=CC=CC(C=3C=CC=CC=3)=C2OC=1[N+]1(COC(=O)CCC(=O)N[C@@H](CCCNC(=N)N)C(=O)NCC(=O)N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C([O-])=O)CCOCC1 SVNJBEMPMKWDCO-KCHLEUMXSA-N 0.000 description 2
- CSGQVNMSRKWUSH-IAGOWNOFSA-N (3r,4r)-4-amino-1-[[4-(3-methoxyanilino)pyrrolo[2,1-f][1,2,4]triazin-5-yl]methyl]piperidin-3-ol Chemical compound COC1=CC=CC(NC=2C3=C(CN4C[C@@H](O)[C@H](N)CC4)C=CN3N=CN=2)=C1 CSGQVNMSRKWUSH-IAGOWNOFSA-N 0.000 description 2
- IAKHMKGGTNLKSZ-INIZCTEOSA-N (S)-colchicine Chemical compound C1([C@@H](NC(C)=O)CC2)=CC(=O)C(OC)=CC=C1C1=C2C=C(OC)C(OC)=C1OC IAKHMKGGTNLKSZ-INIZCTEOSA-N 0.000 description 2
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 2
- QFWCYNPOPKQOKV-UHFFFAOYSA-N 2-(2-amino-3-methoxyphenyl)chromen-4-one Chemical compound COC1=CC=CC(C=2OC3=CC=CC=C3C(=O)C=2)=C1N QFWCYNPOPKQOKV-UHFFFAOYSA-N 0.000 description 2
- GFMMXOIFOQCCGU-UHFFFAOYSA-N 2-(2-chloro-4-iodoanilino)-N-(cyclopropylmethoxy)-3,4-difluorobenzamide Chemical compound C=1C=C(I)C=C(Cl)C=1NC1=C(F)C(F)=CC=C1C(=O)NOCC1CC1 GFMMXOIFOQCCGU-UHFFFAOYSA-N 0.000 description 2
- PDMUGYOXRHVNMO-UHFFFAOYSA-N 2-[4-[3-(6-quinolinylmethyl)-5-triazolo[4,5-b]pyrazinyl]-1-pyrazolyl]ethanol Chemical compound C1=NN(CCO)C=C1C1=CN=C(N=NN2CC=3C=C4C=CC=NC4=CC=3)C2=N1 PDMUGYOXRHVNMO-UHFFFAOYSA-N 0.000 description 2
- XDLYKKIQACFMJG-UHFFFAOYSA-N 2-amino-8-[4-(2-hydroxyethoxy)cyclohexyl]-6-(6-methoxypyridin-3-yl)-4-methylpyrido[2,3-d]pyrimidin-7-one Chemical compound C1=NC(OC)=CC=C1C(C1=O)=CC2=C(C)N=C(N)N=C2N1C1CCC(OCCO)CC1 XDLYKKIQACFMJG-UHFFFAOYSA-N 0.000 description 2
- IUTPJBLLJJNPAJ-UHFFFAOYSA-N 3-(2,5-dioxopyrrol-1-yl)propanoic acid Chemical compound OC(=O)CCN1C(=O)C=CC1=O IUTPJBLLJJNPAJ-UHFFFAOYSA-N 0.000 description 2
- RCLQNICOARASSR-SECBINFHSA-N 3-[(2r)-2,3-dihydroxypropyl]-6-fluoro-5-(2-fluoro-4-iodoanilino)-8-methylpyrido[2,3-d]pyrimidine-4,7-dione Chemical compound FC=1C(=O)N(C)C=2N=CN(C[C@@H](O)CO)C(=O)C=2C=1NC1=CC=C(I)C=C1F RCLQNICOARASSR-SECBINFHSA-N 0.000 description 2
- WUIABRMSWOKTOF-OYALTWQYSA-N 3-[[2-[2-[2-[[(2s,3r)-2-[[(2s,3s,4r)-4-[[(2s,3r)-2-[[6-amino-2-[(1s)-3-amino-1-[[(2s)-2,3-diamino-3-oxopropyl]amino]-3-oxopropyl]-5-methylpyrimidine-4-carbonyl]amino]-3-[(2r,3s,4s,5s,6s)-3-[(2r,3s,4s,5r,6r)-4-carbamoyloxy-3,5-dihydroxy-6-(hydroxymethyl)ox Chemical compound OS([O-])(=O)=O.N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1NC=NC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C WUIABRMSWOKTOF-OYALTWQYSA-N 0.000 description 2
- QGZSANLMUAESBT-UHFFFAOYSA-N 3-piperazin-1-ylpropane-1-sulfonic acid Chemical compound OS(=O)(=O)CCCN1CCNCC1 QGZSANLMUAESBT-UHFFFAOYSA-N 0.000 description 2
- NCPQROHLJFARLL-UHFFFAOYSA-N 4-(2,5-dioxopyrrol-1-yl)butanoic acid Chemical compound OC(=O)CCCN1C(=O)C=CC1=O NCPQROHLJFARLL-UHFFFAOYSA-N 0.000 description 2
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 2
- SUVMJBTUFCVSAD-JTQLQIEISA-N 4-Methylsulfinylbutyl isothiocyanate Natural products C[S@](=O)CCCCN=C=S SUVMJBTUFCVSAD-JTQLQIEISA-N 0.000 description 2
- MOVBBVMDHIRCTG-LJQANCHMSA-N 4-[(3s)-1-azabicyclo[2.2.2]oct-3-ylamino]-3-(1h-benzimidazol-2-yl)-6-chloroquinolin-2(1h)-one Chemical compound C([N@](CC1)C2)C[C@@H]1[C@@H]2NC1=C(C=2NC3=CC=CC=C3N=2)C(=O)NC2=CC=C(Cl)C=C21 MOVBBVMDHIRCTG-LJQANCHMSA-N 0.000 description 2
- XRYJULCDUUATMC-CYBMUJFWSA-N 4-[4-[[(1r)-1-phenylethyl]amino]-7h-pyrrolo[2,3-d]pyrimidin-6-yl]phenol Chemical compound N([C@H](C)C=1C=CC=CC=1)C(C=1C=2)=NC=NC=1NC=2C1=CC=C(O)C=C1 XRYJULCDUUATMC-CYBMUJFWSA-N 0.000 description 2
- XXLPVQZYQCGXOV-UHFFFAOYSA-N 4-amino-5-fluoro-3-[6-(4-methylpiperazin-1-yl)-1H-benzimidazol-2-yl]-1H-quinolin-2-one 2-hydroxypropanoic acid Chemical compound CC(O)C(O)=O.CC(O)C(O)=O.CN1CCN(CC1)c1ccc2nc([nH]c2c1)-c1c(N)c2c(F)cccc2[nH]c1=O XXLPVQZYQCGXOV-UHFFFAOYSA-N 0.000 description 2
- JRWCBEOAFGHNNU-UHFFFAOYSA-N 6-[difluoro-[6-(1-methyl-4-pyrazolyl)-[1,2,4]triazolo[4,3-b]pyridazin-3-yl]methyl]quinoline Chemical compound C1=NN(C)C=C1C1=NN2C(C(F)(F)C=3C=C4C=CC=NC4=CC=3)=NN=C2C=C1 JRWCBEOAFGHNNU-UHFFFAOYSA-N 0.000 description 2
- GMIZZEXBPRLVIV-SECBINFHSA-N 6-bromo-3-(1-methylpyrazol-4-yl)-5-[(3r)-piperidin-3-yl]pyrazolo[1,5-a]pyrimidin-7-amine Chemical compound C1=NN(C)C=C1C1=C2N=C([C@H]3CNCCC3)C(Br)=C(N)N2N=C1 GMIZZEXBPRLVIV-SECBINFHSA-N 0.000 description 2
- HPLNQCPCUACXLM-PGUFJCEWSA-N ABT-737 Chemical compound C([C@@H](CCN(C)C)NC=1C(=CC(=CC=1)S(=O)(=O)NC(=O)C=1C=CC(=CC=1)N1CCN(CC=2C(=CC=CC=2)C=2C=CC(Cl)=CC=2)CC1)[N+]([O-])=O)SC1=CC=CC=C1 HPLNQCPCUACXLM-PGUFJCEWSA-N 0.000 description 2
- OONFNUWBHFSNBT-HXUWFJFHSA-N AEE788 Chemical compound C1CN(CC)CCN1CC1=CC=C(C=2NC3=NC=NC(N[C@H](C)C=4C=CC=CC=4)=C3C=2)C=C1 OONFNUWBHFSNBT-HXUWFJFHSA-N 0.000 description 2
- BUROJSBIWGDYCN-GAUTUEMISA-N AP 23573 Chemical compound C1C[C@@H](OP(C)(C)=O)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 BUROJSBIWGDYCN-GAUTUEMISA-N 0.000 description 2
- IKYJCHYORFJFRR-UHFFFAOYSA-N Alexa Fluor 350 Chemical compound O=C1OC=2C=C(N)C(S(O)(=O)=O)=CC=2C(C)=C1CC(=O)ON1C(=O)CCC1=O IKYJCHYORFJFRR-UHFFFAOYSA-N 0.000 description 2
- JLDSMZIBHYTPPR-UHFFFAOYSA-N Alexa Fluor 405 Chemical compound CC[NH+](CC)CC.CC[NH+](CC)CC.CC[NH+](CC)CC.C12=C3C=4C=CC2=C(S([O-])(=O)=O)C=C(S([O-])(=O)=O)C1=CC=C3C(S(=O)(=O)[O-])=CC=4OCC(=O)N(CC1)CCC1C(=O)ON1C(=O)CCC1=O JLDSMZIBHYTPPR-UHFFFAOYSA-N 0.000 description 2
- WEJVZSAYICGDCK-UHFFFAOYSA-N Alexa Fluor 430 Chemical compound CC[NH+](CC)CC.CC1(C)C=C(CS([O-])(=O)=O)C2=CC=3C(C(F)(F)F)=CC(=O)OC=3C=C2N1CCCCCC(=O)ON1C(=O)CCC1=O WEJVZSAYICGDCK-UHFFFAOYSA-N 0.000 description 2
- WHVNXSBKJGAXKU-UHFFFAOYSA-N Alexa Fluor 532 Chemical compound [H+].[H+].CC1(C)C(C)NC(C(=C2OC3=C(C=4C(C(C(C)N=4)(C)C)=CC3=3)S([O-])(=O)=O)S([O-])(=O)=O)=C1C=C2C=3C(C=C1)=CC=C1C(=O)ON1C(=O)CCC1=O WHVNXSBKJGAXKU-UHFFFAOYSA-N 0.000 description 2
- ZAINTDRBUHCDPZ-UHFFFAOYSA-M Alexa Fluor 546 Chemical compound [H+].[Na+].CC1CC(C)(C)NC(C(=C2OC3=C(C4=NC(C)(C)CC(C)C4=CC3=3)S([O-])(=O)=O)S([O-])(=O)=O)=C1C=C2C=3C(C(=C(Cl)C=1Cl)C(O)=O)=C(Cl)C=1SCC(=O)NCCCCCC(=O)ON1C(=O)CCC1=O ZAINTDRBUHCDPZ-UHFFFAOYSA-M 0.000 description 2
- IGAZHQIYONOHQN-UHFFFAOYSA-N Alexa Fluor 555 Chemical compound C=12C=CC(=N)C(S(O)(=O)=O)=C2OC2=C(S(O)(=O)=O)C(N)=CC=C2C=1C1=CC=C(C(O)=O)C=C1C(O)=O IGAZHQIYONOHQN-UHFFFAOYSA-N 0.000 description 2
- 108010024976 Asparaginase Proteins 0.000 description 2
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 2
- 108091006061 AviTagged proteins Proteins 0.000 description 2
- MLDQJTXFUGDVEO-UHFFFAOYSA-N BAY-43-9006 Chemical compound C1=NC(C(=O)NC)=CC(OC=2C=CC(NC(=O)NC=3C=C(C(Cl)=CC=3)C(F)(F)F)=CC=2)=C1 MLDQJTXFUGDVEO-UHFFFAOYSA-N 0.000 description 2
- 244000063299 Bacillus subtilis Species 0.000 description 2
- 235000014469 Bacillus subtilis Nutrition 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 101000708016 Caenorhabditis elegans Sentrin-specific protease Proteins 0.000 description 2
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 2
- GAGWJHPBXLXJQN-UORFTKCHSA-N Capecitabine Chemical compound C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1[C@H]1[C@H](O)[C@H](O)[C@@H](C)O1 GAGWJHPBXLXJQN-UORFTKCHSA-N 0.000 description 2
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 2
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 2
- 108010067225 Cell Adhesion Molecules Proteins 0.000 description 2
- 102000016289 Cell Adhesion Molecules Human genes 0.000 description 2
- 108010024986 Cyclin-Dependent Kinase 2 Proteins 0.000 description 2
- 102100036239 Cyclin-dependent kinase 2 Human genes 0.000 description 2
- ZBNZXTGUTAYRHI-UHFFFAOYSA-N Dasatinib Chemical compound C=1C(N2CCN(CCO)CC2)=NC(C)=NC=1NC(S1)=NC=C1C(=O)NC1=C(C)C=CC=C1Cl ZBNZXTGUTAYRHI-UHFFFAOYSA-N 0.000 description 2
- 238000012286 ELISA Assay Methods 0.000 description 2
- HKVAMNSJSFKALM-GKUWKFKPSA-N Everolimus Chemical compound C1C[C@@H](OCCO)[C@H](OC)C[C@@H]1C[C@@H](C)[C@H]1OC(=O)[C@@H]2CCCCN2C(=O)C(=O)[C@](O)(O2)[C@H](C)CC[C@H]2C[C@H](OC)/C(C)=C/C=C/C=C/[C@@H](C)C[C@@H](C)C(=O)[C@H](OC)[C@H](O)/C(C)=C/[C@@H](C)C(=O)C1 HKVAMNSJSFKALM-GKUWKFKPSA-N 0.000 description 2
- 238000012413 Fluorescence activated cell sorting analysis Methods 0.000 description 2
- AEMRFAOFKBGASW-UHFFFAOYSA-N Glycolic acid Polymers OCC(O)=O AEMRFAOFKBGASW-UHFFFAOYSA-N 0.000 description 2
- 101710154606 Hemagglutinin Proteins 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- 108010093488 His-His-His-His-His-His Proteins 0.000 description 2
- 101001034652 Homo sapiens Insulin-like growth factor 1 receptor Proteins 0.000 description 2
- 101000610605 Homo sapiens Tumor necrosis factor receptor superfamily member 10A Proteins 0.000 description 2
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 2
- VSNHCAURESNICA-UHFFFAOYSA-N Hydroxyurea Chemical compound NC(=O)NO VSNHCAURESNICA-UHFFFAOYSA-N 0.000 description 2
- 102100039688 Insulin-like growth factor 1 receptor Human genes 0.000 description 2
- 102000010638 Kinesin Human genes 0.000 description 2
- 108010063296 Kinesin Proteins 0.000 description 2
- XUJNEKJLAYXESH-REOHCLBHSA-N L-Cysteine Chemical compound SC[C@H](N)C(O)=O XUJNEKJLAYXESH-REOHCLBHSA-N 0.000 description 2
- 229930182816 L-glutamine Natural products 0.000 description 2
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 2
- LRQKBLKVPFOOQJ-YFKPBYRVSA-N L-norleucine Chemical compound CCCC[C@H]([NH3+])C([O-])=O LRQKBLKVPFOOQJ-YFKPBYRVSA-N 0.000 description 2
- 239000005551 L01XE03 - Erlotinib Substances 0.000 description 2
- 239000005511 L01XE05 - Sorafenib Substances 0.000 description 2
- 239000005536 L01XE08 - Nilotinib Substances 0.000 description 2
- 239000002146 L01XE16 - Crizotinib Substances 0.000 description 2
- UCEQXRCJXIVODC-PMACEKPBSA-N LSM-1131 Chemical compound C1CCC2=CC=CC3=C2N1C=C3[C@@H]1C(=O)NC(=O)[C@H]1C1=CNC2=CC=CC=C12 UCEQXRCJXIVODC-PMACEKPBSA-N 0.000 description 2
- OTPNDVKVEAIXTI-UHFFFAOYSA-N LSM-1274 Chemical compound C12=C3C4=C5C=CC=C[C]5N3C(O3)CCC3N2C2=CC=C[CH]C2=C1C1=C4C(=O)NC1=O OTPNDVKVEAIXTI-UHFFFAOYSA-N 0.000 description 2
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 2
- 108090001090 Lectins Proteins 0.000 description 2
- 102000004856 Lectins Human genes 0.000 description 2
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 2
- 239000012097 Lipofectamine 2000 Substances 0.000 description 2
- 239000006142 Luria-Bertani Agar Substances 0.000 description 2
- 102000043136 MAP kinase family Human genes 0.000 description 2
- 108091054455 MAP kinase family Proteins 0.000 description 2
- 102000003792 Metallothionein Human genes 0.000 description 2
- 108090000157 Metallothionein Proteins 0.000 description 2
- 206010027476 Metastases Diseases 0.000 description 2
- YNAVUWVOSKDBBP-UHFFFAOYSA-N Morpholine Chemical compound C1COCCN1 YNAVUWVOSKDBBP-UHFFFAOYSA-N 0.000 description 2
- 241000713333 Mouse mammary tumor virus Species 0.000 description 2
- VIUAUNHCRHHYNE-JTQLQIEISA-N N-[(2S)-2,3-dihydroxypropyl]-3-(2-fluoro-4-iodoanilino)-4-pyridinecarboxamide Chemical compound OC[C@@H](O)CNC(=O)C1=CC=NC=C1NC1=CC=C(I)C=C1F VIUAUNHCRHHYNE-JTQLQIEISA-N 0.000 description 2
- GCIKSSRWRFVXBI-UHFFFAOYSA-N N-[4-[[4-(4-methyl-1-piperazinyl)-6-[(5-methyl-1H-pyrazol-3-yl)amino]-2-pyrimidinyl]thio]phenyl]cyclopropanecarboxamide Chemical compound C1CN(C)CCN1C1=CC(NC2=NNC(C)=C2)=NC(SC=2C=CC(NC(=O)C3CC3)=CC=2)=N1 GCIKSSRWRFVXBI-UHFFFAOYSA-N 0.000 description 2
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 2
- JOCBASBOOFNAJA-UHFFFAOYSA-N N-tris(hydroxymethyl)methyl-2-aminoethanesulfonic acid Chemical compound OCC(CO)(CO)NCCS(O)(=O)=O JOCBASBOOFNAJA-UHFFFAOYSA-N 0.000 description 2
- 101710093908 Outer capsid protein VP4 Proteins 0.000 description 2
- 101710135467 Outer capsid protein sigma-1 Proteins 0.000 description 2
- DXCUKNQANPLTEJ-UHFFFAOYSA-N PD173074 Chemical compound CC(C)(C)NC(=O)NC1=NC2=NC(NCCCCN(CC)CC)=NC=C2C=C1C1=CC(OC)=CC(OC)=C1 DXCUKNQANPLTEJ-UHFFFAOYSA-N 0.000 description 2
- OYONTEXKYJZFHA-SSHUPFPWSA-N PHA-665752 Chemical compound CC=1C(C(=O)N2[C@H](CCC2)CN2CCCC2)=C(C)NC=1\C=C(C1=C2)/C(=O)NC1=CC=C2S(=O)(=O)CC1=C(Cl)C=CC=C1Cl OYONTEXKYJZFHA-SSHUPFPWSA-N 0.000 description 2
- QIUASFSNWYMDFS-NILGECQDSA-N PX-866 Chemical compound CC(=O)O[C@@H]1C[C@]2(C)C(=O)CC[C@H]2C2=C1[C@@]1(C)[C@@H](COC)OC(=O)\C(=C\N(CC=C)CC=C)C1=C(O)C2=O QIUASFSNWYMDFS-NILGECQDSA-N 0.000 description 2
- 108091093037 Peptide nucleic acid Proteins 0.000 description 2
- 229920000954 Polyglycolide Polymers 0.000 description 2
- 101710176177 Protein A56 Proteins 0.000 description 2
- 108091028664 Ribonucleotide Proteins 0.000 description 2
- BCZUAADEACICHN-UHFFFAOYSA-N SGX-523 Chemical compound C1=NN(C)C=C1C1=NN2C(SC=3C=C4C=CC=NC4=CC=3)=NN=C2C=C1 BCZUAADEACICHN-UHFFFAOYSA-N 0.000 description 2
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 2
- 241000607142 Salmonella Species 0.000 description 2
- 241000710961 Semliki Forest virus Species 0.000 description 2
- 108010034546 Serratia marcescens nuclease Proteins 0.000 description 2
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 2
- 108091027967 Small hairpin RNA Proteins 0.000 description 2
- 102100040113 Tumor necrosis factor receptor superfamily member 10A Human genes 0.000 description 2
- VGQOVCHZGQWAOI-UHFFFAOYSA-N UNPD55612 Natural products N1C(O)C2CC(C=CC(N)=O)=CN2C(=O)C2=CC=C(C)C(O)=C12 VGQOVCHZGQWAOI-UHFFFAOYSA-N 0.000 description 2
- XSQUKJJJFZCRTK-UHFFFAOYSA-N Urea Chemical compound NC(N)=O XSQUKJJJFZCRTK-UHFFFAOYSA-N 0.000 description 2
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 2
- 241000700605 Viruses Species 0.000 description 2
- VWQVUPCCIRVNHF-OUBTZVSYSA-N Yttrium-90 Chemical compound [90Y] VWQVUPCCIRVNHF-OUBTZVSYSA-N 0.000 description 2
- CHKFLBOLYREYDO-SHYZEUOFSA-N [[(2s,4r,5r)-5-(4-amino-2-oxopyrimidin-1-yl)-4-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl] phosphono hydrogen phosphate Chemical compound O=C1N=C(N)C=CN1[C@H]1[C@H](O)C[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(O)=O)O1 CHKFLBOLYREYDO-SHYZEUOFSA-N 0.000 description 2
- 238000009825 accumulation Methods 0.000 description 2
- 108010076089 accutase Proteins 0.000 description 2
- 230000002378 acidificating effect Effects 0.000 description 2
- 230000002411 adverse Effects 0.000 description 2
- 230000003281 allosteric effect Effects 0.000 description 2
- 150000001408 amides Chemical class 0.000 description 2
- 230000003321 amplification Effects 0.000 description 2
- 239000012491 analyte Substances 0.000 description 2
- YBBLVLTVTVSKRW-UHFFFAOYSA-N anastrozole Chemical compound N#CC(C)(C)C1=CC(C(C)(C#N)C)=CC(CN2N=CN=C2)=C1 YBBLVLTVTVSKRW-UHFFFAOYSA-N 0.000 description 2
- 229940045799 anthracyclines and related substance Drugs 0.000 description 2
- VGQOVCHZGQWAOI-HYUHUPJXSA-N anthramycin Chemical compound N1[C@@H](O)[C@@H]2CC(\C=C\C(N)=O)=CN2C(=O)C2=CC=C(C)C(O)=C12 VGQOVCHZGQWAOI-HYUHUPJXSA-N 0.000 description 2
- 239000003429 antifungal agent Substances 0.000 description 2
- 229940121375 antifungal agent Drugs 0.000 description 2
- 230000000890 antigenic effect Effects 0.000 description 2
- 239000003124 biologic agent Substances 0.000 description 2
- 230000008512 biological response Effects 0.000 description 2
- 210000001124 body fluid Anatomy 0.000 description 2
- 239000010839 body fluid Substances 0.000 description 2
- 238000010504 bond cleavage reaction Methods 0.000 description 2
- 229940112133 busulfex Drugs 0.000 description 2
- 125000000484 butyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])C([H])([H])[H] 0.000 description 2
- 239000011575 calcium Substances 0.000 description 2
- 229910052791 calcium Inorganic materials 0.000 description 2
- 239000001506 calcium phosphate Substances 0.000 description 2
- 229910000389 calcium phosphate Inorganic materials 0.000 description 2
- 235000011010 calcium phosphates Nutrition 0.000 description 2
- 125000004432 carbon atom Chemical group C* 0.000 description 2
- 150000007942 carboxylates Chemical class 0.000 description 2
- 239000006143 cell culture medium Substances 0.000 description 2
- 230000006037 cell lysis Effects 0.000 description 2
- 230000004663 cell proliferation Effects 0.000 description 2
- 239000002738 chelating agent Substances 0.000 description 2
- 229960004316 cisplatin Drugs 0.000 description 2
- 229960002271 cobimetinib Drugs 0.000 description 2
- BSMCAPRUBJMWDF-KRWDZBQOSA-N cobimetinib Chemical compound C1C(O)([C@H]2NCCCC2)CN1C(=O)C1=CC=C(F)C(F)=C1NC1=CC=C(I)C=C1F BSMCAPRUBJMWDF-KRWDZBQOSA-N 0.000 description 2
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 2
- 230000001143 conditioned effect Effects 0.000 description 2
- 239000006071 cream Substances 0.000 description 2
- 229960005061 crizotinib Drugs 0.000 description 2
- 238000002447 crystallographic data Methods 0.000 description 2
- 125000004122 cyclic group Chemical group 0.000 description 2
- 229960004397 cyclophosphamide Drugs 0.000 description 2
- 235000018417 cysteine Nutrition 0.000 description 2
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 2
- 125000000151 cysteine group Chemical group N[C@@H](CS)C(=O)* 0.000 description 2
- 231100000433 cytotoxic Toxicity 0.000 description 2
- 230000001472 cytotoxic effect Effects 0.000 description 2
- JOGKUKXHTYWRGZ-UHFFFAOYSA-N dactolisib Chemical compound O=C1N(C)C2=CN=C3C=CC(C=4C=C5C=CC=CC5=NC=4)=CC3=C2N1C1=CC=C(C(C)(C)C#N)C=C1 JOGKUKXHTYWRGZ-UHFFFAOYSA-N 0.000 description 2
- 238000013480 data collection Methods 0.000 description 2
- 229960000975 daunorubicin Drugs 0.000 description 2
- 230000002950 deficient Effects 0.000 description 2
- UREBDLICKHMUKA-CXSFZGCWSA-N dexamethasone Chemical compound C1CC2=CC(=O)C=C[C@]2(C)[C@]2(F)[C@@H]1[C@@H]1C[C@@H](C)[C@@](C(=O)CO)(O)[C@@]1(C)C[C@@H]2O UREBDLICKHMUKA-CXSFZGCWSA-N 0.000 description 2
- 229960003957 dexamethasone Drugs 0.000 description 2
- 238000004090 dissolution Methods 0.000 description 2
- 125000002228 disulfide group Chemical group 0.000 description 2
- 229960004679 doxorubicin Drugs 0.000 description 2
- 239000000975 dye Substances 0.000 description 2
- 239000000839 emulsion Substances 0.000 description 2
- 229960005073 erlotinib hydrochloride Drugs 0.000 description 2
- VJJPUSNTGOMMGY-MRVIYFEKSA-N etoposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@H](C)OC[C@H]4O3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 VJJPUSNTGOMMGY-MRVIYFEKSA-N 0.000 description 2
- GIUYCYHIANZCFB-FJFJXFQQSA-N fludarabine phosphate Chemical compound C1=NC=2C(N)=NC(F)=NC=2N1[C@@H]1O[C@H](COP(O)(O)=O)[C@@H](O)[C@@H]1O GIUYCYHIANZCFB-FJFJXFQQSA-N 0.000 description 2
- 229960005304 fludarabine phosphate Drugs 0.000 description 2
- 229960002949 fluorouracil Drugs 0.000 description 2
- 235000019253 formic acid Nutrition 0.000 description 2
- 230000033581 fucosylation Effects 0.000 description 2
- XGALLCVXEZPNRQ-UHFFFAOYSA-N gefitinib Chemical compound C=12C=C(OCCCN3CCOCC3)C(OC)=CC2=NC=NC=1NC1=CC=C(F)C(Cl)=C1 XGALLCVXEZPNRQ-UHFFFAOYSA-N 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 230000002068 genetic effect Effects 0.000 description 2
- 239000011521 glass Substances 0.000 description 2
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 2
- 231100000844 hepatocellular carcinoma Toxicity 0.000 description 2
- 238000013537 high throughput screening Methods 0.000 description 2
- 210000005260 human cell Anatomy 0.000 description 2
- 125000004435 hydrogen atom Chemical group [H]* 0.000 description 2
- 230000002209 hydrophobic effect Effects 0.000 description 2
- HOMGKSMUEGBAAB-UHFFFAOYSA-N ifosfamide Chemical compound ClCCNP1(=O)OCCCN1CCCl HOMGKSMUEGBAAB-UHFFFAOYSA-N 0.000 description 2
- 229960002411 imatinib Drugs 0.000 description 2
- 229940072221 immunoglobulins Drugs 0.000 description 2
- 238000001114 immunoprecipitation Methods 0.000 description 2
- 239000007943 implant Substances 0.000 description 2
- 238000010348 incorporation Methods 0.000 description 2
- APFVFJFRJDLVQX-AHCXROLUSA-N indium-111 Chemical compound [111In] APFVFJFRJDLVQX-AHCXROLUSA-N 0.000 description 2
- 208000015181 infectious disease Diseases 0.000 description 2
- 229940079322 interferon Drugs 0.000 description 2
- 238000001361 intraarterial administration Methods 0.000 description 2
- 230000003834 intracellular effect Effects 0.000 description 2
- 238000004255 ion exchange chromatography Methods 0.000 description 2
- UWKQSNNFCGGAFS-XIFFEERXSA-N irinotecan Chemical compound C1=C2C(CC)=C3CN(C(C4=C([C@@](C(=O)OC4)(O)CC)C=4)=O)C=4C3=NC2=CC=C1OC(=O)N(CC1)CCC1N1CCCCC1 UWKQSNNFCGGAFS-XIFFEERXSA-N 0.000 description 2
- WXUJAQBSBZLVEV-UHFFFAOYSA-N isogranulatimide Chemical compound N1C2=CC=CC=C2C2=C1N1C=NC=C1C1=C2C(=O)NC1=O WXUJAQBSBZLVEV-UHFFFAOYSA-N 0.000 description 2
- 210000003734 kidney Anatomy 0.000 description 2
- 238000011813 knockout mouse model Methods 0.000 description 2
- JVTAAEKCZFNVCJ-UHFFFAOYSA-N lactic acid Chemical compound CC(O)C(O)=O JVTAAEKCZFNVCJ-UHFFFAOYSA-N 0.000 description 2
- 239000008101 lactose Substances 0.000 description 2
- 229960001320 lapatinib ditosylate Drugs 0.000 description 2
- 239000002523 lectin Substances 0.000 description 2
- 210000004185 liver Anatomy 0.000 description 2
- 238000011068 loading method Methods 0.000 description 2
- 239000006210 lotion Substances 0.000 description 2
- 239000012139 lysis buffer Substances 0.000 description 2
- 229960001924 melphalan Drugs 0.000 description 2
- 229960001428 mercaptopurine Drugs 0.000 description 2
- 108020004999 messenger RNA Proteins 0.000 description 2
- 229960000485 methotrexate Drugs 0.000 description 2
- 229960004857 mitomycin Drugs 0.000 description 2
- KKZJGLLVHKMTCM-UHFFFAOYSA-N mitoxantrone Chemical compound O=C1C2=C(O)C=CC(O)=C2C(=O)C2=C1C(NCCNCCO)=CC=C2NCCNCCO KKZJGLLVHKMTCM-UHFFFAOYSA-N 0.000 description 2
- LBWFXVZLPYTWQI-IPOVEDGCSA-N n-[2-(diethylamino)ethyl]-5-[(z)-(5-fluoro-2-oxo-1h-indol-3-ylidene)methyl]-2,4-dimethyl-1h-pyrrole-3-carboxamide;(2s)-2-hydroxybutanedioic acid Chemical compound OC(=O)[C@@H](O)CC(O)=O.CCN(CC)CCNC(=O)C1=C(C)NC(\C=C/2C3=CC(F)=CC=C3NC\2=O)=C1C LBWFXVZLPYTWQI-IPOVEDGCSA-N 0.000 description 2
- 229950004847 navitoclax Drugs 0.000 description 2
- 239000006199 nebulizer Substances 0.000 description 2
- 239000013642 negative control Substances 0.000 description 2
- HHZIURLSWUIHRB-UHFFFAOYSA-N nilotinib Chemical compound C1=NC(C)=CN1C1=CC(NC(=O)C=2C=C(NC=3N=C(C=CN=3)C=3C=NC=CC=3)C(C)=CC=2)=CC(C(F)(F)F)=C1 HHZIURLSWUIHRB-UHFFFAOYSA-N 0.000 description 2
- 229950010203 nimotuzumab Drugs 0.000 description 2
- 229910052757 nitrogen Inorganic materials 0.000 description 2
- 238000003199 nucleic acid amplification method Methods 0.000 description 2
- 229960000435 oblimersen Drugs 0.000 description 2
- MIMNFCVQODTQDP-NDLVEFNKSA-N oblimersen Chemical compound O=C1NC(=O)C(C)=CN1[C@@H]1O[C@H](COP(S)(=O)O[C@@H]2[C@H](O[C@H](C2)N2C3=NC=NC(N)=C3N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C3=C(C(NC(N)=N3)=O)N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C3=C(C(NC(N)=N3)=O)N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(NC(=O)C(C)=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C3=C(C(NC(N)=N3)=O)N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C3=C(C(NC(N)=N3)=O)N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C3=NC=NC(N)=C3N=C2)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(NC(=O)C(C)=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(N=C(N)C=C2)=O)COP(O)(=S)O[C@@H]2[C@H](O[C@H](C2)N2C(NC(=O)C(C)=C2)=O)CO)[C@@H](O)C1 MIMNFCVQODTQDP-NDLVEFNKSA-N 0.000 description 2
- 239000003960 organic solvent Substances 0.000 description 2
- 208000012988 ovarian serous adenocarcinoma Diseases 0.000 description 2
- 201000003709 ovarian serous carcinoma Diseases 0.000 description 2
- 230000002018 overexpression Effects 0.000 description 2
- 238000007911 parenteral administration Methods 0.000 description 2
- 238000002823 phage display Methods 0.000 description 2
- 239000008363 phosphate buffer Substances 0.000 description 2
- BIWOSRSKDCZIFM-UHFFFAOYSA-N piperidin-3-ol Chemical compound OC1CCCNC1 BIWOSRSKDCZIFM-UHFFFAOYSA-N 0.000 description 2
- 230000003169 placental effect Effects 0.000 description 2
- 229920000747 poly(lactic acid) Polymers 0.000 description 2
- 229920002401 polyacrylamide Polymers 0.000 description 2
- PHXJVRSECIGDHY-UHFFFAOYSA-N ponatinib Chemical compound C1CN(C)CCN1CC(C(=C1)C(F)(F)F)=CC=C1NC(=O)C1=CC=C(C)C(C#CC=2N3N=CC=CC3=NC=2)=C1 PHXJVRSECIGDHY-UHFFFAOYSA-N 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 230000002062 proliferating effect Effects 0.000 description 2
- 239000003380 propellant Substances 0.000 description 2
- AQHHHDLHHXJYJD-UHFFFAOYSA-N propranolol Chemical compound C1=CC=C2C(OCC(O)CNC(C)C)=CC=CC2=C1 AQHHHDLHHXJYJD-UHFFFAOYSA-N 0.000 description 2
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 2
- 238000002708 random mutagenesis Methods 0.000 description 2
- ZAHRKKWIAAJSAO-UHFFFAOYSA-N rapamycin Natural products COCC(O)C(=C/C(C)C(=O)CC(OC(=O)C1CCCCN1C(=O)C(=O)C2(O)OC(CC(OC)C(=CC=CC=CC(C)CC(C)C(=O)C)C)CCC2C)C(C)CC3CCC(O)C(C3)OC)C ZAHRKKWIAAJSAO-UHFFFAOYSA-N 0.000 description 2
- 238000003259 recombinant expression Methods 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 210000003994 retinal ganglion cell Anatomy 0.000 description 2
- 239000002336 ribonucleotide Substances 0.000 description 2
- 238000011218 seed culture Methods 0.000 description 2
- 239000006152 selective media Substances 0.000 description 2
- 238000002864 sequence alignment Methods 0.000 description 2
- 238000012163 sequencing technique Methods 0.000 description 2
- 238000001542 size-exclusion chromatography Methods 0.000 description 2
- 239000001509 sodium citrate Substances 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 238000007920 subcutaneous administration Methods 0.000 description 2
- KDYFGRWQOYBRFD-UHFFFAOYSA-L succinate(2-) Chemical compound [O-]C(=O)CCC([O-])=O KDYFGRWQOYBRFD-UHFFFAOYSA-L 0.000 description 2
- 239000004094 surface-active agent Substances 0.000 description 2
- 239000003826 tablet Substances 0.000 description 2
- 229940120982 tarceva Drugs 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- XOAAWQZATWQOTB-UHFFFAOYSA-N taurine Chemical compound NCCS(O)(=O)=O XOAAWQZATWQOTB-UHFFFAOYSA-N 0.000 description 2
- 229950003046 tesevatinib Drugs 0.000 description 2
- 150000003573 thiols Chemical class 0.000 description 2
- 210000001685 thyroid gland Anatomy 0.000 description 2
- 229960003087 tioguanine Drugs 0.000 description 2
- 239000006208 topical dosage form Substances 0.000 description 2
- UCFGDBYHRUNTLO-QHCPKHFHSA-N topotecan Chemical compound C1=C(O)C(CN(C)C)=C2C=C(CN3C4=CC5=C(C3=O)COC(=O)[C@]5(O)CC)C4=NC2=C1 UCFGDBYHRUNTLO-QHCPKHFHSA-N 0.000 description 2
- 230000001988 toxicity Effects 0.000 description 2
- 231100000419 toxicity Toxicity 0.000 description 2
- 238000012546 transfer Methods 0.000 description 2
- 230000009466 transformation Effects 0.000 description 2
- 238000003146 transient transfection Methods 0.000 description 2
- 238000013519 translation Methods 0.000 description 2
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 2
- 125000002023 trifluoromethyl group Chemical group FC(F)(F)* 0.000 description 2
- 229940094060 tykerb Drugs 0.000 description 2
- 241000701161 unidentified adenovirus Species 0.000 description 2
- UHTHHESEBZOYNR-UHFFFAOYSA-N vandetanib Chemical compound COC1=CC(C(/N=CN2)=N/C=3C(=CC(Br)=CC=3)F)=C2C=C1OCC1CCN(C)CC1 UHTHHESEBZOYNR-UHFFFAOYSA-N 0.000 description 2
- GPXBXXGIAQBQNI-UHFFFAOYSA-N vemurafenib Chemical compound CCCS(=O)(=O)NC1=CC=C(F)C(C(=O)C=2C3=CC(=CN=C3NC=2)C=2C=CC(Cl)=CC=2)=C1F GPXBXXGIAQBQNI-UHFFFAOYSA-N 0.000 description 2
- 238000011179 visual inspection Methods 0.000 description 2
- 238000001262 western blot Methods 0.000 description 2
- PVBORIXVWRTHOZ-UHFFFAOYSA-N (2,5-dioxopyrrol-1-yl)methyl cyclohexanecarboxylate Chemical compound C1CCCCC1C(=O)OCN1C(=O)C=CC1=O PVBORIXVWRTHOZ-UHFFFAOYSA-N 0.000 description 1
- IHVODYOQUSEYJJ-UHFFFAOYSA-N (2,5-dioxopyrrolidin-1-yl) 6-[[4-[(2,5-dioxopyrrol-1-yl)methyl]cyclohexanecarbonyl]amino]hexanoate Chemical compound O=C1CCC(=O)N1OC(=O)CCCCCNC(=O)C(CC1)CCC1CN1C(=O)C=CC1=O IHVODYOQUSEYJJ-UHFFFAOYSA-N 0.000 description 1
- USPAHWKEPYXFBE-UHFFFAOYSA-N (2-fluoro-5-methylphenyl)urea Chemical compound CC1=CC=C(F)C(NC(N)=O)=C1 USPAHWKEPYXFBE-UHFFFAOYSA-N 0.000 description 1
- WCWUXEGQKLTGDX-LLVKDONJSA-N (2R)-1-[[4-[(4-fluoro-2-methyl-1H-indol-5-yl)oxy]-5-methyl-6-pyrrolo[2,1-f][1,2,4]triazinyl]oxy]-2-propanol Chemical compound C1=C2NC(C)=CC2=C(F)C(OC2=NC=NN3C=C(C(=C32)C)OC[C@H](O)C)=C1 WCWUXEGQKLTGDX-LLVKDONJSA-N 0.000 description 1
- HSHPBORBOJIXSQ-HARLFGEKSA-N (2s)-1-[(2s)-2-cyclohexyl-2-[[(2s)-2-(methylamino)propanoyl]amino]acetyl]-n-[2-(1,3-oxazol-2-yl)-4-phenyl-1,3-thiazol-5-yl]pyrrolidine-2-carboxamide Chemical compound C1([C@H](NC(=O)[C@H](C)NC)C(=O)N2[C@@H](CCC2)C(=O)NC2=C(N=C(S2)C=2OC=CN=2)C=2C=CC=CC=2)CCCCC1 HSHPBORBOJIXSQ-HARLFGEKSA-N 0.000 description 1
- MHFUWOIXNMZFIW-WNQIDUERSA-N (2s)-2-hydroxypropanoic acid;n-[4-[4-(4-methylpiperazin-1-yl)-6-[(5-methyl-1h-pyrazol-3-yl)amino]pyrimidin-2-yl]sulfanylphenyl]cyclopropanecarboxamide Chemical compound C[C@H](O)C(O)=O.C1CN(C)CCN1C1=CC(NC2=NNC(C)=C2)=NC(SC=2C=CC(NC(=O)C3CC3)=CC=2)=N1 MHFUWOIXNMZFIW-WNQIDUERSA-N 0.000 description 1
- UFPFGVNKHCLJJO-SSKFGXFMSA-N (2s)-n-[(1s)-1-cyclohexyl-2-[(2s)-2-[4-(4-fluorobenzoyl)-1,3-thiazol-2-yl]pyrrolidin-1-yl]-2-oxoethyl]-2-(methylamino)propanamide Chemical compound C1([C@H](NC(=O)[C@H](C)NC)C(=O)N2[C@@H](CCC2)C=2SC=C(N=2)C(=O)C=2C=CC(F)=CC=2)CCCCC1 UFPFGVNKHCLJJO-SSKFGXFMSA-N 0.000 description 1
- NAALWFYYHHJEFQ-ZASNTINBSA-N (2s,5r,6r)-6-[[(2r)-2-[[6-[4-[bis(2-hydroxyethyl)sulfamoyl]phenyl]-2-oxo-1h-pyridine-3-carbonyl]amino]-2-(4-hydroxyphenyl)acetyl]amino]-3,3-dimethyl-7-oxo-4-thia-1-azabicyclo[3.2.0]heptane-2-carboxylic acid Chemical compound N([C@@H](C(=O)N[C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C=1C=CC(O)=CC=1)C(=O)C(C(N1)=O)=CC=C1C1=CC=C(S(=O)(=O)N(CCO)CCO)C=C1 NAALWFYYHHJEFQ-ZASNTINBSA-N 0.000 description 1
- JXOCDHNKTVLZLD-ITYLOYPMSA-N (3z)-n-(3-chlorophenyl)-3-[[3,5-dimethyl-4-(3-morpholin-4-ylpropyl)-1h-pyrrol-2-yl]methylidene]-n-methyl-2-oxo-1h-indole-5-sulfonamide Chemical compound C=1C=C2NC(=O)\C(=C/C3=C(C(CCCN4CCOCC4)=C(C)N3)C)C2=CC=1S(=O)(=O)N(C)C1=CC=CC(Cl)=C1 JXOCDHNKTVLZLD-ITYLOYPMSA-N 0.000 description 1
- RPHHPHGATVNVCI-UHFFFAOYSA-N (5-methylpyridin-2-yl)urea Chemical compound CC1=CC=C(NC(N)=O)N=C1 RPHHPHGATVNVCI-UHFFFAOYSA-N 0.000 description 1
- VNTHYLVDGVBPOU-QQYBVWGSSA-N (7s,9s)-9-acetyl-7-[(2r,4s,5s,6s)-4-amino-5-hydroxy-6-methyloxan-2-yl]oxy-6,9,11-trihydroxy-4-methoxy-8,10-dihydro-7h-tetracene-5,12-dione;2-hydroxypropane-1,2,3-tricarboxylic acid Chemical compound OC(=O)CC(O)(C(O)=O)CC(O)=O.O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 VNTHYLVDGVBPOU-QQYBVWGSSA-N 0.000 description 1
- FPVKHBSQESCIEP-UHFFFAOYSA-N (8S)-3-(2-deoxy-beta-D-erythro-pentofuranosyl)-3,6,7,8-tetrahydroimidazo[4,5-d][1,3]diazepin-8-ol Natural products C1C(O)C(CO)OC1N1C(NC=NCC2O)=C2N=C1 FPVKHBSQESCIEP-UHFFFAOYSA-N 0.000 description 1
- FDKXTQMXEQVLRF-ZHACJKMWSA-N (E)-dacarbazine Chemical compound CN(C)\N=N\c1[nH]cnc1C(N)=O FDKXTQMXEQVLRF-ZHACJKMWSA-N 0.000 description 1
- LKJPYSCBVHEWIU-KRWDZBQOSA-N (R)-bicalutamide Chemical compound C([C@@](O)(C)C(=O)NC=1C=C(C(C#N)=CC=1)C(F)(F)F)S(=O)(=O)C1=CC=C(F)C=C1 LKJPYSCBVHEWIU-KRWDZBQOSA-N 0.000 description 1
- SUVMJBTUFCVSAD-SNVBAGLBSA-N (R)-sulforaphane Chemical compound C[S@@](=O)CCCCN=C=S SUVMJBTUFCVSAD-SNVBAGLBSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- UKAUYVFTDYCKQA-UHFFFAOYSA-N -2-Amino-4-hydroxybutanoic acid Natural products OC(=O)C(N)CCO UKAUYVFTDYCKQA-UHFFFAOYSA-N 0.000 description 1
- DDMOUSALMHHKOS-UHFFFAOYSA-N 1,2-dichloro-1,1,2,2-tetrafluoroethane Chemical compound FC(F)(Cl)C(F)(F)Cl DDMOUSALMHHKOS-UHFFFAOYSA-N 0.000 description 1
- NKIJBSVPDYIEAT-UHFFFAOYSA-N 1,4,7,10-tetrazacyclododec-10-ene Chemical group C1CNCCN=CCNCCN1 NKIJBSVPDYIEAT-UHFFFAOYSA-N 0.000 description 1
- GLBZSOQDAOLMGC-UHFFFAOYSA-N 1-(2-hydroxy-2-methylpropyl)-n-[5-(7-methoxyquinolin-4-yl)oxypyridin-2-yl]-5-methyl-3-oxo-2-phenylpyrazole-4-carboxamide Chemical compound C=1C=NC2=CC(OC)=CC=C2C=1OC(C=N1)=CC=C1NC(=O)C(C1=O)=C(C)N(CC(C)(C)O)N1C1=CC=CC=C1 GLBZSOQDAOLMGC-UHFFFAOYSA-N 0.000 description 1
- 102100025573 1-alkyl-2-acetylglycerophosphocholine esterase Human genes 0.000 description 1
- PNDPGZBMCMUPRI-HVTJNCQCSA-N 10043-66-0 Chemical compound [131I][131I] PNDPGZBMCMUPRI-HVTJNCQCSA-N 0.000 description 1
- VGONTNSXDCQUGY-RRKCRQDMSA-N 2'-deoxyinosine Chemical group C1[C@H](O)[C@@H](CO)O[C@H]1N1C(N=CNC2=O)=C2N=C1 VGONTNSXDCQUGY-RRKCRQDMSA-N 0.000 description 1
- DVEXZJFMOKTQEZ-UHFFFAOYSA-N 2,3-bis[amino-[(2-aminophenyl)thio]methylidene]butanedinitrile Chemical compound C=1C=CC=C(N)C=1SC(N)=C(C#N)C(C#N)=C(N)SC1=CC=CC=C1N DVEXZJFMOKTQEZ-UHFFFAOYSA-N 0.000 description 1
- QPPUSXURIGJCFQ-UHFFFAOYSA-N 2-[(2-iodoacetyl)amino]propanoic acid Chemical compound OC(=O)C(C)NC(=O)CI QPPUSXURIGJCFQ-UHFFFAOYSA-N 0.000 description 1
- RTQWWZBSTRGEAV-PKHIMPSTSA-N 2-[[(2s)-2-[bis(carboxymethyl)amino]-3-[4-(methylcarbamoylamino)phenyl]propyl]-[2-[bis(carboxymethyl)amino]propyl]amino]acetic acid Chemical compound CNC(=O)NC1=CC=C(C[C@@H](CN(CC(C)N(CC(O)=O)CC(O)=O)CC(O)=O)N(CC(O)=O)CC(O)=O)C=C1 RTQWWZBSTRGEAV-PKHIMPSTSA-N 0.000 description 1
- FZDFGHZZPBUTGP-UHFFFAOYSA-N 2-[[2-[bis(carboxymethyl)amino]-3-(4-isothiocyanatophenyl)propyl]-[2-[bis(carboxymethyl)amino]propyl]amino]acetic acid Chemical compound OC(=O)CN(CC(O)=O)C(C)CN(CC(O)=O)CC(N(CC(O)=O)CC(O)=O)CC1=CC=C(N=C=S)C=C1 FZDFGHZZPBUTGP-UHFFFAOYSA-N 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- ZBMRKNMTMPPMMK-UHFFFAOYSA-N 2-amino-4-[hydroxy(methyl)phosphoryl]butanoic acid;azane Chemical compound [NH4+].CP(O)(=O)CCC(N)C([O-])=O ZBMRKNMTMPPMMK-UHFFFAOYSA-N 0.000 description 1
- DWXQWHJDTRJORM-UHFFFAOYSA-N 2-hydroxy-6-sulfobenzoic acid Chemical compound OC(=O)C1=C(O)C=CC=C1S(O)(=O)=O DWXQWHJDTRJORM-UHFFFAOYSA-N 0.000 description 1
- UAIUNKRWKOVEES-UHFFFAOYSA-N 3,3',5,5'-tetramethylbenzidine Chemical compound CC1=C(N)C(C)=CC(C=2C=C(C)C(N)=C(C)C=2)=C1 UAIUNKRWKOVEES-UHFFFAOYSA-N 0.000 description 1
- JYRHBYQGZJZANB-UHFFFAOYSA-N 3-[[2-[[2-[[2-[4-(2,5-dioxopyrrol-1-yl)butanoylamino]acetyl]amino]acetyl]amino]acetyl]amino]propanoic acid Chemical compound OC(=O)CCNC(=O)CNC(=O)CNC(=O)CNC(=O)CCCN1C(=O)C=CC1=O JYRHBYQGZJZANB-UHFFFAOYSA-N 0.000 description 1
- 125000004179 3-chlorophenyl group Chemical group [H]C1=C([H])C(*)=C([H])C(Cl)=C1[H] 0.000 description 1
- GUPXYSSGJWIURR-UHFFFAOYSA-N 3-octoxypropane-1,2-diol Chemical compound CCCCCCCCOCC(O)CO GUPXYSSGJWIURR-UHFFFAOYSA-N 0.000 description 1
- ALUXPRDJABOEAG-UHFFFAOYSA-N 4-(2,4-dichloro-5-methoxyanilino)-6-methoxy-7-[3-(4-methylpiperazin-1-yl)propoxy]quinoline-2-carbonitrile Chemical compound C1=C(Cl)C(OC)=CC(NC=2C3=CC(OC)=C(OCCCN4CCN(C)CC4)C=C3N=C(C=2)C#N)=C1Cl ALUXPRDJABOEAG-UHFFFAOYSA-N 0.000 description 1
- YLUGQOCWOPDIMD-UHFFFAOYSA-N 4-(dimethylamino)but-2-enamide Chemical compound CN(C)CC=CC(N)=O YLUGQOCWOPDIMD-UHFFFAOYSA-N 0.000 description 1
- NNPUPCNWEHWRPW-UHFFFAOYSA-N 4-(pyridin-2-yldisulfanyl)-2-sulfobutanoic acid Chemical compound OC(=O)C(S(O)(=O)=O)CCSSC1=CC=CC=N1 NNPUPCNWEHWRPW-UHFFFAOYSA-N 0.000 description 1
- GWEVSJHKQHAKNW-UHFFFAOYSA-N 4-[[1-(2,5-dioxopyrrolidin-1-yl)-2h-pyridin-2-yl]disulfanyl]-2-sulfobutanoic acid Chemical compound OC(=O)C(S(O)(=O)=O)CCSSC1C=CC=CN1N1C(=O)CCC1=O GWEVSJHKQHAKNW-UHFFFAOYSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- MMBZCFJKAQZVNI-VPENINKCSA-N 4-amino-5,6-difluoro-1-[(2r,4s,5r)-4-hydroxy-5-(hydroxymethyl)oxolan-2-yl]pyrimidin-2-one Chemical compound FC1=C(F)C(N)=NC(=O)N1[C@@H]1O[C@H](CO)[C@@H](O)C1 MMBZCFJKAQZVNI-VPENINKCSA-N 0.000 description 1
- ZRHDKBOBHHFLBW-UHFFFAOYSA-N 4-amino-5-fluoro-3-[6-(4-methylpiperazin-1-yl)-1H-benzimidazol-2-yl]-1H-quinolin-2-one 2-hydroxypropanoic acid Chemical compound CC(O)C(O)=O.C1CN(C)CCN1C1=CC=C(NC(=N2)C=3C(NC4=CC=CC(F)=C4C=3N)=O)C2=C1 ZRHDKBOBHHFLBW-UHFFFAOYSA-N 0.000 description 1
- ALKJNCZNEOTEMP-UHFFFAOYSA-N 4-n-(5-cyclopropyl-1h-pyrazol-3-yl)-6-(4-methylpiperazin-1-yl)-2-n-[(3-propan-2-yl-1,2-oxazol-5-yl)methyl]pyrimidine-2,4-diamine Chemical compound O1N=C(C(C)C)C=C1CNC1=NC(NC=2NN=C(C=2)C2CC2)=CC(N2CCN(C)CC2)=N1 ALKJNCZNEOTEMP-UHFFFAOYSA-N 0.000 description 1
- DEZJGRPRBZSAKI-KMGSDFBDSA-N 565434-85-7 Chemical compound C([C@@H](N)C(=O)N[C@H](CO)C(=O)N[C@H](CC=1C2=CC=CC=C2NC=1)C(=O)N[C@H](CO)C(=O)N[C@H](CC=1C(=C(F)C(F)=C(F)C=1F)F)C(=O)N[C@H](CC1CCCCC1)C(=O)N[C@H](CCCNC(N)=N)C(=O)N[C@H](CCCNC(N)=N)C(=O)N[C@H](CCCNC(N)=N)C(=O)N[C@H](CCC(N)=O)C(=O)N[C@H](CCCNC(N)=N)C(=O)N[C@H](CCCNC(N)=N)C(O)=O)C(C=C1)=CC=C1C(=O)C1=CC=CC=C1 DEZJGRPRBZSAKI-KMGSDFBDSA-N 0.000 description 1
- YXHLJMWYDTXDHS-IRFLANFNSA-N 7-aminoactinomycin D Chemical compound C[C@H]1OC(=O)[C@H](C(C)C)N(C)C(=O)CN(C)C(=O)[C@@H]2CCCN2C(=O)[C@@H](C(C)C)NC(=O)[C@H]1NC(=O)C1=C(N)C(=O)C(C)=C2OC(C(C)=C(N)C=C3C(=O)N[C@@H]4C(=O)N[C@@H](C(N5CCC[C@H]5C(=O)N(C)CC(=O)N(C)[C@@H](C(C)C)C(=O)O[C@@H]4C)=O)C(C)C)=C3N=C21 YXHLJMWYDTXDHS-IRFLANFNSA-N 0.000 description 1
- 108700012813 7-aminoactinomycin D Proteins 0.000 description 1
- CJIJXIFQYOPWTF-UHFFFAOYSA-N 7-hydroxycoumarin Natural products O1C(=O)C=CC2=CC(O)=CC=C21 CJIJXIFQYOPWTF-UHFFFAOYSA-N 0.000 description 1
- PBCZSGKMGDDXIJ-HQCWYSJUSA-N 7-hydroxystaurosporine Chemical compound N([C@H](O)C1=C2C3=CC=CC=C3N3C2=C24)C(=O)C1=C2C1=CC=CC=C1N4[C@H]1C[C@@H](NC)[C@@H](OC)[C@]3(C)O1 PBCZSGKMGDDXIJ-HQCWYSJUSA-N 0.000 description 1
- ZCYVEMRRCGMTRW-UHFFFAOYSA-N 7553-56-2 Chemical compound [I] ZCYVEMRRCGMTRW-UHFFFAOYSA-N 0.000 description 1
- PBCZSGKMGDDXIJ-UHFFFAOYSA-N 7beta-hydroxystaurosporine Natural products C12=C3N4C5=CC=CC=C5C3=C3C(O)NC(=O)C3=C2C2=CC=CC=C2N1C1CC(NC)C(OC)C4(C)O1 PBCZSGKMGDDXIJ-UHFFFAOYSA-N 0.000 description 1
- DALMAZHDNFCDRP-VMPREFPWSA-N 9h-fluoren-9-ylmethyl n-[(2s)-1-[[(2s)-5-(carbamoylamino)-1-[4-(hydroxymethyl)anilino]-1-oxopentan-2-yl]amino]-3-methyl-1-oxobutan-2-yl]carbamate Chemical compound O=C([C@H](CCCNC(N)=O)NC(=O)[C@@H](NC(=O)OCC1C2=CC=CC=C2C2=CC=CC=C21)C(C)C)NC1=CC=C(CO)C=C1 DALMAZHDNFCDRP-VMPREFPWSA-N 0.000 description 1
- KVLFRAWTRWDEDF-IRXDYDNUSA-N AZD-8055 Chemical compound C1=C(CO)C(OC)=CC=C1C1=CC=C(C(=NC(=N2)N3[C@H](COCC3)C)N3[C@H](COCC3)C)C2=N1 KVLFRAWTRWDEDF-IRXDYDNUSA-N 0.000 description 1
- VRQMAABPASPXMW-HDICACEKSA-N AZD4547 Chemical compound COC1=CC(OC)=CC(CCC=2NN=C(NC(=O)C=3C=CC(=CC=3)N3C[C@@H](C)N[C@@H](C)C3)C=2)=C1 VRQMAABPASPXMW-HDICACEKSA-N 0.000 description 1
- QTBSBXVTEAMEQO-UHFFFAOYSA-M Acetate Chemical compound CC([O-])=O QTBSBXVTEAMEQO-UHFFFAOYSA-M 0.000 description 1
- 108010022752 Acetylcholinesterase Proteins 0.000 description 1
- 102000012440 Acetylcholinesterase Human genes 0.000 description 1
- 239000012103 Alexa Fluor 488 Substances 0.000 description 1
- 239000012104 Alexa Fluor 500 Substances 0.000 description 1
- 239000012105 Alexa Fluor 514 Substances 0.000 description 1
- 239000012109 Alexa Fluor 568 Substances 0.000 description 1
- 239000012110 Alexa Fluor 594 Substances 0.000 description 1
- 239000012111 Alexa Fluor 610 Substances 0.000 description 1
- 239000012112 Alexa Fluor 633 Substances 0.000 description 1
- 239000012114 Alexa Fluor 647 Substances 0.000 description 1
- 239000012115 Alexa Fluor 660 Substances 0.000 description 1
- 239000012116 Alexa Fluor 680 Substances 0.000 description 1
- 239000012117 Alexa Fluor 700 Substances 0.000 description 1
- 239000012118 Alexa Fluor 750 Substances 0.000 description 1
- 239000012099 Alexa Fluor family Substances 0.000 description 1
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 1
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 1
- 108700028369 Alleles Proteins 0.000 description 1
- 102000013455 Amyloid beta-Peptides Human genes 0.000 description 1
- 108010090849 Amyloid beta-Peptides Proteins 0.000 description 1
- 108010032595 Antibody Binding Sites Proteins 0.000 description 1
- 229930182536 Antimycin Natural products 0.000 description 1
- 239000004475 Arginine Substances 0.000 description 1
- 102000015790 Asparaginase Human genes 0.000 description 1
- DCXYFEDJOCDNAF-UHFFFAOYSA-N Asparagine Natural products OC(=O)C(N)CC(N)=O DCXYFEDJOCDNAF-UHFFFAOYSA-N 0.000 description 1
- 208000023275 Autoimmune disease Diseases 0.000 description 1
- 108090001008 Avidin Proteins 0.000 description 1
- LQVXSNNAFNGRAH-QHCPKHFHSA-N BMS-754807 Chemical compound C([C@@]1(C)C(=O)NC=2C=NC(F)=CC=2)CCN1C(=NN1C=CC=C11)N=C1NC(=NN1)C=C1C1CC1 LQVXSNNAFNGRAH-QHCPKHFHSA-N 0.000 description 1
- 108010079882 Bax protein (53-86) Proteins 0.000 description 1
- 102100026189 Beta-galactosidase Human genes 0.000 description 1
- 102100023995 Beta-nerve growth factor Human genes 0.000 description 1
- PKWRMUKBEYJEIX-DXXQBUJASA-N Birinapant Chemical compound CN[C@@H](C)C(=O)N[C@@H](CC)C(=O)N1C[C@@H](O)C[C@H]1CC1=C(C2=C(C3=CC=C(F)C=C3N2)C[C@H]2N(C[C@@H](O)C2)C(=O)[C@H](CC)NC(=O)[C@H](C)NC)NC2=CC(F)=CC=C12 PKWRMUKBEYJEIX-DXXQBUJASA-N 0.000 description 1
- 208000019838 Blood disease Diseases 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 1
- 239000012619 Butyl Sepharose® Substances 0.000 description 1
- SRJDUPOPZMPQLK-UHFFFAOYSA-N C1(O)=NC(=C2C(=N1)N=CC=C2)C Chemical compound C1(O)=NC(=C2C(=N1)N=CC=C2)C SRJDUPOPZMPQLK-UHFFFAOYSA-N 0.000 description 1
- KAJYHKRSEVHEGX-HCQSHQBASA-N C1[C@@]([N+]([O-])=O)(C)[C@@H](NC(=O)OC)[C@@H](C)O[C@H]1O[C@@H]1C(/C)=C/[C@@H]2[C@@H](O)CC(C=O)=CC2(C2=O)OC(O)=C2C(=O)[C@@]2(C)[C@@H]3[C@@H](C)C[C@H](C)[C@H](O[C@@H]4O[C@@H](C)[C@H](OC(C)=O)[C@H](O[C@@H]5O[C@@H](C)[C@H](O[C@@H]6O[C@@H](C)[C@H](O[C@@H]7O[C@@H](C)[C@H](O)CC7)[C@H](O)C6)CC5)C4)[C@H]3C=C[C@H]2\C(C)=C/C1 Chemical compound C1[C@@]([N+]([O-])=O)(C)[C@@H](NC(=O)OC)[C@@H](C)O[C@H]1O[C@@H]1C(/C)=C/[C@@H]2[C@@H](O)CC(C=O)=CC2(C2=O)OC(O)=C2C(=O)[C@@]2(C)[C@@H]3[C@@H](C)C[C@H](C)[C@H](O[C@@H]4O[C@@H](C)[C@H](OC(C)=O)[C@H](O[C@@H]5O[C@@H](C)[C@H](O[C@@H]6O[C@@H](C)[C@H](O[C@@H]7O[C@@H](C)[C@H](O)CC7)[C@H](O)C6)CC5)C4)[C@H]3C=C[C@H]2\C(C)=C/C1 KAJYHKRSEVHEGX-HCQSHQBASA-N 0.000 description 1
- UXVMQQNJUSDDNG-UHFFFAOYSA-L Calcium chloride Chemical compound [Cl-].[Cl-].[Ca+2] UXVMQQNJUSDDNG-UHFFFAOYSA-L 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- GAGWJHPBXLXJQN-UHFFFAOYSA-N Capecitabine Natural products C1=C(F)C(NC(=O)OCCCCC)=NC(=O)N1C1C(O)C(O)C(C)O1 GAGWJHPBXLXJQN-UHFFFAOYSA-N 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 102000014914 Carrier Proteins Human genes 0.000 description 1
- 108010089388 Cdc25C phosphatase (211-221) Proteins 0.000 description 1
- 102000000844 Cell Surface Receptors Human genes 0.000 description 1
- 108010001857 Cell Surface Receptors Proteins 0.000 description 1
- VEXZGXHMUGYJMC-UHFFFAOYSA-M Chloride anion Chemical compound [Cl-] VEXZGXHMUGYJMC-UHFFFAOYSA-M 0.000 description 1
- 206010008631 Cholera Diseases 0.000 description 1
- PTOAARAWEBMLNO-KVQBGUIXSA-N Cladribine Chemical compound C1=NC=2C(N)=NC(Cl)=NC=2N1[C@H]1C[C@H](O)[C@@H](CO)O1 PTOAARAWEBMLNO-KVQBGUIXSA-N 0.000 description 1
- 241000759568 Corixa Species 0.000 description 1
- 241000938605 Crocodylia Species 0.000 description 1
- 102100021906 Cyclin-O Human genes 0.000 description 1
- 102100025698 Cytosolic carboxypeptidase 4 Human genes 0.000 description 1
- 101710112752 Cytotoxin Proteins 0.000 description 1
- IGXWBGJHJZYPQS-SSDOTTSWSA-N D-Luciferin Chemical compound OC(=O)[C@H]1CSC(C=2SC3=CC=C(O)C=C3N=2)=N1 IGXWBGJHJZYPQS-SSDOTTSWSA-N 0.000 description 1
- SHZGCJCMOBCMKK-UHFFFAOYSA-N D-mannomethylose Natural products CC1OC(O)C(O)C(O)C1O SHZGCJCMOBCMKK-UHFFFAOYSA-N 0.000 description 1
- 230000004544 DNA amplification Effects 0.000 description 1
- 230000006820 DNA synthesis Effects 0.000 description 1
- WEAHRLBPCANXCN-UHFFFAOYSA-N Daunomycin Natural products CCC1(O)CC(OC2CC(N)C(O)C(C)O2)c3cc4C(=O)c5c(OC)cccc5C(=O)c4c(O)c3C1 WEAHRLBPCANXCN-UHFFFAOYSA-N 0.000 description 1
- CYCGRDQQIOGCKX-UHFFFAOYSA-N Dehydro-luciferin Natural products OC(=O)C1=CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 CYCGRDQQIOGCKX-UHFFFAOYSA-N 0.000 description 1
- 102100037101 Deoxycytidylate deaminase Human genes 0.000 description 1
- 241000702421 Dependoparvovirus Species 0.000 description 1
- 239000004338 Dichlorodifluoromethane Substances 0.000 description 1
- 108090000204 Dipeptidase 1 Proteins 0.000 description 1
- MWWSFMDVAYGXBV-RUELKSSGSA-N Doxorubicin hydrochloride Chemical compound Cl.O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 MWWSFMDVAYGXBV-RUELKSSGSA-N 0.000 description 1
- 101100507451 Drosophila melanogaster sip3 gene Proteins 0.000 description 1
- 101150029707 ERBB2 gene Proteins 0.000 description 1
- 206010014733 Endometrial cancer Diseases 0.000 description 1
- 206010014759 Endometrial neoplasm Diseases 0.000 description 1
- 241000588914 Enterobacter Species 0.000 description 1
- 241000701959 Escherichia virus Lambda Species 0.000 description 1
- 108091008794 FGF receptors Proteins 0.000 description 1
- 229940125831 FGFR2 inhibitor Drugs 0.000 description 1
- 102100037362 Fibronectin Human genes 0.000 description 1
- 102000002090 Fibronectin type III Human genes 0.000 description 1
- 108050009401 Fibronectin type III Proteins 0.000 description 1
- 108010067306 Fibronectins Proteins 0.000 description 1
- BJGNCJDXODQBOB-UHFFFAOYSA-N Fivefly Luciferin Natural products OC(=O)C1CSC(C=2SC3=CC(O)=CC=C3N=2)=N1 BJGNCJDXODQBOB-UHFFFAOYSA-N 0.000 description 1
- PXGOKWXKJXAPGV-UHFFFAOYSA-N Fluorine Chemical compound FF PXGOKWXKJXAPGV-UHFFFAOYSA-N 0.000 description 1
- PNNNRSAQSRJVSB-SLPGGIOYSA-N Fucose Natural products C[C@H](O)[C@@H](O)[C@H](O)[C@H](O)C=O PNNNRSAQSRJVSB-SLPGGIOYSA-N 0.000 description 1
- 102000006471 Fucosyltransferases Human genes 0.000 description 1
- 108010019236 Fucosyltransferases Proteins 0.000 description 1
- GYHNNYVSQQEPJS-UHFFFAOYSA-N Gallium Chemical compound [Ga] GYHNNYVSQQEPJS-UHFFFAOYSA-N 0.000 description 1
- 241000287828 Gallus gallus Species 0.000 description 1
- 108010010803 Gelatin Proteins 0.000 description 1
- 108700028146 Genetic Enhancer Elements Proteins 0.000 description 1
- WHUUTDBJXJRKMK-UHFFFAOYSA-N Glutamic acid Natural products OC(=O)C(N)CCC(O)=O WHUUTDBJXJRKMK-UHFFFAOYSA-N 0.000 description 1
- SXRSQZLOMIGNAQ-UHFFFAOYSA-N Glutaraldehyde Chemical group O=CCCCC=O SXRSQZLOMIGNAQ-UHFFFAOYSA-N 0.000 description 1
- 102000051366 Glycosyltransferases Human genes 0.000 description 1
- 108700023372 Glycosyltransferases Proteins 0.000 description 1
- 108010026389 Gramicidin Proteins 0.000 description 1
- 239000007996 HEPPS buffer Substances 0.000 description 1
- GIZQLVPDAOBAFN-UHFFFAOYSA-N HEPPSO Chemical group OCCN1CCN(CC(O)CS(O)(=O)=O)CC1 GIZQLVPDAOBAFN-UHFFFAOYSA-N 0.000 description 1
- 229940125497 HER2 kinase inhibitor Drugs 0.000 description 1
- 241001622557 Hesperia Species 0.000 description 1
- 241000282412 Homo Species 0.000 description 1
- 101000897441 Homo sapiens Cyclin-O Proteins 0.000 description 1
- 101000955042 Homo sapiens Deoxycytidylate deaminase Proteins 0.000 description 1
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 102000003839 Human Proteins Human genes 0.000 description 1
- 241000701044 Human gammaherpesvirus 4 Species 0.000 description 1
- AVXURJPOCDRRFD-UHFFFAOYSA-N Hydroxylamine Chemical compound ON AVXURJPOCDRRFD-UHFFFAOYSA-N 0.000 description 1
- PMMYEEVYMWASQN-DMTCNVIQSA-N Hydroxyproline Chemical compound O[C@H]1CN[C@H](C(O)=O)C1 PMMYEEVYMWASQN-DMTCNVIQSA-N 0.000 description 1
- XQFRJNBWHJMXHO-RRKCRQDMSA-N IDUR Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C(I)=C1 XQFRJNBWHJMXHO-RRKCRQDMSA-N 0.000 description 1
- XDXDZDZNSLXDNA-TZNDIEGXSA-N Idarubicin Chemical compound C1[C@H](N)[C@H](O)[C@H](C)O[C@H]1O[C@@H]1C2=C(O)C(C(=O)C3=CC=CC=C3C3=O)=C3C(O)=C2C[C@@](O)(C(C)=O)C1 XDXDZDZNSLXDNA-TZNDIEGXSA-N 0.000 description 1
- 102100026120 IgG receptor FcRn large subunit p51 Human genes 0.000 description 1
- 101710177940 IgG receptor FcRn large subunit p51 Proteins 0.000 description 1
- 108700005091 Immunoglobulin Genes Proteins 0.000 description 1
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 1
- 206010061218 Inflammation Diseases 0.000 description 1
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 1
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical compound C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 1
- UKAUYVFTDYCKQA-VKHMYHEASA-N L-homoserine Chemical compound OC(=O)[C@@H](N)CCO UKAUYVFTDYCKQA-VKHMYHEASA-N 0.000 description 1
- AGPKZVBTJJNPAG-WHFBIAKZSA-N L-isoleucine Chemical compound CC[C@H](C)[C@H](N)C(O)=O AGPKZVBTJJNPAG-WHFBIAKZSA-N 0.000 description 1
- QEFRNWWLZKMPFJ-ZXPFJRLXSA-N L-methionine (R)-S-oxide Chemical compound C[S@@](=O)CC[C@H]([NH3+])C([O-])=O QEFRNWWLZKMPFJ-ZXPFJRLXSA-N 0.000 description 1
- QEFRNWWLZKMPFJ-UHFFFAOYSA-N L-methionine sulphoxide Natural products CS(=O)CCC(N)C(O)=O QEFRNWWLZKMPFJ-UHFFFAOYSA-N 0.000 description 1
- COLNVLDHVKWLRT-QMMMGPOBSA-N L-phenylalanine Chemical compound OC(=O)[C@@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-QMMMGPOBSA-N 0.000 description 1
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 1
- KZSNJWFQEVHDMF-BYPYZUCNSA-N L-valine Chemical compound CC(C)[C@H](N)C(O)=O KZSNJWFQEVHDMF-BYPYZUCNSA-N 0.000 description 1
- 239000005411 L01XE02 - Gefitinib Substances 0.000 description 1
- 239000002147 L01XE04 - Sunitinib Substances 0.000 description 1
- 239000002067 L01XE06 - Dasatinib Substances 0.000 description 1
- 239000003798 L01XE11 - Pazopanib Substances 0.000 description 1
- 239000002118 L01XE12 - Vandetanib Substances 0.000 description 1
- 239000002176 L01XE26 - Cabozantinib Substances 0.000 description 1
- NNJVILVZKWQKPM-UHFFFAOYSA-N Lidocaine Chemical compound CCN(CC)CC(=O)NC1=C(C)C=CC=C1C NNJVILVZKWQKPM-UHFFFAOYSA-N 0.000 description 1
- GQYIWUVLTXOXAJ-UHFFFAOYSA-N Lomustine Chemical compound ClCCN(N=O)C(=O)NC1CCCCC1 GQYIWUVLTXOXAJ-UHFFFAOYSA-N 0.000 description 1
- 108060001084 Luciferase Proteins 0.000 description 1
- 239000005089 Luciferase Substances 0.000 description 1
- DDWFXDSYGUXRAY-UHFFFAOYSA-N Luciferin Natural products CCc1c(C)c(CC2NC(=O)C(=C2C=C)C)[nH]c1Cc3[nH]c4C(=C5/NC(CC(=O)O)C(C)C5CC(=O)O)CC(=O)c4c3C DDWFXDSYGUXRAY-UHFFFAOYSA-N 0.000 description 1
- 206010025323 Lymphomas Diseases 0.000 description 1
- 241001441512 Maytenus serrata Species 0.000 description 1
- VFKZTMPDYBFSTM-KVTDHHQDSA-N Mitobronitol Chemical compound BrC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CBr VFKZTMPDYBFSTM-KVTDHHQDSA-N 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 239000004909 Moisturizer Substances 0.000 description 1
- ZOKXTWBITQBERF-AKLPVKDBSA-N Molybdenum Mo-99 Chemical compound [99Mo] ZOKXTWBITQBERF-AKLPVKDBSA-N 0.000 description 1
- 101000856426 Mus musculus Cullin-7 Proteins 0.000 description 1
- 241001553014 Myrsine salicina Species 0.000 description 1
- LKJPYSCBVHEWIU-UHFFFAOYSA-N N-[4-cyano-3-(trifluoromethyl)phenyl]-3-[(4-fluorophenyl)sulfonyl]-2-hydroxy-2-methylpropanamide Chemical compound C=1C=C(C#N)C(C(F)(F)F)=CC=1NC(=O)C(O)(C)CS(=O)(=O)C1=CC=C(F)C=C1 LKJPYSCBVHEWIU-UHFFFAOYSA-N 0.000 description 1
- XKFTZKGMDDZMJI-HSZRJFAPSA-N N-[5-[(2R)-2-methoxy-1-oxo-2-phenylethyl]-4,6-dihydro-1H-pyrrolo[3,4-c]pyrazol-3-yl]-4-(4-methyl-1-piperazinyl)benzamide Chemical compound O=C([C@H](OC)C=1C=CC=CC=1)N(CC=12)CC=1NN=C2NC(=O)C(C=C1)=CC=C1N1CCN(C)CC1 XKFTZKGMDDZMJI-HSZRJFAPSA-N 0.000 description 1
- OVRNDRQMDRJTHS-RTRLPJTCSA-N N-acetyl-D-glucosamine Chemical group CC(=O)N[C@H]1C(O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-RTRLPJTCSA-N 0.000 description 1
- GHAZCVNUKKZTLG-UHFFFAOYSA-N N-ethyl-succinimide Natural products CCN1C(=O)CCC1=O GHAZCVNUKKZTLG-UHFFFAOYSA-N 0.000 description 1
- HDFGOPSGAURCEO-UHFFFAOYSA-N N-ethylmaleimide Chemical compound CCN1C(=O)C=CC1=O HDFGOPSGAURCEO-UHFFFAOYSA-N 0.000 description 1
- 230000004988 N-glycosylation Effects 0.000 description 1
- 238000005481 NMR spectroscopy Methods 0.000 description 1
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 1
- 108091061960 Naked DNA Proteins 0.000 description 1
- 108010025020 Nerve Growth Factor Proteins 0.000 description 1
- 239000004677 Nylon Substances 0.000 description 1
- 239000012124 Opti-MEM Substances 0.000 description 1
- 239000012570 Opti-MEM I medium Substances 0.000 description 1
- 241000282320 Panthera leo Species 0.000 description 1
- 241001631646 Papillomaviridae Species 0.000 description 1
- 229930040373 Paraformaldehyde Natural products 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 241000233805 Phoenix Species 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000235648 Pichia Species 0.000 description 1
- 229920001054 Poly(ethylene‐co‐vinyl acetate) Polymers 0.000 description 1
- 229920002732 Polyanhydride Polymers 0.000 description 1
- 229920002562 Polyethylene Glycol 3350 Polymers 0.000 description 1
- 239000002202 Polyethylene glycol Substances 0.000 description 1
- 229920001710 Polyorthoester Polymers 0.000 description 1
- 239000004743 Polypropylene Substances 0.000 description 1
- 239000004793 Polystyrene Substances 0.000 description 1
- 229940124158 Protease/peptidase inhibitor Drugs 0.000 description 1
- 239000012614 Q-Sepharose Substances 0.000 description 1
- 108020005067 RNA Splice Sites Proteins 0.000 description 1
- JQYMGXZJTCOARG-UHFFFAOYSA-N Reactive blue 2 Chemical compound C1=2C(=O)C3=CC=CC=C3C(=O)C=2C(N)=C(S(O)(=O)=O)C=C1NC(C=C1S(O)(=O)=O)=CC=C1NC(N=1)=NC(Cl)=NC=1NC1=CC=CC(S(O)(=O)=O)=C1 JQYMGXZJTCOARG-UHFFFAOYSA-N 0.000 description 1
- 101710100969 Receptor tyrosine-protein kinase erbB-3 Proteins 0.000 description 1
- 102100029986 Receptor tyrosine-protein kinase erbB-3 Human genes 0.000 description 1
- 108020005091 Replication Origin Proteins 0.000 description 1
- 206010039491 Sarcoma Diseases 0.000 description 1
- 239000012506 Sephacryl® Substances 0.000 description 1
- MTCFGRXMJLQNBG-UHFFFAOYSA-N Serine Natural products OCC(N)C(O)=O MTCFGRXMJLQNBG-UHFFFAOYSA-N 0.000 description 1
- 241000607720 Serratia Species 0.000 description 1
- 241000607715 Serratia marcescens Species 0.000 description 1
- 108020004459 Small interfering RNA Proteins 0.000 description 1
- 229920002125 Sokalan® Polymers 0.000 description 1
- 229920002472 Starch Polymers 0.000 description 1
- 108091081024 Start codon Proteins 0.000 description 1
- ZSJLQEPLLKMAKR-UHFFFAOYSA-N Streptozotocin Natural products O=NN(C)C(=O)NC1C(O)OC(CO)C(O)C1O ZSJLQEPLLKMAKR-UHFFFAOYSA-N 0.000 description 1
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 1
- QAOWNCQODCNURD-UHFFFAOYSA-N Sulfuric acid Chemical compound OS(O)(=O)=O QAOWNCQODCNURD-UHFFFAOYSA-N 0.000 description 1
- 210000001744 T-lymphocyte Anatomy 0.000 description 1
- PZBFGYYEXUXCOF-UHFFFAOYSA-N TCEP Chemical compound OC(=O)CCP(CCC(O)=O)CCC(O)=O PZBFGYYEXUXCOF-UHFFFAOYSA-N 0.000 description 1
- 229940123237 Taxane Drugs 0.000 description 1
- 101710192266 Tegument protein VP22 Proteins 0.000 description 1
- WDLRUFUQRNWCPK-UHFFFAOYSA-N Tetraxetan Chemical compound OC(=O)CN1CCN(CC(O)=O)CCN(CC(O)=O)CCN(CC(O)=O)CC1 WDLRUFUQRNWCPK-UHFFFAOYSA-N 0.000 description 1
- QHOPXUFELLHKAS-UHFFFAOYSA-N Thespesin Natural products CC(C)c1c(O)c(O)c2C(O)Oc3c(c(C)cc1c23)-c1c2OC(O)c3c(O)c(O)c(C(C)C)c(cc1C)c23 QHOPXUFELLHKAS-UHFFFAOYSA-N 0.000 description 1
- FOCVUCIESVLUNU-UHFFFAOYSA-N Thiotepa Chemical compound C1CN1P(N1CC1)(=S)N1CC1 FOCVUCIESVLUNU-UHFFFAOYSA-N 0.000 description 1
- AYFVYJQAPQTCCC-UHFFFAOYSA-N Threonine Natural products CC(O)C(N)C(O)=O AYFVYJQAPQTCCC-UHFFFAOYSA-N 0.000 description 1
- 239000004473 Threonine Substances 0.000 description 1
- 208000033781 Thyroid carcinoma Diseases 0.000 description 1
- 102000003978 Tissue Plasminogen Activator Human genes 0.000 description 1
- 108090000373 Tissue Plasminogen Activator Proteins 0.000 description 1
- 101710120037 Toxin CcdB Proteins 0.000 description 1
- 108700019146 Transgenes Proteins 0.000 description 1
- 241000223259 Trichoderma Species 0.000 description 1
- YZCKVEUIGOORGS-NJFSPNSNSA-N Tritium Chemical compound [3H] YZCKVEUIGOORGS-NJFSPNSNSA-N 0.000 description 1
- 239000013504 Triton X-100 Substances 0.000 description 1
- 229920004890 Triton X-100 Polymers 0.000 description 1
- 108060008682 Tumor Necrosis Factor Proteins 0.000 description 1
- 102100033019 Tyrosine-protein phosphatase non-receptor type 11 Human genes 0.000 description 1
- 101710116241 Tyrosine-protein phosphatase non-receptor type 11 Proteins 0.000 description 1
- 241000700618 Vaccinia virus Species 0.000 description 1
- KZSNJWFQEVHDMF-UHFFFAOYSA-N Valine Natural products CC(C)C(N)C(O)=O KZSNJWFQEVHDMF-UHFFFAOYSA-N 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- 108010087302 Viral Structural Proteins Proteins 0.000 description 1
- 238000002441 X-ray diffraction Methods 0.000 description 1
- FHNFHKCVQCLJFQ-NJFSPNSNSA-N Xenon-133 Chemical compound [133Xe] FHNFHKCVQCLJFQ-NJFSPNSNSA-N 0.000 description 1
- CBPNZQVSJQDFBE-SREVRWKESA-N [(1S,2R,4S)-4-[(2R)-2-[(1R,9S,12S,15R,16E,18R,19R,21R,23S,24E,26E,28E,30S,32R,35R)-1,18-dihydroxy-19,30-dimethoxy-15,17,21,23,29,35-hexamethyl-2,3,10,14,20-pentaoxo-11,36-dioxa-4-azatricyclo[30.3.1.04,9]hexatriaconta-16,24,26,28-tetraen-12-yl]propyl]-2-methoxycyclohexyl] 3-hydroxy-2-(hydroxymethyl)-2-methylpropanoate Chemical compound C[C@@H]1CC[C@@H]2C[C@@H](/C(=C/C=C/C=C/[C@H](C[C@H](C(=O)[C@@H]([C@@H](/C(=C/[C@H](C(=O)C[C@H](OC(=O)[C@@H]3CCCCN3C(=O)C(=O)[C@@]1(O2)O)[C@H](C)C[C@@H]4CC[C@@H]([C@@H](C4)OC)OC(=O)C(C)(CO)CO)C)/C)O)OC)C)C)/C)OC CBPNZQVSJQDFBE-SREVRWKESA-N 0.000 description 1
- LTEJRLHKIYCEOX-PUODRLBUSA-N [(2r)-1-[4-[(4-fluoro-2-methyl-1h-indol-5-yl)oxy]-5-methylpyrrolo[2,1-f][1,2,4]triazin-6-yl]oxypropan-2-yl] 2-aminopropanoate Chemical compound C1=C2NC(C)=CC2=C(F)C(OC2=NC=NN3C=C(C(=C32)C)OC[C@@H](C)OC(=O)C(C)N)=C1 LTEJRLHKIYCEOX-PUODRLBUSA-N 0.000 description 1
- BTKMJKKKZATLBU-UHFFFAOYSA-N [2-(1,3-benzothiazol-2-yl)-1,3-benzothiazol-6-yl] dihydrogen phosphate Chemical compound C1=CC=C2SC(C3=NC4=CC=C(C=C4S3)OP(O)(=O)O)=NC2=C1 BTKMJKKKZATLBU-UHFFFAOYSA-N 0.000 description 1
- KDSLOZQCGWJINB-UHFFFAOYSA-N [F].ClN(C1=NN=NC=C1)Cl Chemical compound [F].ClN(C1=NN=NC=C1)Cl KDSLOZQCGWJINB-UHFFFAOYSA-N 0.000 description 1
- IQPTVTKRJHUUGI-UHFFFAOYSA-N [[1-(2,5-dioxopyrrolidin-1-yl)-2h-pyridin-2-yl]disulfanyl] propanoate Chemical compound CCC(=O)OSSC1C=CC=CN1N1C(=O)CCC1=O IQPTVTKRJHUUGI-UHFFFAOYSA-N 0.000 description 1
- 230000002159 abnormal effect Effects 0.000 description 1
- 239000008351 acetate buffer Substances 0.000 description 1
- NIOHNDKHQHVLKA-UHFFFAOYSA-N acetic acid;7-(8-formyl-1,6,7-trihydroxy-3-methyl-5-propan-2-ylnaphthalen-2-yl)-2,3,8-trihydroxy-6-methyl-4-propan-2-ylnaphthalene-1-carbaldehyde Chemical compound CC(O)=O.CC(C)C1=C(O)C(O)=C(C=O)C2=C(O)C(C=3C(O)=C4C(C=O)=C(O)C(O)=C(C4=CC=3C)C(C)C)=C(C)C=C21 NIOHNDKHQHVLKA-UHFFFAOYSA-N 0.000 description 1
- 229940022698 acetylcholinesterase Drugs 0.000 description 1
- 125000003668 acetyloxy group Chemical group [H]C([H])([H])C(=O)O[*] 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 229930183665 actinomycin Natural products 0.000 description 1
- 239000008186 active pharmaceutical agent Substances 0.000 description 1
- 238000011374 additional therapy Methods 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 229940009456 adriamycin Drugs 0.000 description 1
- 229940064305 adrucil Drugs 0.000 description 1
- 229960001686 afatinib Drugs 0.000 description 1
- ULXXDDBFHOBEHA-CWDCEQMOSA-N afatinib Chemical compound N1=CN=C2C=C(O[C@@H]3COCC3)C(NC(=O)/C=C/CN(C)C)=CC2=C1NC1=CC=C(F)C(Cl)=C1 ULXXDDBFHOBEHA-CWDCEQMOSA-N 0.000 description 1
- 229940042992 afinitor Drugs 0.000 description 1
- 238000013019 agitation Methods 0.000 description 1
- QNAYBMKLOCPYGJ-UHFFFAOYSA-M alaninate Chemical compound CC(N)C([O-])=O QNAYBMKLOCPYGJ-UHFFFAOYSA-M 0.000 description 1
- 150000001298 alcohols Chemical class 0.000 description 1
- 229940098174 alkeran Drugs 0.000 description 1
- 125000002947 alkylene group Chemical group 0.000 description 1
- HSFWRNGVRCDJHI-UHFFFAOYSA-N alpha-acetylene Natural products C#C HSFWRNGVRCDJHI-UHFFFAOYSA-N 0.000 description 1
- 150000001370 alpha-amino acid derivatives Chemical class 0.000 description 1
- 235000008206 alpha-amino acids Nutrition 0.000 description 1
- VREFGVBLTWBCJP-UHFFFAOYSA-N alprazolam Chemical compound C12=CC(Cl)=CC=C2N2C(C)=NN=C2CN=C1C1=CC=CC=C1 VREFGVBLTWBCJP-UHFFFAOYSA-N 0.000 description 1
- 229910052782 aluminium Inorganic materials 0.000 description 1
- XAGFODPZIPBFFR-UHFFFAOYSA-N aluminium Chemical compound [Al] XAGFODPZIPBFFR-UHFFFAOYSA-N 0.000 description 1
- 125000003368 amide group Chemical group 0.000 description 1
- 229960002932 anastrozole Drugs 0.000 description 1
- 229940035674 anesthetics Drugs 0.000 description 1
- 239000004037 angiogenesis inhibitor Substances 0.000 description 1
- 210000004102 animal cell Anatomy 0.000 description 1
- 230000001093 anti-cancer Effects 0.000 description 1
- 229940121363 anti-inflammatory agent Drugs 0.000 description 1
- 239000002260 anti-inflammatory agent Substances 0.000 description 1
- 230000000340 anti-metabolite Effects 0.000 description 1
- 239000000043 antiallergic agent Substances 0.000 description 1
- 229940088710 antibiotic agent Drugs 0.000 description 1
- 238000009175 antibody therapy Methods 0.000 description 1
- 229940100197 antimetabolite Drugs 0.000 description 1
- 239000002256 antimetabolite Substances 0.000 description 1
- 239000003080 antimitotic agent Substances 0.000 description 1
- CQIUKKVOEOPUDV-IYSWYEEDSA-N antimycin Chemical compound OC1=C(C(O)=O)C(=O)C(C)=C2[C@H](C)[C@@H](C)OC=C21 CQIUKKVOEOPUDV-IYSWYEEDSA-N 0.000 description 1
- 229940041181 antineoplastic drug Drugs 0.000 description 1
- 239000003096 antiparasitic agent Substances 0.000 description 1
- 229940125687 antiparasitic agent Drugs 0.000 description 1
- 239000003443 antiviral agent Substances 0.000 description 1
- 239000012736 aqueous medium Substances 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- PYMYPHUHKUWMLA-WDCZJNDASA-N arabinose Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)C=O PYMYPHUHKUWMLA-WDCZJNDASA-N 0.000 description 1
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 1
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 1
- 229940078010 arimidex Drugs 0.000 description 1
- 206010003246 arthritis Diseases 0.000 description 1
- 235000009582 asparagine Nutrition 0.000 description 1
- 229960001230 asparagine Drugs 0.000 description 1
- 235000003704 aspartic acid Nutrition 0.000 description 1
- 239000005441 aurora Substances 0.000 description 1
- 230000001363 autoimmune Effects 0.000 description 1
- 210000003050 axon Anatomy 0.000 description 1
- 230000003376 axonal effect Effects 0.000 description 1
- VSRXQHXAPYXROS-UHFFFAOYSA-N azanide;cyclobutane-1,1-dicarboxylic acid;platinum(2+) Chemical compound [NH2-].[NH2-].[Pt+2].OC(=O)C1(C(O)=O)CCC1 VSRXQHXAPYXROS-UHFFFAOYSA-N 0.000 description 1
- BWKOZPVPARTQIV-UHFFFAOYSA-N azanium;hydron;2-hydroxypropane-1,2,3-tricarboxylate Chemical compound [NH4+].OC(=O)CC(O)(C(O)=O)CC([O-])=O BWKOZPVPARTQIV-UHFFFAOYSA-N 0.000 description 1
- 210000003719 b-lymphocyte Anatomy 0.000 description 1
- 244000052616 bacterial pathogen Species 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- 230000008901 benefit Effects 0.000 description 1
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 1
- 108010005774 beta-Galactosidase Proteins 0.000 description 1
- OQFSQFPPLPISGP-UHFFFAOYSA-N beta-carboxyaspartic acid Natural products OC(=O)C(N)C(C(O)=O)C(O)=O OQFSQFPPLPISGP-UHFFFAOYSA-N 0.000 description 1
- 102000006635 beta-lactamase Human genes 0.000 description 1
- 229960000997 bicalutamide Drugs 0.000 description 1
- 229940108502 bicnu Drugs 0.000 description 1
- 230000001588 bifunctional effect Effects 0.000 description 1
- 108091008324 binding proteins Proteins 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 239000000090 biomarker Substances 0.000 description 1
- 238000001815 biotherapy Methods 0.000 description 1
- 108091006004 biotinylated proteins Proteins 0.000 description 1
- 108010063132 birinapant Proteins 0.000 description 1
- 229960001561 bleomycin Drugs 0.000 description 1
- OYVAGSVQBOHSSS-UAPAGMARSA-O bleomycin A2 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC=C(N=1)C=1SC=C(N=1)C(=O)NCCC[S+](C)C)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C OYVAGSVQBOHSSS-UAPAGMARSA-O 0.000 description 1
- 229960004395 bleomycin sulfate Drugs 0.000 description 1
- 230000036765 blood level Effects 0.000 description 1
- 229960003736 bosutinib Drugs 0.000 description 1
- UBPYILGKFZZVDX-UHFFFAOYSA-N bosutinib Chemical compound C1=C(Cl)C(OC)=CC(NC=2C3=CC(OC)=C(OCCCN4CCN(C)CC4)C=C3N=CC=2C#N)=C1Cl UBPYILGKFZZVDX-UHFFFAOYSA-N 0.000 description 1
- 210000004556 brain Anatomy 0.000 description 1
- 244000309464 bull Species 0.000 description 1
- 229950003628 buparlisib Drugs 0.000 description 1
- 239000001110 calcium chloride Substances 0.000 description 1
- 229910001628 calcium chloride Inorganic materials 0.000 description 1
- 238000004364 calculation method Methods 0.000 description 1
- 229930195731 calicheamicin Natural products 0.000 description 1
- HXCHCVDVKSCDHU-LULTVBGHSA-N calicheamicin Chemical compound C1[C@H](OC)[C@@H](NCC)CO[C@H]1O[C@H]1[C@H](O[C@@H]2C\3=C(NC(=O)OC)C(=O)C[C@](C/3=C/CSSSC)(O)C#C\C=C/C#C2)O[C@H](C)[C@@H](NO[C@@H]2O[C@H](C)[C@@H](SC(=O)C=3C(=C(OC)C(O[C@H]4[C@@H]([C@H](OC)[C@@H](O)[C@H](C)O4)O)=C(I)C=3C)OC)[C@@H](O)C2)[C@@H]1O HXCHCVDVKSCDHU-LULTVBGHSA-N 0.000 description 1
- 229940088954 camptosar Drugs 0.000 description 1
- 208000035269 cancer or benign tumor Diseases 0.000 description 1
- 229950002826 canertinib Drugs 0.000 description 1
- 229960004117 capecitabine Drugs 0.000 description 1
- 229940056434 caprelsa Drugs 0.000 description 1
- 239000004202 carbamide Substances 0.000 description 1
- 239000001569 carbon dioxide Substances 0.000 description 1
- 229910002092 carbon dioxide Inorganic materials 0.000 description 1
- 229960004424 carbon dioxide Drugs 0.000 description 1
- 125000002915 carbonyl group Chemical group [*:2]C([*:1])=O 0.000 description 1
- 125000003178 carboxy group Chemical group [H]OC(*)=O 0.000 description 1
- UHBYWPGGCSDKFX-UHFFFAOYSA-N carboxyglutamic acid Chemical compound OC(=O)C(N)CC(C(O)=O)C(O)=O UHBYWPGGCSDKFX-UHFFFAOYSA-N 0.000 description 1
- 150000001732 carboxylic acid derivatives Chemical class 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 229940097647 casodex Drugs 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 230000017455 cell-cell adhesion Effects 0.000 description 1
- 238000012054 celltiter-glo Methods 0.000 description 1
- 230000005889 cellular cytotoxicity Effects 0.000 description 1
- 230000036755 cellular response Effects 0.000 description 1
- 239000001913 cellulose Substances 0.000 description 1
- 229920002678 cellulose Polymers 0.000 description 1
- 210000003169 central nervous system Anatomy 0.000 description 1
- 239000000919 ceramic Substances 0.000 description 1
- 210000001638 cerebellum Anatomy 0.000 description 1
- 229960005395 cetuximab Drugs 0.000 description 1
- 230000008859 change Effects 0.000 description 1
- NDAYQJDHGXTBJL-MWWSRJDJSA-N chembl557217 Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](C(C)C)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](C(C)C)NC(=O)[C@H](C)NC(=O)[C@H](NC(=O)CNC(=O)[C@@H](NC=O)C(C)C)CC(C)C)C(=O)NCCO)=CNC2=C1 NDAYQJDHGXTBJL-MWWSRJDJSA-N 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 235000013330 chicken meat Nutrition 0.000 description 1
- JCKYGMPEJWAADB-UHFFFAOYSA-N chlorambucil Chemical compound OC(=O)CCCC1=CC=C(N(CCCl)CCCl)C=C1 JCKYGMPEJWAADB-UHFFFAOYSA-N 0.000 description 1
- 229960001265 ciclosporin Drugs 0.000 description 1
- 239000007979 citrate buffer Substances 0.000 description 1
- 229950006647 cixutumumab Drugs 0.000 description 1
- 229960002436 cladribine Drugs 0.000 description 1
- 238000004140 cleaning Methods 0.000 description 1
- 229960001338 colchicine Drugs 0.000 description 1
- 230000004154 complement system Effects 0.000 description 1
- 230000009918 complex formation Effects 0.000 description 1
- 238000007906 compression Methods 0.000 description 1
- 230000006835 compression Effects 0.000 description 1
- 238000009833 condensation Methods 0.000 description 1
- 230000005494 condensation Effects 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 239000000599 controlled substance Substances 0.000 description 1
- 238000007796 conventional method Methods 0.000 description 1
- 238000009109 curative therapy Methods 0.000 description 1
- 230000001086 cytosolic effect Effects 0.000 description 1
- 231100000050 cytotoxic potential Toxicity 0.000 description 1
- 230000003013 cytotoxicity Effects 0.000 description 1
- 231100000135 cytotoxicity Toxicity 0.000 description 1
- 229950006418 dactolisib Drugs 0.000 description 1
- 229960002448 dasatinib Drugs 0.000 description 1
- 229940052372 daunorubicin citrate liposome Drugs 0.000 description 1
- 229960003109 daunorubicin hydrochloride Drugs 0.000 description 1
- 229940107841 daunoxome Drugs 0.000 description 1
- 230000034994 death Effects 0.000 description 1
- 230000003247 decreasing effect Effects 0.000 description 1
- 230000007547 defect Effects 0.000 description 1
- 230000007850 degeneration Effects 0.000 description 1
- 230000003412 degenerative effect Effects 0.000 description 1
- 230000022811 deglycosylation Effects 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 239000005547 deoxyribonucleotide Substances 0.000 description 1
- 125000002637 deoxyribonucleotide group Chemical group 0.000 description 1
- 230000001419 dependent effect Effects 0.000 description 1
- 230000000779 depleting effect Effects 0.000 description 1
- 230000002074 deregulated effect Effects 0.000 description 1
- 230000036576 dermal application Effects 0.000 description 1
- 238000013461 design Methods 0.000 description 1
- 230000001627 detrimental effect Effects 0.000 description 1
- UQLDLKMNUJERMK-UHFFFAOYSA-L di(octadecanoyloxy)lead Chemical compound [Pb+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O UQLDLKMNUJERMK-UHFFFAOYSA-L 0.000 description 1
- NIJJYAXOARWZEE-UHFFFAOYSA-N di-n-propyl-acetic acid Natural products CCCC(C(O)=O)CCC NIJJYAXOARWZEE-UHFFFAOYSA-N 0.000 description 1
- 238000002405 diagnostic procedure Methods 0.000 description 1
- PXBRQCKWGAHEHS-UHFFFAOYSA-N dichlorodifluoromethane Chemical compound FC(F)(Cl)Cl PXBRQCKWGAHEHS-UHFFFAOYSA-N 0.000 description 1
- 235000019404 dichlorodifluoromethane Nutrition 0.000 description 1
- 229940042935 dichlorodifluoromethane Drugs 0.000 description 1
- 229940087091 dichlorotetrafluoroethane Drugs 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 238000009792 diffusion process Methods 0.000 description 1
- 239000003085 diluting agent Substances 0.000 description 1
- 238000006471 dimerization reaction Methods 0.000 description 1
- 125000002147 dimethylamino group Chemical group [H]C([H])([H])N(*)C([H])([H])[H] 0.000 description 1
- 206010013023 diphtheria Diseases 0.000 description 1
- 238000009826 distribution Methods 0.000 description 1
- PMMYEEVYMWASQN-UHFFFAOYSA-N dl-hydroxyproline Natural products OC1C[NH2+]C(C([O-])=O)C1 PMMYEEVYMWASQN-UHFFFAOYSA-N 0.000 description 1
- 229960003668 docetaxel Drugs 0.000 description 1
- 230000007783 downstream signaling Effects 0.000 description 1
- 229960002918 doxorubicin hydrochloride Drugs 0.000 description 1
- 239000000890 drug combination Substances 0.000 description 1
- 238000012377 drug delivery Methods 0.000 description 1
- 229940126534 drug product Drugs 0.000 description 1
- 239000012893 effector ligand Substances 0.000 description 1
- 229940099302 efudex Drugs 0.000 description 1
- 229940121647 egfr inhibitor Drugs 0.000 description 1
- 230000005611 electricity Effects 0.000 description 1
- 229940073038 elspar Drugs 0.000 description 1
- 239000003480 eluent Substances 0.000 description 1
- 238000010828 elution Methods 0.000 description 1
- 239000012149 elution buffer Substances 0.000 description 1
- 201000003914 endometrial carcinoma Diseases 0.000 description 1
- 230000002357 endometrial effect Effects 0.000 description 1
- 210000001163 endosome Anatomy 0.000 description 1
- RDYMFSUJUZBWLH-UHFFFAOYSA-N endosulfan Chemical compound C12COS(=O)OCC2C2(Cl)C(Cl)=C(Cl)C1(Cl)C2(Cl)Cl RDYMFSUJUZBWLH-UHFFFAOYSA-N 0.000 description 1
- 230000006862 enzymatic digestion Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 210000000981 epithelium Anatomy 0.000 description 1
- 238000011067 equilibration Methods 0.000 description 1
- 229940082789 erbitux Drugs 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 125000000031 ethylamino group Chemical group [H]C([H])([H])C([H])([H])N([H])[*] 0.000 description 1
- LYCAIKOWRPUZTN-UHFFFAOYSA-N ethylene glycol Natural products OCCO LYCAIKOWRPUZTN-UHFFFAOYSA-N 0.000 description 1
- 125000002534 ethynyl group Chemical group [H]C#C* 0.000 description 1
- 229960005420 etoposide Drugs 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 229960005167 everolimus Drugs 0.000 description 1
- 230000029142 excretion Effects 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 231100000776 exotoxin Toxicity 0.000 description 1
- 239000002095 exotoxin Substances 0.000 description 1
- 239000013613 expression plasmid Substances 0.000 description 1
- 230000002550 fecal effect Effects 0.000 description 1
- 102000052178 fibroblast growth factor receptor activity proteins Human genes 0.000 description 1
- 229940125829 fibroblast growth factor receptor inhibitor Drugs 0.000 description 1
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 1
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 1
- 239000011737 fluorine Substances 0.000 description 1
- 229910052731 fluorine Inorganic materials 0.000 description 1
- 239000011888 foil Substances 0.000 description 1
- 229940014144 folate Drugs 0.000 description 1
- OVBPIULPVIDEAO-LBPRGKRZSA-N folic acid Chemical compound C=1N=C2NC(N)=NC(=O)C2=NC=1CNC1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 OVBPIULPVIDEAO-LBPRGKRZSA-N 0.000 description 1
- 235000019152 folic acid Nutrition 0.000 description 1
- 239000011724 folic acid Substances 0.000 description 1
- 230000008014 freezing Effects 0.000 description 1
- 238000007710 freezing Methods 0.000 description 1
- 125000002446 fucosyl group Chemical group C1([C@@H](O)[C@H](O)[C@H](O)[C@@H](O1)C)* 0.000 description 1
- 238000002825 functional assay Methods 0.000 description 1
- 125000000524 functional group Chemical group 0.000 description 1
- 230000002538 fungal effect Effects 0.000 description 1
- 101150023212 fut8 gene Proteins 0.000 description 1
- 229910052733 gallium Inorganic materials 0.000 description 1
- 239000007789 gas Substances 0.000 description 1
- 239000007792 gaseous phase Substances 0.000 description 1
- 210000001156 gastric mucosa Anatomy 0.000 description 1
- 229960002584 gefitinib Drugs 0.000 description 1
- 239000008273 gelatin Substances 0.000 description 1
- 229920000159 gelatin Polymers 0.000 description 1
- 235000019322 gelatine Nutrition 0.000 description 1
- 235000011852 gelatine desserts Nutrition 0.000 description 1
- 229960005277 gemcitabine Drugs 0.000 description 1
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 1
- 239000003193 general anesthetic agent Substances 0.000 description 1
- 229940080856 gleevec Drugs 0.000 description 1
- 229940084910 gliadel Drugs 0.000 description 1
- 239000003862 glucocorticoid Substances 0.000 description 1
- 235000013922 glutamic acid Nutrition 0.000 description 1
- 239000004220 glutamic acid Substances 0.000 description 1
- ZDXPYRJPNDTMRX-UHFFFAOYSA-N glutamine Natural products OC(=O)C(N)CCC(N)=O ZDXPYRJPNDTMRX-UHFFFAOYSA-N 0.000 description 1
- 150000004676 glycans Chemical class 0.000 description 1
- XDDAORKBJWWYJS-UHFFFAOYSA-N glyphosate Chemical compound OC(=O)CNCP(O)(O)=O XDDAORKBJWWYJS-UHFFFAOYSA-N 0.000 description 1
- 229930000755 gossypol Natural products 0.000 description 1
- 229950005277 gossypol Drugs 0.000 description 1
- 230000005484 gravity Effects 0.000 description 1
- 230000035876 healing Effects 0.000 description 1
- 239000000185 hemagglutinin Substances 0.000 description 1
- 208000014951 hematologic disease Diseases 0.000 description 1
- 208000018706 hematopoietic system disease Diseases 0.000 description 1
- 229940022353 herceptin Drugs 0.000 description 1
- 125000004051 hexyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])* 0.000 description 1
- 238000004128 high performance liquid chromatography Methods 0.000 description 1
- 238000007825 histological assay Methods 0.000 description 1
- 210000000688 human artificial chromosome Anatomy 0.000 description 1
- 239000003906 humectant Substances 0.000 description 1
- 229940096120 hydrea Drugs 0.000 description 1
- 125000001183 hydrocarbyl group Chemical group 0.000 description 1
- 150000002431 hydrogen Chemical class 0.000 description 1
- 150000002433 hydrophilic molecules Chemical class 0.000 description 1
- 125000004356 hydroxy functional group Chemical group O* 0.000 description 1
- WGCNASOHLSPBMP-UHFFFAOYSA-N hydroxyacetaldehyde Natural products OCC=O WGCNASOHLSPBMP-UHFFFAOYSA-N 0.000 description 1
- 229960001330 hydroxycarbamide Drugs 0.000 description 1
- 229960002591 hydroxyproline Drugs 0.000 description 1
- 229960001001 ibritumomab tiuxetan Drugs 0.000 description 1
- 229940099279 idamycin Drugs 0.000 description 1
- 229940090411 ifex Drugs 0.000 description 1
- 229960001101 ifosfamide Drugs 0.000 description 1
- 238000003384 imaging method Methods 0.000 description 1
- 229960003685 imatinib mesylate Drugs 0.000 description 1
- YLMAHDNUQAMNNX-UHFFFAOYSA-N imatinib methanesulfonate Chemical compound CS(O)(=O)=O.C1CN(C)CCN1CC1=CC=C(C(=O)NC=2C=C(NC=3N=C(C=CN=3)C=3C=NC=CC=3)C(C)=CC=2)C=C1 YLMAHDNUQAMNNX-UHFFFAOYSA-N 0.000 description 1
- 238000007654 immersion Methods 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 238000002649 immunization Methods 0.000 description 1
- 230000001571 immunoadjuvant effect Effects 0.000 description 1
- 238000003125 immunofluorescent labeling Methods 0.000 description 1
- 239000000568 immunological adjuvant Substances 0.000 description 1
- 239000002955 immunomodulating agent Substances 0.000 description 1
- 229960003444 immunosuppressant agent Drugs 0.000 description 1
- 239000003018 immunosuppressive agent Substances 0.000 description 1
- 230000001771 impaired effect Effects 0.000 description 1
- 239000012535 impurity Substances 0.000 description 1
- 238000011078 in-house production Methods 0.000 description 1
- 229910052738 indium Inorganic materials 0.000 description 1
- APFVFJFRJDLVQX-UHFFFAOYSA-N indium atom Chemical compound [In] APFVFJFRJDLVQX-UHFFFAOYSA-N 0.000 description 1
- 229940055742 indium-111 Drugs 0.000 description 1
- 239000000411 inducer Substances 0.000 description 1
- 230000002757 inflammatory effect Effects 0.000 description 1
- 230000004054 inflammatory process Effects 0.000 description 1
- 206010022000 influenza Diseases 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000000977 initiatory effect Effects 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 239000000138 intercalating agent Substances 0.000 description 1
- 238000005305 interferometry Methods 0.000 description 1
- 238000010212 intracellular staining Methods 0.000 description 1
- 238000007913 intrathecal administration Methods 0.000 description 1
- 239000011630 iodine Substances 0.000 description 1
- 229910052740 iodine Inorganic materials 0.000 description 1
- PGLTVOMIXTUURA-UHFFFAOYSA-N iodoacetamide Chemical compound NC(=O)CI PGLTVOMIXTUURA-UHFFFAOYSA-N 0.000 description 1
- 239000003456 ion exchange resin Substances 0.000 description 1
- 229920003303 ion-exchange polymer Polymers 0.000 description 1
- 150000002500 ions Chemical class 0.000 description 1
- 229940084651 iressa Drugs 0.000 description 1
- 229960004768 irinotecan Drugs 0.000 description 1
- 125000000959 isobutyl group Chemical group [H]C([H])([H])C([H])(C([H])([H])[H])C([H])([H])* 0.000 description 1
- 238000002955 isolation Methods 0.000 description 1
- 229960000310 isoleucine Drugs 0.000 description 1
- AGPKZVBTJJNPAG-UHFFFAOYSA-N isoleucine Natural products CCC(C)C(N)C(O)=O AGPKZVBTJJNPAG-UHFFFAOYSA-N 0.000 description 1
- 125000001449 isopropyl group Chemical group [H]C([H])([H])C([H])(*)C([H])([H])[H] 0.000 description 1
- 229930027917 kanamycin Natural products 0.000 description 1
- 229960000318 kanamycin Drugs 0.000 description 1
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 1
- 229930182823 kanamycin A Natural products 0.000 description 1
- 230000002147 killing effect Effects 0.000 description 1
- 101150066555 lacZ gene Proteins 0.000 description 1
- 229960000448 lactic acid Drugs 0.000 description 1
- 229960004891 lapatinib Drugs 0.000 description 1
- 108010034897 lentil lectin Proteins 0.000 description 1
- 208000032839 leukemia Diseases 0.000 description 1
- 229940063725 leukeran Drugs 0.000 description 1
- 229960004194 lidocaine Drugs 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 238000012417 linear regression Methods 0.000 description 1
- MPVGZUGXCQEXTM-UHFFFAOYSA-N linifanib Chemical compound CC1=CC=C(F)C(NC(=O)NC=2C=CC(=CC=2)C=2C=3C(N)=NNC=3C=CC=2)=C1 MPVGZUGXCQEXTM-UHFFFAOYSA-N 0.000 description 1
- 229950001762 linsitinib Drugs 0.000 description 1
- PKCDDUHJAFVJJB-VLZXCDOPSA-N linsitinib Chemical compound C1[C@](C)(O)C[C@@H]1C1=NC(C=2C=C3N=C(C=CC3=CC=2)C=2C=CC=CC=2)=C2N1C=CN=C2N PKCDDUHJAFVJJB-VLZXCDOPSA-N 0.000 description 1
- 150000002632 lipids Chemical class 0.000 description 1
- 238000001638 lipofection Methods 0.000 description 1
- 239000007791 liquid phase Substances 0.000 description 1
- 210000005229 liver cell Anatomy 0.000 description 1
- 239000003589 local anesthetic agent Substances 0.000 description 1
- 230000033001 locomotion Effects 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- HWYHZTIRURJOHG-UHFFFAOYSA-N luminol Chemical compound O=C1NNC(=O)C2=C1C(N)=CC=C2 HWYHZTIRURJOHG-UHFFFAOYSA-N 0.000 description 1
- 210000004072 lung Anatomy 0.000 description 1
- OHSVLFRHMCKCQY-NJFSPNSNSA-N lutetium-177 Chemical compound [177Lu] OHSVLFRHMCKCQY-NJFSPNSNSA-N 0.000 description 1
- 239000008176 lyophilized powder Substances 0.000 description 1
- 125000003588 lysine group Chemical group [H]N([H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C([H])(N([H])[H])C(*)=O 0.000 description 1
- 230000002132 lysosomal effect Effects 0.000 description 1
- 210000003712 lysosome Anatomy 0.000 description 1
- 230000001868 lysosomic effect Effects 0.000 description 1
- 239000012516 mab select resin Substances 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 229950001869 mapatumumab Drugs 0.000 description 1
- 229950008001 matuzumab Drugs 0.000 description 1
- 230000001404 mediated effect Effects 0.000 description 1
- 238000002483 medication Methods 0.000 description 1
- 229910052751 metal Inorganic materials 0.000 description 1
- 239000002184 metal Substances 0.000 description 1
- 150000002739 metals Chemical class 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 230000001394 metastastic effect Effects 0.000 description 1
- 125000004184 methoxymethyl group Chemical group [H]C([H])([H])OC([H])([H])* 0.000 description 1
- CPMDPSXJELVGJG-UHFFFAOYSA-N methyl 2-hydroxy-3-[N-[4-[methyl-[2-(4-methylpiperazin-1-yl)acetyl]amino]phenyl]-C-phenylcarbonimidoyl]-1H-indole-6-carboxylate Chemical compound OC=1NC2=CC(=CC=C2C=1C(=NC1=CC=C(C=C1)N(C(CN1CCN(CC1)C)=O)C)C1=CC=CC=C1)C(=O)OC CPMDPSXJELVGJG-UHFFFAOYSA-N 0.000 description 1
- YACKEPLHDIMKIO-UHFFFAOYSA-N methylphosphonic acid Chemical compound CP(O)(O)=O YACKEPLHDIMKIO-UHFFFAOYSA-N 0.000 description 1
- LSDPWZHWYPCBBB-UHFFFAOYSA-O methylsulfide anion Chemical compound [SH2+]C LSDPWZHWYPCBBB-UHFFFAOYSA-O 0.000 description 1
- 125000004170 methylsulfonyl group Chemical group [H]C([H])([H])S(*)(=O)=O 0.000 description 1
- 230000000813 microbial effect Effects 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 238000000386 microscopy Methods 0.000 description 1
- 238000013508 migration Methods 0.000 description 1
- 230000005012 migration Effects 0.000 description 1
- 235000013336 milk Nutrition 0.000 description 1
- 239000008267 milk Substances 0.000 description 1
- 210000004080 milk Anatomy 0.000 description 1
- 231100000324 minimal toxicity Toxicity 0.000 description 1
- 239000007758 minimum essential medium Substances 0.000 description 1
- 239000003595 mist Substances 0.000 description 1
- 229960005485 mitobronitol Drugs 0.000 description 1
- 229960001156 mitoxantrone Drugs 0.000 description 1
- 108091005601 modified peptides Proteins 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- 230000001333 moisturizer Effects 0.000 description 1
- 230000004001 molecular interaction Effects 0.000 description 1
- 239000003068 molecular probe Substances 0.000 description 1
- 238000012544 monitoring process Methods 0.000 description 1
- 239000000178 monomer Substances 0.000 description 1
- 238000010172 mouse model Methods 0.000 description 1
- ZTFBIUXIQYRUNT-MDWZMJQESA-N mubritinib Chemical compound C1=CC(C(F)(F)F)=CC=C1\C=C\C1=NC(COC=2C=CC(CCCCN3N=NC=C3)=CC=2)=CO1 ZTFBIUXIQYRUNT-MDWZMJQESA-N 0.000 description 1
- 238000002703 mutagenesis Methods 0.000 description 1
- 231100000350 mutagenesis Toxicity 0.000 description 1
- 229940090009 myleran Drugs 0.000 description 1
- AZBFJBJXUQUQLF-UHFFFAOYSA-N n-(1,5-dimethylpyrrolidin-3-yl)pyrrolidine-1-carboxamide Chemical compound C1N(C)C(C)CC1NC(=O)N1CCCC1 AZBFJBJXUQUQLF-UHFFFAOYSA-N 0.000 description 1
- RDSACQWTXKSHJT-UHFFFAOYSA-N n-[3,4-difluoro-2-(2-fluoro-4-iodoanilino)-6-methoxyphenyl]-1-(2,3-dihydroxypropyl)cyclopropane-1-sulfonamide Chemical compound C1CC1(CC(O)CO)S(=O)(=O)NC=1C(OC)=CC(F)=C(F)C=1NC1=CC=C(I)C=C1F RDSACQWTXKSHJT-UHFFFAOYSA-N 0.000 description 1
- RDSACQWTXKSHJT-NSHDSACASA-N n-[3,4-difluoro-2-(2-fluoro-4-iodoanilino)-6-methoxyphenyl]-1-[(2s)-2,3-dihydroxypropyl]cyclopropane-1-sulfonamide Chemical compound C1CC1(C[C@H](O)CO)S(=O)(=O)NC=1C(OC)=CC(F)=C(F)C=1NC1=CC=C(I)C=C1F RDSACQWTXKSHJT-NSHDSACASA-N 0.000 description 1
- 125000004108 n-butyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])C([H])([H])* 0.000 description 1
- 125000004123 n-propyl group Chemical group [H]C([H])([H])C([H])([H])C([H])([H])* 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 229940086322 navelbine Drugs 0.000 description 1
- 210000005170 neoplastic cell Anatomy 0.000 description 1
- 230000001613 neoplastic effect Effects 0.000 description 1
- 229950008835 neratinib Drugs 0.000 description 1
- JWNPDZNEKVCWMY-VQHVLOKHSA-N neratinib Chemical compound C=12C=C(NC(=O)\C=C\CN(C)C)C(OCC)=CC2=NC=C(C#N)C=1NC(C=C1Cl)=CC=C1OCC1=CC=CC=N1 JWNPDZNEKVCWMY-VQHVLOKHSA-N 0.000 description 1
- 229940053128 nerve growth factor Drugs 0.000 description 1
- 210000000653 nervous system Anatomy 0.000 description 1
- 230000001537 neural effect Effects 0.000 description 1
- 210000002569 neuron Anatomy 0.000 description 1
- 108010087904 neutravidin Proteins 0.000 description 1
- 229940080607 nexavar Drugs 0.000 description 1
- 229960001346 nilotinib Drugs 0.000 description 1
- 229960004378 nintedanib Drugs 0.000 description 1
- XZXHXSATPCNXJR-ZIADKAODSA-N nintedanib Chemical compound O=C1NC2=CC(C(=O)OC)=CC=C2\C1=C(C=1C=CC=CC=1)\NC(C=C1)=CC=C1N(C)C(=O)CN1CCN(C)CC1 XZXHXSATPCNXJR-ZIADKAODSA-N 0.000 description 1
- 208000037916 non-allergic rhinitis Diseases 0.000 description 1
- 238000012758 nuclear staining Methods 0.000 description 1
- 229920001778 nylon Polymers 0.000 description 1
- QYSGYZVSCZSLHT-UHFFFAOYSA-N octafluoropropane Chemical compound FC(F)(F)C(F)(F)C(F)(F)F QYSGYZVSCZSLHT-UHFFFAOYSA-N 0.000 description 1
- 238000011275 oncology therapy Methods 0.000 description 1
- 230000003287 optical effect Effects 0.000 description 1
- 210000000056 organ Anatomy 0.000 description 1
- 229940127084 other anti-cancer agent Drugs 0.000 description 1
- 210000001672 ovary Anatomy 0.000 description 1
- 125000003854 p-chlorophenyl group Chemical group [H]C1=C([H])C(*)=C([H])C([H])=C1Cl 0.000 description 1
- 238000010979 pH adjustment Methods 0.000 description 1
- 229910052763 palladium Inorganic materials 0.000 description 1
- 210000000496 pancreas Anatomy 0.000 description 1
- 229960001972 panitumumab Drugs 0.000 description 1
- 229920002866 paraformaldehyde Polymers 0.000 description 1
- 230000005298 paramagnetic effect Effects 0.000 description 1
- CUIHSIWYWATEQL-UHFFFAOYSA-N pazopanib Chemical compound C1=CC2=C(C)N(C)N=C2C=C1N(C)C(N=1)=CC=NC=1NC1=CC=C(C)C(S(N)(=O)=O)=C1 CUIHSIWYWATEQL-UHFFFAOYSA-N 0.000 description 1
- WVUNYSQLFKLYNI-AATRIKPKSA-N pelitinib Chemical compound C=12C=C(NC(=O)\C=C\CN(C)C)C(OCC)=CC2=NC=C(C#N)C=1NC1=CC=C(F)C(Cl)=C1 WVUNYSQLFKLYNI-AATRIKPKSA-N 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 229960002340 pentostatin Drugs 0.000 description 1
- FPVKHBSQESCIEP-JQCXWYLXSA-N pentostatin Chemical compound C1[C@H](O)[C@@H](CO)O[C@H]1N1C(N=CNC[C@H]2O)=C2N=C1 FPVKHBSQESCIEP-JQCXWYLXSA-N 0.000 description 1
- 125000001147 pentyl group Chemical group C(CCCC)* 0.000 description 1
- 239000000137 peptide hydrolase inhibitor Substances 0.000 description 1
- 102000013415 peroxidase activity proteins Human genes 0.000 description 1
- 108040007629 peroxidase activity proteins Proteins 0.000 description 1
- 229960002087 pertuzumab Drugs 0.000 description 1
- 239000000825 pharmaceutical preparation Substances 0.000 description 1
- 230000000144 pharmacologic effect Effects 0.000 description 1
- 230000000079 pharmacotherapeutic effect Effects 0.000 description 1
- COLNVLDHVKWLRT-UHFFFAOYSA-N phenylalanine Natural products OC(=O)C(N)CC1=CC=CC=C1 COLNVLDHVKWLRT-UHFFFAOYSA-N 0.000 description 1
- 125000003356 phenylsulfanyl group Chemical group [*]SC1=C([H])C([H])=C([H])C([H])=C1[H] 0.000 description 1
- 150000004713 phosphodiesters Chemical class 0.000 description 1
- PTMHPRAIXMAOOB-UHFFFAOYSA-L phosphoramidate Chemical compound NP([O-])([O-])=O PTMHPRAIXMAOOB-UHFFFAOYSA-L 0.000 description 1
- LFGREXWGYUGZLY-UHFFFAOYSA-N phosphoryl Chemical group [P]=O LFGREXWGYUGZLY-UHFFFAOYSA-N 0.000 description 1
- BZQFBWGGLXLEPQ-REOHCLBHSA-N phosphoserine Chemical compound OC(=O)[C@@H](N)COP(O)(O)=O BZQFBWGGLXLEPQ-REOHCLBHSA-N 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 230000006461 physiological response Effects 0.000 description 1
- YJGVMLPVUAXIQN-HAEOHBJNSA-N picropodophyllotoxin Chemical compound COC1=C(OC)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@H](O)[C@@H]3[C@H]2C(OC3)=O)=C1 YJGVMLPVUAXIQN-HAEOHBJNSA-N 0.000 description 1
- LHNIIDJUOCFXAP-UHFFFAOYSA-N pictrelisib Chemical compound C1CN(S(=O)(=O)C)CCN1CC1=CC2=NC(C=3C=4C=NNC=4C=CC=3)=NC(N3CCOCC3)=C2S1 LHNIIDJUOCFXAP-UHFFFAOYSA-N 0.000 description 1
- 210000002381 plasma Anatomy 0.000 description 1
- 230000036470 plasma concentration Effects 0.000 description 1
- 229940063179 platinol Drugs 0.000 description 1
- 229920000191 poly(N-vinyl pyrrolidone) Polymers 0.000 description 1
- 229920001200 poly(ethylene-vinyl acetate) Polymers 0.000 description 1
- 229920003229 poly(methyl methacrylate) Polymers 0.000 description 1
- 230000008488 polyadenylation Effects 0.000 description 1
- 229920002338 polyhydroxyethylmethacrylate Polymers 0.000 description 1
- 239000004926 polymethyl methacrylate Substances 0.000 description 1
- 229920001155 polypropylene Polymers 0.000 description 1
- 229920001282 polysaccharide Polymers 0.000 description 1
- 239000005017 polysaccharide Substances 0.000 description 1
- 229920002223 polystyrene Polymers 0.000 description 1
- 229920002451 polyvinyl alcohol Polymers 0.000 description 1
- 239000004800 polyvinyl chloride Substances 0.000 description 1
- 229920000915 polyvinyl chloride Polymers 0.000 description 1
- 229960001131 ponatinib Drugs 0.000 description 1
- 238000010837 poor prognosis Methods 0.000 description 1
- 238000002600 positron emission tomography Methods 0.000 description 1
- 230000004481 post-translational protein modification Effects 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 239000003755 preservative agent Substances 0.000 description 1
- 150000003141 primary amines Chemical class 0.000 description 1
- 238000012545 processing Methods 0.000 description 1
- 239000000047 product Substances 0.000 description 1
- 210000001236 prokaryotic cell Anatomy 0.000 description 1
- 230000000644 propagated effect Effects 0.000 description 1
- 238000011321 prophylaxis Methods 0.000 description 1
- 229960003712 propranolol Drugs 0.000 description 1
- 230000001681 protective effect Effects 0.000 description 1
- 239000003197 protein kinase B inhibitor Substances 0.000 description 1
- 238000000455 protein structure prediction Methods 0.000 description 1
- 230000002685 pulmonary effect Effects 0.000 description 1
- 229940117820 purinethol Drugs 0.000 description 1
- 229950010131 puromycin Drugs 0.000 description 1
- 125000002206 pyridazin-3-yl group Chemical group [H]C1=C([H])C([H])=C(*)N=N1 0.000 description 1
- 125000004527 pyrimidin-4-yl group Chemical group N1=CN=C(C=C1)* 0.000 description 1
- 125000004943 pyrimidin-6-yl group Chemical group N1=CN=CC=C1* 0.000 description 1
- 239000002096 quantum dot Substances 0.000 description 1
- 125000004550 quinolin-6-yl group Chemical group N1=CC=CC2=CC(=CC=C12)* 0.000 description 1
- 230000005855 radiation Effects 0.000 description 1
- 239000012857 radioactive material Substances 0.000 description 1
- 238000003127 radioimmunoassay Methods 0.000 description 1
- 230000003439 radiotherapeutic effect Effects 0.000 description 1
- 230000035484 reaction time Effects 0.000 description 1
- 230000009257 reactivity Effects 0.000 description 1
- 229940044601 receptor agonist Drugs 0.000 description 1
- 239000000018 receptor agonist Substances 0.000 description 1
- 230000006798 recombination Effects 0.000 description 1
- 238000005215 recombination Methods 0.000 description 1
- 238000011084 recovery Methods 0.000 description 1
- 230000008929 regeneration Effects 0.000 description 1
- 238000011069 regeneration method Methods 0.000 description 1
- FNHKPVJBJVTLMP-UHFFFAOYSA-N regorafenib Chemical compound C1=NC(C(=O)NC)=CC(OC=2C=C(F)C(NC(=O)NC=3C=C(C(Cl)=CC=3)C(F)(F)F)=CC=2)=C1 FNHKPVJBJVTLMP-UHFFFAOYSA-N 0.000 description 1
- 201000010174 renal carcinoma Diseases 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 108091008146 restriction endonucleases Proteins 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 238000004366 reverse phase liquid chromatography Methods 0.000 description 1
- 238000004007 reversed phase HPLC Methods 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 1
- 125000002652 ribonucleotide group Chemical group 0.000 description 1
- 229960001302 ridaforolimus Drugs 0.000 description 1
- BPRHUIZQVSMCRT-VEUZHWNKSA-N rosuvastatin Chemical compound CC(C)C1=NC(N(C)S(C)(=O)=O)=NC(C=2C=CC(F)=CC=2)=C1\C=C\[C@@H](O)C[C@@H](O)CC(O)=O BPRHUIZQVSMCRT-VEUZHWNKSA-N 0.000 description 1
- 229960000672 rosuvastatin Drugs 0.000 description 1
- 239000012146 running buffer Substances 0.000 description 1
- 239000012898 sample dilution Substances 0.000 description 1
- 238000005070 sampling Methods 0.000 description 1
- 229930195734 saturated hydrocarbon Natural products 0.000 description 1
- 238000013341 scale-up Methods 0.000 description 1
- 125000002914 sec-butyl group Chemical group [H]C([H])([H])C([H])([H])C([H])(*)C([H])([H])[H] 0.000 description 1
- 230000003248 secreting effect Effects 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 238000010187 selection method Methods 0.000 description 1
- 230000035945 sensitivity Effects 0.000 description 1
- 230000001953 sensory effect Effects 0.000 description 1
- 229950008834 seribantumab Drugs 0.000 description 1
- 229960001153 serine Drugs 0.000 description 1
- 239000012679 serum free medium Substances 0.000 description 1
- 239000002453 shampoo Substances 0.000 description 1
- 230000035939 shock Effects 0.000 description 1
- 238000003998 size exclusion chromatography high performance liquid chromatography Methods 0.000 description 1
- 238000005549 size reduction Methods 0.000 description 1
- 239000002002 slurry Substances 0.000 description 1
- 239000004055 small Interfering RNA Substances 0.000 description 1
- 150000003384 small molecules Chemical class 0.000 description 1
- NLJMYIDDQXHKNR-UHFFFAOYSA-K sodium citrate Chemical compound O.O.[Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O NLJMYIDDQXHKNR-UHFFFAOYSA-K 0.000 description 1
- JJGWLCLUQNFDIS-GTSONSFRSA-M sodium;1-[6-[5-[(3as,4s,6ar)-2-oxo-1,3,3a,4,6,6a-hexahydrothieno[3,4-d]imidazol-4-yl]pentanoylamino]hexanoyloxy]-2,5-dioxopyrrolidine-3-sulfonate Chemical compound [Na+].O=C1C(S(=O)(=O)[O-])CC(=O)N1OC(=O)CCCCCNC(=O)CCCC[C@H]1[C@H]2NC(=O)N[C@H]2CS1 JJGWLCLUQNFDIS-GTSONSFRSA-M 0.000 description 1
- YCLWMUYXEGEIGD-UHFFFAOYSA-M sodium;2-hydroxy-3-[4-(2-hydroxyethyl)piperazin-1-yl]propane-1-sulfonate Chemical compound [Na+].OCCN1CCN(CC(O)CS([O-])(=O)=O)CC1 YCLWMUYXEGEIGD-UHFFFAOYSA-M 0.000 description 1
- 210000004872 soft tissue Anatomy 0.000 description 1
- 239000006104 solid solution Substances 0.000 description 1
- 230000000392 somatic effect Effects 0.000 description 1
- 229960003787 sorafenib Drugs 0.000 description 1
- 238000009987 spinning Methods 0.000 description 1
- 230000010473 stable expression Effects 0.000 description 1
- 239000012128 staining reagent Substances 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 239000008107 starch Substances 0.000 description 1
- 235000019698 starch Nutrition 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 230000000707 stereoselective effect Effects 0.000 description 1
- 150000003431 steroids Chemical class 0.000 description 1
- 229940090374 stivarga Drugs 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 229960001052 streptozocin Drugs 0.000 description 1
- ZSJLQEPLLKMAKR-GKHCUFPYSA-N streptozocin Chemical compound O=NN(C)C(=O)N[C@H]1[C@@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O ZSJLQEPLLKMAKR-GKHCUFPYSA-N 0.000 description 1
- 229960004793 sucrose Drugs 0.000 description 1
- NVBFHJWHLNUMCV-UHFFFAOYSA-N sulfamide Chemical compound NS(N)(=O)=O NVBFHJWHLNUMCV-UHFFFAOYSA-N 0.000 description 1
- 125000000446 sulfanediyl group Chemical group *S* 0.000 description 1
- 229960005559 sulforaphane Drugs 0.000 description 1
- 235000015487 sulforaphane Nutrition 0.000 description 1
- 239000011593 sulfur Substances 0.000 description 1
- 235000011149 sulphuric acid Nutrition 0.000 description 1
- 229960002812 sunitinib malate Drugs 0.000 description 1
- 108010033090 surfactant protein A receptor Proteins 0.000 description 1
- 238000001356 surgical procedure Methods 0.000 description 1
- 238000004114 suspension culture Methods 0.000 description 1
- 230000002459 sustained effect Effects 0.000 description 1
- 229940034785 sutent Drugs 0.000 description 1
- 210000005222 synovial tissue Anatomy 0.000 description 1
- 230000001839 systemic circulation Effects 0.000 description 1
- 230000009885 systemic effect Effects 0.000 description 1
- 229940037128 systemic glucocorticoids Drugs 0.000 description 1
- FQZYTYWMLGAPFJ-OQKDUQJOSA-N tamoxifen citrate Chemical compound [H+].[H+].[H+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O.C=1C=CC=CC=1C(/CC)=C(C=1C=CC(OCCN(C)C)=CC=1)/C1=CC=CC=C1 FQZYTYWMLGAPFJ-OQKDUQJOSA-N 0.000 description 1
- 229960003454 tamoxifen citrate Drugs 0.000 description 1
- 229940069905 tasigna Drugs 0.000 description 1
- 229960003080 taurine Drugs 0.000 description 1
- RCINICONZNJXQF-XAZOAEDWSA-N taxol® Chemical compound O([C@@H]1[C@@]2(CC(C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3(C21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-XAZOAEDWSA-N 0.000 description 1
- 229940063683 taxotere Drugs 0.000 description 1
- 229910052713 technetium Inorganic materials 0.000 description 1
- GKLVYJBZJHMRIY-UHFFFAOYSA-N technetium atom Chemical compound [Tc] GKLVYJBZJHMRIY-UHFFFAOYSA-N 0.000 description 1
- NRUKOCRGYNPUPR-QBPJDGROSA-N teniposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@@H](OC[C@H]4O3)C=3SC=CC=3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 NRUKOCRGYNPUPR-QBPJDGROSA-N 0.000 description 1
- 125000000999 tert-butyl group Chemical group [H]C([H])([H])C(*)(C([H])([H])[H])C([H])([H])[H] 0.000 description 1
- GKCBAIGFKIBETG-UHFFFAOYSA-N tetracaine Chemical compound CCCCNC1=CC=C(C(=O)OCCN(C)C)C=C1 GKCBAIGFKIBETG-UHFFFAOYSA-N 0.000 description 1
- 229960002372 tetracaine Drugs 0.000 description 1
- 229910052716 thallium Inorganic materials 0.000 description 1
- BKVIYDNLLOSFOA-UHFFFAOYSA-N thallium Chemical compound [Tl] BKVIYDNLLOSFOA-UHFFFAOYSA-N 0.000 description 1
- 238000011285 therapeutic regimen Methods 0.000 description 1
- 230000004797 therapeutic response Effects 0.000 description 1
- RYYWUUFWQRZTIU-UHFFFAOYSA-K thiophosphate Chemical compound [O-]P([O-])([O-])=S RYYWUUFWQRZTIU-UHFFFAOYSA-K 0.000 description 1
- 229960001196 thiotepa Drugs 0.000 description 1
- 208000013077 thyroid gland carcinoma Diseases 0.000 description 1
- ORYDPOVDJJZGHQ-UHFFFAOYSA-N tirapazamine Chemical compound C1=CC=CC2=[N+]([O-])C(N)=N[N+]([O-])=C21 ORYDPOVDJJZGHQ-UHFFFAOYSA-N 0.000 description 1
- 238000012090 tissue culture technique Methods 0.000 description 1
- 229960000187 tissue plasminogen activator Drugs 0.000 description 1
- 229950005976 tivantinib Drugs 0.000 description 1
- 238000011200 topical administration Methods 0.000 description 1
- 229960002190 topotecan hydrochloride Drugs 0.000 description 1
- 229960005267 tositumomab Drugs 0.000 description 1
- LIRYPHYGHXZJBZ-UHFFFAOYSA-N trametinib Chemical compound CC(=O)NC1=CC=CC(N2C(N(C3CC3)C(=O)C3=C(NC=4C(=CC(I)=CC=4)F)N(C)C(=O)C(C)=C32)=O)=C1 LIRYPHYGHXZJBZ-UHFFFAOYSA-N 0.000 description 1
- 229960001308 trametinib dimethyl sulfoxide Drugs 0.000 description 1
- OQUFJVRYDFIQBW-UHFFFAOYSA-N trametinib dimethyl sulfoxide Chemical compound CS(C)=O.CC(=O)NC1=CC=CC(N2C(N(C3CC3)C(=O)C3=C(NC=4C(=CC(I)=CC=4)F)N(C)C(=O)C(C)=C32)=O)=C1 OQUFJVRYDFIQBW-UHFFFAOYSA-N 0.000 description 1
- FGMPLJWBKKVCDB-UHFFFAOYSA-N trans-L-hydroxy-proline Natural products ON1CCCC1C(O)=O FGMPLJWBKKVCDB-UHFFFAOYSA-N 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 230000002103 transcriptional effect Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 230000001052 transient effect Effects 0.000 description 1
- 230000010474 transient expression Effects 0.000 description 1
- 229960000575 trastuzumab Drugs 0.000 description 1
- CYRMSUTZVYGINF-UHFFFAOYSA-N trichlorofluoromethane Chemical compound FC(Cl)(Cl)Cl CYRMSUTZVYGINF-UHFFFAOYSA-N 0.000 description 1
- 229940029284 trichlorofluoromethane Drugs 0.000 description 1
- HRXKRNGNAMMEHJ-UHFFFAOYSA-K trisodium citrate Chemical compound [Na+].[Na+].[Na+].[O-]C(=O)CC(O)(CC([O-])=O)C([O-])=O HRXKRNGNAMMEHJ-UHFFFAOYSA-K 0.000 description 1
- 229940038773 trisodium citrate Drugs 0.000 description 1
- 229910052722 tritium Inorganic materials 0.000 description 1
- 102000003390 tumor necrosis factor Human genes 0.000 description 1
- 238000013413 tumor xenograft mouse model Methods 0.000 description 1
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 1
- 150000004917 tyrosine kinase inhibitor derivatives Chemical class 0.000 description 1
- 238000004704 ultra performance liquid chromatography Methods 0.000 description 1
- ORHBXUUXSCNDEV-UHFFFAOYSA-N umbelliferone Chemical compound C1=CC(=O)OC2=CC(O)=CC=C21 ORHBXUUXSCNDEV-UHFFFAOYSA-N 0.000 description 1
- HFTAFOQKODTIJY-UHFFFAOYSA-N umbelliferone Natural products Cc1cc2C=CC(=O)Oc2cc1OCC=CC(C)(C)O HFTAFOQKODTIJY-UHFFFAOYSA-N 0.000 description 1
- 241000701447 unidentified baculovirus Species 0.000 description 1
- 241001529453 unidentified herpesvirus Species 0.000 description 1
- 241001430294 unidentified retrovirus Species 0.000 description 1
- 239000004474 valine Substances 0.000 description 1
- MSRILKIQRXUYCT-UHFFFAOYSA-M valproate semisodium Chemical compound [Na+].CCCC(C(O)=O)CCC.CCCC(C([O-])=O)CCC MSRILKIQRXUYCT-UHFFFAOYSA-M 0.000 description 1
- 229960000604 valproic acid Drugs 0.000 description 1
- 229960000241 vandetanib Drugs 0.000 description 1
- 239000003981 vehicle Substances 0.000 description 1
- 229960003862 vemurafenib Drugs 0.000 description 1
- 238000012795 verification Methods 0.000 description 1
- GBABOYUKABKIAF-IELIFDKJSA-N vinorelbine Chemical compound C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC GBABOYUKABKIAF-IELIFDKJSA-N 0.000 description 1
- 229960002066 vinorelbine Drugs 0.000 description 1
- CILBMBUYJCWATM-PYGJLNRPSA-N vinorelbine ditartrate Chemical compound OC(=O)[C@H](O)[C@@H](O)C(O)=O.OC(=O)[C@H](O)[C@@H](O)C(O)=O.C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC CILBMBUYJCWATM-PYGJLNRPSA-N 0.000 description 1
- 210000002845 virion Anatomy 0.000 description 1
- 239000000277 virosome Substances 0.000 description 1
- 230000000007 visual effect Effects 0.000 description 1
- 229940069559 votrient Drugs 0.000 description 1
- 239000008215 water for injection Substances 0.000 description 1
- 230000003442 weekly effect Effects 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 229940049068 xalkori Drugs 0.000 description 1
- 229940053867 xeloda Drugs 0.000 description 1
- 210000005253 yeast cell Anatomy 0.000 description 1
Images
Classifications
-
- A61K47/48384—
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6801—Drug-antibody or immunoglobulin conjugates defined by the pharmacologically or therapeutically active agent
- A61K47/6803—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/41—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having five-membered rings with two or more ring hetero atoms, at least one of which being nitrogen, e.g. tetrazole
- A61K31/425—Thiazoles
- A61K31/428—Thiazoles condensed with carbocyclic rings
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/435—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with one nitrogen as the only ring hetero atom
- A61K31/47—Quinolines; Isoquinolines
- A61K31/4738—Quinolines; Isoquinolines ortho- or peri-condensed with heterocyclic ring systems
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/495—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with two or more nitrogen atoms as the only ring heteroatoms, e.g. piperazine or tetrazines
- A61K31/505—Pyrimidines; Hydrogenated pyrimidines, e.g. trimethoprim
- A61K31/519—Pyrimidines; Hydrogenated pyrimidines, e.g. trimethoprim ortho- or peri-condensed with heterocyclic rings
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/33—Heterocyclic compounds
- A61K31/395—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins
- A61K31/535—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with at least one nitrogen and one oxygen as the ring hetero atoms, e.g. 1,2-oxazines
- A61K31/537—Heterocyclic compounds having nitrogen as a ring hetero atom, e.g. guanethidine or rifamycins having six-membered rings with at least one nitrogen and one oxygen as the ring hetero atoms, e.g. 1,2-oxazines spiro-condensed or forming part of bridged ring systems
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K31/00—Medicinal preparations containing organic active ingredients
- A61K31/63—Compounds containing para-N-benzenesulfonyl-N-groups, e.g. sulfanilamide, p-nitrobenzenesulfonyl hydrazide
- A61K31/635—Compounds containing para-N-benzenesulfonyl-N-groups, e.g. sulfanilamide, p-nitrobenzenesulfonyl hydrazide having a heterocyclic ring, e.g. sulfadiazine
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K38/00—Medicinal preparations containing peptides
- A61K38/04—Peptides having up to 20 amino acids in a fully defined sequence; Derivatives thereof
- A61K38/05—Dipeptides
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K39/395—Antibodies; Immunoglobulins; Immune serum, e.g. antilymphocytic serum
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K45/00—Medicinal preparations containing active ingredients not provided for in groups A61K31/00 - A61K41/00
- A61K45/06—Mixtures of active ingredients without chemical characterisation, e.g. antiphlogistics and cardiaca
-
- A61K47/48561—
-
- A61K47/48715—
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6801—Drug-antibody or immunoglobulin conjugates defined by the pharmacologically or therapeutically active agent
- A61K47/6803—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates
- A61K47/68033—Drugs conjugated to an antibody or immunoglobulin, e.g. cisplatin-antibody conjugates the drug being a maytansine
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6835—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site
- A61K47/6849—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a receptor, a cell surface antigen or a cell surface determinant
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6835—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site
- A61K47/6851—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell
- A61K47/6861—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell the tumour determinant being from kidney or bladder cancer cell
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6835—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site
- A61K47/6851—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell
- A61K47/6869—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment the modifying agent being an antibody or an immunoglobulin bearing at least one antigen-binding site the antibody targeting a determinant of a tumour cell the tumour determinant being from a cell of the reproductive system: ovaria, uterus, testes, prostate
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K47/00—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient
- A61K47/50—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates
- A61K47/51—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent
- A61K47/68—Medicinal preparations characterised by the non-active ingredients used, e.g. carriers or inert additives; Targeting or modifying agents chemically bound to the active ingredient the non-active ingredient being chemically bound to the active ingredient, e.g. polymer-drug conjugates the non-active ingredient being a modifying agent the modifying agent being an antibody, an immunoglobulin or a fragment thereof, e.g. an Fc-fragment
- A61K47/6889—Conjugates wherein the antibody being the modifying agent and wherein the linker, binder or spacer confers particular properties to the conjugates, e.g. peptidic enzyme-labile linkers or acid-labile linkers, providing for an acid-labile immuno conjugate wherein the drug may be released from its antibody conjugated part in an acidic, e.g. tumoural or environment
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
- C07K16/3038—Kidney, bladder
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/30—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants from tumour cells
- C07K16/3069—Reproductive system, e.g. ovaria, uterus, testes, prostate
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/574—Immunoassay; Biospecific binding assay; Materials therefor for cancer
- G01N33/57484—Immunoassay; Biospecific binding assay; Materials therefor for cancer involving compounds serving as markers for tumor, cancer, neoplasia, e.g. cellular determinants, receptors, heat shock/stress proteins, A-protein, oligosaccharides, metabolites
- G01N33/57492—Immunoassay; Biospecific binding assay; Materials therefor for cancer involving compounds serving as markers for tumor, cancer, neoplasia, e.g. cellular determinants, receptors, heat shock/stress proteins, A-protein, oligosaccharides, metabolites involving compounds localized on the membrane of tumor or cancer cells
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/21—Immunoglobulins specific features characterized by taxonomic origin from primates, e.g. man
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/33—Crossreactivity, e.g. for species or epitope, or lack of said crossreactivity
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/34—Identification of a linear epitope shorter than 20 amino acid residues or of a conformational epitope defined by amino acid residues
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/40—Immunoglobulins specific features characterized by post-translational modification
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/51—Complete heavy chain or Fd fragment, i.e. VH + CH1
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/515—Complete light chain, i.e. VL + CL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/55—Fab or Fab'
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/73—Inducing cell death, e.g. apoptosis, necrosis or inhibition of cell proliferation
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2333/00—Assays involving biological materials from specific organisms or of a specific nature
- G01N2333/435—Assays involving biological materials from specific organisms or of a specific nature from animals; from humans
- G01N2333/705—Assays involving receptors, cell surface antigens or cell surface determinants
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Medicinal Chemistry (AREA)
- General Health & Medical Sciences (AREA)
- Engineering & Computer Science (AREA)
- Immunology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Epidemiology (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Cell Biology (AREA)
- Organic Chemistry (AREA)
- Molecular Biology (AREA)
- Biochemistry (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Urology & Nephrology (AREA)
- Genetics & Genomics (AREA)
- Biophysics (AREA)
- Oncology (AREA)
- Hematology (AREA)
- Biomedical Technology (AREA)
- Microbiology (AREA)
- Reproductive Health (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Physics & Mathematics (AREA)
- Pathology (AREA)
- Hospice & Palliative Care (AREA)
- Biotechnology (AREA)
- Food Science & Technology (AREA)
- Physics & Mathematics (AREA)
- Analytical Chemistry (AREA)
- Gastroenterology & Hepatology (AREA)
- Pregnancy & Childbirth (AREA)
- Gynecology & Obstetrics (AREA)
- Mycology (AREA)
Abstract
본 발명은 항-CDH6 항체, 항체 단편, 항체 약물 접합체 및 암의 치료를 위한 그의 용도에 관한 것이다.
Description
본 개시내용은 항-CDH6 항체, 항체 단편, 항체 약물 접합체 및 암의 치료를 위한 그의 용도에 관한 것이다.
카드헤린은 세포-세포 접촉에 수반되는 세포 부착 분자의 패밀리이다. 개별 결합 특이성을 갖는 하위부류로 이루어진 패밀리에 30종 초과의 카드헤린 분자가 존재한다. 상피와 같은 조직의 발생 및 유지에서 주요 역할을 하는 전형적 카드헤린, 예컨대 상피 (E) 카드헤린, 신경 (N) 카드헤린 및 태반 (P) 카드헤린이 널리 연구되어 있다. 카드헤린 패밀리는 또한 카드헤린을 2개의 군, 유형 I 및 유형 II로 분리하는 그의 아미노산 유사성에 의해 분류되었다. 이에 따라, 조직 특이적 카드헤린, 예컨대 카드헤린-6 (CDH6)이 존재한다.
CDH6은 1995년에 최초로 클로닝 및 특징화되었다 (Shimoyama et al., Cancer Res. 1995: 55 (10)2206-2211). 시모야마(Shimoyama)에 의해 단리된 CDH6 cDNA는 4315개의 뉴클레오티드이고, 790개의 아미노산의 카드헤린 단백질을 코딩하고, 래트 K-카드헤린과 97% 상동성을 나타냈다 (Shimoyama, 상기 문헌). CDH6은 5개의 세포외 카드헤린 반복부, 막횡단 도메인 및 세포질 도메인을 갖는 유형 II 카드헤린이다. 정상 조직 발현을 프로빙한 경우에, CDH6은 뇌, 소뇌 및 신장에서 검출되었고, 폐, 췌장 및 위 점막에서는 보다 약하게 발현되었다. 신경계에서, CDH6은 청각계 및 체성감각계의 경계를 정하는 것으로 밝혀졌다 (Inoue et al., Mol. Cell Neuro. 2008: (39) 95-104). CDH6이 눈에서 이러한 시각 품질, 예컨대 밝기, 운동의 방향 또는 에지를 담당하는 망막 신경절 세포 (RGC)의 하위세트에 의해 발현된다는 것이 입증되었다 (Osterhout et al., Neuron 2011: 71(4)632-639). CDH6 녹아웃 마우스에서, CDH6의 부재는 축삭 표적화의 상실을 야기한다. CDH6 돌연변이체 축삭은 적절하게 신장되었고, 그의 표적으로 적절하게 안내되었지만, 이어서 그의 연결부를 통해 그를 지나 성장하였다 (Osterhout, 상기 문헌). 또 다른 연구에서, CDH6 녹아웃 마우스는 발성에서의 결함을 나타냈다 (Nakagawa eta la., PLOS 2012: 7(11) e49233).
암에서의 그의 발현으로 돌아가, CDH6 cDNA를 간세포성 암종 세포주로부터 클로닝하였으며, 이는 이러한 유형의 암에 수반될 수 있는 초기 지표였다 (Shimoyama, 상기 문헌). 초기 연구는 여러 간세포암 세포주에서의 CDH6 발현을 밝혀냈지만, 정산 간에서의 발현은 밝혀내지 못했다. 보다 나중의 연구에서, 한 군이 신세포 암종을 갖는 216명의 환자를 분석하였고, CDH6 발현이 이러한 유형의 암과 상관관계가 있다는 것을 밝혀냈다 (Paul et al., J Urology 2004 (171): 97-101). 시마주이(Shimazui)는 CDH6을 발현하며 E-카드헤린 발현이 결핍된 신세포 암종 환자가 불량한 예후를 가진다는 것을 밝혀냈다 (Shimazui et al., Clin. Cancer Res. 1998 (4): 2419-2424). 또한, CDH6이 TGF-B 표적이며 갑상선암에서 역할을 한다는 것이 발견되었다 (Sancisi et al., PLoS One 2013; 8(9): e75489). 마지막으로, CDH6은 난소암에 대한 바이오마커인 것으로 제시되었다 (Kobel et al., PLoS One 2008 5(12): e232).
항체 약물 접합체 ("ADC")는 암의 치료에서 세포독성제의 국부 전달에 사용되었다 (예를 들어, 문헌 [Lambert, Curr. Opinion In Pharmacology 5:543-549, 2005] 참조). ADC는 약물 모이어티의 표적화 전달을 가능하게 하여, 최소 독성과 함께 최대 효능이 달성될 수 있다. 보다 많은 ADC가 유망한 임상 결과를 나타냄에 따라, 암 요법을 위한 새로운 치료제를 개발하는 것에 대한 요구가 증가한다.
하기 화학식의 항체 약물 접합체 또는 그의 제약상 허용되는 염:
Ab-(L-(D)m)n
여기서
Ab는 인간 CDH6의 에피토프에 특이적으로 결합하는 항체 또는 그의 항원 결합 단편이고;
L은 링커이고;
D는 약물 모이어티이고;
m은 1 내지 8의 정수이고;
n은 1 내지 10의 정수이다.
임의의 상기 실시양태에 있어서, 상기 n이 3 또는 4인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 CDH6의 세포외 도메인 (서열식별번호(SEQ ID NO): 4)에 특이적으로 결합하는 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 항원 결합 단편이 서열식별번호: 534에서의 인간 CDH6의 에피토프에 특이적으로 결합하는 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것은 항체 약물 접합체:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역.
임의의 상기 실시양태에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, CDR 내의 1 또는 2개의 아미노산이 변형, 결실 또는 치환된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL).
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 234를 포함하는 중쇄, 및 서열식별번호: 235를 포함하는 경쇄;
(ii) 서열식별번호: 220을 포함하는 중쇄, 및 서열식별번호: 221을 포함하는 경쇄;
(iii) 서열식별번호: 276을 포함하는 중쇄, 및 서열식별번호: 277을 포함하는 경쇄;
(iv) 서열식별번호: 318을 포함하는 중쇄, 및 서열식별번호: 319를 포함하는 경쇄;
(v) 서열식별번호: 24를 포함하는 중쇄, 및 서열식별번호: 25를 포함하는 경쇄;
(vi) 서열식별번호: 38을 포함하는 중쇄, 및 서열식별번호: 39를 포함하는 경쇄;
(vii) 서열식별번호: 52를 포함하는 중쇄, 및 서열식별번호: 53을 포함하는 경쇄;
(viii) 서열식별번호: 66을 포함하는 중쇄, 및 서열식별번호: 67을 포함하는 경쇄;
(ix) 서열식별번호: 80을 포함하는 중쇄, 및 서열식별번호: 81을 포함하는 경쇄;
(x) 서열식별번호: 94를 포함하는 중쇄, 및 서열식별번호: 95를 포함하는 경쇄;
(xi) 서열식별번호: 108을 포함하는 중쇄, 및 서열식별번호: 109를 포함하는 경쇄;
(xii) 서열식별번호: 122를 포함하는 중쇄, 및 서열식별번호: 123을 포함하는 경쇄;
(xiii) 서열식별번호: 136을 포함하는 중쇄, 및 서열식별번호: 137을 포함하는 경쇄;
(xiv) 서열식별번호: 150을 포함하는 중쇄, 및 서열식별번호: 151을 포함하는 경쇄;
(xv) 서열식별번호: 164를 포함하는 중쇄, 및 서열식별번호: 165를 포함하는 경쇄;
(xvi) 서열식별번호: 178을 포함하는 중쇄, 및 서열식별번호: 179를 포함하는 경쇄;
(xvii) 서열식별번호: 192를 포함하는 중쇄, 및 서열식별번호: 193을 포함하는 경쇄;
(xviii) 서열식별번호: 206을 포함하는 중쇄, 및 서열식별번호: 207을 포함하는 경쇄;
(xix) 서열식별번호: 248을 포함하는 중쇄, 및 서열식별번호: 249를 포함하는 경쇄;
(xx) 서열식별번호: 262를 포함하는 중쇄, 및 서열식별번호: 263을 포함하는 경쇄;
(xxi) 서열식별번호: 290을 포함하는 중쇄, 및 서열식별번호: 291을 포함하는 경쇄;
(xxii) 서열식별번호: 304를 포함하는 중쇄, 및 서열식별번호: 305를 포함하는 경쇄; 또는
(xxiii) 서열식별번호: 332를 포함하는 중쇄, 및 서열식별번호: 333을 포함하는 경쇄.
임의의 상기 실시양태에 있어서, 가변 경쇄 또는 가변 중쇄 영역에 걸쳐 적어도 90, 91, 92, 93, 94, 95, 96, 97, 98 또는 99% 동일성을 보유하는 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 항체가 모노클로날 항체, 키메라 항체, 인간화 항체, 인간 조작된 항체, 인간 항체, 단일 쇄 항체(scFv) 또는 항체 단편인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 링커 (L)가 절단가능한 링커, 비-절단가능한 링커, 친수성 링커, 사전하전된 링커 및 디카르복실산 기반 링커로 이루어진 군으로부터 선택된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 링커가 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB), N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP), N-숙신이미딜 4-(2-피리딜디티오)펜타노에이트 (SPP), N-숙신이미딜 4-(2-피리딜디티오)부타노에이트 (SPDB), N-숙신이미딜 아이오도아세테이트 (SIA), N-숙신이미딜(4-아이오도아세틸)아미노벤조에이트 (SIAB), 말레이미드 PEG NHS, N-숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (SMCC), N-술포숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (술포-SMCC) 또는 2,5-디옥소피롤리딘-1-일 17-(2,5-디옥소-2,5-디히드로-1H-피롤-1-일)-5,8,11,14-테트라옥소-4,7,10,13-테트라아자헵타데칸-1-오에이트 (CX1-1)로 이루어진 군으로부터 선택된 가교 시약으로부터 유래된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 링커가 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)로부터 유래된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 약물 모이어티 (D)가 메이탄시노이드, V-ATPase 억제제, 아폽토시스촉진제, Bcl2 억제제, MCL1 억제제, HSP90 억제제, IAP 억제제, mTor 억제제, 미세관 안정화제, 미세관 탈안정화제, 아우리스타틴, 돌라스타틴, MetAP (메티오닌 아미노펩티다제), 단백질 CRM1의 핵 유출의 억제제, DPPIV 억제제, 프로테아솜 억제제, 미토콘드리아에서의 포스포릴 전달 반응의 억제제, 단백질 합성 억제제, 키나제 억제제, CDK2 억제제, CDK9 억제제, 키네신 억제제, HDAC 억제제, DNA 손상 작용제, DNA 알킬화제, DNA 삽입제, DNA 작은 홈 결합제 및 DHFR 억제제로 이루어진 군으로부터 선택된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 약물 모이어티가 메이탄시노이드인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 메이탄시노이드가 N(2')-데아세틸-N2-(4-메르캅토-4-메틸-1-옥소펜틸)-메이탄신 (DM4) 또는 N(2')-데아세틸-N(2')-(3-메르캅토-1-옥소프로필)-메이탄신 (DM1)인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 또 다른 치료제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 표 18에 열거된 치료제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
하기 화학식의 항체 약물 접합체 또는 그의 제약상 허용되는 염:
여기서
Ab는 인간 CDH6에 특이적으로 결합하는 항체 또는 그의 항원 결합 단편이고, n은 1 내지 10의 정수이다.
임의의 상기 실시양태에 있어서, 상기 항체 또는 항원 결합 단편이 (서열식별번호: 4)에서의 인간 CDH6의 에피토프에 특이적으로 결합하는 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 (서열식별번호: 534)에서의 인간 CDH6에 특이적으로 결합하는 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 Ab가 다음을 포함하는 항체 또는 그의 항원 결합 단편인 항체 약물 접합체:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역.
임의의 상기 실시양태에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, CDR 내의 1 또는 2개의 아미노산이 변형, 결실 또는 치환된 것인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL).
임의의 상기 실시양태에 있어서, 가변 경쇄 또는 가변 중쇄 영역에 걸쳐 적어도 90, 91, 92, 93, 94, 95, 96, 97, 98 또는 99% 동일성을 보유하는 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 항체가 모노클로날 항체, 키메라 항체, 인간화 항체, 인간 조작된 항체, 인간 항체, 단일 쇄 항체(scFv) 또는 항체 단편인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 n이 2 내지 8의 정수인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 상기 n이 3 내지 4의 정수인 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 또 다른 치료제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 표 18에 열거된 치료제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
임의의 상기 실시양태의 항체 약물 접합체 및 제약상 허용되는 담체를 포함하는 제약 조성물.
임의의 상기 실시양태에 있어서, 동결건조물로서 제조되는 제약 조성물.
임의의 상기 실시양태에 있어서, 상기 동결건조물이 임의의 상기 실시양태의 항체 약물 접합체, 숙신산나트륨 및 폴리소르베이트 20을 포함하는 것인 제약 조성물.
CDH6 양성 암의 치료를 필요로 하는 환자에게 항체 약물 접합체 또는 제약 조성물을 투여하는 것을 포함하는, 상기 환자에서 CDH6 양성 암을 치료하는 방법.
상기 암이 난소암, 신암, 간암, 연부조직암, CNS암, 갑상선암 및 담관암종으로 이루어진 군으로부터 선택된 것인 방법.
항체 약물 접합체 또는 제약 조성물이 또 다른 치료제와 조합되어 투여되는 것인 방법.
항체 약물 접합체 또는 제약 조성물이 표 18에 열거된 치료제와 조합되어 투여되는 것인 방법.
임의의 상기 실시양태에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 의약으로서 사용하기 위한 항체 약물 접합체.
CDH6 양성 암의 치료에 사용하기 위한, 항체 약물 접합체 또는 그의 제약 조성물.
임의의 상기 실시양태에 있어서, 또 다른 치료제와 조합되어 투여되는 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 표 18에 열거된 치료제와 조합되어 투여되는 항체 약물 접합체.
임의의 상기 실시양태에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
임의의 상기 실시양태에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
임의의 상기 실시양태의 항체 또는 항원 결합 단편을 코딩하는 핵산.
임의의 상기 실시양태의 핵산을 포함하는 벡터.
임의의 상기 실시양태에 따른 벡터를 포함하는 숙주 세포.
숙주 세포를 배양하는 단계 및 배양물로부터 항체를 회수하는 단계를 포함하는, 항체 또는 항원 결합 단편을 생산하는 방법.
(a) N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)를 약물 모이어티 DM-4에 화학적으로 연결시키는 단계;
(b) 상기 링커-약물을 세포 배양물로부터 회수된 항체에 접합시키는 단계; 및
(c) 항체 약물 접합체를 정제하는 단계
를 포함하는, 항-CDH6 항체 약물 접합체를 생산하는 방법.
UV 분광광도계로 측정된 평균 항체에 대한 메이탄시노이드 비 (MAR)가 약 3.5인, 임의의 상기 실시양태에 따라 제조된 항체 약물 접합체.
다음을 포함하는 항체 또는 그의 항원 결합 단편:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역.
임의의 상기 실시양태에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 또는 그의 항원 결합 단편.
임의의 상기 실시양태에 있어서, 다음을 포함하는 항체 또는 그의 항원 결합 단편:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL).
표지되어 있는 임의의 상기 실시양태의 항체 또는 그의 항원 결합 단편을 포함하는 진단 시약.
임의의 상기 실시양태에 있어서, 표지가 방사성표지, 형광단, 발색단, 영상화제 및 금속 이온으로 이루어진 군으로부터 선택된 것인 진단 시약.
정의
달리 언급되지 않는 한, 본원에 사용된 하기 용어 및 어구는 하기 의미를 갖도록 의도된다.
용어 "알킬"은 명시된 수의 탄소 원자를 갖는 1가 포화 탄화수소 쇄를 지칭한다. 예를 들어, C1-6 알킬은 1 내지 6개의 탄소 원자를 갖는 알킬 기를 지칭한다. 알킬 기는 직쇄형 또는 분지형일 수 있다. 대표적인 분지형 알킬 기는 1, 2 또는 3개의 분지를 갖는다. 알킬 기의 예는 메틸, 에틸, 프로필 (n-프로필 및 이소프로필), 부틸 (n-부틸, 이소부틸, sec-부틸 및 t-부틸), 펜틸 (n-펜틸, 이소펜틸 및 네오펜틸) 및 헥실을 포함하나 이에 제한되지는 않는다.
본원에 사용된 용어 "항체"는 상응하는 항원에 비-공유적으로, 가역적으로 및 특이적 방식으로 결합할 수 있는 이뮤노글로불린 패밀리의 폴리펩티드를 지칭한다. 예를 들어, 자연 발생 IgG 항체는 디술피드 결합에 의해 상호-연결된 적어도 2개의 중 (H) 쇄 및 2개의 경 (L) 쇄를 포함하는 사량체이다. 각각의 중쇄는 중쇄 가변 영역 (본원에서 VH로 약칭됨) 및 중쇄 불변 영역으로 구성된다. 중쇄 불변 영역은 3개의 도메인 CH1, CH2 및 CH3으로 구성된다. 각각의 경쇄는 경쇄 가변 영역 (본원에서 VL로 약칭됨) 및 경쇄 불변 영역으로 구성된다. 경쇄 불변 영역은 1개의 도메인 CL로 구성된다. VH 및 VL 영역은 프레임워크 영역 (FR)으로 불리는 보다 보존된 영역이 산재되어 있는 상보성 결정 영역 (CDR)으로 불리는 초가변 영역으로 추가로 세분될 수 있다. 각각의 VH 및 VL은 아미노-말단으로부터 카르복시-말단으로 하기 순서로 배열된 3개의 CDR 및 4개의 FR로 구성된다: FR1, CDR1, FR2, CDR2, FR3, CDR3 및 FR4. 중쇄 및 경쇄의 가변 영역은 항원과 상호작용하는 결합 도메인을 함유한다. 항체의 불변 영역은 면역계의 다양한 세포 (예를 들어, 이펙터 세포) 및 전형적 보체계의 제1 성분 (Clq)을 포함한 숙주 조직 또는 인자에 대한 이뮤노글로불린의 결합을 매개할 수 있다.
용어 "항체"는 모노클로날 항체, 인간 항체, 인간화 항체, 낙타류 항체, 키메라 항체 및 항-이디오타입 (항-Id) 항체 (예를 들어 본 개시내용의 항체에 대한 항-Id 항체를 포함함)를 포함한다. 항체는 임의의 이소형/부류 (예를 들어, IgG, IgE, IgM, IgD, IgA 및 IgY) 또는 하위부류 (예를 들어, IgG1, IgG2, IgG3, IgG4, IgA1 및 IgA2)일 수 있다.
"상보성-결정 도메인" 또는 "상보성-결정 영역 ("CDR")은 상호교환가능하게 VL 및 VH의 초가변 영역을 지칭한다. CDR은 항체 쇄의 표적 단백질-결합 부위이며, 이러한 표적 단백질에 대한 특이성을 보유한다. 각각의 인간 VL 또는 VH에 3개의 CDR (N-말단으로부터 순차적으로 넘버링된 CDR1-3)이 있고, 이들은 가변 도메인의 약 15-20%를 구성한다. CDR은 그의 영역 및 순서에 의해 지칭될 수 있다. 예를 들어, "VHCDR1" 또는 "HCDR1"은 둘 다 중쇄 가변 영역의 제1 CDR을 지칭한다. CDR은 표적 단백질의 에피토프에 대해 구조적으로 상보적이며, 따라서 결합 특이성의 직접적인 원인이 된다. VL 또는 VH의 나머지 스트레치, 소위 프레임워크 영역은 아미노산 서열에 있어서 보다 적은 변화를 나타낸다 (Kuby, Immunology, 4th ed., Chapter 4. W.H. Freeman & Co., New York, 2000).
CDR 및 프레임워크 영역의 위치는 관련 기술분야에서의 다양한 널리 공지된 정의, 예를 들어 카바트(Kabat), 코티아(Chothia) 및 AbM을 사용하여 결정될 수 있다 (예를 들어, 문헌 [Johnson et al., Nucleic Acids Res., 29:205-206 (2001); Chothia and Lesk, J. Mol. Biol., 196:901-917 (1987); Chothia et al., Nature, 342:877-883 (1989); Chothia et al., J. Mol. Biol., 227:799-817 (1992); Al-Lazikani et al., J.Mol.Biol., 273:927-748 (1997)] 참조). 항원 결합 부위의 정의는 또한 하기에 기재되어 있다: 문헌 [Ruiz et al., Nucleic Acids Res., 28:219-221 (2000); 및 Lefranc, M.P., Nucleic Acids Res., 29:207-209 (2001); MacCallum et al., J. Mol. Biol., 262:732-745 (1996); 및 Martin et al., Proc. Natl. Acad. Sci. USA, 86:9268-9272 (1989); Martin et al., Methods Enzymol., 203:121-153 (1991); 및 Rees et al., In Sternberg M.J.E. (ed.), Protein Structure Prediction, Oxford University Press, Oxford, 141-172 (1996)].
경쇄 및 중쇄는 둘 다 구조적 및 기능적 상동성 영역으로 분류될 수 있다. 용어 "불변" 및 "가변"은 기능적으로 사용된다. 이와 관련하여, 경쇄 (VL) 및 중쇄 (VH) 부분 둘 다의 가변 도메인은 항원 인식 및 특이성을 결정하는 것으로 인지될 것이다. 반대로, 경쇄의 불변 도메인 (CL) 및 중쇄의 불변 도메인 (CH1, CH2 또는 CH3)은 중요한 생물학적 특성, 예컨대 분비, 경태반 이동성, Fc 수용체 결합, 보체 결합 등을 부여한다. 통상적으로, 불변 영역 도메인의 넘버링은 항체의 항원 결합 부위 또는 아미노-말단에서 더욱 멀어질수록 증가한다. N-말단은 가변 영역이고, C-말단에 불변 영역이 있고; CH3 및 CL 도메인은 실제로 각각 중쇄 및 경쇄의 카르복시-말단 도메인을 포함한다.
본원에 사용된 용어 "항원 결합 단편"은 항원의 에피토프와 (예를 들어, 결합, 입체 장애, 안정화/탈안정화, 공간 분포에 의해) 특이적으로 상호작용하는 능력을 보유하는 항체의 1개 이상의 부분을 포함하는 폴리펩티드를 지칭한다. 결합 단편의 예는 단일-쇄 Fv (scFv), 디술피드-연결 Fv (sdFv), Fab 단편, F(ab') 단편, VL, VH, CL 및 CH1 도메인으로 이루어진 1가 단편; 힌지 영역에서 디술피드 가교에 의해 연결된 2개의 Fab 단편을 포함하는 2가 단편인 F(ab)2 단편; VH 및 CH1 도메인으로 이루어진 Fd 단편; 항체의 단일 아암의 VL 및 VH 도메인으로 이루어진 Fv 단편; VH 도메인으로 이루어진 dAb 단편 (Ward et al., Nature 341:544-546, 1989); 및 단리된 상보성 결정 영역 (CDR), 또는 항체의 다른 에피토프-결합 단편을 포함하나 이에 제한되지는 않는다.
또한, Fv 단편의 2개의 도메인 VL 및 VH가 개별 유전자에 의해 코딩되더라도, 재조합 방법을 사용하여, 이들이 VL 및 VH 영역이 쌍을 이루어 1가 분자를 형성한 단일 단백질 쇄 (단일 쇄 Fv ("scFv")로 공지됨; 예를 들어, 문헌 [Bird et al., Science 242:423-426, 1988; 및 Huston et al., Proc. Natl. Acad. Sci. 85:5879-5883, 1988] 참조)로 만들어지게 할 수 있는 합성 링커에 의해 이들이 연결될 수 있다. 이러한 단일 쇄 항체는 또한 용어 "항원 결합 단편" 내에 포괄되도록 의도된다. 이들 항원 결합 단편은 관련 기술분야의 통상의 기술자에게 공지된 통상적인 기술을 사용하여 수득되고, 단편은 무손상 항체와 동일한 방식으로 유용성에 대해 스크리닝된다.
항원 결합 단편은 또한 단일 도메인 항체, 맥시바디, 미니바디, 나노바디, 인트라바디, 디아바디, 트리아바디, 테트라바디, v-NAR 및 bis-scFv 내로 혼입될 수 있다 (예를 들어, 문헌 [Hollinger and Hudson, Nature Biotechnology 23:1126-1136, 2005] 참조). 항원 결합 단편이 피브로넥틴 유형 III (Fn3)과 같은 폴리펩티드에 기반한 스캐폴드 내로 이식될 수 있다 (피브로넥틴 폴리펩티드 모노바디를 기재하는 미국 특허 번호 6,703,199 참조).
항원 결합 단편은 한 쌍의 탠덤 Fv 절편 (VH-CH1-VH-CH1)을 포함하는 단일 쇄 분자 내로 혼입될 수 있고, 이는 상보적 경쇄 폴리펩티드와 함께 한 쌍의 항원 결합 영역을 형성한다 (문헌 [Zapata et al., Protein Eng. 8:1057-1062, 1995]; 및 미국 특허 번호 5,641,870).
본원에 사용된 용어 "모노클로날 항체" 또는 "모노클로날 항체 조성물"은 실질적으로 동일한 아미노산 서열을 갖거나 또는 동일한 유전자 공급원으로부터 유래된 항체 및 항원 결합 단편을 포함한 폴리펩티드를 지칭한다. 이러한 용어는 또한 단일 분자 조성물의 항체 분자의 제제를 포함한다. 모노클로날 항체 조성물은 특정한 에피토프에 대해 단일 결합 특이성 및 친화도를 디스플레이한다.
본원에 사용된 용어 "인간 항체"는 프레임워크 및 CDR 영역이 둘 다 인간 기원의 서열로부터 유래된 것인 가변 영역을 갖는 항체를 포함한다. 또한, 항체가 불변 영역을 함유하는 경우에, 불변 영역은 또한 이러한 인간 서열, 예를 들어 인간 배선 서열, 또는 인간 배선 서열의 돌연변이된 형태, 또는 인간 프레임워크 서열 분석으로부터 유래된 컨센서스 프레임워크 서열을 함유하는 항체 (예를 들어, 문헌 [Knappik et al., J. Mol. Biol. 296:57-86, 2000]에 기재됨)로부터 유래된다.
본 개시내용의 인간 항체는 인간 서열에 의해 코딩되지 않는 아미노산 잔기 (예를 들어, 시험관내에서의 무작위 또는 부위-특이적 돌연변이유발에 의해 또는 생체내에서의 체세포 돌연변이에 의해 도입된 돌연변이, 또는 안정성 또는 제조를 촉진하기 위한 보존적 치환)를 포함할 수 있다.
본원에 사용된 용어 "인식하다"는 에피토프가 선형인지 입체적인지에 관계없이, 에피토프를 발견하고 그와 상호작용하는 (예를 들어, 결합하는) 항체 또는 그의 항원 결합 단편을 지칭한다. 용어 "에피토프"는 본 개시내용의 항체 또는 항원 결합 단편이 특이적으로 결합하는 항원 상의 부위를 지칭한다. 에피토프는 인접 아미노산 또는 단백질의 3차 폴딩에 의해 병치된 비인접 아미노산 둘 다로부터 형성될 수 있다. 인접 아미노산으로부터 형성되는 에피토프는 전형적으로 변성 용매에 대한 노출시에 유지되고, 반면 3차 폴딩에 의해 형성되는 에피토프는 전형적으로 변성 용매를 사용한 처리시에 상실된다. 에피토프는 전형적으로 적어도 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 또는 15개의 아미노산을 고유한 공간 입체형태로 포함한다. 에피토프의 공간 입체형태를 결정하는 방법은 관련 기술분야의 기술, 예를 들어 X선 결정학 및 2-차원 핵 자기 공명을 포함한다 (예를 들어, 문헌 [Epitope Mapping Protocols in Methods in Molecular Biology, Vol. 66, G. E. Morris, Ed. (1996)] 참조). "파라토프"는 항원의 에피토프를 인식하는 항체의 일부이다.
항원 (예를 들어, 단백질)과 항체, 항체 단편 또는 항체-유래 결합제 사이의 상호작용을 기재하는 문맥에서 사용되는 경우에 어구 "특이적으로 결합하다" 또는 "선택적으로 결합하다"는 단백질 및 다른 생물제제의 비균질 집단, 예를 들어 생물학적 샘플, 예를 들어 혈액, 혈청, 혈장 또는 조직 샘플 내의 항원의 존재를 결정짓는 결합 반응을 지칭한다. 따라서, 특정 지정된 면역검정 조건 하에서, 특정한 결합 특이성을 갖는 항체 또는 결합제는 특정한 항원에 배경값의 적어도 2배로 결합하고, 샘플에 존재하는 다른 항원에 유의한 양으로 실질적으로 결합하지 않는다. 한 측면에서, 지정된 면역검정 조건 하에서, 특정한 결합 특이성을 갖는 항체 또는 결합제는 특정한 항원에 배경값의 적어도 10배로 결합하고, 샘플에 존재하는 다른 항원에 유의한 양으로 실질적으로 결합하지 않는다. 이러한 조건 하에서 항체 또는 결합제에 대한 특이적 결합은 항체 또는 결합제가 특정한 단백질에 대한 그의 특이성에 대해 선택된 것을 필요로 할 수 있다. 원하는 경우 또는 적절한 경우, 이러한 선택은 다른 종 (예를 들어, 마우스 또는 래트) 또는 다른 하위유형으로부터의 분자와 교차-반응하는 항체를 제외시킴으로써 달성될 수 있다. 대안적으로, 일부 측면에서, 특정 목적 분자와 교차-반응하는 항체 또는 항체 단편이 선택된다.
본원에 사용된 용어 "친화도"는 단일 항원 부위에서의 항체와 항원 사이의 상호작용의 강도를 지칭한다. 각각의 항원 부위 내에서, 항체 "아암"의 가변 영역은 수많은 부위에서의 항원과 약한 비-공유결합력을 통해 상호작용하고; 상호작용이 더 많을수록 친화도가 더 강해진다.
용어 "단리된 항체"는 상이한 항원 특이성을 갖는 다른 항체가 실질적으로 없는 항체를 지칭한다. 그러나, 한 항원에 특이적으로 결합하는 단리된 항체는 다른 항원에 대한 교차-반응성을 가질 수 있다. 또한, 단리된 항체는 다른 세포 물질 및/또는 화학물질이 실질적으로 없을 수 있다.
용어 "상응하는 인간 배선 서열"은 인간 배선 이뮤노글로불린 가변 영역 서열에 의해 코딩되는 모든 다른 공지된 가변 영역 아미노산 서열과 비교하여 참조 가변 영역 아미노산 서열 또는 하위서열과 가장 높게 결정된 아미노산 서열 동일성을 공유하는 인간 가변 영역 아미노산 서열 또는 하위서열을 코딩하는 핵산 서열을 지칭한다. 상응하는 인간 배선 서열은 또한 모든 다른 평가된 가변 영역 아미노산 서열과 비교하여 참조 가변 영역 아미노산 서열 또는 하위서열과 가장 높은 아미노산 서열 동일성을 갖는 인간 가변 영역 아미노산 서열 또는 하위서열을 지칭할 수 있다. 상응하는 인간 배선 서열은 프레임워크 영역 단독, 상보성 결정 영역 단독, 프레임워크 및 상보성 결정 영역, 가변 절편 (상기 정의된 바와 같음), 또는 가변 영역을 포함하는 서열 또는 하위서열의 다른 조합일 수 있다. 서열 동일성은 본원에 기재된 방법을 사용하여, 예를 들어 BLAST, ALIGN 또는 관련 기술분야에 공지된 또 다른 정렬 알고리즘을 사용하여 2개의 서열을 정렬시켜 결정될 수 있다. 상응하는 인간 배선 핵산 또는 아미노산 서열은 참조 가변 영역 핵산 또는 아미노산 서열과 적어도 약 90%, 91, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 가질 수 있다.
다양한 면역검정 포맷을 사용하여 특정한 단백질과 특이적으로 면역반응성인 항체를 선택할 수 있다. 예를 들어, 고체-상 ELISA 면역검정을 상용적으로 사용하여 단백질과 특이적으로 면역반응성인 항체를 선택한다 (특이적 면역반응성을 결정하는데 사용될 수 있는 면역검정 포맷 및 조건의 기재에 대해서는, 예를 들어 문헌 [Harlow & Lane, Using Antibodies, A Laboratory Manual (1998)] 참조). 전형적으로, 특이적 또는 선택적 결합 반응은 배경 신호에 대해 적어도 2배, 보다 전형적으로는 배경 신호에 대해 적어도 10 내지 100배의 신호를 생성할 것이다.
용어 "평형 해리 상수 (KD, M)"는 해리율 상수 (kd, 시간-1)를 회합률 상수 (ka, 시간-1, M-1)로 나눈 것을 지칭한다. 평형 해리 상수는 관련 기술분야에서의 임의의 공지된 방법을 사용하여 측정될 수 있다. 본 개시내용의 항체는 일반적으로 평형 해리 상수가 약 10-7 또는 10-8 M 미만, 예를 들어 약 10-9 M 또는 10-10 M 미만, 일부 측면에서 약 10-11 M, 10-12 M 또는 10-13 M 미만일 것이다.
용어 "생체이용률"은 환자에게 투여된 소정량의 약물의 전신 이용률 (즉, 혈액/혈장 수준)을 지칭한다. 생체이용률은 투여된 투여 형태로부터 전신 순환에 도달하는 약물의 시간 (속도) 및 총량 (정도) 둘 다의 측정치를 나타내는 절대적인 용어이다.
본원에 사용된 어구 "로 본질적으로 이루어진"은 방법 또는 조성물에 포함되는 활성 제약 작용제의 종류 또는 종, 뿐만 아니라 방법 또는 조성물의 의도되는 목적에 대해 불활성인 임의의 부형제를 지칭한다. 일부 측면에서, 어구 "로 본질적으로 이루어진"은 본 개시내용의 항체 약물 접합체 이외의 1종 이상의 추가의 활성제의 포함을 명백하게 배제한다. 일부 측면에서, 어구 "로 본질적으로 이루어진"은 본 개시내용의 항체 약물 접합체 및 제2 공-투여 작용제 이외의 1종 이상의 추가의 활성제의 포함을 명백하게 배제한다.
용어 "아미노산"은 자연 발생, 합성 및 비천연 아미노산, 뿐만 아니라 자연 발생 아미노산과 유사한 방식으로 기능하는 아미노산 유사체 및 아미노산 모방체를 지칭한다. 자연 발생 아미노산은 유전자 코드에 의해 코딩된 것, 뿐만 아니라 나중에 변형되는 그러한 아미노산, 예를 들어 히드록시프롤린, γ-카르복시글루타메이트 및 O-포스포세린이다. 아미노산 유사체는 자연 발생 아미노산과 동일한 기본 화학 구조, 즉 수소, 카르복실 기, 아미노 기 및 R 기에 결합된 α-탄소를 갖는 화합물, 예를 들어 호모세린, 노르류신, 메티오닌 술폭시드, 메티오닌 메틸 술포늄을 지칭한다. 이러한 유사체는 변형된 R 기 (예를 들어, 노르류신) 또는 변형된 펩티드 백본을 갖지만, 자연 발생 아미노산과 동일한 기본 화학 구조를 보유한다. 아미노산 모방체는 아미노산의 일반 화학 구조와 상이한 구조를 갖지만, 자연 발생 아미노산과 유사한 방식으로 기능하는 화학적 화합물을 지칭한다.
용어 "보존적으로 변형된 변이체"는 아미노산 및 핵산 서열 둘 다에 적용된다. 특정한 핵산 서열과 관련하여, 보존적으로 변형된 변이체는 동일한 또는 본질적으로 동일한 아미노산 서열을 코딩하는 그러한 핵산을 지칭하거나, 핵산이 아미노산 서열을 코딩하지 않는 경우에는 본질적으로 동일한 서열을 지칭한다. 유전자 코드의 축중성으로 인해, 다수의 기능적으로 동일한 핵산은 임의의 주어진 단백질을 코딩한다. 예를 들어, 코돈 GCA, GCC, GCG 및 GCU는 모두 아미노산 알라닌을 코딩한다. 따라서, 알라닌이 코돈에 의해 명시되는 모든 위치에서, 코돈은 코딩되는 폴리펩티드를 변경하지 않으면서, 기재된 상응하는 코돈 중 임의의 것으로 변경될 수 있다. 이러한 핵산 변이는 보존적으로 변형된 변이의 일종인 "침묵 변이"이다. 폴리펩티드를 코딩하는 본원의 모든 핵산 서열은 또한 핵산의 모든 가능한 침묵 변이를 기재한다. 관련 기술분야의 통상의 기술자는 핵산 내의 각 코돈 (통상적으로 메티오닌에 대한 유일한 코돈인 AUG, 및 통상적으로 트립토판에 대한 유일한 코돈인 TGG 제외)이 기능적으로 동일한 분자를 생성하도록 변형될 수 있음을 인식할 것이다. 따라서, 폴리펩티드를 코딩하는 핵산의 각각의 침묵 변이는 각각의 기재된 서열에 내포된다.
폴리펩티드 서열의 경우, "보존적으로 변형된 변이체"는 아미노산을 화학적으로 유사한 아미노산으로 치환시키는, 폴리펩티드 서열에 대한 개별 치환, 결실 또는 부가를 포함한다. 기능적으로 유사한 아미노산을 제공하는 보존적 치환 표가 관련 기술분야에 널리 공지되어 있다. 이러한 보존적으로 변형된 변이체는 다형성 변이체, 종간 상동체 및 대립유전자에 추가적이며 이들을 배제하지 않는다. 하기 8개의 군은 서로에 대해 보존적 치환인 아미노산을 함유한다: 1) 알라닌 (A), 글리신 (G); 2) 아스파르트산 (D), 글루탐산 (E); 3) 아스파라긴 (N), 글루타민 (Q); 4) 아르기닌 (R), 리신 (K); 5) 이소류신 (I), 류신 (L), 메티오닌 (M), 발린 (V); 6) 페닐알라닌 (F), 티로신 (Y), 트립토판 (W); 7) 세린 (S), 트레오닌 (T); 및 8) 시스테인 (C), 메티오닌 (M) (예를 들어, 문헌 [Creighton, Proteins (1984)] 참조). 일부 측면에서, 용어 "보존적 서열 변형"은 이러한 아미노산 서열을 함유하는 항체의 결합 특징에 유의하게 영향을 미치거나 변경시키지 않는 아미노산 변형을 지칭하는 것으로 사용된다.
본원에 사용된 용어 "최적화된"은 생산 세포 또는 유기체, 일반적으로는 진핵 세포, 예를 들어 효모 세포, 피키아(Pichia) 세포, 진균 세포, 트리코더마(Trichoderma) 세포, 차이니즈 햄스터 난소 세포 (CHO) 또는 인간 세포에서 선호되는 코돈을 사용하여 아미노산 서열을 코딩하도록 변경된 뉴클레오티드 서열을 지칭한다. 최적화된 뉴클레오티드 서열은 "모" 서열로도 또한 공지되어 있는 출발 뉴클레오티드 서열에 의해 본래 코딩되는 아미노산 서열을 완전하게 또는 가능한 한 많이 보유하도록 조작된다.
2개 이상의 핵산 또는 폴리펩티드 서열과 관련하여 용어 "퍼센트 동일한" 또는 "퍼센트 동일성"은 2개 이상의 서열 또는 하위서열이 동일한 정도를 지칭한다. 2개의 서열은 비교되는 영역에 걸쳐 아미노산 또는 뉴클레오티드의 동일한 서열을 갖는 경우에 "동일"하다. 2개의 서열은, 하기 서열 비교 알고리즘 중 하나를 사용하거나 또는 수동 정렬 및 육안 검사에 의한 측정시에 비교 윈도우 또는 지정된 영역에 걸쳐 최대 상응도로 비교 및 정렬하였을 때, 2개의 서열이 동일한 아미노산 잔기 또는 뉴클레오티드의 명시된 백분율 (즉, 명시된 영역에 걸쳐, 또는 명시되지 않은 경우에는 전체 서열에 걸쳐 60% 동일성, 임의로 65%, 70%, 75%, 80%, 85%, 90%, 95% 또는 99% 동일성)을 갖는 경우에 "실질적으로 동일하다". 임의로, 동일성은 적어도 약 30개 뉴클레오티드 (또는 10개 아미노산) 길이인 영역에 걸쳐, 또는 보다 바람직하게는 100 내지 500개 또는 1000개 또는 그 초과의 뉴클레오티드 (또는 20개, 50개, 200개 또는 그 초과의 아미노산) 길이인 영역에 걸쳐 존재한다.
서열 비교를 위해, 전형적으로 한 서열이 시험 서열과 비교되는 참조 서열로서 역할을 한다. 서열 비교 알고리즘을 사용하는 경우에, 시험 및 참조 서열을 컴퓨터 내로 입력하고, 필요한 경우에 하위서열 좌표를 지정하고, 서열 알고리즘 프로그램 파라미터를 지정한다. 디폴트 프로그램 파라미터를 사용할 수 있거나, 또는 대안적 파라미터를 지정할 수 있다. 이어서, 서열 비교 알고리즘은 프로그램 파라미터에 기반하여 참조 서열에 대한 시험 서열의 퍼센트 서열 동일성을 계산한다.
본원에 사용된 "비교 윈도우"는 2개의 서열을 최적으로 정렬시킨 후에 서열을 인접 위치의 동일한 수의 참조 서열과 비교할 수 있는, 20 내지 600개, 통상적으로는 약 50 내지 약 200개, 보다 통상적으로는 약 100 내지 약 150개로 이루어진 군으로부터 선택된 인접 위치의 수 중 어느 하나의 절편에 대한 언급을 포함한다. 비교를 위한 서열 정렬 방법은 관련 기술분야에 널리 공지되어 있다. 비교를 위한 최적의 서열 정렬은, 예를 들어 문헌 [Smith and Waterman, Adv. Appl. Math. 2:482c (1970)]의 국부 상동성 알고리즘, 문헌 [Needleman and Wunsch, J. Mol. Biol. 48:443 (1970)]의 상동성 정렬 알고리즘, 문헌 [Pearson and Lipman, Proc. Natl. Acad. Sci. USA 85:2444 (1988)]의 유사성 검색 방법, 이들 알고리즘의 컴퓨터 구현 (위스콘신주 매디슨 사이언스 드라이브 575 소재의 제네틱스 컴퓨터 그룹(Genetics Computer Group)의 위스콘신 제네틱스 소프트웨어 패키지(Wisconsin Genetics Software Package)의 GAP, BESTFIT, FASTA 및 TFASTA), 또는 수동 정렬 및 육안 검사 (예를 들어, 문헌 [Brent et al., Current Protocols in Molecular Biology, 2003] 참조)에 의해 수행될 수 있다.
퍼센트 서열 동일성 및 서열 유사성을 결정하는데 적합한 알고리즘의 두 예는 BLAST 및 BLAST 2.0 알고리즘이고, 이들은 각각 문헌 [Altschul et al., Nuc. Acids Res. 25:3389-3402, 1977] 및 [Altschul et al., J. Mol. Biol. 215:403-410, 1990]에 기재되어 있다. BLAST 분석을 수행하기 위한 소프트웨어는 국립 생물 정보 센터(National Center for Biotechnology Information)를 통해 공중 이용가능하다. 이러한 알고리즘은 먼저 데이터베이스 서열 내 동일한 길이의 워드와 정럴하였을 때 어떤 양의 값의 역치 점수 T에 매칭되거나 이를 충족시키는, 질의 서열 내의 길이 W의 짧은 워드를 확인함으로써 높은 점수의 서열 쌍 (HSP)을 확인하는 것을 수반한다. T는 이웃 워드 점수 역치로 지칭된다 (Altschul et al., 상기 문헌). 이들 초기 이웃 워드 히트는 이를 함유하는 보다 긴 HSP를 찾는 검색을 개시하기 위한 시드로서 작용한다. 워드 히트는 누적 정렬 점수가 증가할 수 있는 한, 각각의 서열을 따라 두 방향으로 연장된다. 누적 점수는, 뉴클레오티드 서열의 경우, 파라미터 M (매칭 잔기의 쌍에 대한 보상 점수; 항상 > 0) 및 N (미스매칭 잔기에 대한 페널티 점수; 항상 < 0)을 사용하여 계산된다. 아미노산 서열의 경우, 누적 점수를 계산하기 위해 점수화 매트릭스가 사용된다. 워드 히트의 각각의 방향으로의 확장은 다음 경우일 때 중지된다: 누적 정렬 점수가 그의 최대 달성 값으로부터 X 양만큼 하락한 경우; 누적 점수가 1개 이상의 음으로 점수화된 잔기 정렬의 축적으로 인해 0 이하로 떨어진 경우; 또는 어느 한 서열의 말단에 도달한 경우. BLAST 알고리즘 파라미터 W, T 및 X는 정렬의 감도 및 속도를 결정한다. BLASTN 프로그램 (뉴클레오티드 서열의 경우)은 디폴트로서 워드 길이 (W) 11, 예상값 (E) 10, M=5, N=-4 및 두 가닥의 비교를 사용한다. 아미노산 서열의 경우, BLASTP 프로그램은 디폴트로서 워드 길이 3, 및 예상값 (E) 10, 및 BLOSUM62 점수화 매트릭스 (문헌 [Henikoff and Henikoff, (1989) Proc. Natl. Acad. Sci. USA 89:10915] 참조) 정렬 (B) 50, 예상값 (E) 10, M=5, N=-4, 및 두 가닥의 비교를 사용한다.
BLAST 알고리즘은 또한 2개의 서열 사이의 유사성의 통계적 분석을 수행한다 (예를 들어, 문헌 [Karlin and Altschul, Proc. Natl. Acad. Sci. USA 90:5873-5787, 1993] 참조). BLAST 알고리즘에 의해 제공되는 유사성의 한 척도는 2개의 뉴클레오티드 또는 아미노산 서열 사이의 매칭이 우연히 발생할 확률의 지표를 제공하는 최소 합계 확률 (P(N))이다. 예를 들어, 핵산은 참조 핵산에 대한 시험 핵산의 비교에서 최소 합계 확률이 약 0.2 미만, 보다 바람직하게는 약 0.01 미만, 가장 바람직하게는 약 0.001 미만인 경우에 참조 서열과 유사한 것으로 간주된다.
2개의 아미노산 서열 사이의 퍼센트 동일성은 또한, PAM120 가중치 잔기 표, 갭 길이 페널티 12 및 캡 페널티 4를 사용하여 ALIGN 프로그램 (버전 2.0) 내로 혼입된 문헌 [E. Meyers and W. Miller, Comput. Appl. Biosci. 4:11-17, 1988]의 알고리즘을 사용하여 결정될 수 있다. 추가로, 2개의 아미노산 서열 사이의 퍼센트 동일성은 Blossom 62 매트릭스 또는 PAM250 매트릭스, 및 갭 가중치 16, 14, 12, 10, 8, 6 또는 4 및 길이 가중치 1, 2, 3, 4, 5 또는 6을 사용하여 GCG 소프트웨어 패키지 내의 GAP 프로그램 내로 혼입된 문헌 [Needleman and Wunsch, J. Mol. Biol. 48:444-453, 1970]의 알고리즘을 사용하여 결정될 수 있다.
상기 나타낸 서열 동일성의 백분율 이외에, 2개의 핵산 서열 또는 폴리펩티드가 실질적으로 동일하다는 또 다른 지표는, 제1 핵산에 의해 코딩되는 폴리펩티드가 하기 기재된 바와 같이 제2 핵산에 의해 코딩되는 폴리펩티드에 대해 생성된 항체와 면역학적으로 교차 반응성이라는 것이다. 따라서, 폴리펩티드는 전형적으로, 예를 들어 2개의 펩티드가 단지 보존적 치환에 의해서만 상이한 경우에 제2 폴리펩티드와 실질적으로 동일하다. 2개의 핵산 서열이 실질적으로 동일하다는 또 다른 지표는, 2개의 분자 또는 그의 상보체가 하기 기재된 바와 같이 엄격한 조건 하에서 서로 혼성화된다는 것이다. 2개의 핵산 서열이 실질적으로 동일하다는 또 다른 지표는, 동일한 프라이머가 서열을 증폭시키는데 사용될 수 있다는 것이다.
용어 "핵산"은 본원에서 용어 "폴리뉴클레오티드"와 상호교환가능하게 사용되고, 단일- 또는 이중-가닥 형태의 데옥시리보뉴클레오티드 또는 리보뉴클레오티드 및 그의 중합체를 지칭한다. 용어는 합성, 자연 발생 및 비-자연 발생이고, 참조 핵산과 유사한 결합 특성을 갖고, 참조 뉴클레오티드와 유사한 방식으로 대사되는, 공지된 뉴클레오티드 유사체 또는 변형된 백본 잔기 또는 연결부를 함유하는 핵산을 포괄한다. 이러한 유사체의 예는 비제한적으로, 포스포로티오에이트, 포스포르아미데이트, 메틸 포스포네이트, 키랄-메틸 포스포네이트, 2-O-메틸 리보뉴클레오티드, 펩티드-핵산 (PNA)을 포함한다.
달리 나타내지 않는 한, 특정한 핵산 서열은 또한, 명백하게 나타낸 서열, 뿐만 아니라 그의 보존적으로 변형된 변이체 (예를 들어, 축중성 코돈 치환) 및 상보적 서열도 함축적으로 포괄한다. 구체적으로, 하기에서 상술되는 바와 같이, 1개 이상의 선택된 (또는 모든) 코돈의 제3 위치가 혼합-염기 및/또는 데옥시이노신 잔기로 치환된 서열을 생성시킴으로써 축중성 코돈 치환이 달성될 수 있다 (Batzer et al., (1991) Nucleic Acid Res. 19:5081; Ohtsuka et al., (1985) J. Biol. Chem. 260:2605-2608; 및 Rossolini et al., (1994) Mol. Cell. Probes 8:91-98).
핵산과 관련하여 용어 "작동가능하게 연결된"은 2개 이상의 폴리뉴클레오티드 (예를 들어, DNA) 절편 사이의 기능적 관계를 지칭한다. 전형적으로, 이는 전사된 서열에 대한 전사 조절 서열의 기능적 관계를 지칭한다. 예를 들어, 프로모터 또는 인핸서 서열은 그가 적절한 숙주 세포 또는 다른 발현 시스템에서 코딩 서열의 전사를 자극하거나 조정하는 경우에 코딩 서열에 작동가능하게 연결되어 있다. 일반적으로, 전사된 서열에 작동가능하게 연결되어 있는 프로모터 전사 조절 서열은 전사된 서열에 물리적으로 인접하며, 즉 이는 시스-작용성이다. 그러나, 일부 전사 조절 서열, 예컨대 인핸서는 전사를 증진시키는 코딩 서열에 물리적으로 인접하거나 근접하여 위치할 필요가 없다.
용어 "폴리펩티드" 또는 "단백질"은 아미노산 잔기의 중합체를 지칭하는 것으로 본원에서 상호교환가능하게 사용된다. 상기 용어는 자연 발생 아미노산 중합체 및 비-자연 발생 아미노산 중합체 뿐만 아니라, 1개 이상의 아미노산 잔기가 상응하는 자연 발생 아미노산의 인공 화학적 모방체인 아미노산 중합체에도 적용된다. 달리 나타내지 않는 한, 특정한 폴리펩티드 서열은 또한 그의 보존적으로 변형된 변이체도 함축적으로 포괄한다.
본원에 사용된 용어 "면역접합체" 또는 "항체 약물 접합체"는 항체 또는 그의 항원 결합 단편과 또 다른 작용제, 예컨대 페이로드, 약물 모이어티, 화학요법제, 독소, 면역요법제, 영상화 프로브 등과의 연결을 지칭한다. 연결은 공유 결합, 또는 비-공유 상호작용, 예컨대 정전기력을 통한 상호작용일 수 있다. 관련 기술분야에 공지된 다양한 링커가 면역접합체를 형성하기 위해 사용될 수 있다. 추가로, 면역접합체는 면역접합체를 코딩하는 폴리뉴클레오티드로부터 발현될 수 있는 융합 단백질 형태로 제공될 수 있다. 본원에 사용된 "융합 단백질"은 본래 개별 단백질 (펩티드 및 폴리펩티드 포함)을 코딩하는 2종 이상의 유전자 또는 유전자 단편의 연결을 통해 생성된 단백질을 지칭한다. 융합 유전자의 번역은 각각의 본래 단백질로부터 유래된 기능적 특성을 갖는 단일 단백질을 생성한다.
용어 "대상체"는 인간 및 비-인간 동물을 포함한다. 비-인간 동물은 모든 척추동물, 예를 들어 포유동물 및 비-포유동물, 예컨대 비-인간 영장류, 양, 개, 소, 닭, 양서류 및 파충류를 포함한다. 언급된 경우를 제외하고, 용어 "환자" 또는 "대상체"는 본원에서 상호교환가능하게 사용된다.
본원에 사용된 용어 "독소", "세포독소" 또는 "세포독성제"는 세포의 성장 및 증식에 유해하며 세포 또는 악성종양을 감소시키거나 억제하거나 또는 파괴하는 작용을 할 수 있는 임의의 작용제를 지칭한다.
본원에 사용된 용어 "항암제"는 세포독성제, 화학요법제, 방사선요법 및 방사선요법제, 표적화 항암제 및 면역요법제를 포함하나 이에 제한되지는 않는, 암과 같은 세포 증식성 장애를 치료하는데 사용될 수 있는 임의의 작용제를 지칭한다.
본원에 사용된 용어 "약물 모이어티" 또는 "페이로드"는 항체 또는 항원 결합 단편에 접합된 화학적 모이어티를 지칭하며, 임의의 치료제 또는 진단제, 예를 들어 항암제, 항염증제, 항감염제 (예를 들어, 항진균제, 항박테리아제, 항기생충제, 항바이러스제) 또는 마취제를 포함할 수 있다. 특정 측면에서, 약물 모이어티는 V-ATPase 억제제, HSP90 억제제, IAP 억제제, mTor 억제제, 미세관 안정화제, 미세관 탈안정화제, 아우리스타틴, 돌라스타틴, 메이탄시노이드, MetAP (메티오닌 아미노펩티다제), 단백질 CRM1의 핵 유출의 억제제, DPPIV 억제제, 미토콘드리아에서의 포스포릴 전달 반응의 억제제, 단백질 합성 억제제, 키나제 억제제, CDK2 억제제, CDK9 억제제, 프로테아솜 억제제, 키네신 억제제, HDAC 억제제, DNA 손상 작용제, DNA 알킬화제, DNA 삽입제, DNA 작은 홈 결합제 및 DHFR 억제제로부터 선택된다. 이들 각각을 본 개시내용의 항체 및 방법과 상용성인 링커에 부착시키는 방법은 관련 기술분야에 공지되어 있다. 예를 들어, 문헌 [Singh et al., (2009) Therapeutic Antibodies: Methods and Protocols, vol. 525, 445-457]을 참조한다. 추가로, 페이로드는 생물물리학적 프로브, 형광단, 스핀 표지, 적외선 프로브, 친화도 프로브, 킬레이트화제, 분광학적 프로브, 방사성 프로브, 지질 분자, 폴리에틸렌 글리콜, 중합체, 스핀 표지, DNA, RNA, 단백질, 펩티드, 표면, 항체, 항체 단편, 나노입자, 양자점, 리포솜, PLGA 입자, 사카라이드 또는 폴리사카라이드일 수 있다.
용어 "메이탄시노이드 약물 모이어티"는 메이탄시노이드 화합물의 구조를 갖는 항체-약물 접합체의 하위구조를 의미한다. 메이탄신은 동아프리카 관목 메이테누스 세라타(Maytenus serrata)에서 최초로 단리되었다 (미국 특허 번호 3,896,111). 이후, 특정 미생물이 메이탄시노이드, 예컨대 메이탄시놀 및 C-3 메이탄시놀 에스테르를 또한 생성한다는 것이 발견되었다 (미국 특허 번호 4,151,042). 합성 메이탄시놀 및 메이탄시놀 유사체가 보고되었다. 미국 특허 번호 4,137,230; 4,248,870; 4,256,746; 4,260,608; 4,265,814; 4,294,757; 4,307,016; 4,308,268; 4,308,269; 4,309,428; 4,313,946; 4,315,929; 4,317,821; 4,322,348; 4,331,598; 4,361,650; 4,364,866; 4,424,219; 4,450,254; 4,362,663; 및 4,371,533, 및 문헌 [Kawai et al. (1984) Chem. Pharm. Bull. 3441-3451]을 참조한다 (이들 각각은 참조로 명백하게 포함됨). 접합에 유용한 메이탄시노이드의 구체적 예는 DM1, DM3 및 DM4를 포함한다.
"종양"은 신생물성 세포 성장 및 증식 (악성인지 양성인지에 관계없이), 및 모든 전암성 및 암성 세포 및 조직을 지칭한다.
용어 "항종양 활성"은 종양 세포 증식, 생존율 또는 전이 활성의 비율의 감소를 의미한다. 항종양 활성을 나타내는 가능한 방식은 종양 세포의 성장 속도의 저하, 종양 크기 정지 또는 종양 크기 감소를 나타내는 것이다. 이러한 활성은 이종이식편 모델, 동종이식편 모델, MMTV 모델, 및 항종양 활성을 조사하기 위한 관련 기술분야에 공지된 다른 공지된 모델을 포함하나 이에 제한되지는 않는 허용된 시험관내 또는 생체내 종양 모델을 사용하여 평가될 수 있다.
용어 "악성종양"은 비-양성 종양 또는 암을 지칭한다. 본원에 사용된 용어 "암"은 탈조절되거나 비제어된 세포 성장을 특징으로 하는 악성종양을 포함한다. 예시적인 암은 다음을 포함한다: 암종, 육종, 백혈병 및 림프종.
용어 "암"은 원발성 악성 종양 (예를 들어, 세포가 대상체의 신체에서 본래 종양의 부위 이외의 다른 부위로 이동하지 않은 종양) 및 속발성 악성 종양 (예를 들어, 종양 세포가 본래 종양의 부위와 상이한 속발성 부위로 이동하는 전이로부터 발생하는 종양)을 포함한다.
용어 "카드헤린 6" 또는 "CDH6"은 세포-세포 부착 분자의 카드헤린 패밀리의 구성원인 세포 부착 분자를 지칭한다. CDH6의 핵산 및 아미노산 서열은 공지되어 있으며, 또한 진뱅크(GenBank) 수탁번호 AK291290 (단백질 수탁번호 BAF83979.1)로 공개되어 있다. 또한, 인간 CDH6 cDNA에 대해 서열식별번호: 1 및 인간 CDH6 단백질 서열에 대해 서열식별번호: 2를 참조한다. 구조적으로, CDH6 수용체는 5개의 세포외 카드헤린 반복부를 갖는 유형 II 카드헤린이고, 그의 전장에 걸쳐 서열식별번호: 2의 아미노산 서열과 적어도 약 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다. 구조적으로, CDH6 핵산 서열은 그의 전장에 걸쳐 서열식별번호: 1의 핵산 서열과 적어도 약 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 서열 동일성을 갖는다.
용어 "CDH6 발현 암" 또는 "CDH6 양성 암"은 암 세포의 표면 상에 CDH6 및/또는 CDH6의 돌연변이체 형태를 발현하는 암을 지칭한다.
본원에 사용된, 임의의 질환 또는 장애에 대한 용어 "치료하다", "치료하는" 또는 "치료"는, 한 측면에서, 질환 또는 장애를 개선하는 것 (즉, 질환 또는 그의 임상 증상 중 적어도 1종의 발달을 둔화시키거나 저지하거나 감소시키는 것)을 지칭한다. 또 다른 측면에서, "치료하다", "치료하는" 또는 "치료"는 환자가 식별할 수 없는 것을 포함하는 적어도 1종의 신체 파라미터를 완화시키거나 개선하는 것을 지칭한다. 또 다른 측면에서, "치료하다", "치료하는" 또는 "치료"는 신체적으로 (예를 들어, 식별가능한 증상의 안정화), 생리학적으로 (예를 들어, 신체 파라미터의 안정화), 또는 둘 다로 질환 또는 장애를 조정하는 것을 지칭한다. 또 다른 측면에서, "치료하다", "치료하는" 또는 "치료"는 질환 또는 장애의 발병 또는 발달 또는 진행을 방지하거나 지연시키는 것을 지칭한다.
용어 "치료상 허용되는 양" 또는 "치료 유효 용량"은 상호교환가능하게, 목적하는 결과 (즉, 종양 크기의 감소, 종양 성장의 억제, 전이의 방지, 바이러스, 박테리아, 진균 또는 기생충 감염의 억제 또는 방지)를 일으키는데 충분한 양을 지칭한다. 일부 측면에서, 치료상 허용되는 양은 바람직하지 않은 부작용을 유도하거나 야기하지 않는다. 치료상 허용되는 양은 먼저 저용량을 투여하고, 이어서 목적하는 효과가 달성될 때까지 그 용량을 점차 증가시킴으로써 결정될 수 있다. 본 개시내용의 분자의 "예방 유효 투여량" 및 "치료 유효 투여량"은 각각 질환 증상 (암과 연관된 증상 포함)의 발병을 방지하거나 또는 그의 중증도의 감소를 유발할 수 있다.
용어 "공-투여하다"는 개체의 혈액 중 2종의 활성제의 동시 존재를 지칭한다. 공-투여되는 활성제는 공동으로 또는 순차적으로 전달될 수 있다.
도 1은 OVCAR3에서의 항-CDH6 항체의 세포 결합 EC50의 그래프 도시이다.
도 2는 항-CDH6 항체의 에피토프 비닝을 보여준다.
도 3은 CDH6의 5개의 EC 도메인의 중수소 맵핑을 나타낸다.
도 4는 CDH2 ECD의 공지된 구조 상에 오버레이된 CDH6 EC5 도메인에 결합하는 NOV0710의 결정 구조이다.
도 5는 CDH6에 결합하는 NOV0712의 결정 구조를 도시한다.
도 6은 CDH6에 결합하는 NOV0710의 결정 구조를 도시한다.
도 7은 CDH6의 돌연변이체 형태에 결합하는 3종의 항-CDH6 항체의 능력을 보여주는 ELISA 검정이다.
도 8은 인간, 시노몰구스, 래트 또는 마우스 CDH6을 발현하는 CHO 세포주에 결합하는, 비접합 또는 술포-SPDB-DM4 링커/페이로드와 접합된 항-CDH6 항체의 FACS 데이터이다.
도 9는 CDH6 형질감염된 세포주 및 CDH6 siRNA 녹다운을 갖는 세포주를 포함한 난소암 세포주에서의 CDH6 발현의 수준을 보여주는 FACS 그래프이다.
도 10은 난소암 세포주의 패널에서 비접합 또는 술포-SPDB-DM4 링커/페이로드와 접합된 항-CDH6 항체를 보여준다.
도 11은 난소암 세포주에 대한 DM1 또는 DM4에 접합되었을 때의 항-CDH6 항체의 시험관내 활성을 보여준다.
도 12는 비접합 항-CDH6 항체 또는 술포-SPDB-DM4에 접합된 항-CDH6 항체의 시험관내 활성을 보여준다.
도 13은 난소암 이종이식편 마우스 모델에서 시험된, DM1에 접합되었을 때의 항-CDH6 항체의 효능을 입증한다.
도 14는 난소암 이종이식편 마우스 모델에서 시험된, DM1에 접합되었을 때의 항-CDH6 항체의 효능을 입증한다.
도 15는 난소암 이종이식편 마우스 모델에서 시험된, SPDB-DM4가 접합되었을 때의 항-CDH6 항체의 효능을 보여준다.
도 16은 난소암 이종이식편 마우스 모델에서 NOV0712-SPDB-DM4 및 NOV0712-SMCC-DM1의 효능을 비교한다.
도 17은 난소암의 환자-유래된 원발성 종양 이종이식편 (PTX) 마우스 모델에서 SPDB-DM4, 술포-SPDB-DM4 및 SMCC-DM1 링커/페이로드 조합을 갖는 NOV0712의 효능을 비교한다.
도 18은 난소암 이종이식편 마우스 모델에서 SPDB-DM4 또는 술포-SPDB-DM4에 접합되었을 때의 NOV0712의 상이한 용량의 효능을 보여준다.
도 19는 환자-유래된 원발성 난소 종양 이종이식편 마우스 모델에서 술포-SPDB-DM4 또는 SMCC-DM1 링커 페이로드 조합을 갖는 NOV0712를 비교한다.
도 20은 환자 유래된 원발성 난소암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 입증한다.
도 21은 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 22는 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 23은 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 24는 CDH6-표적화 ADC와 BCL2/BCL-Xl, BCL-Xl, IAP 및 MEK 억제제와의 조합 활성에 대한 로에베(Loewe) 상승작용 점수를 제공한다.
도 25는 암성 조직에서 CDH6 mRNA 발현의 그래프 표현이다.
도 26은 난소암, 자궁내막암, 신암 및 담관암종 조직에서 면역조직화학에 의한 CDH6 단백질 발현의 그래프 표현이다.
도 2는 항-CDH6 항체의 에피토프 비닝을 보여준다.
도 3은 CDH6의 5개의 EC 도메인의 중수소 맵핑을 나타낸다.
도 4는 CDH2 ECD의 공지된 구조 상에 오버레이된 CDH6 EC5 도메인에 결합하는 NOV0710의 결정 구조이다.
도 5는 CDH6에 결합하는 NOV0712의 결정 구조를 도시한다.
도 6은 CDH6에 결합하는 NOV0710의 결정 구조를 도시한다.
도 7은 CDH6의 돌연변이체 형태에 결합하는 3종의 항-CDH6 항체의 능력을 보여주는 ELISA 검정이다.
도 8은 인간, 시노몰구스, 래트 또는 마우스 CDH6을 발현하는 CHO 세포주에 결합하는, 비접합 또는 술포-SPDB-DM4 링커/페이로드와 접합된 항-CDH6 항체의 FACS 데이터이다.
도 9는 CDH6 형질감염된 세포주 및 CDH6 siRNA 녹다운을 갖는 세포주를 포함한 난소암 세포주에서의 CDH6 발현의 수준을 보여주는 FACS 그래프이다.
도 10은 난소암 세포주의 패널에서 비접합 또는 술포-SPDB-DM4 링커/페이로드와 접합된 항-CDH6 항체를 보여준다.
도 11은 난소암 세포주에 대한 DM1 또는 DM4에 접합되었을 때의 항-CDH6 항체의 시험관내 활성을 보여준다.
도 12는 비접합 항-CDH6 항체 또는 술포-SPDB-DM4에 접합된 항-CDH6 항체의 시험관내 활성을 보여준다.
도 13은 난소암 이종이식편 마우스 모델에서 시험된, DM1에 접합되었을 때의 항-CDH6 항체의 효능을 입증한다.
도 14는 난소암 이종이식편 마우스 모델에서 시험된, DM1에 접합되었을 때의 항-CDH6 항체의 효능을 입증한다.
도 15는 난소암 이종이식편 마우스 모델에서 시험된, SPDB-DM4가 접합되었을 때의 항-CDH6 항체의 효능을 보여준다.
도 16은 난소암 이종이식편 마우스 모델에서 NOV0712-SPDB-DM4 및 NOV0712-SMCC-DM1의 효능을 비교한다.
도 17은 난소암의 환자-유래된 원발성 종양 이종이식편 (PTX) 마우스 모델에서 SPDB-DM4, 술포-SPDB-DM4 및 SMCC-DM1 링커/페이로드 조합을 갖는 NOV0712의 효능을 비교한다.
도 18은 난소암 이종이식편 마우스 모델에서 SPDB-DM4 또는 술포-SPDB-DM4에 접합되었을 때의 NOV0712의 상이한 용량의 효능을 보여준다.
도 19는 환자-유래된 원발성 난소 종양 이종이식편 마우스 모델에서 술포-SPDB-DM4 또는 SMCC-DM1 링커 페이로드 조합을 갖는 NOV0712를 비교한다.
도 20은 환자 유래된 원발성 난소암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 입증한다.
도 21은 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 22는 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 23은 환자 유래된 원발성 신암의 이종이식편 마우스 모델에서 술포-SPDB-DM4에 접합된 항-CDH6 항체의 생체내 효능을 보여준다.
도 24는 CDH6-표적화 ADC와 BCL2/BCL-Xl, BCL-Xl, IAP 및 MEK 억제제와의 조합 활성에 대한 로에베(Loewe) 상승작용 점수를 제공한다.
도 25는 암성 조직에서 CDH6 mRNA 발현의 그래프 표현이다.
도 26은 난소암, 자궁내막암, 신암 및 담관암종 조직에서 면역조직화학에 의한 CDH6 단백질 발현의 그래프 표현이다.
본 개시내용은 CDH6에 결합하는 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체를 제공한다. 특히, 본 개시내용은 CDH6에 결합하고 이러한 결합 시에 내재화되는 항체 및 항체 단편 (예를 들어, 항원 결합 단편)에 관한 것이다. 본 개시내용의 항체 및 항체 단편 (예를 들어, 항원 결합 단편)은 항체 약물 접합체를 생산하는데 사용될 수 있다. 또한, 본 개시내용은 바람직한 약동학 특징 및 다른 바람직한 속성을 갖는 항체 약물 접합체를 제공하고, 따라서 CDH6을 발현하는 암, 제한 없이 예를 들어: 난소암, 신암, 간암, 연부조직암, CNS암, 갑상선암 및 담관암종을 치료하는데 사용될 수 있다. 본 개시내용은 항체 약물 접합체를 포함하는 제약 조성물 및 암의 치료를 위해 이러한 제약 조성물을 제조 및 사용하는 방법을 추가로 제공한다.
항체 약물 접합체
본 개시내용은 CDH6에 특이적으로 결합하는 항체, 항원 결합 단편 또는 그의 기능적 등가물이 약물 모이어티에 연결된 항체 약물 접합체를 제공한다. 한 측면에서, 항체, 항원 결합 단편 또는 그의 기능적 등가물은 링커에 의한 공유 부착을 통해 항암제인 약물 모이어티에 연결된다. 항체 약물 접합체는 유효 용량의 항암제 (예를 들어, 세포독성제)를 CDH6을 발현하는 종양 조직에 선택적으로 전달할 수 있고, 이에 의해 보다 큰 선택성 (및 보다 낮은 유효 용량)이 달성될 수 있다.
한 측면에서, 본 개시내용은 하기 화학식 I의 면역접합체를 제공한다:
Ab-(L-(D)m)n
여기서 Ab는 본원에 기재된 CDH6 결합 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 나타내고;
L은 링커이고;
D는 약물 모이어티이고;
m은 1-8의 정수이고;
n은 1 내지 20의 정수이다. 한 측면에서, n은 1 내지 10, 2 내지 8, 또는 2 내지 5의 정수이다. 구체적 측면에서, n은 3 내지 4이다. 일부 측면에서, m은 1이다. 일부 측면에서, m은 2, 3 또는 4이다.
항체에 대한 약물 모이어티 비가 특정 접합체 분자에 대한 정확한 값 (예를 들어, 화학식 I에서 n 곱하기 m)을 갖는 경우에, 값은 종종 많은 분자를 함유하는 샘플을 기재하기 위해 사용될 때 전형적으로 접합 단계와 연관된 어느 정도의 불균질성으로 인해 평균 값일 것으로 이해된다. 면역접합체의 샘플에 대한 평균 로딩은 본원에서 항체에 대한 약물 모이어티 비 또는 "DAR"로 지칭된다. 메이탄시노이드의 측면에서, 이는 항체에 대한 메이탄시노이드 비 또는 "MAR"로서 지칭될 수 있다. 일부 측면에서, DAR은 약 1 내지 약 5이고, 전형적으로 약 3, 3.5, 4, 4.5 또는 5이다. 일부 측면에서, 샘플의 적어도 50 중량%는 평균 DAR ± 2를 갖는 화합물이고, 바람직하게는 샘플의 적어도 50%는 평균 DAR ± 1을 함유하는 접합체이다. 다른 측면은 DAR이 약 3.5인 면역접합체를 포함한다. 일부 측면에서, '약 n'의 DAR은 DAR에 대한 측정된 값이 n의 20% 이내인 것을 의미한다.
본 개시내용은 약물 모이어티에 연결 또는 접합된 본원에 개시된 바와 같은 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 그의 기능적 등가물을 포함하는 면역접합체를 제공한다. 한 측면에서, 약물 모이어티 D는 하기 구조를 갖는 것을 포함한, 메이탄시노이드 약물 모이어티이다:
여기서, 파상선은 항체 약물 접합체의 링커에 대한 메이탄시노이드의 황 원자의 공유 부착을 나타낸다. R은 각 경우에 독립적으로 H 또는 C1-C6 알킬이다. 황 원자에 아미드 기를 부착시키는 알킬렌 기는 메타닐, 에탄일 또는 프로파닐일 수 있고, 즉 m은 1, 2, 또는 3이다. (미국 특허 번호 633,410, 미국 특허 번호 5,208,020, 문헌 [Chari et al. (1992) Cancer Res. 52;127-131, Lui et al. (1996) Proc. Natl. Acad. Sci. 93:8618-8623]).
개시된 면역접합체에 대해 메이탄시노이드 약물 모이어티의 모든 입체이성질체, 즉 메이탄시노이드의 키랄 탄소에서의 R 및 S 배위의 임의의 조합이 고려된다. 한 측면에서, 메이탄시노이드 약물 모이어티는 하기 입체화학을 갖는다.
한 측면에서, 메이탄시노이드 약물 모이어티는 N2'-데아세틸-N2'-(3-메르캅토-1-옥소프로필)-메이탄신 (또한 DM1로도 공지됨)이다. DM1은 하기 구조 화학식에 의해 나타내어진다.
또 다른 측면에서, 메이탄시노이드 약물 모이어티는 N2'-데아세틸-N2'-(4-메르캅토-1-옥소펜틸)-메이탄신 (또한 DM3으로도 공지됨)이다. DM3은 하기 구조 화학식에 의해 나타내어진다.
또 다른 측면에서, 메이탄시노이드 약물 모이어티는 N2'-데아세틸-N2'-(4-메틸-4-메르캅토-1-옥소펜틸)-메이탄신 (또한 DM4로도 공지됨)이다. DM4는 하기 구조 화학식에 의해 나타내어진다.
약물 모이어티 D는 링커 L을 통해 항체 Ab에 연결될 수 있다. L은 항체 Ab를 약물 모이어티 D에 연결시킬 수 있는 임의의 화학적 모이어티이다. 링커 L은 공유 결합(들)을 통해 항체 Ab를 약물 모이어티 D에 부착시킨다. 링커 시약은 약물 모이어티 D 및 항체 Ab를 연결시켜 항체 약물 접합체를 형성시키는데 사용될 수 있는 이관능성 또는 다관능성 모이어티이다. 항체 약물 접합체는 약물 모이어티 D 및 항체 Ab에 대한 결합을 위한 반응성 관능기를 갖는 링커를 사용하여 제조될 수 있다. 시스테인, 티올 또는 아민, 예를 들어 N-말단 또는 아미노산 측쇄, 예컨대 항체의 리신이 링커 시약의 관능기와 결합을 형성할 수 있다.
한 측면에서, L은 절단가능한 링커이다. 또 다른 측면에서, L은 비-절단가능한 링커이다. 일부 측면에서, L은 산-불안정성 링커, 광-불안정성 링커, 펩티다제 절단가능한 링커, 에스테라제 절단가능한 링커, 디술피드 결합 환원성 링커, 친수성 링커, 사전하전된 링커 또는 디카르복실산-기반 링커이다.
약물 모이어티 D, 예를 들어 메이탄시노이드와 항체 Ab 사이의 비-절단가능한 링커를 형성하는 적합한 가교 시약은 관련 기술분야에 널리 공지되어 있고, 황 원자를 포함하는 비-절단가능한 링커 (예컨대 SMCC) 또는 황 원자가 없는 것을 형성할 수 있다. 약물 모이어티 D, 예를 들어 메이탄시노이드와 항체 Ab 사이의 비-절단가능한 링커를 형성하는 바람직한 가교 시약은 말레이미도- 또는 할로아세틸-기반 모이어티를 포함한다. 본 개시내용에 따르면, 이러한 비-절단가능한 링커는 말레이미도- 또는 할로아세틸-기반 모이어티로부터 유래된다고 한다.
말레이미도-기반 모이어티를 포함하는 가교 시약은 N-숙신이미딜-4-(말레이미도메틸)시클로헥산카르복실레이트 (SMCC), 술포숙신이미딜 4-(N-말레이미도메틸) 시클로헥산-1-카르복실레이트 (술포-SMCC), N-숙신이미딜-4-(말레이미도메틸)시클로헥산-1-카르복시-(6-아미노카프로에이트) (SMCC의 "장쇄" 유사체임 (LC-SMCC)), κ-말레이미도운데칸산 N-숙신이미딜 에스테르 (KMUA), γ-말레이미도부티르산 N-숙신이미딜 에스테르 (GMBS), ε-말레이미도카프로산 N-숙신이미딜 에스테르 (EMCS), m-말레이미도벤조일-N-히드록시숙신이미드 에스테르 (MBS), N-(α-말레이미도아세톡시)-숙신이미드 에스테르 (AMSA), 숙신이미딜-6-(β-말레이미도프로피온아미도)헥사노에이트 (SMPH), N-숙신이미딜-4-(p-말레이미도페닐)-부티레이트 (SMPB), N-(-p-말레이미도페닐)이소시아네이트 (PMIP) 및 폴리에틸렌 글리콜 스페이서, 예컨대 MAL-PEG-NHS를 포함하나 이에 제한되지는 않는다. 이들 가교 시약은 말레이미도-기반 모이어티로부터 유래된 비-절단가능한 링커를 형성한다. 말레이미도-기반 가교 시약의 대표적인 구조가 하기에 제시된다.
또 다른 측면에서, 링커 L은 N-숙신이미딜-4-(말레이미도메틸)시클로헥산카르복실레이트 (SMCC), 술포숙신이미딜 4-(N-말레이미도메틸) 시클로헥산-1-카르복실레이트 (술포-SMCC) 또는 MAL-PEG-NHS로부터 유래된다.
할로아세틸-기반 모이어티를 포함하는 가교 시약은 N-숙신이미딜 아이오도아세테이트 (SIA), N-숙신이미딜(4-아이오도아세틸)아미노벤조에이트 (SIAB), N-숙신이미딜 브로모아세테이트 (SBA) 및 N-숙신이미딜 3-(브로모아세트아미도)프로피오네이트 (SBAP)를 포함한다. 이들 가교 시약은 할로아세틸-기반 모이어티로부터 유래된 비-절단가능한 링커를 형성한다. 할로아세틸-기반 가교 시약의 대표적인 구조가 하기에 제시된다.
한 측면에서, 링커 L은 N-숙신이미딜 아이오도아세테이트 (SIA) 또는 N-숙신이미딜(4-아이오도아세틸)아미노벤조에이트 (SIAB)로부터 유래된다.
약물 모이어티 D, 예를 들어 메이탄시노이드와 항체 Ab 사이의 절단가능한 링커를 형성하는 적합한 가교 시약은 관련 기술분야에 널리 공지되어 있다. 디술피드 함유 링커는 생리학적 조건 하에 발생할 수 있는 디술피드 교환을 통해 절단가능한 링커이다. 본 개시내용에 따르면, 이러한 절단가능한 링커는 디술피드-기반 모이어티로부터 유래된다고 한다. 적합한 디술피드 가교 시약은 N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP), N-숙신이미딜-4-(2-피리딜디티오)펜타노에이트 (SPP), N-숙신이미딜-4-(2-피리딜디티오)부타노에이트 (SPDB) 및 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)를 포함하며, 이들의 구조가 하기에 제시된다. 이들 디술피드 가교 시약은 디술피드-기반 모이어티로부터 유래된 절단가능한 링커를 형성한다.
N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP),
N-숙신이미딜-4-(2-피리딜디티오)펜타노에이트 (SPP),
N-숙신이미딜-4-(2-피리딜디티오)부타노에이트 (SPDB) 및
N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB).
한 측면에서, 링커 L은 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)으로부터 유래된다.
약물 모이어티 D, 예를 들어 메이탄시노이드와 항체 Ab 사이의 하전된 링커를 형성하는 적합한 가교 시약은 사전하전된 가교 시약으로서 공지되어 있다. 한 측면에서, 링커 L은 사전하전된 가교 시약 CX1-1로부터 유래된다. CX1-1의 구조는 하기와 같다.
2,5-디옥소피롤리딘-1-일 17-(2,5-디옥소-2,5-디히드로-1H-피롤-1-일)-5,8,11,14-테트라옥소-4,7,10,13-테트라아자헵타데칸-1-오에이트 (CX1-1)
본 개시내용에 의해 제공된 한 측면에서, 접합체는 하기 구조 화학식 중 어느 하나로 나타내어진다:
여기서
Ab는 인간 CDH6에 특이적으로 결합하는 항체 또는 그의 항원 결합 단편이고;
n은, Ab의 1급 아민과의 아미드 결합의 형성을 통해 Ab에 부착된 D-L 기의 수를 나타내며, 1 내지 20의 정수이다. 한 측면에서, n은 1 내지 10, 2 내지 8 또는 2 내지 5의 정수이다. 구체적 측면에서, n은 3 또는 4이다.
한 측면에서, 접합체 내 항체에 대한 약물 모이어티 (예를 들어, DM1 또는 DM4)의 평균 몰비 (즉, 메이탄시노이드 항체 비 (MAR)로 또한 공지된 평균 w 값)은 약 1 내지 약 10, 약 2 내지 약 8 (예를 들어, 1.9, 2.0, 2.1, 2.2, 2.3, 2.4, 2.5, 2.6, 2.7, 2.8, 2.9, 3.0, 3.1, 3.2, 3.3, 3.4, 3.5, 3.6, 3.7, 3.8, 3.9, 4.0, 4.1, 4.2, 4.3, 4.4, 4.5, 4.6, 4.7, 4.8, 4.9, 5.0, 5.1, 5.2, 5.3, 5.4, 5.5, 5.6, 5.7, 5.8, 5.9, 6.0, 6.1, 6.2, 6.3, 6.4, 6.5, 6.6, 6.7, 6.8, 6.9, 7.0, 7.1, 7.2, 7.3, 7.4, 7.5, 7.6, 7.7, 7.8, 7.9, 8.0, 또는 8.1), 약 2.5 내지 약 7, 약 3 내지 약 5, 약 2.5 내지 약 4.5 (예를 들어, 약 2.5, 약 2.6, 약 2.7, 약 2.8, 약 2.9, 약 3.0, 약 3.1, 약 3.3, 약 3.4, 약 3.5, 약 3.6, 약 3.7, 약 3.8, 약 3.9, 약 4.0, 약 4.1, 약 4.2, 약 4.3, 약 4.4, 약 4.5), 약 3.0 내지 약 4.0, 약 3.2 내지 약 4.2, 또는 약 4.5 내지 5.5 (예를 들어, 약 4.5, 약 4.6, 약 4.7, 약 4.8, 약 4.9, 약 5.0, 약 5.1, 약 5.2, 약 5.3, 약 5.4, 또는 약 5.5)이다.
본 개시내용에 의해 제공된 한 측면에서, 접합체는 실질적으로 높은 순도를 갖고, 하기 특색 중 하나 이상을 갖는다: (a) 접합체 종의 약 90% 초과 (예를 들어, 약 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 이상), 바람직하게는 약 95% 초과가 단량체임, (b) 접합체 제제 내 비접합 링커 수준이 약 10% 미만 (예를 들어, 약 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2%, 1% 또는 0% 이하)임 (총 링커에 비해), (c) 접합체 종의 10% 미만이 가교됨 (예를 들어, 약 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2%, 1% 또는 0% 이하), (d) 접합체 제제 내 유리 약물 모이어티 (예를 들어, DM1 또는 DM4) 수준이 약 2% 미만 (예를 들어, 약 1.5%, 1.4%, 1.3%, 1.2%, 1.1%, 1.0%, 0.9%, 0.8%, 0.7%, 0.6%, 0.5%, 0.4%, 0.3%, 0.2%, 0.1% 또는 0% 이하)임 (총 세포독성제에 비해 mol/mol).
본원에 사용된 용어 "비접합 링커"는 항체가 링커를 통해 약물 모이어티 (예를 들어, DM1 또는 DM4)에 공유 커플링되지 않은, 가교 시약 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)으로부터 유래된 링커와 공유 연결된 항체를 지칭한다 (즉, "비접합 링커"는 Ab-SMCC, Ab-SPDB, Ab-술포-SPDB 또는 Ab-CX1-1로 나타내어질 수 있음).
1. 약물 모이어티
본 개시내용은 CDH6에 특이적으로 결합하는 면역접합체를 제공한다. 본 개시내용의 면역접합체는 약물 모이어티, 예를 들어 항암제, 항-혈액 장애 작용제, 자가면역 치료제, 항염증제, 항진균제, 항박테리아제, 항기생충제, 항바이러스제 또는 마취제에 접합된 항-CDH6 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물을 포함한다. 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 관련 기술분야에 공지된 임의의 방법을 사용하여 동일하거나 상이한 여러 약물 모이어티에 접합될 수 있다.
특정 측면에서, 본 개시내용의 면역접합체의 약물 모이어티는 다음으로 이루어진 군으로부터 선택된다: 메이탄시노이드, V-ATPase 억제제, 아폽토시스촉진제, Bcl2 억제제, MCL1 억제제, HSP90 억제제, IAP 억제제, mTor 억제제, 미세관 안정화제, 미세관 탈안정화제, 아우리스타틴, 돌라스타틴, MetAP (메티오닌 아미노펩티다제), 단백질 CRM1의 핵 유출의 억제제, DPPIV 억제제, 프로테아솜 억제제, 미토콘드리아에서의 포스포릴 전달 반응의 억제제, 단백질 합성 억제제, 키나제 억제제, CDK2 억제제, CDK9 억제제, 키네신 억제제, HDAC 억제제, DNA 손상 작용제, DNA 알킬화제, DNA 삽입제, DNA 작은 홈 결합제 및 DHFR 억제제.
한 측면에서, 본 개시내용의 면역접합체의 약물 모이어티는 DM1, DM3 또는 DM4와 같은, 그러나 이에 제한되지 않는 메이탄시노이드 약물 모이어티이다.
추가로, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 소정의 생물학적 반응을 변형시키는 약물 모이어티에 접합될 수 있다. 약물 모이어티는 전형적 화학 치료제로 제한되는 것으로 해석되지 않아야 한다. 예를 들어, 약물 모이어티는 목적하는 생물학적 활성을 보유하는 단백질, 펩티드 또는 폴리펩티드일 수 있다. 이러한 단백질은, 예를 들어 독소, 예컨대 아브린, 리신 A, 슈도모나스 외독소, 콜레라 독소, 또는 디프테리아 독소, 단백질, 예컨대 종양 괴사 인자, α-인터페론, β-인터페론, 신경 성장 인자, 혈소판 유래 성장 인자, 조직 플라스미노겐 활성화제, 시토카인, 아폽토시스제, 항혈관신생제, 또는 생물학적 반응 조절제, 예컨대, 예를 들어 림포카인을 포함할 수 있다.
한 측면에서, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 세포독소, 약물 (예를 들어, 면역억제제) 또는 방사성독소와 같은 약물 모이어티에 접합된다. 세포독소의 예는 탁산 (예를 들어, 국제 (PCT) 특허 출원 번호 WO 01/38318 및 PCT/US03/02675 참조), DNA-알킬화제 (예를 들어, CC-1065 유사체), 안트라시클린, 튜부리신 유사체, 두오카르마이신 유사체, 아우리스타틴 E, 아우리스타틴 F, 메이탄시노이드, 및 반응성 폴리에틸렌 글리콜 모이어티를 포함하는 세포독성제 (예를 들어, 문헌 [Sasse et al., J. Antibiot. (Tokyo), 53, 879-85 (2000), Suzawa et al., Bioorg. Med. Chem., 8, 2175-84 (2000), Ichimura et al., J. Antibiot. (Tokyo), 44, 1045-53 (1991), Francisco et al., Blood 2003 15;102(4):1458-65], 미국 특허 번호 5,475,092, 6,340,701, 6,372,738, 및 6,436,931, 미국 특허 출원 공개 번호 2001/0036923 A1, 펜딩 미국 특허 출원 일련 번호 10/024,290 및 10/116,053, 및 국제 (PCT) 특허 출원 번호 WO 01/49698 참조), 탁손, 시토칼라신 B, 그라미시딘 D, 브로민화에티듐, 에메틴, 미토마이신, 에토포시드, 테노포시드, 빈크리스틴, 빈블라스틴, 콜키신, 독소루비신, 다우노루비신, 디히드록시 안트라신 디온, 미톡산트론, 미트라마이신, 악티노마이신 D, 1-데히드로테스토스테론, 글루코코르티코이드, 프로카인, 테트라카인, 리도카인, 프로프라놀롤, 및 퓨로마이신 및 그의 유사체 또는 상동체를 포함하나 이에 제한되지는 않는다. 치료제는 또한, 예를 들어 항대사물 (예를 들어, 메토트렉세이트, 6-메르캅토퓨린, 6-티오구아닌, 시타라빈, 5-플루오로우라실 데카르바진), 제거제 (예를 들어, 메클로레타민, 티오테파 클로람부실, 멜팔란, 카르무스틴 (BSNU) 및 로무스틴 (CCNU), 시클로포스파미드, 부술판, 디브로모만니톨, 스트렙토조토신, 미토마이신 C, 및 시스-디클로로디아민 백금 (II) (DDP) 시스플라틴, 안트라시클린 (예를 들어, 다우노루비신 (이전에 다우노마이신) 및 독소루비신), 항생제 (예를 들어, 닥티노마이신 (이전에 악티노마이신), 블레오마이신, 미트라마이신, 및 안트라마이신 (AMC)), 및 항유사분열제 (예를 들어, 빈크리스틴 및 빈블라스틴)을 포함한다. (예를 들어, 시애틀 제네틱스(Seattle Genetics) US20090304721 참조).
본 개시내용의 항체, 항체 단편 (항원 결합 단편) 또는 기능적 등가물에 접합될 수 있는 세포독소의 다른 예는 두오카르마이신, 칼리케아미신, 메이탄신 및 아우리스타틴, 및 그의 유도체를 포함한다.
다양한 유형의 세포독소, 링커, 및 치료제를 항체에 접합시키는 방법이 관련 기술분야에 공지되어 있고, 예를 들어 문헌 [Saito et al., (2003) Adv. Drug Deliv. Rev. 55:199-215; Trail et al., (2003) Cancer Immunol. Immunother. 52:328-337; Payne, (2003) Cancer Cell 3:207-212; Allen, (2002) Nat. Rev. Cancer 2:750-763; Pastan and Kreitman, (2002) Curr. Opin. Investig. Drugs 3:1089-1091; Senter and Springer, (2001) Adv. Drug Deliv. Rev. 53:247-264]을 참조한다.
본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 또한 방사성면역접합체로 지칭되는 세포독성 방사성제약이 생성되도록 방사성 동위원소에 접합될 수도 있다. 진단적으로 또는 치료적으로 사용하기 위해 항체에 접합될 수 있는 방사성 동위원소의 예는 아이오딘-131, 인듐-111, 이트륨-90 및 루테튬-177을 포함하나 이에 제한되지는 않는다. 방사성면역접합체를 제조하는 방법은 관련 기술분야에 확립되어 있다. 제발린(Zevalin)™ (아이덱 파마슈티칼스(IDEC Pharmaceuticals)) 및 벡사르(Bexxar)™ (코릭사 파마슈티칼스(Corixa Pharmaceuticals))를 포함한 방사성면역접합체의 예가 상업적으로 입수가능하고, 유사한 방법이 본원에 개시된 항체를 사용하여 방사성면역접합체를 제조하는데 사용될 수 있다. 특정 측면에서, 마크로시클릭 킬레이트화제는 1,4,7,10-테트라아자시클로도데칸-N,N',N",N"'-테트라아세트산 (DOTA)이며, 이는 링커 분자를 통해 항체에 부착될 수 있다. 이러한 링커 분자는 관련 기술분야에 통상적으로 공지되어 있고, 문헌 [Denardo et al., (1998) Clin Cancer Res. 4(10):2483-90; Peterson et al., (1999) Bioconjug. Chem. 10(4):553-7; 및 Zimmerman et al., (1999) Nucl. Med. Biol. 26(8):943-50]에 기재되어 있으며, 이들 각각은 그 전문이 참조로 포함된다.
본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 또한 이종 단백질 또는 폴리펩티드 (또는 그의 단편, 바람직하게는 적어도 10, 적어도 20, 적어도 30, 적어도 40, 적어도 50, 적어도 60, 적어도 70, 적어도 80, 적어도 90 또는 적어도 100개 아미노산의 폴리펩티드)에 접합되어 융합 단백질이 생성될 수도 있다. 특히, 본 개시내용은 본원에 기재된 항체 단편 (예를 들어, 항원 결합 단편) (예를 들어, Fab 단편, Fd 단편, Fv 단편, F(ab)2 단편, VH 도메인, VH CDR, VL 도메인 또는 VL CDR) 및 이종 단백질, 폴리펩티드 또는 펩티드를 포함하는 융합 단백질을 제공한다.
추가의 융합 단백질은 유전자-셔플링, 모티프-셔플링, 엑손-셔플링 및/또는 코돈-셔플링 (집합적으로 "DNA 셔플링"으로 지칭됨)의 기술을 통해 생성될 수 있다. DNA 셔플링은 본 개시내용의 항체 또는 그의 단편의 활성을 변경시키는데 사용될 수 있다 (예를 들어, 보다 높은 친화도 및 보다 낮은 해리율을 갖는 항체 또는 그의 단편). 일반적으로, 미국 특허 번호 5,605,793, 5,811,238, 5,830,721, 5,834,252 및 5,837,458; 문헌 [Patten et al., (1997) Curr. Opinion Biotechnol. 8:724-33; Harayama, (1998) Trends Biotechnol. 16(2):76-82; Hansson et al., (1999) J. Mol. Biol. 287:265-76; 및 Lorenzo and Blasco, (1998) Biotechniques 24(2):308- 313]을 참조한다 (각각의 이들 특허 및 공보는 그 전문이 본원에 참조로 포함됨). 항체 또는 그의 단편, 또는 코딩된 항체 또는 그의 단편은 재조합 전에 오류-유발 PCR, 무작위 뉴클레오티드 삽입 또는 다른 방법에 의한 무작위 돌연변이유발에 적용시킴으로써 변경될 수 있다. 항원에 특이적으로 결합하는 항체 또는 그의 단편을 코딩하는 폴리뉴클레오티드는 1종 이상의 이종 분자의 1종 이상의 성분, 모티프, 절편, 부분, 도메인, 단편 등과 재조합될 수 있다.
또한, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 마커 서열, 예컨대 펩티드에 접합되어 정제를 용이하게 할 수 있다. 바람직한 측면에서, 마커 아미노산 서열은 헥사-히스티딘 펩티드, 예컨대 특히 pQE 벡터 (퀴아젠(QIAGEN), 캘리포니아주 채츠워스)에 제공된 태그이며, 이들 중 다수는 상업적으로 입수가능하다. 문헌 [Gentz et al., (1989) Proc. Natl. Acad. Sci. USA 86:821-824]에 기재된 바와 같이, 예를 들어 헥사-히스티딘은 융합 단백질의 편리한 정제를 제공한다. 정제에 유용한 다른 펩티드 태그는 인플루엔자 헤마글루티닌 단백질로부터 유래된 에피토프에 상응하는 헤마글루티닌 ("HA") 태그 (Wilson et al., (1984) Cell 37:767), 및 "FLAG" 태그 (A. Einhauer et al., J. Biochem. Biophys. Methods 49: 455-465, 2001)를 포함하나 이에 제한되지는 않는다. 본 개시내용에 기재된 바와 같이, 항체 또는 항원 결합 단편은 또한 종양-투과 펩티드에, 그의 효능을 강화시키기 위해 접합될 수도 있다.
다른 측면에서, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물은 진단제 또는 검출가능한 작용제에 접합된다. 이러한 면역접합체는 특정한 요법의 효능을 결정하는 것과 같은 임상 시험 절차의 일부로서 질환 또는 장애의 발병, 발달, 진행 및/또는 중증도를 모니터링하거나 예후하는데 유용할 수 있다. 이러한 진단 및 검출은 양고추냉이 퍼옥시다제, 알칼리성 포스파타제, 베타-갈락토시다제 또는 아세틸콜린에스테라제와 같은, 그러나 이에 제한되지 않는 다양한 효소; 스트렙타비딘/비오틴 및 아비딘/비오틴과 같은, 그러나 이에 제한되지 않는 보결분자단; 알렉사 플루오르(Alexa Fluor) 350, 알렉사 플루오르 405, 알렉사 플루오르 430, 알렉사 플루오르 488, 알렉사 플루오르 500, 알렉사 플루오르 514, 알렉사 플루오르 532, 알렉사 플루오르 546, 알렉사 플루오르 555, 알렉사 플루오르 568, 알렉사 플루오르 594, 알렉사 플루오르 610, 알렉사 플루오르 633, 알렉사 플루오르 647, 알렉사 플루오르 660, 알렉사 플루오르 680, 알렉사 플루오르 700, 알렉사 플루오르 750, 움벨리페론, 플루오레세인, 플루오레세인 이소티오시아네이트, 로다민, 디클로로트리아지닐아민 플루오레세인, 단실 클로라이드 또는 피코에리트린과 같은, 그러나 이에 제한되지 않는 형광 물질; 루미놀과 같은, 그러나 이에 제한되지 않는 발광 물질; 루시페라제, 루시페린 및 에쿼린과 같은, 그러나 이에 제한되지 않는 생물발광 물질; 아이오딘 (131I, 125I, 123I 및 121I), 탄소 (14C), 황 (35S), 삼중수소 (3H), 인듐 (115In, 113In, 112In 및 111In), 테크네튬 (99Tc), 탈륨 (201Ti), 갈륨 (68Ga, 67Ga), 팔라듐 (103Pd), 몰리브데넘 (99Mo), 크세논 (133Xe), 플루오린 (18F), 153Sm, 177Lu, 159Gd, 149Pm, 140La, 175Yb, 166Ho, 90Y, 47Sc, 186Re, 188Re, 142 Pr, 105Rh, 97Ru, 68Ge, 57Co, 65Zn, 85Sr, 32P, 153Gd, 169Yb, 51Cr, 54Mn, 75Se, 64Cu, 113Sn 및 117Sn과 같은, 그러나 이에 제한되지 않는 방사성 물질; 및 다양한 양전자 방출 단층촬영을 사용하는 양전자 방출 금속, 및 비-방사성 상자성 금속 이온을 포함하나 이에 제한되지 않는 검출가능한 물질에 항체를 커플링시킴으로써 달성될 수 있다.
본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물이 고체 지지체에 부착될 수도 있고, 이는 표적 항원의 정제 또는 면역검정에 특히 유용하다. 이러한 고체 지지체는 유리, 셀룰로스, 폴리아크릴아미드, 나일론, 폴리스티렌, 폴리비닐 클로라이드 또는 폴리프로필렌을 포함하나 이에 제한되지는 않는다.
2. 링커
본원에 사용된 "링커"는 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 기능적 등가물을 또 다른 모이어티, 예컨대 약물 모이어티에 연결시킬 수 있는 임의의 화학적 모이어티이다. 링커는 화합물 또는 항체가 활성으로 남아있는 조건에서, 절단, 예컨대 산-유도된 절단, 광-유도된 절단, 펩티다제-유도된 절단, 에스테라제-유도된 절단 및 디술피드 결합 절단에 대해 링커가 감수성일 수 있다 (절단가능한 링커). 대안적으로, 링커는 절단에 대해 실질적으로 저항성일 수 있다 (예를 들어, 안정한 링커 또는 비-절단가능한 링커). 일부 측면에서, 링커는 사전하전된 링커, 친수성 링커 또는 디카르복실산-기반 링커이다.
한 측면에서, 사용되는 링커는 가교 시약, 예컨대 N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP), N-숙신이미딜 4-(2-피리딜디티오)펜타노에이트 (SPP), N-숙신이미딜 4-(2-피리딜디티오)부타노에이트 (SPDB), N-숙신이미딜-4-(2-피리딜디티오)-2-술포-부타노에이트 (술포-SPDB), N-숙신이미딜 아이오도아세테이트 (SIA), N-숙신이미딜(4-아이오도아세틸)아미노벤조에이트 (SIAB), 말레이미드 PEG NHS, N-숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (SMCC), N-술포숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (술포-SMCC) 또는 2,5-디옥소피롤리딘-1-일 17-(2,5-디옥소-2,5-디히드로-1H-피롤-1-일)-5,8,11,14-테트라옥소-4,7,10,13-테트라아자헵타데칸-1-오에이트 (CX1-1)로부터 유래된다. 또 다른 측면에서, 사용되는 링커는 가교제, 예컨대 N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP), N-숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (SMCC), N-술포숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (술포-SMCC), N-숙신이미딜-4-(2-피리딜디티오)-2-술포-부타노에이트 (술포-SPDB) 또는 2,5-디옥소피롤리딘-1-일 17-(2,5-디옥소-2,5-디히드로-1H-피롤-1-일)-5,8,11,14-테트라옥소-4,7,10,13-테트라아자헵타데칸-1-오에이트 (CX1-1)로부터 유래된다.
비-절단가능한 링커는 안정한 공유결합 방식으로 약물 모이어티, 예컨대 메이탄시노이드를 항체에 연결시킬 수 있는 임의의 화학적 모이어티이고, 절단가능한 링커에 대해 상기 열거된 카테고리 하에 속하지 않는다. 따라서, 비-절단가능한 링커는 산-유도된 절단, 광-유도된 절단, 펩티다제-유도된 절단, 에스테라제-유도된 절단 및 디술피드 결합 절단에 대해 실질적으로 저항성이다. 또한, 비-절단가능한이란 약물 모이어티, 예컨대 메이탄시노이드 또는 항체가 그의 활성을 상실하지 않는 조건에서, 산, 광불안정성 절단제, 펩티다제, 에스테라제, 또는 디술피드 결합을 절단하는 화학적 또는 생리학적 화합물에 의해 유도되는 절단을 견디는, 링커 내이거나 링커에 인접한 화학 결합의 능력을 지칭한다.
산-불안정성 링커는 산성 pH에서 절단가능한 링커이다. 예를 들어, 특정 세포내 구획, 예컨대 엔도솜 및 리소좀은 산성 pH (pH 4-5)이고, 산-불안정성 링커를 절단하는데 적합한 조건을 제공한다.
광-불안정성 링커는 신체 표면 및 광에 접근가능한 많은 체강에서 유용한 링커이다. 또한, 적외선이 조직을 침투할 수 있다.
일부 링커는 펩티다제에 의해 절단될 수 있고, 즉 펩티다제 절단가능한 링커일 수 있다. 단지 특정 펩티드만이 세포 내부 또는 외부에서 용이하게 절단되며, 예를 들어 문헌 [Trout et al., 79 Proc. Natl. Acad.Sci. USA, 626-629 (1982) 및 Umemoto et al. 43 Int. J. Cancer, 677-684 (1989)]을 참조한다. 또한, 펩티드는 α-아미노산, 및 화학적으로 한 아미노산의 카르복실레이트와 제2 아미노산의 아미노 기 사이의 아미드 결합인 펩티드 결합으로 구성된다. 다른 아미드 결합, 예컨대 카르복실레이트와 리신의 ε-아미노기 사이의 결합은 펩티드 결합이 아닌 것으로 이해되고, 비-절단가능한 것으로 간주된다.
일부 링커는 에스테라제에 의해 절단될 수 있고, 즉 에스테라제 절단가능한 링커일 수 있다. 다시, 단지 특정 에스테르만이 세포 내부 또는 외부에 존재하는 에스테라제에 의해 절단될 수 있다. 에스테르는 카르복실산과 알콜의 축합에 의해 형성된다. 단순 에스테르는 단순 알콜, 예컨대 지방족 알콜, 및 소형 시클릭 및 소형 방향족 알콜로 생산된 에스테르이다.
사전하전된 링커는 항체 약물 접합체 내로의 혼입 후에 자신의 전하를 유지하는 하전된 가교 시약으로부터 유래된다. 사전하전된 링커의 예은 US 2009/0274713에서 찾을 수 있다.
3. 접합 및 ADC 제조
본 개시내용의 접합체는 관련 기술분야에 공지된 임의의 방법, 예컨대 미국 특허 번호 7,811,572, 6,411,163, 7,368,565, 및 8,163,888, 및 미국 출원 공개 2011/0003969, 2011/0166319, 2012/0253021 및 2012/0259100에 기재된 방법에 의해 제조될 수 있다. 이들 특허 및 특허 출원 공개의 전체 교시는 본원에 참조로 포함된다.
1-단계 공정
한 측면에서, 본 개시내용의 접합체는 1-단계 공정에 의해 제조될 수 있다. 공정은 적합한 pH에서 임의로 1종 이상의 공-용매를 함유하는 실질적으로 수성 매질 중에서 항체, 약물 및 가교제를 합하는 것을 포함한다. 한 측면에서, 공정은 본 개시내용의 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시켜 항체 및 약물을 포함하는 제1 혼합물을 형성시키고, 이어서 pH가 약 4 내지 약 9인 용액 중에서 항체 및 약물을 포함하는 제1 혼합물을 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시켜 (i) 접합체 (예를 들어, Ab-MCC-DM1, Ab-SPDB-DM4, 술포-SPDB-DM4, 또는 Ab-CX1-1-DM1), (ii) 유리 약물 (예를 들어, DM1 또는 DM4), 및 (iii) 반응 부산물을 포함하는 혼합물을 제공하는 단계를 포함한다.
한 측면에서, 1-단계 공정은 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 pH가 약 6 이상 (예를 들어, 약 6 내지 약 9, 약 6 내지 약 7, 약 7 내지 약 9, 약 7 내지 약 8.5, 약 7.5 내지 약 8.5, 약 7.5 내지 약 8.0, 약 8.0 내지 약 9.0, 또는 약 8.5 내지 약 9.0)인 용액 중에서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함한다. 예를 들어, 공정은 세포 결합제를 약물 (DM1 또는 DM4)과 접촉시키고, 이어서 pH가 약 6.0, 약 6.1, 약 6.2, 약 6.3, 약 6.4, 약 6.5, 약 6.6, 약 6.7, 약 6.8, 약 6.9, 약 7.0, 약 7.1, 약 7.2, 약 7.3, 약 7.4, 약 7.5, 약 7.6, 약 7.7, 약 7.8, 약 7.9, 약 8.0, 약 8.1, 약 8.2, 약 8.3, 약 8.4, 약 8.5, 약 8.6, 약 8.7, 약 8.8, 약 8.9, 또는 약 9.0인 용액 중에서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함한다. 또 다른 측면에서, 공정은 세포 결합제를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 pH가 약 7.8인 (예를 들어, pH가 7.6 내지 8.0이거나 또는 pH가 7.7 내지 7.9인) 용액 중에서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함한다.
1-단계 공정 (즉, 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것)은 관련 기술분야에 공지된 임의의 적합한 온도에서 수행될 수 있다. 예를 들어, 약 20℃ 이하 (예를 들어, 약 -10℃ (단 용액은, 예를 들어 세포독성제 및 이관능성 가교 시약을 용해시키도록 사용된 유기 용매의 존재에 의해 동결되는 것이 방지됨) 내지 약 20℃, 약 0℃ 내지 약 18℃, 약 4℃ 내지 약 16℃)에서, 실온 (예를 들어, 약 20℃ 내지 약 30℃ 또는 약 20℃ 내지 약 25℃)에서, 또는 승온 (예를 들어, 약 30℃ 내지 약 37℃)에서 1-단계 공정이 이루어질 수 있다. 한 측면에서, 1-단계 공정은 약 16℃ 내지 약 24℃ (예를 들어, 약 16℃, 약 17℃, 약 18℃, 약 19℃, 약 20℃, 약 21℃, 약 22℃, 약 23℃, 약 24℃, 또는 약 25℃)의 온도에서 이루어진다. 또 다른 측면에서, 1-단계 공정은 약 15℃ 이하 (예를 들어, 약 -10℃ 내지 약 15℃, 또는 약 0℃ 내지 약 15℃)의 온도에서 수행된다. 예를 들어, 공정은 약 15℃, 약 14℃, 약 13℃, 약 12℃, 약 11℃, 약 10℃, 약 9℃, 약 8℃, 약 7℃, 약 6℃, 약 5℃, 약 4℃, 약 3℃, 약 2℃, 약 1℃, 약 0℃, 약 -1℃, 약 -2℃, 약 -3℃, 약 -4℃, 약 -5℃, 약 -6℃, 약 -7℃, 약 -8℃, 약 -9℃, 또는 약 -10℃의 온도에서 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함하며, 단 용액은, 예를 들어 가교제 (예를 들어, SMCC, 술포-SMCC, 술포-SPDB SPDB, 또는 CX1-1)를 용해시키도록 사용된 유기 용매(들)의 존재에 의해 동결되는 것이 방지된다. 한 측면에서, 공정은 약 -10℃ 내지 약 15℃, 약 0℃ 내지 약 15℃, 약 0℃ 내지 약 10℃, 약 0℃ 내지 약 5℃, 약 5℃ 내지 약 15℃, 약 10℃ 내지 약 15℃, 또는 약 5℃ 내지 약 10℃의 온도에서 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함한다. 또 다른 측면에서, 공정은 약 10℃의 온도 (예를 들어, 8℃ 내지 12℃의 온도 또는 9℃ 내지 11℃의 온도)에서 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것을 포함한다.
한 측면에서, 상기 기재된 접촉은 항체를 제공하고, 이어서 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시켜 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 제1 혼합물을 형성시키고, 이어서 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 제1 혼합물을 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것에 의해 실시된다. 예를 들어, 한 측면에서, 항체가 반응 용기 내에 제공되고, 약물 (예를 들어, DM1 또는 DM4)이 반응 용기에 첨가되고 (이에 의해 항체와 접촉함), 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)가 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물에 첨가된다 (이에 의해 항체 및 약물을 포함하는 혼합물과 접촉함). 한 측면에서, 항체가 반응 용기 내에 제공되고, 항체를 용기에 제공한 직후에 약물 (예를 들어, DM1 또는 DM4)이 반응 용기에 첨가된다. 또 다른 측면에서, 항체가 반응 용기 내에 제공되고, 항체를 용기에 제공한 후의 시간 간격 (예를 들어, 세포 결합체를 공간에 제공한 후 약 5분, 약 10분, 약 20분, 약 30분, 약 40분, 약 50분, 약 1시간, 약 1일, 또는 더 긴 기간) 이후에 약물 (예를 들어, DM1 또는 DM4)이 반응 용기에 첨가된다. 약물 (예를 들어, DM1 또는 DM4)이 빠르게 (즉, 짧은 시간 간격, 예컨대 약 5분, 약 10분 이내에) 또는 느리게 (예컨대 펌프를 사용함으로써) 첨가될 수 있다.
이어서, 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시킨 직후에 또는 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고 나서 약간의 나중 시점 (예를 들어, 약 5분 내지 약 8시간 또는 더 긴 시점)에 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물이 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉될 수 있다. 예를 들어, 한 측면에서, 약물 (예를 들어, DM1 또는 DM4)을 항체를 포함하는 반응 용기에 첨가한 직후에 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)가 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물에 첨가된다. 대안적으로, 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고 나서 약 5분, 약 10분, 약 20분, 약 30분, 약 1시간, 약 2시간, 약 3시간, 약 4시간, 약 5시간, 약 6시간, 약 7시간, 약 8시간, 또는 더 긴 기간 후에 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물이 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉될 수 있다.
항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물이 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉된 후, 반응이 약 1시간, 약 2시간, 약 3시간, 약 4시간, 약 5시간, 약 6시간, 약 7시간, 약 8시간, 약 9시간, 약 10시간, 약 11시간, 약 12시간, 약 13시간, 약 14시간, 약 15시간, 약 16시간, 약 17시간, 약 18시간, 약 19시간, 약 20시간, 약 21시간, 약 22시간, 약 23시간, 약 24시간, 또는 더 긴 기간 (예를 들어, 약 30시간, 약 35시간, 약 40시간, 약 45시간, 또는 약 48시간) 동안 진행되도록 한다.
한 측면에서, 1-단계 공정은 임의의 미반응 약물 (예를 들어, DM1 또는 DM4) 및/또는 미반응 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)를 켄칭시키는 켄칭 단계를 추가로 포함한다. 켄칭 단계는 전형적으로 접합체의 정제 전에 수행된다. 한 측면에서, 혼합물은 혼합물을 켄칭 시약과 접촉시킴으로써 켄칭된다. 본원에 사용된 "켄칭 시약"은 유리 약물 (예를 들어, DM1 또는 DM4) 및/또는 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 반응하는 시약을 지칭한다. 한 측면에서, 말레이미드 또는 할로아세트아미드 켄칭 시약, 예컨대 4-말레이미도부티르산, 3-말레이미도프로피온산, N-에틸말레이미드, 아이오도아세트아미드, 또는 아이오도아세트아미도프로피온산이 약물 (예를 들어, DM1 또는 DM4) 내의 임의의 미반응 기 (예컨대 티올)가 켄칭되는 것을 보장하도록 사용될 수 있다. 켄칭 단계는 약물 (예를 들어, DM1)의 이량체화를 방지하는 것을 도울 수 있다. 이량체화된 DM1은 제거하기가 어려울 수 있다. 극성의 하전된 티올-켄칭 시약 (예컨대 4-말레이미도부티르산 또는 3-말레이미도프로피온산)으로의 켄칭 시에, 과량의 미반응 DM1이 극성의 하전된 수용성 부가물로 전환되고, 이는 정제 단계 동안 공유 연결된 접합체로부터 용이하게 분리될 수 있다. 비극성 및 중성 티올-켄칭 시약으로의 켄칭이 또한 사용될 수 있다. 한 측면에서, 혼합물은 혼합물을 미반응 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 반응하는 켄칭 시약과 접촉시킴으로써 켄칭된다. 예를 들어, 임의의 미반응 SMCC를 켄칭시키기 위해 친핵체가 혼합물에 첨가될 수 있다. 바람직하게는 친핵체는 아미노 기를 함유하는 친핵체, 예컨대 리신, 타우린 및 히드록실아민이다.
또 다른 측면에서, 혼합물이 켄칭 시약과 접촉되기 전에 반응 (즉, 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시키고, 이어서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시키는 것)이 완료까지 진행되도록 한다. 이와 관련하여, 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물이 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉되고 나서 약 1시간 내지 약 48시간 (예를 들어, 약 1시간, 약 2시간, 약 3시간, 약 4시간, 약 5시간, 약 6시간, 약 7시간, 약 8시간, 약 9시간, 약 10시간, 약 11시간, 약 12시간, 약 13시간, 약 14시간, 약 15시간, 약 16시간, 약 17시간, 약 18시간, 약 19시간, 약 20시간, 약 21시간, 약 22시간, 약 23시간, 약 24시간, 또는 약 25시간 내지 약 48시간) 후에 켄칭 시약이 혼합물에 첨가된다.
대안적으로, 혼합물은 혼합물의 pH를 약 5.0 (예를 들어, 4.8, 4.9, 5.0, 5.1 또는 5.2)으로 저하시킴으로써 켄칭된다. 또 다른 측면에서, 혼합물은 pH를 6.0 미만, 5.5 미만, 5.0 미만, 4.8 미만, 4.6 미만, 4.4 미만, 4.2 미만, 4.0 미만으로 저하시킴으로써 켄칭된다. 대안적으로, pH는 약 4.0 (예를 들어, 3.8, 3.9, 4.0, 4.1 또는 4.2) 내지 약 6.0 (예를 들어, 5.8, 5.9, 6.0, 6.1 또는 6.2), 약 4.0 내지 약 5.0, 약 4.5 (예를 들어, 4.3, 4.4, 4.5, 4.6 또는 4.7) 내지 약 5.0으로 저하된다. 한 측면에서, 혼합물은 혼합물의 pH를 4.8로 저하시킴으로써 켄칭된다. 또 다른 측면에서, 혼합물은 혼합물의 pH를 5.5로 저하시킴으로써 켄칭된다.
한 측면에서, 1-단계 공정은 불안정하게 결합된 링커를 항체로부터 방출시키는 유지 단계를 추가로 포함한다. 유지 단계는 접합체의 정제 전에 (예를 들어, 반응 단계 후에, 반응 단계와 켄칭 단계 사이에, 또는 켄칭 단계 후에) 혼합물을 유지시키는 것을 포함한다. 예를 들어, 공정은 (a) 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시켜 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물을 형성시키고, 이어서 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물을 pH가 약 4 내지 약 9인 용액 중에서 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시켜 (i) 접합체 (예를 들어, Ab-MCC-DM1, Ab-SPDB-DM4, 술포-SPDB-DM4 또는 Ab-CX1-1-DM1), (ii) 유리 약물 (예를 들어, DM1 또는 DM4), 및 (iii) 반응 부산물을 포함하는 혼합물을 제공하는 단계, (b) 단계 (a)에서 제조된 혼합물을 유지시켜, 불안정하게 결합된 링커를 세포 결합제로부터 방출시키는 단계, 및 (c) 혼합물을 정제하여 정제된 접합체를 제공하는 단계를 포함한다.
또 다른 측면에서, 공정은 (a) 항체를 약물 (예를 들어, DM1 또는 DM4)과 접촉시켜 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물을 형성하고, 이어서 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물을 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 pH가 약 4 내지 약 9인 용액 중에서 접촉시켜 (i) 접합체, (ii) 유리 약물 (예를 들어, DM1 또는 DM4), 및 (iii) 반응 부산물을 포함하는 혼합물을 제공하는 단계, (b) 단계 (a)에서 제조된 혼합물을 켄칭시켜 임의의 미반응 약물 (예를 들어, DM1 또는 DM4) 및/또는 미반응 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)를 켄칭시키는 단계, (c) 단계 (b)에서 제조된 혼합물을 유지시켜, 불안정하게 결합된 링커를 세포-결합제로부터 방출시키는 단계, 및 (d) 혼합물을 정제하여 정제된 접합체 (예를 들어, Ab-MCC-DM1, Ab-SPDB-DM4, Ab-술포-SPDB-DM4 또는 Ab-CX1-1-DM1)를 제공하는 단계를 포함한다.
대안적으로, 유지 단계는 접합체의 정제 후에 수행될 수 있고, 추가의 정제 단계가 이어질 수 있다.
또 다른 측면에서, 유지 단계 전에 반응이 완료까지 진행되도록 한다. 이와 관련하여, 항체 및 약물 (예를 들어, DM1 또는 DM4)을 포함하는 혼합물이 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉되고 나서 약 1시간 내지 약 48시간 (예를 들어, 약 1시간, 약 2시간, 약 3시간, 약 4시간, 약 5시간, 약 6시간, 약 7시간, 약 8시간, 약 9시간, 약 10시간, 약 11시간, 약 12시간, 약 13시간, 약 14시간, 약 15시간, 약 16시간, 약 17시간, 약 18시간, 약 19시간, 약 20시간, 약 21시간, 약 22시간, 약 23시간, 약 24시간, 또는 약 24시간 내지 약 48시간) 후에 유지 단계가 수행될 수 있다.
유지 단계는 용액을 적합한 온도 (예를 들어, 약 0℃ 내지 약 37℃)에서 적합한 기간 (예를 들어, 약 1시간 내지 약 1주, 약 1시간 내지 약 24시간, 약 1시간 내지 약 8시간, 또는 약 1시간 내지 약 4시간) 동안 유지시켜, 안정하게 결합된 링커를 항체로부터 실질적으로 방출시키지 않으면서 불안정하게 결합된 링커를 항체로부터 방출시키는 것을 포함한다. 한 측면에서, 유지 단계는 용액을 약 20℃ 이하 (예를 들어, 약 0℃ 내지 약 18℃, 약 4℃ 내지 약 16℃)에서, 실온 (예를 들어, 약 20℃ 내지 약 30℃ 또는 약 20℃ 내지 약 25℃)에서, 또는 승온 (예를 들어, 약 30℃ 내지 약 37℃)에서 유지시키는 것을 포함한다. 한 측면에서, 유지 단계는 용액을 약 16℃ 내지 약 24℃ (예를 들어, 약 15℃, 약 16℃, 약 17℃, 약 18℃, 약 19℃, 약 20℃, 약 21℃, 약 22℃, 약 23℃, 약 24℃, 또는 약 25℃)의 온도에서 유지시키는 것을 포함한다. 또 다른 측면에서, 유지 단계는 용액을 약 2℃ 내지 약 8℃ (예를 들어, 약 0℃, 약 1℃, 약 2℃, 약 3℃, 약 4℃, 약 5℃, 약 6℃, 약 7℃, 약 8℃, 약 9℃, 또는 약 10℃)의 온도에서 유지시키는 것을 포함한다. 또 다른 측면에서, 유지 단계는 용액을 약 37℃ (예를 들어, 약 34℃, 약 35℃, 약 36℃, 약 37℃, 약 38℃, 약 39℃, 또는 약 40℃)의 온도에서 유지시키는 것을 포함한다.
유지 단계의 지속기간은 유지 단계가 수행되는 온도 및 pH에 좌우된다. 예를 들어, 유지 단계의 지속기간은 유지 단계를 승온에서 수행함으로써 실질적으로 감소될 수 있고, 이때 세포 결합제-세포독성제 접합체의 안정성에 의해 최대 온도가 제한된다. 유지 단계는 용액을 약 1시간 내지 약 1일 (예를 들어, 약 1시간, 약 2시간, 약 3시간, 약 4시간, 약 5시간, 약 6시간, 약 7시간, 약 8시간, 약 9시간, 약 10시간, 약 12시간, 약 14시간, 약 16시간, 약 18시간, 약 20시간, 약 22시간, 또는 약 24시간), 약 10시간 내지 약 24시간, 약 12시간 내지 약 24시간, 약 14시간 내지 약 24시간, 약 16시간 내지 약 24시간, 약 18시간 내지 약 24시간, 약 20시간 내지 약 24시간, 약 5시간 내지 약 1주, 약 20시간 내지 약 1주, 약 12시간 내지 약 1주 (예를 들어, 약 12시간, 약 16시간, 약 20시간, 약 24시간, 약 2일, 약 3일, 약 4일, 약 5일, 약 6일, 또는 약 7일), 또는 약 1일 내지 약 1주 동안 유지시키는 것을 포함할 수 있다.
한 측면에서, 유지 단계는 용액을 약 2℃ 내지 약 8℃의 온도에서 적어도 약 12시간 내지 1주까지의 기간 동안 유지시키는 것을 포함한다. 또 다른 측면에서, 유지 단계는 용액을 약 2℃ 내지 약 8℃의 온도에서 밤새 (예를 들어, 약 12 내지 약 24시간, 바람직하게는 약 20시간 동안) 유지시키는 것을 포함한다.
유지 단계에 대한 pH 값은 바람직하게는 약 4 내지 약 10이다. 한 측면에서, 유지 단계에 대한 pH 값은 약 4 이상, 그러나 약 6 미만 (예를 들어, 4 내지 5.9) 또는 약 5 이상, 그러나 약 6 미만 (예를 들어, 5 내지 5.9)이다. 또 다른 측면에서, 유지 단계에 대한 pH 값은 약 6 내지 약 10 (예를 들어, 약 6.5 내지 약 9, 약 6 내지 약 8) 범위이다. 예를 들어, 유지 단계에 대한 pH 값은 약 6, 약 6.5, 약 7, 약 7.5, 약 8, 약 8.5, 약 9, 약 9.5, 또는 약 10일 수 있다.
다른 측면에서, 유지 단계는 혼합물을 25℃에서 약 6-7.5의 pH에서 약 12시간 내지 약 1주 동안 인큐베이션하는 것, 혼합물을 4℃에서 약 4.5-5.9의 pH에서 약 5시간 내지 약 5일 동안 인큐베이션하는 것, 또는 혼합물을 25℃에서 약 4.5-5.9의 pH에서 약 5시간 내지 약 1일 동안 인큐베이션하는 것을 포함할 수 있다.
1-단계 공정은 접합체의 용해도 및 회수를 증가시키기 위해 수크로스를 반응 단계에 첨가하는 것을 임의로 포함할 수 있다. 바람직하게는, 수크로스는 약 0.1% (w/v) 내지 약 20% (w/v) (예를 들어, 약 0.1% (w/v), 1% (w/v), 5% (w/v), 10% (w/v), 15% (w/v), 또는 20% (w/v))의 농도로 첨가된다. 바람직하게는, 수크로스는 약 1% (w/v) 내지 약 10% (w/v) (예를 들어, 약 0.5% (w/v), 약 1% (w/v), 약 1.5% (w/v), 약 2% (w/v), 약 3% (w/v), 약 4% (w/v), 약 5% (w/v), 약 6% (w/v), 약 7% (w/v), 약 8% (w/v), 약 9% (w/v), 약 10% (w/v), 또는 약 11% (w/v))의 농도로 첨가된다. 추가로, 반응 단계는 완충제의 첨가를 또한 포함할 수 있다. 관련 기술분야에 공지된 임의의 적합한 완충제가 사용될 수 있다. 적합한 완충제는, 예를 들어 시트레이트 완충제, 아세테이트 완충제, 숙시네이트 완충제 및 포스페이트 완충제를 포함한다. 한 측면에서, 완충제는 HEPPSO (N-(2-히드록시에틸)피페라진-N'-(2- 히드록시프로판술폰산)), POPSO (피페라진-1,4-비스-(2-히드록시-프로판-술폰산)탈수물), HEPES (4-(2-히드록시에틸)피페라진-1-에탄술폰산), HEPPS (EPPS) (4-(2-히드록시에틸)피페라진-1-프로판술폰산), TES (N-[트리스(히드록시메틸)메틸]-2-아미노에탄술폰산) 및 그의 조합으로 이루어진 군으로부터 선택된다.
1-단계 공정은 정제된 접합체 (예를 들어, Ab-MCC-DM1, Ab-SPDB-DM4, Ab-술포-SPDB-DM4 또는 Ab-CX1-1-DM1)를 제공하도록 혼합물을 정제하는 단계를 추가로 포함할 수 있다. 관련 기술분야에 공지된 임의의 정제 방법이 본 개시내용의 접합체를 정제하는데 사용될 수 있다. 한 측면에서, 본 개시내용의 접합체는 접선 유동 여과 (TFF), 비-흡착 크로마토그래피, 흡착 크로마토그래피, 흡착 여과, 선택적 침전, 또는 임의의 다른 적합한 정제 공정, 뿐만 아니라 그의 조합을 사용한다. 또 다른 측면에서, 접합체를 상기 기재된 정제 공정에 적용하기 전에, 접합체가 먼저 1종 이상의 PVDF 막을 통해 여과된다. 대안적으로, 접합체를 상기 기재된 정제 공정에 적용한 후에, 접합체가 1종 이상의 PVDF 막을 통해 여과된다. 예를 들어, 한 측면에서, 접합체가 1종 이상의 PVDF 막을 통해 여과되고, 이어서 접선 유동 여과를 사용하여 정제된다. 대안적으로, 접합체가 접선 유동 여과를 사용하여 정제되고, 이어서 1종 이상의 PVDF 막을 통해 여과된다.
펠리콘(Pellicon)® 유형 시스템 (밀리포어(Millipore), 매사추세츠주 빌러리카), 사르토콘(Sartocon)® 카세트 시스템 (사르토리우스 아게(Sartorius AG), 뉴욕주 에지우드), 및 센트라세트(Centrasette)® 유형 시스템 (폴 코포레이션(Pall Corp.), 뉴욕주 이스트 힐스)을 포함한 임의의 적합한 TFF 시스템이 정제에 이용될 수 있다.
임의의 적합한 흡착 크로마토그래피 수지가 정제에 이용될 수 있다. 바람직한 흡착 크로마토그래피 수지는 히드록시아파타이트 크로마토그래피, 소수성 전하 유도 크로마토그래피 (HCIC), 소수성 상호작용 크로마토그래피 (HIC), 이온 교환 크로마토그래피, 혼합 모드 이온 교환 크로마토그래피, 고정화된 금속 친화성 크로마토그래피 (IMAC), 염료 리간드 크로마토그래피, 친화성 크로마토그래피, 역상 크로마토그래피 및 그의 조합을 포함한다. 적합한 히드록시아파타이트 수지의 예는 세라믹 히드록시아파타이트 (CHT 유형 I 및 유형 II, 바이오-라드 래버러토리즈(Bio-Rad Laboratories) (캘리포니아주 허큘레스)), HA 울트로겔(Ultrogel)® 히드록시아파타이트 (폴 코포레이션, 뉴욕주 이스트 힐스), 및 세라믹 플루오로아파타이트 (CFT 유형 I 및 유형 II, 바이오-라드 래버러토리즈 (캘리포니아주 허큘레스))를 포함한다. 적합한 HCIC 수지의 예는 MEP 하이퍼셀(MEP Hypercel)® 수지 (폴 코포레이션, 뉴욕주 이스트 힐스)이다. 적합한 HIC 수지의 예는 부틸-세파로스, 헥실-세파로스, 페닐-세파로스 및 옥틸 세파로스 수지 (모두 지이 헬스케어(GE Healthcare), 뉴저지주 피스카타웨이), 뿐만 아니라 마크로-프렙(Macro-prep)® 메틸 및 마크로-프렙® t-부틸 수지 (바이오라드 래버러토리즈, 캘리포니아주 허큘레스)를 포함한다. 적합한 이온 교환 수지의 예는 SP-세파로스®, CM-세파로스® 및 Q-세파로스® 수지 (모두 지이 헬스케어, 뉴저지주 피스카타웨이), 및 우노스피어(Unosphere)® S 수지 (바이오-라드 래버러토리즈, 캘리포니아주 허큘레스)를 포함한다. 적합한 혼합 모드 이온 교환체의 예는 베이커본드(Bakerbond)® ABx 수지 (제이티 베이커(JT Baker), 뉴저지주 필립스버그)를 포함한다. 적합한 IMAC 수지의 예는 킬레이팅 세파로스(Chelating Sepharose)® 수지 (지이 헬스케어, 뉴저지주 피스카타웨이) 및 프로피니티(Profinity)® IMAC 수지 (바이오-라드 래버러토리즈, 캘리포니아주 허큘레스)를 포함한다. 적합한 염료 리간드 수지의 예는 블루 세파로스(Blue Sepharose) 수지 (지이 헬스케어, 뉴저지주 피스카타웨이) 및 아피-겔 블루(Affi-gel Blue) 수지 (바이오-라드 래버러토리즈, 캘리포니아주 허큘레스)를 포함한다. 적합한 친화성 수지의 예는 단백질 A 세파로스 수지 (예를 들어, 맙셀렉트(MabSelect), 지이 헬스케어, 뉴저지주 피스카타웨이) 및 렉틴 친화성 수지, 예를 들어 렌틸 렉틴 세파로스(Lentil Lectin Sepharose)® 수지 (지이 헬스케어, 뉴저지주 피스카타웨이)를 포함하며, 여기서 항체는 적절한 렉틴 결합 부위를 보유한다. 적합한 역상 수지의 예는 C4, C8 및 C18 수지 (그레이스 비닥(Grace Vydac), 캘리포니아주 헤스페리아)를 포함한다.
임의의 적합한 비-흡착 크로마토그래피 수지가 정제에 이용될 수 있다. 적합한 비-흡착 크로마토그래피 수지의 예는 세파덱스(SEPHADEX)™ G-25, G-50, G-100, 세파크릴(SEPHACRYL)™ 수지 (예를 들어, S-200 및 S-300), 슈퍼덱스(SUPERDEX)™ 수지 (예를 들어, 슈퍼덱스™ 75 및 슈퍼덱스™ 200), 바이오-겔(BIO-GEL)® 수지 (예를 들어, P-6, P-10, P-30, P-60 및 P-100), 및 관련 기술분야의 통상의 기술자에게 공지된 다른 것을 포함하나 이에 제한되지는 않는다.
2-단계 공정 및 원-포트 공정
한 측면에서, 본 개시내용의 접합체는 미국 특허 7,811,572 및 미국 특허 출원 공개 번호 2006/0182750에 기재된 바와 같이 제조될 수 있다. 공정은 (a) 본 개시내용의 항체를 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 접촉시켜 링커 (즉 Ab-SMCC, Ab-SPDB 또는 Ab-CX1-1)를 항체에 공유 부착시키고, 이에 의해 링커가 결합되어 있는 항체를 포함하는 제1 혼합물을 제조하는 단계; (b) 제1 혼합물을 정제 방법에 임의로 적용하여 링커가 결합되어 있는 항체의 정제된 제1 혼합물을 제조하는 단계; (c) 링커가 결합되어 있는 항체를 약물 (예를 들어, DM1 또는 DM4)과 약 4 내지 약 9의 pH를 갖는 용액 중에서 반응시킴으로써 약물 (예를 들어, DM1 또는 DM4)을 링커가 결합되어 있는 항체와 제1 혼합물 중에서 접합시켜 (i) 접합체 (예를 들어, Ab-MCC-DM1, Ab-SPDB-DM4, Ab-술포-SPDB-DM4 또는 Ab-CX1-1-DM1), (ii) 유리 약물 (예를 들어, DM1 또는 DM4); 및 (iii) 반응 부산물을 포함하는 제2 혼합물을 제조하는 단계; 및 (d) 제2 혼합물을 정제 공정에 적용하여 접합체를 제2 혼합물의 다른 성분으로부터 정제하는 단계를 포함한다. 대안적으로, 정제 단계 (b)가 생략될 수 있다. 본원에 기재된 임의의 정제 방법이 단계 (b) 및 (d)에 사용될 수 있다. 한 실시양태에서, TFF가 단계 (b) 및 (d) 둘 다에 사용된다. 또 다른 실시양태에서, TFF가 단계 (b)에 사용되고, 흡착 크로마토그래피 (예를 들어, CHT)가 단계 (d)에 사용된다.
1-단계 시약 및 계내 공정
한 측면에서, 본 개시내용의 접합체는 미국 특허 6,441,163 및 미국 특허 출원 공개 번호 2011/0003969 및 2008/0145374에 기재된 바와 같이 미리 형성된 약물-링커 화합물 (예를 들어, SMCC-DM1, 술포-SMCC-DM1, SPDB-DM4, 술포-SPDB-DM4 또는 CX1-1-DM1)을 본 개시내용의 항체에 접합시키고, 이어서 정제 단계를 수행함으로써 제조될 수 있다. 본원에 기재된 임의의 정제 방법이 사용될 수 있다. 약물-링커 화합물은 약물 (예를 들어, DM1 또는 DM4)을 가교제 (예를 들어, SMCC, 술포-SMCC, SPDB, 술포-SPDB 또는 CX1-1)와 반응시킴으로써 제조된다. 약물-링커 화합물 (예를 들어, SMCC-DM1, 술포-SMCC-DM1, SPDB-DM4, 술포-SPDB-DM4 또는 CX1-1-DM1)은 항체에 접합되기 전에 임의로 정제에 적용된다.
4. 바람직한 항체 및 항체 약물 접합체의 특징화 및 선택
본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체는 관련 기술분야에 공지된 다양한 검정에 의해 이들의 물리적/화학적 성질 및/또는 생물학적 활성에 대해 특징화 및 선택될 수 있다.
예를 들어, 본 개시내용의 항체는 ELISA, FACS, 비아코어(Biacore) 또는 웨스턴 블롯과 같은 공지된 방법에 의해 그의 항원 결합 활성에 대해 시험될 수 있다.
트랜스제닉 동물 및 세포주는 종양-연관 항원 및 세포 표면 수용체를 과다발현하는 암의 예방적 또는 치유적 치료로서의 잠재력을 갖는 항체 약물 접합체 (ADC)를 스크리닝하는데 특히 유용하다. 유용한 ADC에 대한 스크리닝은 트랜스제닉 동물에게 후보 ADC를 소정 범위의 용량에 걸쳐 투여하고, 평가되는 질환 또는 장애에 대한 ADC의 효과(들)에 대해 다양한 시점에서 검정하는 것을 수반할 수 있다. 대안적으로 또는 추가로, 약물은 질환 유도자 (적용가능한 경우)에의 노출 전에 또는 그와 동시에 투여될 수 있다. 후보 ADC는 연속으로 및 개별적으로, 또는 증 또는 고-처리량 스크리닝 포맷 하에 병행하여 스크리닝될 수 있다.
한 측면은 (a) CDH6을 발현하는 안정한 암 세포주 또는 인간 환자 종양으로부터의 세포 (예를 들어, 난소 세포주 또는 종양 단편, 신세포주 또는 종양 단편, 간 세포주 또는 종양 단편, 갑상선 세포주 또는 종양 단편, CNS암 세포주 또는 종양 단편, 담관암종 암 세포주 또는 종양 단편, 난소, 신장, 간, 연부 조직, CNS, 갑상선 또는 담관암종 1차 세포)를 비-인간 동물 내로 이식하는 단계; (b) ADC 약물 후보를 비-인간 동물에게 투여하는 단계; 및 (c) 이식된 세포주로부터의 종양의 성장을 억제하는 후보의 능력을 결정하는 단계를 포함하는 스크리닝 방법이다. 본 개시내용은 (a) CDH6을 발현하는 안정한 암 세포주로부터의 세포를 약물 후보와 접촉시키는 단계, 및 (b) 안정한 세포주의 성장을 억제하는 ADC 후보의 능력을 평가하는 단계를 포함하는, CDH6의 과다발현을 특징으로 하는 질환 또는 장애의 치료를 위한 ADC 후보를 스크리닝하는 방법을 또한 포괄한다.
추가의 측면은 (a) CDH6을 발현하는 안정한 암 세포주로부터의 세포를 ADC 약물 후보와 접촉시키는 단계 및 (b) 세포 사멸을 유도하는 ADC 후보의 능력을 평가하는 단계를 포함하는 스크리닝 방법이다. 한 측면에서, 아폽토시스를 유도하는 ADC 후보의 능력이 평가된다.
후보 ADC는 트랜스제닉 동물에게 소정 범위의 용량에 걸쳐 투여하고, 시간 경과에 따른 화합물에 대한 동물의 생리학적 반응을 평가함으로써 스크리닝될 수 있다. 일부 경우에, 화합물을 화합물의 효능을 증진시킬 보조인자와 함께 투여하는 것이 적절할 수 있다. 대상체 트랜스제닉 동물로부터 유래된 세포주가 CDH6 과다발현과 연관된 다양한 장애를 치료하는데 유용한 ADC를 스크리닝하는데 사용되는 경우에, 시험 ADC는 적절한 시간에 세포 배양 배지에 첨가되고, ADC에 대한 세포 반응은 적절한 생화학적 및/또는 조직학적 검정을 사용하여 시간 경과에 따라 평가된다.
따라서, 본 개시내용은 CDH6을 특이적으로 표적화하고 그에 결합하는 ADC 및 종양 세포 상에 발현된 CDH6을 확인하기 위한 검정을 제공한다.
CDH6 항체
본 개시내용은 인간 CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다. 본 개시내용의 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)은 하기 실시예에서 기재된 바와 같이 단리된 인간 모노클로날 항체 또는 그의 단편을 포함하나 이에 제한되지는 않는다.
본 개시내용은 특정 측면에서, 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이 서열식별번호: 20, 34, 48, 62, 76, 90, 104, 118, 132, 146, 160, 174, 188, 202, 216, 230, 244, 258, 272, 286, 300, 314 또는 328의 아미노산 서열을 갖는 VH 도메인을 포함하는 (표 5 및 6), CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다. 본 개시내용은 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이 표 5 및 6에 열거된 VH CDR 중 어느 하나의 아미노산 서열을 갖는 VH CDR을 포함하는, CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 또한 제공한다. 특정한 측면에서, 본 개시내용은 항체가 표 5 및 6에 열거된 VH CDR 중 어느 것의 아미노산 서열을 갖는 VH CDR을 1개, 2개, 3개, 4개, 5개 또는 그 초과로 포함하는 (또는 대안적으로는 그로 이루어지는), CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다.
본 개시내용은 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이 서열식별번호: 21, 35, 49, 63, 77, 91, 105, 119, 133, 147, 161, 175, 189, 203, 217, 231, 245, 259, 273, 287, 301, 315 또는 329의 아미노산 서열을 갖는 VL 도메인을 포함하는 (표 5 및 6), CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다. 본 개시내용은 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이 하기 표 5 및 6에 열거된 VL CDR 중 어느 하나의 아미노산 서열을 갖는 VL CDR을 포함하는, CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 또한 제공한다. 특히, 본 개시내용은 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이 표 5 및 6에 열거된 VL CDR 중 어느 것의 아미노산 서열을 갖는 VL CDR을 1개, 2개, 3개 또는 그 초과로 포함하는 (또는 대안적으로는 그로 이루어지는), CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다.
본 개시내용의 다른 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)은 돌연변이되었지만 표 5 및 6에 기재된 서열에서 도시된 CDR 영역과 CDR 영역에서의 동일성이 적어도 60, 70, 80, 90 또는 95 퍼센트인 아미노산을 포함한다. 일부 측면에서, 이는 표 5 및 6에 기재된 서열에서 도시된 CDR 영역과 비교하였을 때 CDR 영역에서 1개, 2개, 3개, 4개 또는 5개 이하의 아미노산이 돌연변이된 돌연변이체 아미노산 서열을 포함한다.
본 개시내용은 CDH6에 특이적으로 결합하는 항체의 VH, VL, 전장 중쇄 및 전장 경쇄를 코딩하는 핵산 서열을 또한 제공한다. 이러한 핵산 서열은 포유동물 세포에서의 발현에 대해 최적화될 수 있다.
본 개시내용의 다른 항체는 아미노산 또는 아미노산을 코딩하는 핵산이 돌연변이되었지만 표 5 및 6에 기재된 서열에 대한 동일성이 적어도 60, 70, 80, 90 또는 95 퍼센트인 것을 포함한다. 일부 측면에서, 이는 표 5 및 6에 기재된 서열에 도시된 가변 영역과 비교하였을 때 가변 영역에서 1, 2, 3, 4 또는 5개 이하의 아미노산이 돌연변이되면서 실질적으로 동일한 치료 활성을 보유하는 돌연변이체 아미노산 서열을 포함한다.
각각의 이들 항체가 CDH6에 결합할 수 있기 때문에, VH, VL, 전장 경쇄 및 전장 중쇄 서열 (아미노산 서열 및 아미노산 서열을 코딩하는 뉴클레오티드 서열)이 "혼합 및 매칭"되어 다른 CDH6-결합 항체가 생성될 수 있다. 이러한 "혼합 및 매칭"된 CDH6-결합 항체는 관련 기술분야에 공지된 결합 검정 (예를 들어, ELISA, 및 실시예 섹션에 기재된 다른 검정)을 사용하여 시험될 수 있다. 이들 쇄가 혼합 및 매칭되는 경우에, 특정한 VH/VL 쌍형성으로부터의 VH 서열은 구조적으로 유사한 VH 서열로 대체되어야 한다. 마찬가지로, 특정한 전장 중쇄 / 전장 경쇄 쌍형성으로부터의 전장 중쇄 서열은 구조적으로 유사한 전장 중쇄 서열로 대체되어야 한다. 마찬가지로, 특정한 VH/VL 쌍형성으로부터의 VL 서열은 구조적으로 유사한 VL 서열로 대체되어야 한다. 마찬가지로, 특정한 전장 중쇄 / 전장 경쇄 쌍으로부터의 전장 경쇄 서열이 구조적으로 유사한 전장 경쇄 서열로 대체되어야 한다. 따라서, 한 측면에서, 본 개시내용은 서열식별번호: 20, 34, 48, 62, 76, 90, 104, 118, 132, 146, 160, 174, 188, 202, 216, 230, 244, 258, 272, 286, 300, 314 또는 328로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 중쇄 가변 영역 (표 5 및 6); 및 서열식별번호: 21, 35, 49, 63, 77, 91, 105, 119, 133, 147, 161, 175, 189, 203, 217, 231, 245, 259, 273, 287, 301, 315 또는 329로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 경쇄 가변 영역 (표 5 및 6)을 가지며, CDH6에 특이적으로 결합하는 단리된 모노클로날 항체 또는 그의 항원 결합 영역을 제공한다.
또 다른 측면에서, 본 개시내용은 (i) 서열식별번호: 24, 38, 52, 66, 80, 94, 108, 122, 136, 150, 164, 178, 192, 206, 220, 234, 248, 262, 276, 290, 304, 318 또는 332로 이루어진 군으로부터 선택된, 포유동물의 세포에서의 발현에 대해 최적화된 아미노산 서열을 포함하는 전장 중쇄; 및 서열식별번호: 25, 39, 53, 67, 81, 95, 109, 123, 137, 151, 165, 179, 193, 207, 221, 235, 249, 263, 277, 291, 305, 319 또는 333으로 이루어진 군으로부터 선택된, 포유동물의 세포에서의 발현에 대해 최적화된 아미노산 서열을 포함하는 전장 경쇄를 갖는 단리된 모노클로날 항체; 또는 (ii) 그의 항원 결합 부분을 포함하는 기능적 단백질을 제공한다
또 다른 측면에서, 본 개시내용은 표 5 및 6에 기재된 바와 같은 중쇄 및 경쇄 CDR1, CDR2 및 CDR3 또는 그의 조합을 포함하는 CDH6-결합 항체를 제공한다. 항체의 VH CDR1의 아미노산 서열은 서열식별번호: 17, 31, 45, 59, 73, 87, 101, 115, 129, 143, 157, 171, 185, 199, 213, 227, 241, 255, 269, 283, 297, 311 및 325에 제시된다. 항체의 VH CDR2의 아미노산 서열은 서열식별번호: 18, 32, 46, 60, 74, 88, 102,116, 130, 144, 158, 172, 186, 200, 214, 228, 242, 256, 270, 284, 298, 312 및 326에 제시된다. 항체의 VH CDR3의 아미노산 서열은 서열식별번호: 19, 33, 47, 61, 75, 89, 103, 117, 131, 145, 159, 173, 187, 201, 215, 229, 243, 257, 271, 285, 299, 313 및 327에 제시된다. 항체의 VL CDR1의 아미노산 서열은 서열식별번호: 14, 28, 42, 56, 70, 84, 98, 112, 126, 140, 154, 168, 182, 196, 210, 224, 238, 252, 266, 280, 294, 308 및 322에 제시된다. 항체의 VL CDR2의 아미노산 서열은 서열식별번호: 15, 29, 43, 57, 71, 85, 99, 113, 127, 141, 155, 169, 183, 197, 211, 225, 239, 253, 267, 281, 295, 309 및 323에 제시된다. 항체의 VL CDR3의 아미노산 서열은 서열식별번호: 16, 30, 44, 58, 72, 86, 100, 114, 128, 142, 156, 170, 184, 198, 212, 226, 240, 254, 268, 282, 296, 310 및 324에 제시된다.
각각의 이들 항체가 CDH6에 결합할 수 있다는 것 및 항원-결합 특이성이 주로 CDR1, 2 및 3 영역에 의해 제공된다는 것을 고려하여, VH CDR1, 2 및 3 서열 및 VL CDR1, 2 및 3 서열이 "혼합 및 매칭"될 수 있다 (즉, 다른 CDH6-결합 분자를 생성하기 위해 각각의 항체가 VH CDR1, 2 및 3 및 VL CDR1, 2 및 3을 함유하여야 하지만, 상이한 항체로부터의 CDR이 혼합 및 매칭될 수 있음). 이러한 "혼합 및 매칭"된 CDH6-결합 항체는 관련 기술분야에 공지된 결합 검정 및 실시예에 기재된 것 (예를 들어, ELISA)을 사용하여 시험될 수 있다. VH CDR 서열이 혼합 및 매칭되는 경우에, 특정한 VH 서열로부터의 CDR1, CDR2 및/또는 CDR3 서열은 구조적으로 유사한 CDR 서열(들)로 대체되어야 한다. 마찬가지로, VL CDR 서열이 혼합 및 매칭되는 경우에, 특정한 VL 서열로부터의 CDR1, CDR2 및/또는 CDR3 서열은 구조적으로 유사한 CDR 서열(들)로 대체되어야 한다. 1개 이상의 VH 및/또는 VL CDR 영역 서열을 본 개시내용의 모노클로날 항체에 대해 본원에 제시된 CDR 서열로부터의 구조적으로 유사한 서열로 치환함으로써 신규 VH 및 VL 서열이 생성될 수 있다는 것이 통상의 기술자에게 용이하게 자명할 것이다.
따라서, 본 개시내용은 서열식별번호: 17, 31, 45, 59, 73, 87, 101, 115, 129, 143, 157, 171, 185, 199, 213, 227, 241, 255, 269, 283, 297, 311 및 325로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 중쇄 CDR1; 서열식별번호: 18, 32, 46, 60, 74, 88, 102,116, 130, 144, 158, 172, 186, 200, 214, 228, 242, 256, 270, 284, 298, 312 및 326으로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 중쇄 CDR2; 서열식별번호: 19, 33, 47, 61, 75, 89, 103, 117, 131, 145, 159, 173, 187, 201, 215, 229, 243, 257, 271, 285, 299, 313 및 327로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 중쇄 CDR3; 서열식별번호: 14, 28, 42, 56, 70, 84, 98, 112, 126, 140, 154, 168, 182, 196, 210, 224, 238, 252, 266, 280, 294, 308 및 322로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 경쇄 CDR1; 서열식별번호: 15, 29, 43, 57, 71, 85, 99, 113, 127, 141, 155, 169, 183, 197, 211, 225, 239, 253, 267, 281, 295, 309 및 323으로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 경쇄 CDR2; 및 서열식별번호: 16, 30, 44, 58, 72, 86, 100, 114, 128, 142, 156, 170, 184, 198, 212, 226, 240, 254, 268, 282, 296, 310 및 324로 이루어진 군으로부터 선택된 아미노산 서열을 포함하는 경쇄 CDR3을 포함하며, CDH6에 특이적으로 결합하는 단리된 모노클로날 항체 또는 항원 결합 영역을 제공한다.
구체적 측면에서, CDH6에 특이적으로 결합하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)은 다음을 포함한다:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역.
특정 측면에서, CDH6에 특이적으로 결합하는 항체는 표 5 및/또는 6에 기재된 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)이다.
1. 에피토프 및 동일한 에피토프에 결합하는 항체의 확인
본 개시내용은 CDH6의 세포외 도메인 내의 에피토프에 결합하는 항체 및 항체 단편 (예를 들어, 항원 결합 단편)을 제공한다. 특정 측면에서, 항체 및 항체 단편은 CDH6 세포외 도메인의 도메인 내의 에피토프에 결합할 수 있다.
본 개시내용은 표 5 및 6에 기재된 항-CDH6 항체와 동일한 에피토프에 결합하는 항체 및 항체 단편 (예를 들어, 항원 결합 단편)을 또한 제공한다. 따라서, 추가의 항체 및 항체 단편 (예를 들어, 항원 결합 단편)이 CDH6 결합 검정에서 다른 항체와 교차-경쟁하는 (예를 들어, 통계적으로 유의한 방식으로 다른 항체의 결합을 경쟁적으로 억제하는) 그의 능력을 기반으로 하여 확인될 수 있다. 본 개시내용의 항체 및 항체 단편 (예를 들어, 항원 결합 단편)이 CDH6 단백질 (예를 들어, 인간 CDH6)에 결합하는 것을 억제하는 시험 항체의 능력은, 시험 항체가 CDH6에 결합하는 것에 대해 상기 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)과 경쟁할 수 있다는 것을 입증하고; 비제한적 이론에 따르면, 이러한 항체는 자신이 경쟁하는 항체 또는 항체 단편 (예를 들어, 항원 결합 단편)과 동일하거나 관련된 (예를 들어, 구조적으로 유사하거나 공간적으로 근접한) CDH6 단백질 상의 에피토프에 결합할 수 있다. 특정 측면에서, 본 개시내용의 항체 및 항체 단편 (예를 들어, 항원 결합 단편)과 동일한 CDH6 상의 에피토프에 결합하는 항체는 인간 또는 인간화 모노클로날 항체이다. 이러한 인간 또는 인간화 모노클로날 항체는 본원에 기재된 바와 같이 제조 및 단리될 수 있다.
2. Fc 영역의 프레임워크의 추가의 변경
본 개시내용은 부위-특이적으로 표지된 면역접합체를 제공한다. 이들 면역접합체는, 예를 들어 항체의 특성을 개선하기 위해, VH 및/또는 VL 내의 프레임워크 잔기에 대한 변형을 추가로 포함하는 변형된 항체 또는 그의 항원 결합 단편을 포함할 수 있다. 전형적으로 이러한 프레임워크 변형은 항체의 면역원성을 감소시키기 위해 이루어진다. 예를 들어, 하나의 접근법은 1개 이상의 프레임워크 잔기를 상응하는 배선 서열로 "역-돌연변이"시키는 것이다. 보다 구체적으로, 체세포 돌연변이를 겪은 항체는 항체가 유래된 배선 서열과는 상이한 프레임워크 잔기를 함유할 수 있다. 이러한 잔기는 항체 프레임워크 서열을 항체가 유래된 배선 서열과 비교함으로써 확인될 수 있다. 프레임워크 영역 서열을 그의 배선 형상으로 되돌리기 위해, 예를 들어 부위-지정 돌연변이유발에 의해, 체세포 돌연변이가 배선 서열로 "역-돌연변이"될 수 있다. 이러한 "역-돌연변이"된 항체가 또한 포괄되는 것으로 의도된다.
또 다른 유형의 프레임워크 변형은 프레임워크 영역 내 또는 심지어 1개 이상의 CDR 영역 내의 1개 이상의 잔기를 돌연변이시켜 T-세포 에피토프를 제거함으로써 항체의 잠재적인 면역원성을 감소시키는 것을 수반한다. 이러한 접근법은 "탈면역화"로 또한 지칭되고, 미국 특허 공개 번호 2003/0153043 (Carr et al.)에 보다 상세히 기재되어 있다.
프레임워크 또는 CDR 영역 내에서 이루어진 변형에 추가로 또는 대안적으로, 전형적으로는 항체의 1종 이상의 기능적 특성, 예컨대 혈청 반감기, 보체 고정, Fc 수용체 결합 및/또는 항원-의존성 세포성 세포독성을 변경시키기 위해, 항체가 Fc 영역 내에 변형을 포함하도록 조작될 수 있다. 또한, 다시 항체의 1종 이상의 기능적 특성을 변경시키기 위해, 항체가 화학적으로 변형될 수 있거나 (예를 들어, 1종 이상의 화학적 모이어티가 항체에 부착될 수 있음), 또는 그의 글리코실화가 변경되도록 변형될 수 있다. 각각의 이들 측면은 하기에 추가로 상세히 기재되어 있다.
한 측면에서, CH1의 힌지 영역은 힌지 영역 내의 시스테인 잔기의 수가 변경되도록, 예를 들어 증가 또는 감소되도록 변형된다. 이러한 접근법은 미국 특허 번호 5,677,425 (Bodmer et al.)에 추가로 기재되어 있다. CH1의 힌지 영역 내의 시스테인 잔기의 수는, 예를 들어 경쇄 및 중쇄의 조립을 용이하게 하거나 또는 항체의 안정성을 증가 또는 감소시키도록 변경된다.
또 다른 측면에서, 항체의 Fc 힌지 영역은 항체의 생물학적 반감기를 감소시키도록 돌연변이된다. 보다 구체적으로, 1개 이상의 아미노산 돌연변이가 Fc-힌지 단편의 CH2-CH3 도메인 계면 영역 내로 도입되어, 항체가 천연 Fc-힌지 도메인 스타필로코실 단백질 A (SpA) 결합에 비해 손상된 SpA 결합을 갖도록 한다. 이러한 접근법은 미국 특허 번호 6,165,745 (Ward et al.)에 추가로 상세히 기재되어 있다.
또 다른 측면에서, 1개 이상의 아미노산 잔기를 상이한 아미노산 잔기로 대체함으로써 Fc 영역이 변경되어 항체의 이펙터 기능이 변경된다. 예를 들어, 1개 이상의 아미노산이 상이한 아미노산 잔기로 대체되어, 항체가 이펙터 리간드에 대해 변경된 친화도를 갖지만 모 항체의 항원-결합 능력은 보유하도록 할 수 있다. 친화도가 변경된 이펙터 리간드는, 예를 들어 Fc 수용체 또는 보체의 C1 성분일 수 있다. 이러한 접근법은 미국 특허 번호 5,624,821 및 5,648,260 (둘 다 Winter et al.)에 기재되어 있다.
또 다른 측면에서, 아미노산 잔기로부터 선택된 1개 이상의 아미노산이 상이한 아미노산 잔기로 대체되어, 항체가 변경된 C1q 결합 및/또는 감소 또는 폐지된 보체 의존성 세포독성 (CDC)을 갖도록 할 수 있다. 이러한 접근법은, 예를 들어 미국 특허 번호 6,194,551 (Idusogie et al.)에 기재되어 있다.
또 다른 측면에서, 1개 이상의 아미노산 잔기가 변경됨으로써 보체를 고정하는 항체의 능력이 변경된다. 이러한 접근법은, 예를 들어 PCT 공개 WO 94/29351 (Bodmer et al.)에 기재되어 있다. 구체적 측면에서, 본 개시내용의 항체 또는 그의 항원 결합 단편의 1개 이상의 아미노산이 IgG1 하위부류 및 카파 이소형에 대한 1개 이상의 동종이형 아미노산 잔기로 대체된다. 동종이형 아미노산 잔기는 또한 문헌 [Jefferis et al., MAbs. 1:332-338 (2009)]에 기재된 바와 같이, IgG1, IgG2 및 IgG3 하위부류의 중쇄의 불변 영역, 뿐만 아니라 카파 이소형의 경쇄의 불변 영역을 포함하나 이에 제한되지는 않는다.
또 다른 측면에서, Fc 영역은 1개 이상의 아미노산을 변형시킴으로써 항체 의존성 세포성 세포독성 (ADCC)을 매개하는 항체의 능력을 증가시고/거나 Fcγ 수용체에 대한 항체의 친화도를 증가시키도록 변형된다. 이러한 접근법은, 예를 들어 PCT 공개 WO 00/42072 (Presta)에 기재되어 있다. 또한, FcγRI, FcγRII, FcγRIII 및 FcRn에 대한 인간 IgG1 상의 결합 부위가 맵핑되었고, 개선된 결합을 갖는 변이체가 기재되어 있다 (문헌 [Shields et al., J. Biol. Chem. 276:6591-6604, 2001] 참조).
또 다른 측면에서, 항체의 글리코실화가 변형된다. 예를 들어, 비-글리코실화 항체가 제조될 수 있다 (즉, 항체에 글리코실화가 결핍됨). 글리코실화는, 예를 들어 "항원"에 대한 항체의 친화도를 증가시키도록 변경될 수 있다. 이러한 탄수화물 변형은, 예를 들어 항체 서열 내의 1개 이상의 글리코실화 부위를 변경시킴으로써 달성될 수 있다. 예를 들어, 1개 이상의 가변 영역 프레임워크 글리코실화 부위의 제거를 유발하는 1개 이상의 아미노산 치환이 이루짐으로써 그 부위에서 글리코실화를 제거할 수 있다. 이러한 비-글리코실화는 항원에 대한 항체의 친화도를 증가시킬 수 있다. 이러한 접근법은, 예를 들어 미국 특허 번호 5,714,350 및 6,350,861 (Co et al.)에 기재되어 있다.
추가로 또는 대안적으로, 변경된 유형의 글리코실화를 갖는 항체, 예컨대 감소된 양의 푸코실 잔기를 갖는 저푸코실화 항체 또는 증가된 양분성 GlcNac 구조를 갖는 항체가 제조될 수 있다. 이러한 변경된 글리코실화 패턴은 항체의 ADCC 능력을 증가시키는 것으로 입증되었다. 이러한 탄수화물 변형은, 예를 들어 변경된 글리코실화 기구를 갖는 숙주 세포 내에서 항체를 발현시킴으로써 달성될 수 있다. 변경된 글리코실화 기구를 갖는 세포는 관련 기술분야에 기재되어 있고, 재조합 항체를 발현시키는 숙주 세포로서 사용됨으로써 변경된 글리코실화를 갖는 항체가 생산된다. 예를 들어, EP 1,176,195 (Hang et al.)는 푸코실 트랜스퍼라제를 코딩하는, 기능적으로 파괴된 FUT8 유전자를 갖는 세포주를 기재하며, 이러한 세포주에서 발현된 항체는 저푸코실화를 나타낸다. PCT 공개 WO 03/035835 (Presta)는 Asn(297)-연결된 탄수화물에 푸코스를 부착시키는 능력이 감소되어 숙주 세포 내에서 발현된 항체에서 저푸코실화가 유발되는 변이체 CHO 세포주인 Lecl3 세포를 기재한다 (또한, 문헌 [Shields et al., (2002) J. Biol. Chem. 277:26733-26740] 참조). PCT 공개 WO 99/54342 (Umana et al.)는 당단백질-변형 글리코실 트랜스퍼라제 (예를 들어, 베타(1,4)-N 아세틸글루코스아미닐트랜스퍼라제 III (GnTIII))를 발현하도록 조작된 세포주를 기재하며, 조작된 세포주에서 발현된 항체는 증가된 양분성 GlcNac 구조를 나타내고, 이는 항체의 증가된 ADCC 활성을 유발한다 (또한 문헌 [Umana et al., Nat. Biotech. 17:176-180, 1999] 참조).
또 다른 측면에서, 항체는 그의 생물학적 반감기가 증가되도록 변형된다. 다양한 접근법이 가능하다. 예를 들어, 미국 특허 번호 6,277,375 (Ward)에 기재된 바와 같은 하기 돌연변이 중 1개 이상이 도입될 수 있다: T252L, T254S, T256F. 대안적으로, 생물학적 반감기를 증가시키기 위해, 항체는 미국 특허 번호 5,869,046 및 6,121,022 (Presta et al.)에 기재된 바와 같이 IgG의 Fc 영역의 CH2 도메인의 2개의 루프로부터 얻은 샐비지 수용체 결합 에피토프를 함유하도록 CH1 또는 CL 영역 내에서 변경될 수 있다.
항체의 ADCC 활성을 최소화하기 위해, Fc 영역에서의 특정한 돌연변이가 이펙터 세포와의 상호작용이 최소인 "Fc 침묵" 항체를 생성한다. 일반적으로, "IgG Fc 영역"은 천연 서열 Fc 영역 및 변이체 Fc 영역을 포함하는, 이뮤노글로불린 중쇄의 C-말단 영역을 규정하도록 사용된다. 인간 IgG 중쇄 Fc 영역은 일반적으로 IgG 항체의 위치 C226 또는 P230으로부터 카르복실-말단까지의 아미노산 잔기를 포함하는 것으로 규정된다. Fc 영역에서 잔기의 넘버링은 카바트의 EU 인덱스의 것이다. 예를 들어, 항체의 생산 또는 정제 동안, Fc 영역의 C-말단 리신 (잔기 K447)이 제거될 수 있다.
침묵 이펙터 기능은 항체의 Fc 영역에서의 돌연변이에 의해 수득될 수 있고, 관련 기술분야에 기재되었다: LALA 및 N297A (Strohl, W., 2009, Curr. Opin. Biotechnol. vol. 20(6):685-691); 및 D265A (Baudino et al., 2008, J. Immunol. 181 : 6664-69). 또한, WO2012065950 (Heusser et al.)을 참조한다. 침묵 Fc IgG1 항체의 예는 IgG1 Fc 아미노산 서열 내의 L234A 및 L235A 돌연변이를 포함하는 LALA 돌연변이체이다. 침묵 IgG1 항체의 또 다른 예는 DAPA (D265A, P329A) 돌연변이 (US 6,737,056)이다. 침묵 IgG1 항체의 또 다른 예는 N297A 돌연변이를 포함하고, 이는 무-글리코실화/비-글리코실화 항체를 생성한다.
Fc 침묵 항체는 무 또는 저 ADCC 활성을 나타내며, 후자는 Fc 침묵 항체가 50% 미만의 특이적 세포 용해인 ADCC 활성을 나타낸다는 것을 의미하고, 무 ADCC 활성은 Fc 침묵 항체가 1% 미만인 ADCC 활성 (특이적 세포 용해)을 나타낸다는 것을 의미한다.
3. CDH6 항체의 생산
항-CDH6 항체 및 그의 항체 단편 (예를 들어, 항원 결합 단편)은, 재조합 발현, 화학적 합성 및 항체 사량체의 효소적 소화를 포함하나 이에 제한되지는 않는 관련 기술분야에 공지된 임의의 수단에 의해 생산될 수 있고, 반면에 전장 모노클로날 항체는, 예를 들어 하이브리도마 또는 재조합 생산에 의해 수득될 수 있다. 재조합 발현은 관련 기술분야에 공지된 임의의 적합한 숙주 세포, 예를 들어 포유동물 숙주 세포, 박테리아 숙주 세포, 효모 숙주 세포, 곤충 숙주 세포 등으로부터 일어날 수 있다.
본 개시내용은 본원에 기재된 항체를 코딩하는 폴리뉴클레오티드, 예를 들어 본원에 기재된 바와 같은 상보성 결정 영역을 포함하는 중쇄 또는 경쇄 가변 영역 또는 절편을 코딩하는 폴리뉴클레오티드를 추가로 제공한다. 일부 측면에서, 중쇄 가변 영역을 코딩하는 폴리뉴클레오티드는 서열식별번호: 22, 36, 50, 64, 78, 92, 106, 120, 134, 148, 162, 176, 190, 204, 218, 232, 246, 260, 274, 288, 302, 316 및 330으로 이루어진 군으로부터 선택된 폴리뉴클레오티드와 적어도 85%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 핵산 서열 동일성을 갖는다. 일부 측면에서, 경쇄 가변 영역을 코딩하는 폴리뉴클레오티드는 서열식별번호: 23, 37, 51, 65, 79, 93, 107, 121, 135, 149, 163, 177, 191, 205, 219, 233, 247, 261, 275, 289, 303, 317 및 331로 이루어진 군으로부터 선택된 폴리뉴클레오티드와 적어도 85%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 핵산 서열 동일성을 갖는다.
일부 측면에서, 중쇄를 코딩하는 폴리뉴클레오티드는 서열식별번호: 26, 40, 54, 68, 82, 96, 110, 124, 138, 152, 166, 180, 194, 208, 222, 236, 250, 264, 278, 292, 306, 320 및 334의 폴리뉴클레오티드와 적어도 85%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 핵산 서열 동일성을 갖는다. 일부 측면에서, 경쇄를 코딩하는 폴리뉴클레오티드는 서열식별번호: 27, 41, 55, 69, 83, 97, 111, 125, 139, 153, 167, 181, 195, 209, 223, 237, 251, 265, 279, 293, 307, 321 및 335의 폴리뉴클레오티드와 적어도 85%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% 또는 100% 핵산 서열 동일성을 갖는다.
본 개시내용의 폴리뉴클레오티드는 단지 항-CDH6 항체의 가변 영역 서열만을 코딩할 수 있다. 이들은 또한 항체의 가변 영역 및 불변 영역 둘 다를 코딩할 수 있다. 폴리뉴클레오티드 서열 중 일부는 예시된 항-CDH6 항체 중 하나의 중쇄 및 경쇄 둘 다의 가변 영역을 포함하는 폴리펩티드를 코딩한다. 일부 다른 폴리뉴클레오티드는 각각 마우스 항체 중 하나의 중쇄 및 경쇄의 가변 영역과 실질적으로 동일한 2개의 폴리펩티드 절편을 코딩한다.
폴리뉴클레오티드 서열은 항-CDH6 항체 또는 그의 결합 단편을 코딩하는 기존의 서열 (예를 들어, 하기 실시예에 기재된 바와 같은 서열)의 신생 고체-상 DNA 합성 또는 PCR 돌연변이유발에 의해 생산될 수 있다. 핵산의 직접적인 화학적 합성은 관련 기술분야에 공지된 방법, 예컨대 [Narang et al., Meth. Enzymol. 68:90, 1979; the phosphodiester method of Brown et al., Meth. Enzymol. 68:109, 1979]의 포스포디에스테르 방법; [Beaucage et al., Tetra. Lett., 22:1859, 1981]의 디에틸포스포르아미다이트 방법; 및 미국 특허 번호 4,458,066의 고체 지지체 방법에 의해 달성될 수 있다. PCR에 의해 폴리뉴클레오티드 서열에 돌연변이를 도입하는 것은, 예를 들어 [PCR Technology: Principles and Applications for DNA Amplification, H.A. Erlich (Ed.), Freeman Press, NY, NY, 1992; PCR Protocols: A Guide to Methods and Applications, Innis et al. (Ed.), Academic Press, San Diego, CA, 1990; Mattila et al., Nucleic Acids Res. 19:967, 1991; 및 Eckert et al., PCR Methods and Applications 1:17, 1991]에 기재된 바와 같이 수행될 수 있다.
또한, 본 개시내용에서는 상기 기재된 항-CDH6 항체를 생산하기 위한 발현 벡터 및 숙주 세포가 제공된다. 다양한 발현 벡터가 항-CDH6 항체 쇄 또는 결합 단편을 코딩하는 폴리뉴클레오티드를 발현시키는데 사용될 수 있다. 포유동물 숙주 세포에서 항체를 생산하기 위해 바이러스-기반 및 비-바이러스 발현 벡터가 둘 다 사용될 수 있다. 비-바이러스 벡터 및 시스템은 플라스미드, 에피솜 벡터 (전형적으로, 단백질 또는 RNA 발현을 위한 발현 카세트를 가짐), 및 인간 인공 염색체 (예를 들어, 문헌 [Harrington et al., Nat Genet 15:345, 1997] 참조)를 포함한다. 예를 들어, 포유동물 (예를 들어, 인간) 세포에서 항-CDH6 폴리뉴클레오티드 및 폴리펩티드를 발현시키는데 유용한 비-바이러스 벡터는 pThioHis A, B & C, pcDNA3.1/His, pEBVHis A, B & C (인비트로젠(Invitrogen), 캘리포니아주 샌디에고), MPSV 벡터, 및 다른 단백질을 발현시키는 것으로 관련 기술분야에 공지된 수많은 다른 벡터를 포함한다. 유용한 바이러스 벡터는 레트로바이러스, 아데노바이러스, 아데노연관 바이러스, 헤르페스 바이러스를 기반으로 하는 벡터, SV40, 유두종 바이러스, HBP 엡스타인 바르 바이러스를 기반으로 하는 벡터, 백시니아 바이러스 벡터 및 셈리키 포레스트 바이러스 (SFV)를 포함한다. 문헌 [Brent et al., 상기 문헌; Smith, Annu. Rev. Microbiol. 49:807, 1995; 및 Rosenfeld et al., Cell 68:143, 1992]을 참조한다.
발현 벡터의 선택은 벡터를 발현시키고자 의도되는 숙주 세포에 좌우된다. 전형적으로, 발현 벡터는 항-CDH6 항체 쇄 또는 단편을 코딩하는 폴리뉴클레오티드에 작동가능하게 연결된 프로모터 및 다른 조절 서열 (예를 들어, 인핸서)을 포함한다. 일부 측면에서, 유도성 프로모터가 유도 조건 하인 경우를 제외하고는 삽입된 서열의 발현을 방지하도록 사용된다. 유도성 프로모터는, 예를 들어 아라비노스, lacZ, 메탈로티오네인 프로모터 또는 열 쇼크 프로모터를 포함한다. 형질전환된 유기체의 배양물은, 발현 산물이 숙주 세포에 의해 보다 우수하게 허용되는 코딩 서열에 대한 집단으로 편향되지 않으면서 비유도 조건 하에서 증대될 수 있다. 프로모터에 추가로, 항-CDH6 항체 쇄 또는 단편의 효율적인 발현을 위해 다른 조절 요소가 또한 요구되거나 바람직할 수 있다. 이들 요소는 전형적으로 ATG 개시 코돈 및 인접한 리보솜 결합 부위 또는 다른 서열을 포함한다. 추가로, 발현의 효율은 사용 시에 세포계에 적절한 인핸서의 포함에 의해 의해 증진될 수 있다 (예를 들어 문헌 [Scharf et al., Results Probl. Cell Differ. 20:125, 1994; 및 Bittner et al., Meth. Enzymol., 153:516, 1987] 참조). 예를 들어, SV40 인핸서 또는 CMV 인핸서가 포유동물 숙주 세포에서의 발현을 증가시키기 위해 사용될 수 있다.
발현 벡터는 또한 삽입된 항-CDH6 항체 서열에 의해 코딩되는 폴리펩티드와의 융합 단백질을 형성하는 분비 신호 서열 위치를 제공할 수 있다. 보다 종종, 삽입된 항-CDH6 항체 서열은 신호 서열에 연결된 후 벡터에 포함된다. 항-CDH6 항체 경쇄 및 중쇄 가변 도메인을 코딩하는 서열을 수용하는데 사용되는 벡터는 때때로 또한 불변 영역 또는 그의 일부를 코딩한다. 이러한 벡터는 불변 영역과의 융합 단백질로서 가변 영역의 발현을 가능하게 함으로써 무손상 항체 또는 그의 단편이 생산되게 한다. 전형적으로, 이러한 불변 영역은 인간의 것이다.
항-CDH6 항체 쇄를 보유하고 발현하는 숙주 세포는 원핵 또는 진핵 세포일 수 있다. 이. 콜라이(E. coli)는 본 개시내용의 폴리뉴클레오티드를 클로닝하고 발현하는데 유용한 한 원핵 숙주이다. 사용하기에 적합한 다른 미생물 숙주는 바실루스, 예컨대 바실루스 서브틸리스(Bacillus subtilis), 및 다른 엔테로박테리아세아에, 예컨대 살모넬라(Salmonella), 세라티아(Serratia) 및 다양한 슈도모나스(Pseudomonas) 종을 포함한다. 이들 원핵 숙주에서, 전형적으로 숙주 세포와 상용성인 발현 제어 서열 (예를 들어, 복제 기점)을 함유하는 발현 벡터를 또한 제조할 수 있다. 추가로, 임의의 수의 다양한 널리 공지된 프로모터, 예컨대 락토스 프로모터 시스템, 트립토판 (trp) 프로모터 시스템, 베타-락타마제 프로모터 시스템, 또는 파지 람다로부터의 프로모터 시스템이 존재할 것이다. 프로모터는 임의로 오퍼레이터 서열과 함께 전형적으로 발현을 제어하고, 전사 및 번역을 개시하고 완료하기 위한 리보솜 결합 부위 서열 등을 갖는다. 다른 미생물, 예컨대 효모가 또한 항-CDH6 폴리펩티드를 발현시키는데 사용될 수 있다. 바큘로바이러스 벡터와 조합되어 곤충 세포가 또한 사용될 수 있다.
한 측면에서, 포유동물 숙주 세포가 본 개시내용의 항-CDH6 폴리펩티드를 발현시키고 제조하는데 사용된다. 예를 들어, 이들은 내인성 이뮤노글로불린 유전자를 발현하는 하이브리도마 세포주 (예를 들어, 골수종 하이브리도마 클론), 또는 외인성 발현 벡터를 보유하는 포유동물 세포주 (예를 들어, SP2/0 골수종 세포)일 수 있다. 이들은 임의의 정상적인 사멸, 또는 정상적이거나 비정상적인 불멸 동물 또는 인간 세포를 포함한다. 예를 들어, CHO 세포주, 다양한 COS 세포주, HeLa 세포, 골수종 세포주, 형질전환된 B-세포 및 하이브리도마를 포함한, 무손상 이뮤노글로불린을 분비할 수 있는 다수의 적합한 숙주 세포주가 개발되었다. 폴리펩티드를 발현시키기 위한 포유동물 조직 세포 배양의 사용은, 예를 들어 문헌 [Winnacker, From Genes to Clones, VCH Publishers, N.Y., N.Y., 1987]에서 일반적으로 논의된다. 포유동물 숙주 세포를 위한 발현 벡터는 발현 제어 서열, 예컨대 복제 기점, 프로모터, 및 인핸서 (예를 들어, 문헌 [Queen et al., Immunol. Rev. 89:49-68, 1986] 참조), 및 필수적인 프로세싱 정보 부위, 예컨대 리보솜 결합 부위, RNA 스플라이스 부위, 폴리아데닐화 부위, 및 전사 종결인자 서열을 포함할 수 있다. 이들 발현 벡터는 통상적으로 포유동물 유전자 또는 포유동물 바이러스로부터 유래된 프로모터를 함유한다. 적합한 프로모터는 구성적, 세포 유형-특이적, 단계-특이적 및/또는 조정가능하거나 조절가능한 것일 수 있다. 유용한 프로모터는 메탈로티오네인 프로모터, 구성적 아데노바이러스 주요 후기 프로모터, 덱사메타손-유도성 MMTV 프로모터, SV40 프로모터, MRP polIII 프로모터, 구성적 MPSV 프로모터, 테트라시클린-유도성 CMV 프로모터 (예컨대, 인간 극초기 CMV 프로모터), 구성적 CMV 프로모터, 및 관련 기술분야에 공지된 프로모터-인핸서 조합을 포함하나 이에 제한되지는 않는다.
관심 폴리뉴클레오티드 서열을 함유하는 발현 벡터를 도입하는 방법은 세포 숙주의 유형에 따라 달라진다. 예를 들어, 염화칼슘 형질감염은 통상적으로 원핵 세포에 이용되고, 한편 인산칼슘 처리 또는 전기천공은 다른 세포 숙주에 사용될 수 있다 (일반적으로 [Sambrook et al., 상기 문헌] 참조). 다른 방법, 예를 들어 전기천공, 인산칼슘 처리, 리포솜-매개 형질전환, 주사 및 미세주사, 탄도적 방법, 비로솜, 이뮤노리포솜, 다가양이온:핵산 접합체, 네이키드 DNA, 인공 비리온, 헤르페스 바이러스 구조 단백질 VP22에 대한 융합 (Elliot and O'Hare, Cell 88:223, 1997), DNA의 작용제-증진 흡수 및 생체외 형질도입을 포함한다. 재조합 단백질의 장기, 고수율 생산을 위해, 안정한 발현이 종종 요구될 것이다. 예를 들어, 항-CDH6 항체 쇄 또는 결합 단편을 안정하게 발현하는 세포주는 바이러스 복제 기점 또는 내인성 발현 요소 및 선택가능한 마커 유전자를 함유하는 발현 벡터를 사용하여 제조될 수 있다. 벡터의 도입 후에, 세포는 선택 배지로 교체하기 전에 풍부화된 배지에서 1-2일 동안 성장될 수 있다. 선택 마커의 목적은 선택에 대한 저항성을 부여하는 것이고, 그의 존재는 선택 배지에서 도입 서열을 성공적으로 발현하는 세포의 성장을 가능하게 한다. 저항성이 있는, 안정하게 형질감염된 세포는 세포 유형에 적절한 조직 배양 기술을 사용하여 증식될 수 있다.
치료 및 진단 용도
본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체는 암, 예컨대 고형 암의 치료를 포함하나 이에 제한되지는 않는 다양한 용도에서 유용하다. 특정 측면에서, 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체는 종양 성장 억제, 분화 유도, 종양 부피 감소, 및/또는 종양의 종양발생성 감소에 유용하다. 사용 방법은 시험관내, 생체외 또는 생체내 방법일 수 있다.
한 측면에서, 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체는 생물학적 샘플 내의 CDH6의 존재를 검출하는데 유용하다. 본원에 사용된 용어 "검출하는"은 정량적 또는 정성적 검출을 포괄한다. 특정 측면에서, 생물학적 샘플은 세포 또는 조직을 포함한다. 특정 측면에서, 이러한 조직은 CDH6을 다른 조직에 비해 더 높은 수준으로 발현하는 정상 및/또는 암성 조직을 포함한다.
한 측면에서, 본 개시내용은 생물학적 샘플 내의 CDH6의 존재를 검출하는 방법을 제공한다. 특정 측면에서, 방법은 생물학적 샘플을 항-CDH6 항체와, 항체가 항원에 결합하는 것을 허용하는 조건 하에 접촉시키고, 항체와 항원 사이에 복합체가 형성되는지 여부를 검출하는 것을 포함한다.
또한, CDH6의 증가된 발현과 연관된 장애를 진단하는 방법이 제공된다. 특정 측면에서, 방법은 시험 세포를 항-CDH6 항체와 접촉시키고; CDH6 항원에 대한 항-CDH6 항체의 결합을 검출함으로써 시험 세포 상에서의 CDH6의 발현 수준을 (정량적으로 또는 정성적으로) 결정하고; 시험 세포에서의 CDH6의 발현 수준을 대조군 세포 (예를 들어, 시험 세포와 동일한 조직 기원의 정상 세포 또는 이러한 정상 세포에 필적하는 수준으로 CDH6을 발현하는 세포)에서의 CDH6의 발현 수준과 비교하고, 여기서 대조군 세포와 비교 시에 시험 세포 상에서의 CDH6의 더 높은 발현 수준은 CDH6의 증가된 발현과 연관된 장애의 존재를 나타내는 것을 포함한다. 특정 측면에서, 시험 세포는 CDH6의 증가된 발현과 연관된 장애를 갖는 것으로 의심되는 개체로부터 수득된다. 특정 측면에서, 장애는 세포 증식성 장애, 예컨대 암 또는 종양이다.
특정 측면에서, 진단 또는 검출 방법, 예컨대 상기 기재된 것은 세포 표면 상에서 발현된 CDH6 또는 세포 표면 상에 CDH6을 발현하는 세포로부터 수득된 막 제제 내의 CDH6에 대한 항-CDH6 항체의 결합을 검출하는 것을 포함한다. 세포 표면 상에서 발현된 CDH6에 대한 항-CDH6 항체의 결합을 검출하기 위한 예시적인 검정은 "FACS" 검정이다.
특정의 다른 방법이 CDH6에 대한 항-CDH6 항체의 결합을 검출하는데 사용될 수 있다. 이러한 방법은 관련 기술분야에 널리 공지된 항원-결합 검정, 예컨대 웨스턴 블롯, 방사성 면역검정, ELISA (효소 연결된 면역흡착 검정), "샌드위치" 면역검정, 면역침전 검정, 형광 면역검정, 단백질 A 면역검정 및 면역조직화학 (IHC)을 포함하나 이에 제한되지는 않는다.
특정 측면에서, 항-CDH6 항체는 표지된다. 표지는 직접적으로 검출되는 표지 또는 모이어티 (예컨대 형광, 발색, 전자-밀집, 화학발광 및 방사성 표지), 뿐만 아니라 간접적으로, 예를 들어 효소적 반응 또는 분자 상호작용을 통해 검출되는 모이어티, 예컨대 효소 또는 리간드를 포함하나 이에 제한되지는 않는다.
특정 측면에서, 항-CDH6 항체는 불용성 매트릭스 상에 고정화된다. 고정화는 용액 중에 유리 상태로 남아 있는 임의의 CDH6 단백질로부터 항-CDH6 항체를 분리하는 것을 수반한다. 이는 통상적으로 검정 절차 전에, 예컨대 수불용성 매트릭스 또는 표면에 대한 흡착에 의해 (미국 특허 번호 3,720,760 (Bennich et al.)), 또는 공유결합 커플링에 의해 (예를 들어, 글루타르알데히드 가교를 사용함) 항-CDH6 항체를 불용화시킴으로써, 또는 항-CDH6 항체와 CDH6 단백질 사이에 복합체가 형성된 후에, 예를 들어 면역침전에 의해 항-CDH6 항체를 불용화시킴으로써 달성된다.
임의의 상기 진단 또는 검출 측면은 항-CDH6 항체 대신에 또는 이에 추가로 본 개시내용의 면역접합체를 사용하여 수행될 수 있다.
한 측면에서, 본 개시내용은 환자에게 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체를 투여함으로써 질환을 치료하는 것을 포함하는, 질환을 치료하거나, 예방하거나 또는 호전시키는 방법을 제공한다. 특정 측면에서, 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체로 치료되는 질환은 암이다. 치료 및/또는 예방될 수 있는 질환의 예는 난소암, 신암, 간암, 연부조직암, CNS암, 갑상선암 및 담관암종을 포함하나 이에 제한되지는 않는다. 특정 측면에서, 암은 항체, 항체 단편 (예를 들어, 항원 결합 단편) 및 항체 약물 접합체가 특이적으로 결합할 수 있는 CDH6 발현 세포를 특징으로 한다.
본 개시내용은 치료 유효량의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체를 투여하는 것을 포함하는, 암을 치료하는 방법을 제공한다. 특정 측면에서, 암은 고형 암이다. 특정 측면에서, 대상체는 인간이다.
특정 측면에서, 종양 성장을 억제하는 방법은 대상체에게 치료 유효량의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체를 투여하는 것을 포함한다. 특정 측면에서, 대상체는 인간이다. 특정 측면에서, 대상체는 종양을 갖거나 또는 종양이 제거된 적이 있다.
특정 측면에서, 종양은 항-CDH6 항체가 결합하는 CDH6을 발현한다. 특정 측면에서, 종양은 인간 CDH6을 과다발현한다.
질환의 치료를 위해, 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체의 적절한 투여량은 다양한 인자, 예컨대 치료될 질환의 유형, 질환의 중증도 및 경과, 질환의 반응성, 선행 요법, 환자의 임상 병력 등에 좌우된다. 항체 또는 작용제는 한번에 또는 수일 내지 수개월 동안 지속되는 일련의 치료에 걸쳐, 또는 치유가 일어나거나 질환 상태의 축소 (예를 들어, 종양 크기의 감소)가 달성될 때까지 투여될 수 있다. 최적의 투여 일정이 환자 신체 내의 약물 축적의 측정치로부터 계산될 수 있고, 개별적인 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체의 상대적인 효능에 따라 다를 것이다. 특정 측면에서, 투여량은 체중 kg당 0.01 mg 내지 10 mg (예를 들어, 0.01 mg, 0.05mg, 0.lmg, 0.5mg, lmg, 2mg, 3mg, 4mg, 5mg, 7mg, 8mg, 9mg, 또는 10mg)이고, 매일, 매주, 매월 또는 매년 1회 이상 제공될 수 있다. 특정 측면에서, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체는 2주마다 1회 또는 3주마다 1회 제공된다. 치료하는 의사는 체액 또는 조직 내 약물의 측정된 체류 시간 및 농도를 기반으로 하여 투여를 위한 반복률을 추정할 수 있다.
조합 요법
특정 경우에, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체가 다른 치료제, 예컨대 다른 항암제, 항알레르기제, 항오심제 (또는 항구토제), 통증 완화제, 세포보호제 및 그의 조합과 조합된다.
조합 요법에 사용하기 위해 고려되는 일반적 화학요법제는 아나스트로졸 (아리미덱스(Arimidex)®), 비칼루타미드 (카소덱스(Casodex)®), 블레오마이신 술페이트 (블레녹산(Blenoxane)®), 부술판 (밀레란(Myleran)®), 부술판 주사 (부술펙스(Busulfex)®), 카페시타빈 (젤로다(Xeloda)®), N4-펜톡시카르보닐-5-데옥시-5-플루오로시티딘, 카르보플라틴 (파라플라틴(Paraplatin)®), 카르무스틴 (BiCNU®), 클로람부실 (류케란(Leukeran)®), 시스플라틴 (플라티놀(Platinol)®), 클라드리빈 (류스타틴(Leustatin)®), 시클로포스파미드 (시톡산(Cytoxan)® 또는 네오사르(Neosar)®), 시타라빈, 시토신 아라비노시드 (시토사르-U(Cytosar-U)®), 시타라빈 리포솜 주사 (데포사이트(DepoCyt)®), 다카르바진 (DTIC-돔(DTIC-Dome)®), 닥티노마이신 (악티노마이신 D, 코스메간(Cosmegan)®), 다우노루비신 히드로클로라이드 (세루비딘(Cerubidine)®), 다우노루비신 시트레이트 리포솜 주사 (다우녹솜(DaunoXome)®), 덱사메타손, 도세탁셀 (탁소테레(Taxotere)®), 독소루비신 히드로클로라이드 (아드리아마이신(Adriamycin)®, 루벡스(Rubex)®), 에토포시드 (베페시드(Vepesid)®), 플루다라빈 포스페이트 (플루다라(Fludara)®), 5-플루오로우라실 (아드루실(Adrucil)®, 에푸덱스(Efudex)®), 플루타미드 (유렉신(Eulexin)®), 테자시티빈, 겜시타빈 (디플루오로데옥시시티딘), 히드록시우레아 (히드레아(Hydrea)®), 이다루비신 (이다마이신(Idamycin)®), 이포스파미드 (이펙스(IFEX)®), 이리노테칸 (캄프토사르(Camptosar)®), L-아스파라기나제 (엘스파르(ELSPAR)®), 류코보린 칼슘, 멜팔란 (알케란(Alkeran)®), 6-메르캅토퓨린 (퓨린톨(Purinethol)®), 메토트렉세이트 (폴렉스(Folex)®), 미톡산트론 (노반트론(Novantrone)®), 밀로타르그, 파클리탁셀 (탁솔(Taxol)®), 포에닉스 (이트륨90/MX-DTPA), 펜토스타틴, 폴리페프로산 20과 카르무스틴 이식물 (글리아델(Gliadel)®), 타목시펜 시트레이트 (놀바덱스(Nolvadex)®), 테니포시드 (부몬(Vumon)®), 6-티오구아닌, 티오테파, 티라파자민 (티라존(Tirazone)®), 주사를 위한 토포테칸 히드로클로라이드 (하이캄틴(Hycamptin)®), 빈블라스틴 (벨반(Velban)®), 빈크리스틴 (온코빈(Oncovin)®) 및 비노렐빈 (나벨빈(Navelbine)®)을 포함한다.
한 측면에서, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체가 제약 조합 제제에서 또는 조합 요법으로서의 투여 요법에서 항암 특성이 있는 제2 화합물과 조합된다. 제약 조합 제제 또는 투여 요법의 제2 화합물은 서로에 불리하게 영향을 미치지 않도록 조합의 항체 또는 면역접합체에 대한 상보적 활성을 가질 수 있다. 예를 들어, 본 개시내용의 항체, 항체 단편 (예를 들어, 항원 결합 단편) 또는 항체 약물 접합체가, 비제한적으로 화학요법제, 티로신 키나제 억제제, 예를 들어 이마티닙과 조합되어 투여될 수 있다.
본원에 사용된 용어 "제약 조합물"은 한 투여 단위 형태의 고정된 조합물, 또는 2종 이상의 치료제가 독립적으로 동시에 또는 개별적으로 시간 간격을 두고 (특히, 이들 시간 간격이, 조합 파트너가 협력적 효과, 예를 들어 상승작용적 효과를 나타내는 것을 가능하게 하는 경우) 투여될 수 있는 조합 투여를 위한 비-고정 조합물 또는 부분들의 키트를 지칭한다.
용어 "조합 요법"은 본 개시내용에 기재된 치료적 상태 또는 장애를 치료하기 위한 2종 이상의 치료제의 투여를 지칭한다. 이러한 투여는 이들 치료제를 실질적으로 동시인 방식으로, 예컨대 고정 비의 활성 성분을 갖는 단일 캡슐로 공-투여하는 것을 포괄한다. 대안적으로, 이러한 투여는 각각의 활성 성분에 대해 다중 또는 개별 용기 (예를 들어, 캡슐, 분말 및 액체)에서 공-투여하는 것을 포괄한다. 분말 및/또는 액체는 투여 전에 재구성되거나 목적하는 용량으로 희석될 수 있다. 추가로, 이러한 투여는 또한 대략적으로 동일한 시간에 또는 상이한 시간에 각각의 유형의 치료제를 순차적인 방식으로 사용하는 것을 또한 포괄한다. 어느 경우든, 치료 요법은 본원에 기재된 상태 또는 장애를 치료하는데 있어서 약물 조합물의 유익한 효과를 제공할 것이다.
조합 요법은 "상승작용"을 제공하고 "상승작용적"임을, 즉 활성 성분을 함께 사용할 경우 달성되는 효과가 화합물을 개별적으로 사용함으로써 유발되는 효과의 합계보다 더 큼을 입증할 수 있다. 상승작용적 효과는 활성 성분이 다음과 같은 경우에 달성될 수 있다: (1) 공-제제화 및 투여되거나 조합으로 동시에 전달되는 단위 투여 제제; (2) 개별 제제로서 교대로 또는 병행으로 전달됨; 또는 (3) 일부 다른 요법에 의함. 교대 요법으로 전달되는 경우에, 화합물을, 예를 들어 별개의 주사기에서 상이하게 주사함으로써 순차적으로 투여 또는 전달하는 경우에, 상승작용적 효과가 달성될 수 있다. 일반적으로, 교대 요법 동안에, 유효 투여량의 각각의 활성 성분이 순차적으로, 즉 연속으로 투여되고, 반면에 조합 요법에서는 유효 투여량의 2종 이상의 활성 성분이 함께 투여된다.
한 측면에서, 본 개시내용은 암의 치료를 필요로 하는 대상체에게 항체 약물 접합체를 1종 이상의 티로신 키나제 억제제 (EGFR 억제제, Her2 억제제, Her3 억제제, IGFR 억제제 및 Met 억제제를 포함하나 이에 제한되지는 않음)와 조합하여 투여함으로써 암을 치료하는 방법을 제공한다.
예를 들어, 티로신 키나제 억제제는 에를로티닙 히드로클로라이드 (타르세바(Tarceva)®); 리니파닙 (N-[4-(3-아미노-1H-인다졸-4-일)페닐]-N'-(2-플루오로-5-메틸페닐)우레아) (ABT 869로 또한 공지되어 있고, 제넨테크(Genentech)로부터 입수가능함); 수니티닙 말레이트 (수텐트(Sutent)®); 보수티닙 (4-[(2,4-디클로로-5-메톡시페닐)아미노]-6-메톡시-7-[3-(4-메틸피페라진-1-일)프로폭시]퀴놀린-3-카르보니트릴 (SKI-606으로 또한 공지되어 있고, 미국 특허 번호 6,780,996에 기재됨); 다사티닙 (스프리셀(Sprycel)®); 파조파닙 (보트리엔트(Votrient)®); 소라페닙 (넥사바르(Nexavar)®); 작티마 (ZD6474); 닐로티닙 (타시그나(Tasigna)®); 레고라페닙 (스티바르가(Stivarga)®) 및 이마티닙 또는 이마티닙 메실레이트 (글리벡(Gilvec)® 및 글리벡(Gleevec)®)를 포함하나 이에 제한되지는 않는다.
표피 성장 인자 수용체 (EGFR) 억제제는 에를로티닙 히드로클로라이드 (타르세바(Tarceva)®), 게피티닙 (이레사(Iressa)®); N-[4-[(3-클로로-4-플루오로페닐)아미노]-7-[[(3"S")-테트라히드로-3-푸라닐]옥시]-6-퀴나졸리닐]-4(디메틸아미노)-2-부텐아미드 (토복(Tovok)®); 반데타닙 (카프렐사(Caprelsa)®); 라파티닙 (타이커브(Tykerb)®); (3R,4R)-4-아미노-1-((4-((3-메톡시페닐)아미노)피롤로[2,1-f][1,2,4]트리아진-5-일)메틸)피페리딘-3-올 (BMS690514); 카네르티닙 디히드로클로라이드 (CI-1033); 6-[4-[(4-에틸-1-피페라지닐)메틸]페닐]-N-[(1R)-1-페닐에틸]-7H-피롤로[2,3-d]피리미딘-4-아민 (AEE788, CAS 497839-62-0); 무브리티닙 (TAK165); 펠리티닙 (EKB569); 아파티닙 (BIBW2992); 네라티닙 (HKI-272); N-[4-[[1-[(3-플루오로페닐)메틸]-1H-인다졸-5-일]아미노]-5-메틸피롤로[2,1-f][1,2,4]트리아진-6-일]-카르밤산, (3S)-3-모르폴리닐메틸 에스테르 (BMS599626); N-(3,4-디클로로-2-플루오로페닐)-6-메톡시-7-[[(3aα,5β,6aα)-옥타히드로-2-메틸시클로펜타[c]피롤-5-일]메톡시]-4-퀴나졸린아민 (XL647, CAS 781613-23-8); 및 4-[4-[[(1R)-1-페닐에틸]아미노]-7H-피롤로[2,3-d]피리미딘-6-일]-페놀 (PKI166, CAS 187724-61-4)을 포함하나 이에 제한되지는 않는다.
EGFR 항체는 세툭시맙 (에르비툭스(Erbitux)®); 파니투무맙 (벡티빅스(Vectibix)®); 마투주맙 (EMD-72000); 니모투주맙 (hR3); 잘루투무맙; TheraCIM h-R3; MDX0447 (CAS 339151-96-1); 및 ch806 (mAb-806, CAS 946414-09-1)을 포함하나 이에 제한되지는 않는다.
인간 표피 성장 인자 수용체 2 (HER2 수용체) (또한 Neu, ErbB-2, CD340 또는 p185로도 공지됨) 억제제는 트라스투주맙 (헤르셉틴(Herceptin)®); 페르투주맙 (옴니타르그(Omnitarg)®); 네라티닙 (HKI-272, (2E)-N-[4-[[3-클로로-4-[(피리딘-2-일)메톡시]페닐]아미노]-3-시아노-7-에톡시퀴놀린-6-일]-4-(디메틸아미노)부트-2-엔아미드 및 PCT 공개 번호 WO 05/028443에 기재됨); 라파티닙 또는 라파티닙 디토실레이트 (타이커브(Tykerb)®); (3R,4R)-4-아미노-1-((4-((3-메톡시페닐)아미노)피롤로[2,1-f][1,2,4]트리아진-5-일)메틸)피페리딘-3-올 (BMS690514); (2E)-N-[4-[(3-클로로-4-플루오로페닐)아미노]-7-[[(3S)-테트라히드로-3-푸라닐]옥시]-6-퀴나졸리닐]-4-(디메틸아미노)-2-부텐아미드 (BIBW-2992, CAS 850140-72-6); N-[4-[[1-[(3-플루오로페닐)메틸]-1H-인다졸-5-일]아미노]-5-메틸피롤로[2,1-f][1,2,4]트리아진-6-일]-카르밤산, (3S)-3-모르폴리닐메틸 에스테르 (BMS 599626, CAS 714971-09-2); 카네르티닙 디히드로클로라이드 (PD183805 또는 CI-1033); 및 N-(3,4-디클로로-2-플루오로페닐)-6-메톡시-7-[[(3aα,5β,6aα)-옥타히드로-2-메틸시클로펜타[c]피롤-5-일]메톡시]-4-퀴나졸린아민 (XL647, CAS 781613-23-8)을 포함하나 이에 제한되지는 않는다.
HER3 억제제는 LJM716, MM-121, AMG-888, RG7116, REGN-1400, AV-203, MP-RM-1, MM-111, 및 MEHD-7945A를 포함하나 이에 제한되지는 않는다
MET 억제제는 카보잔티닙 (XL184, CAS 849217-68-1); 포레티닙 (GSK1363089, 이전에 XL880, CAS 849217-64-7); 티반티닙 (ARQ197, CAS 1000873-98-2); 1-(2-히드록시-2-메틸프로필)-N-(5-(7-메톡시퀴놀린-4-일옥시)피리딘-2-일)-5-메틸-3-옥소-2-페닐-2,3-디히드로-1H-피라졸-4-카르복스아미드 (AMG 458); 크리조티닙 (잘코리(Xalkori)®, PF-02341066); (3Z)-5-(2,3-디히드로-1H-인돌-1-일술포닐)-3-({3,5-디메틸-4-[(4-메틸피페라진-1-일)카르보닐]-1H-피롤-2-일}메틸렌)-1,3-디히드로-2H-인돌-2-온 (SU11271); (3Z)-N-(3-클로로페닐)-3-({3,5-디메틸-4-[(4-메틸피페라진-1-일)카르보닐]-1H-피롤-2-일}메틸렌)-N-메틸-2-옥소인돌린-5-술폰아미드 (SU11274); (3Z)-N-(3-클로로페닐)-3-{[3,5-디메틸-4-(3-모르폴린-4-일프로필)-1H-피롤-2-일]메틸렌}-N-메틸-2-옥소인돌린-5-술폰아미드 (SU11606); 6-[디플루오로[6-(1-메틸-1H-피라졸-4-일)-1,2,4-트리아졸로[4,3-b]피리다진-3-일]메틸]-퀴놀린 (JNJ38877605, CAS 943540-75-8); 2-[4-[1-(퀴놀린-6-일메틸)-1H-[1,2,3]트리아졸로[4,5-b]피라진-6-일]-1H-피라졸-1-일]에탄올 (PF04217903, CAS 956905-27-4); N-((2R)-1,4-디옥산-2-일메틸)-N-메틸-N'-[3-(1-메틸-1H-피라졸-4-일)-5-옥소-5H-벤조[4,5]시클로헵타[1,2-b]피리딘-7-일]술파미드 (MK2461, CAS 917879-39-1); 6-[[6-(1-메틸-1H-피라졸-4-일)-1,2,4-트리아졸로[4,3-b]피리다진-3-일]티오]-퀴놀린 (SGX523, CAS 1022150-57-7); 및 (3Z)-5-[[(2,6-디클로로페닐)메틸]술포닐]-3-[[3,5-디메틸-4-[[(2R)-2-(1-피롤리디닐메틸)-1-피롤리디닐]카르보닐]-1H-피롤-2-일]메틸렌]-1,3-디히드로-2H-인돌-2-온 (PHA665752, CAS 477575-56-7)을 포함하나 이에 제한되지는 않는다.
IGF1R 억제제는 BMS-754807, XL-228, OSI-906, GSK0904529A, A-928605, AXL1717, KW-2450, MK0646, AMG479, IMCA12, MEDI-573, 및 BI836845를 포함하나 이에 제한되지는 않는다. 예를 들어, 검토를 위해 문헌 [Yee, JNCI, 104; 975 (2012)]을 참조한다.
또 다른 측면에서, 본 개시내용은 암의 치료를 필요로 하는 대상체에게 항체 약물 접합체를 1종 이상의 FGF 하류 신호전달 경로 억제제 (MEK 억제제, Braf 억제제, PI3K/Akt 억제제, SHP2 억제제 및 또한 mTor 억제제를 포함하나 이에 제한되지는 않음)와 조합하여 투여함으로써 암을 치료하는 방법을 제공한다.
예를 들어, 미토겐-활성화 단백질 키나제 (MEK) 억제제는 XL-518 (GDC-0973으로 또한 공지되어 있고, Cas 번호 1029872-29-4이며, 에이씨씨 코포레이션(ACC Corp.)으로부터 입수가능함); 2-[(2-클로로-4-아이오도페닐)아미노]-N-(시클로프로필메톡시)-3,4-디플루오로-벤즈아미드 (CI-1040 또는 PD184352로 또한 공지되어 있고, PCT 공개 번호 WO2000035436에 기재됨); N-[(2R)-2,3-디히드록시프로폭시]-3,4-디플루오로-2-[(2-플루오로-4-아이오도페닐)아미노]-벤즈아미드 (PD0325901로 또한 공지되어 있고, PCT 공개 번호 WO2002006213에 기재됨); 2,3-비스[아미노[(2-아미노페닐)티오]메틸렌]-부탄디니트릴 (U0126으로 또한 공지되어 있고, 미국 특허 번호 2,779,780에 기재됨); N-[3,4-디플루오로-2-[(2-플루오로-4-아이오도페닐)아미노]-6-메톡시페닐]-1-[(2R)-2,3-디히드록시프로필]-시클로프로판술폰아미드 (RDEA119 또는 BAY869766으로 또한 공지되어 있고, PCT 공개 번호 WO2007014011에 기재됨); (3S,4R,5Z,8S,9S,11E)-14-(에틸아미노)-8,9,16-트리히드록시-3,4-디메틸-3,4,9,19-테트라히드로-1H-2-벤족사시클로테트라데신-1,7(8H)-디온] (E6201로 또한 공지되어 있고, PCT 공개 번호 WO2003076424에 기재됨); 2'-아미노-3'-메톡시플라본 (PD98059로 또한 공지되어 있고, 독일의 비아핀 게엠베하 운트 코., 카게(Biaffin GmbH & Co., KG)로부터 입수가능함); 베무라페닙 (PLX-4032, CAS 918504-65-1); (R)-3-(2,3-디히드록시프로필)-6-플루오로-5-(2-플루오로-4-아이오도페닐아미노)-8-메틸피리도[2,3-d]피리미딘-4,7(3H,8H)-디온 (TAK-733, CAS 1035555-63-5); 피마세르팁 (AS-703026, CAS 1204531-26-9); 및 트라메티닙 디메틸 술폭시드 (GSK-1120212, CAS 1204531-25-80)를 포함하나 이에 제한되지는 않는다.
포스포이노시티드 3-키나제 (PI3K) 억제제는 4-[2-(1H-인다졸-4-일)-6-[[4-(메틸술포닐)피페라진-1-일]메틸]티에노[3,2-d]피리미딘-4-일]모르폴린 (GDC 0941로 또한 공지되어 있고, PCT 공개 번호 WO 09/036082 및 WO 09/055730에 기재됨); 2-메틸-2-[4-[3-메틸-2-옥소-8-(퀴놀린-3-일)-2,3-디히드로이미다조[4,5-c]퀴놀린-1-일]페닐]프로피오니트릴 (BEZ 235 또는 NVP-BEZ 235로 또한 공지되어 있고, PCT 공개 번호 WO 06/122806에 기재됨); 4-(트리플루오로메틸)-5-(2,6-디모르폴리노피리미딘-4-일)피리딘-2-아민 (BKM120 또는 NVP-BKM120으로 또한 공지되어 있고, PCT 공개 번호 WO2007/084786에 기재됨); 토자세르팁 (VX680 또는 MK-0457, CAS 639089-54-6); (5Z)-5-[[4-(4-피리디닐)-6-퀴놀리닐]메틸렌]-2,4-티아졸리딘디온 (GSK1059615, CAS 958852-01-2); (1E,4S,4aR,5R,6aS,9aR)-5-(아세틸옥시)-1-[(디-2-프로페닐아미노)메틸렌]-4,4a,5,6,6a,8,9,9a-옥타히드로-11-히드록시-4-(메톡시메틸)-4a,6a-디메틸-시클로펜타[5,6]나프토[1,2-c]피란-2,7,10(1H)-트리온 (PX866, CAS 502632-66-8); 및 8-페닐-2-(모르폴린-4-일)-크로멘-4-온 (LY294002, CAS 154447-36-6)을 포함하나 이에 제한되지는 않는다.
mTor 억제제는 템시롤리무스 (토리셀(Torisel)®); 리다포롤리무스 (이전에 데포롤리무스로 공지되고, (1R,2R,4S)-4-[(2R)-2 [(1R,9S,12S,15R,16E,18R,19R,21R, 23S,24E,26E,28Z,30S,32S,35R)-1,18-디히드록시-19,30-디메톡시-15,17,21,23,29,35-헥사메틸-2,3,10,14,20-펜타옥소-11,36-디옥사-4-아자트리시클로[30.3.1.04,9]헥사트리아콘타-16,24,26,28-테트라엔-12-일]프로필]-2-메톡시시클로헥실 디메틸포스피네이트이고, AP23573 및 MK8669로 또한 공지되어 있고, PCT 공개 번호 WO 03/064383에 기재됨); 에베롤리무스 (아피니토르(Afinitor)® 또는 RAD001); 라파마이신 (AY22989, 시롤리무스(Sirolimus)®); 시마피모드 (CAS 164301-51-3); (5-{2,4-비스[(3S)-3-메틸모르폴린-4-일]피리도[2,3-d]피리미딘-7-일}-2-메톡시페닐)메탄올 (AZD8055); 2-아미노-8-[트랜스-4-(2-히드록시에톡시)시클로헥실]-6-(6-메톡시-3-피리디닐)-4-메틸-피리도[2,3-d]피리미딘-7(8H)-온 (PF04691502, CAS 1013101-36-4); 및 N2-[1,4-디옥소-4-[[4-(4-옥소-8-페닐-4H-1-벤조피란-2-일)모르폴리늄-4-일]메톡시]부틸]-L-아르기닐글리실-L-α-아스파르틸L-세린-, 내부 염 (SF1126, CAS 936487-67-1)을 포함하나 이에 제한되지는 않는다.
또 다른 측면에서, 본 개시내용은 암의 치료를 필요로 하는 대상체에게 항체 약물 접합체를 1종 이상의 아폽토시스촉진제 (IAP 억제제, Bcl2 억제제, MCl1 억제제, Trail 작용제, Chk 억제제를 포함하나 이에 제한되지는 않음)와 조합하여 투여함으로써 암을 치료하는 방법을 제공한다.
예를 들어, IAP 억제제는 NVP-LCL161, GDC-0917, AEG-35156, AT406 및 TL32711을 포함하나 이에 제한되지는 않는다. IAP 억제제의 다른 예는 WO04/005284, WO 04/007529, WO05/097791, WO 05/069894, WO 05/069888, WO 05/094818, US2006/0014700, US2006/0025347, WO 06/069063, WO 06/010118, WO 06/017295, 및 WO08/134679 (이들 모두 본원에 참조로 포함됨)에 개시된 것을 포함하나 이에 제한되지는 않는다.
BCL-2 억제제는 4-[4-[[2-(4-클로로페닐)-5,5-디메틸-1-시클로헥센-1-일]메틸]-1-피페라지닐]-N-[[4-[[(1R)-3-(4-모르폴리닐)-1-[(페닐티오)메틸]프로필]아미노]-3-[(트리플루오로메틸)술포닐]페닐]술포닐]벤즈아미드 (ABT-263으로 또한 공지되어 있고, PCT 공개 번호 WO 09/155386에 기재됨); 테트로카르신 A; 안티마이신; 고시폴 ((-)BL-193); 오바토클락스; 에틸-2-아미노-6-시클로펜틸-4-(1-시아노-2-에톡시-2-옥소에틸)-4H크로몬-3-카르복실레이트 (HA14 - 1); 오블리메르센 (G3139, 게나센스(Genasense)®); Bak BH3 펩티드; (-)-고시폴 아세트산 (AT-101); 4-[4-[(4'-클로로[1,1'-비페닐]-2-일)메틸]-1-피페라지닐]-N-[[4-[[(1R)-3-(디메틸아미노)-1-[(페닐티오)메틸]프로필]아미노]-3-니트로페닐]술포닐]-벤즈아미드 (ABT-737, CAS 852808-04-9); 및 나비토클락스 (ABT-263, CAS 923564-51-6)를 포함하나 이에 제한되지는 않는다.
DR4 (TRAILR1) 및 DR5 (TRAILR2)를 포함하는 아폽토시스촉진 수용체 효능제 (PARA)는 둘라네르민 (AMG-951, RhApo2L/TRAIL); 마파투무맙 (HRS-ETR1, CAS 658052-09-6); 렉사투무맙 (HGS-ETR2, CAS 845816-02-6); 아포맙 (아포맙(Apomab)®); 코나투무맙 (AMG655, CAS 896731-82-1); 및 티가투주맙 (CS1008, CAS 946415-34-5, 다이이치 산쿄(Daiichi Sankyo)로부터 입수가능함)을 포함하나 이에 제한되지는 않는다.
체크포인트 키나제 (CHK) 억제제는 7-히드록시스타우로스포린 (UCN-01); 6-브로모-3-(1-메틸-1H-피라졸-4-일)-5-(3R)-3-피페리디닐-피라졸로[1,5-a]피리미딘-7-아민 (SCH900776, CAS 891494-63-6); 5-(3-플루오로페닐)-3-우레이도티오펜-2-카르복실산 N-[(S)-피페리딘-3-일]아미드 (AZD7762, CAS 860352-01-8); 4-[((3S)-1-아자비시클로[2.2.2]옥트-3-일)아미노]-3-(1H-벤즈이미다졸-2-일)-6-클로로퀴놀린-2(1H)-온 (CHIR 124, CAS 405168-58-3); 7-아미노닥티노마이신 (7-AAD), 이소그라눌라티미드, 데브로모히메니알디신; N-[5-브로모-4-메틸-2-[(2S)-2-모르폴리닐메톡시]-페닐]-N'-(5-메틸-2-피라지닐)우레아 (LY2603618, CAS 911222-45-2); 술포라판 (CAS 4478-93-7, 4-메틸술피닐부틸 이소티오시아네이트); 9,10,11,12-테트라히드로-9,12-에폭시-1H-디인돌로[1,2,3-fg:3',2',1'-kl]피롤로[3,4-i][1,6]벤조디아조신-1,3(2H)-디온 (SB-218078, CAS 135897-06-2); 및 TAT-S216A (Sha et al., Mol. Cancer. Ther 2007; 6(1):147-153), 및 CBP501을 포함하나 이에 제한되지는 않는다.
한 측면에서, 본 개시내용은 암의 치료를 필요로 하는 대상체에게 항체 약물 접합체를 1종 이상의 FGFR 억제제와 조합하여 투여함으로써 암을 치료하는 방법을 제공한다. 예를 들어, FGFR 억제제는 브리바닙 알라니네이트 (BMS-582664, (S)-((R)-1-(4-(4-플루오로-2-메틸-1H-인돌-5-일옥시)-5-메틸피롤로[2,1-f][1,2,4]트리아진-6-일옥시)프로판-2-일)2-아미노프로파노에이트); 바르가테프 (BIBF1120, CAS 928326-83-4); 도비티닙 디락트산 (TKI258, CAS 852433-84-2); 3-(2,6-디클로로-3,5-디메톡시-페닐)-1-{6-[4-(4-에틸-피페라진-1-일)-페닐아미노]-피리미딘-4-일}-1-메틸-우레아 (BGJ398, CAS 872511-34-7); 다누세르팁 (PHA-739358); 및 (PD173074, CAS 219580-11-7)을 포함하나 이에 제한되지는 않는다. 구체적 측면에서, 본 개시내용은 암의 치료를 필요로 하는 대상체에게 항체 약물 접합체를 FGFR2 억제제, 예컨대 3-(2,6-디클로로-3,5-디메톡시페닐)-1-(6((4-(4-에틸피페라진-1-일)페닐)아미노)피리미딘-4-일)-1-메틸우레아 (BGJ-398로 또한 공지됨); 또는 4-아미노-5-플루오로-3-(5-(4-메틸피페라진-1-일)-1H-벤조[d]이미다졸-2-일)퀴놀린-2(1H)-온 (도비티닙 또는 TKI-258로 또한 공지됨), AZD4547 (문헌 [Gavine et al., 2012, Cancer Research 72, 2045-56], N-[5-[2-(3,5-디메톡시페닐)에틸]-2H-피라졸-3-일]-4-(3R,5S)-디메틸피페라진-1-일)벤즈아미드), 포나티닙 (AP24534; 문헌 [Gozgit et al., 2012, Mol Cancer Ther., 11; 690-99]; 3-[2-(이미다조[1,2-b]피리다진-3-일)에티닐]-4-메틸-N-{4-[(4-메틸피페라진-1-일)메틸]-3-(트리플루오로메틸)페닐}벤즈아미드, CAS 943319-70-8)과 조합하여 투여함으로써 암을 치료하는 방법을 제공한다.
제약 조성물
면역접합체를 포함하는 제약 또는 멸균 조성물을 제조하기 위해, 본 개시내용의 면역접합체가 제약상 허용되는 담체 또는 부형제와 혼합된다. 조성물은 추가로 암, 예를 들어 난소암, 신암, 간암, 연부조직암, CNS암, 갑상선암 및 담관암종을 치료 또는 예방하는데 적합한 1종 이상의 다른 치료제를 함유할 수 있다.
치료제 및 진단제의 제제는 생리학상 허용되는 담체, 부형제 또는 안정화제와 혼합되어, 예를 들어 동결건조 분말, 슬러리, 수용액, 로션 또는 현탁액의 형태로 제조될 수 있다 (예를 들어, 문헌 [Hardman et al., Goodman and Gilman's The Pharmacological Basis of Therapeutics, McGraw-Hill, New York, N.Y., 2001; Gennaro, Remington: The Science and Practice of Pharmacy, Lippincott, Williams, and Wilkins, New York, N.Y., 2000; Avis, et al. (eds.), Pharmaceutical Dosage Forms: Parenteral Medications, Marcel Dekker, NY, 1993; Lieberman, et al. (eds.), Pharmaceutical Dosage Forms: Tablets, Marcel Dekker, NY, 1990; Lieberman, et al. (eds.) Pharmaceutical Dosage Forms: Disperse Systems, Marcel Dekker, NY, 1990; Weiner and Kotkoskie, Excipient Toxicity and Safety, Marcel Dekker, Inc., New York, N.Y., 2000] 참조).
구체적 측면에서, 본 개시내용의 항체 약물 접합체의 임상 서비스 형태 (CSF)는 ADC, 숙신산나트륨 및 폴리소르베이트 20을 함유하는 바이알 내의 동결건조물이다. 동결건조물은 주사용수로 재구성될 수 있고, 용액은 약 5.0의 pH에서 ADC, 숙신산나트륨, 수크로스 및 폴리소르베이트 20을 함유한다. 후속 정맥내 투여를 위해, 수득된 용액이 통상적으로 담체 용액 내로 추가로 희석될 것이다.
치료제에 대한 투여 요법을 선택하는 것은 물질의 혈청 또는 조직 전환율, 증상의 수준, 물질의 면역원성 및 생물학적 매트릭스에서의 표적 세포의 접근가능성을 포함한 여러 인자에 좌우된다. 특정 측면에서, 투여 요법은 허용되는 부작용 수준과 일치하게 환자에 전달되는 치료제의 양을 최대화한다. 따라서, 전달되는 생물제제의 양은 부분적으로 특정한 물질 및 치료될 상태의 중증도에 좌우된다. 항체, 시토카인 및 소분자의 적절한 용량을 선택하는데 있어서의 지침이 입수가능하다 (예를 들어, 문헌 [Wawrzynczak, Antibody Therapy, Bios Scientific Pub. Ltd, Oxfordshire, UK, 1996; Kresina (ed.), Monoclonal Antibodies, Cytokines and Arthritis, Marcel Dekker, New York, N.Y., 1991; Bach (ed.), Monoclonal Antibodies and Peptide Therapy in Autoimmune Diseases, Marcel Dekker, New York, N.Y., 1993; Baert et al., New Engl. J. Med. 348:601-608, 2003; Milgrom et al., New Engl. J. Med. 341:1966-1973, 1999; Slamon et al., New Engl. J. Med. 344:783-792, 2001; Beniaminovitz et al., New Engl. J. Med. 342:613-619, 2000; Ghosh et al., New Engl. J. Med. 348:24-32, 2003; Lipsky et al., New Engl. J. Med. 343:1594-1602, 2000] 참조).
적절한 용량의 결정은, 예를 들어 치료에 영향을 미치는 것으로 관련 기술분야에 공지되어 있거나 그러한 것으로 의심되거나, 또는 치료에 영향을 미칠 것으로 예측되는 파라미터 또는 인자를 사용하여 임상의에 의해 이루어진다. 일반적으로, 용량은 최적 용량보다 다소 적은 양에서 시작하며, 그 후 임의의 부정적 부작용에 비해 바람직하거나 최적인 효과가 달성될 때까지 소량 증분으로 증가시킨다. 중요한 진단 척도는, 예를 들어 염증의 증상 또는 생산된 염증성 시토카인의 수준을 포함한다.
제약 조성물 항체 약물 접합체 내의 활성 성분의 실제 투여량 수준은 환자에게 독성이지 않으면서 특정한 환자, 조성물 및 투여 방식에 대한 목적하는 치료 반응을 달성하는데 효과적인 활성 성분의 양이 수득되도록 달라질 수 있다. 선택되는 투여량 수준은 사용되는 본 개시내용의 특정한 조성물 또는 그의 에스테르, 염 또는 아미드의 활성, 투여 경로, 투여 시간, 사용되는 특정한 화합물의 배출 속도, 치료의 지속기간, 사용되는 특정한 조성물과 조합되어 사용되는 다른 약물, 화합물 및/또는 물질, 치료되는 환자의 연령, 성별, 체중, 상태, 전반적 건강 및 과거 병력, 및 의학 기술분야에 공지된 유사 인자를 포함한 다양한 약동학적 인자에 좌우될 것이다.
항체 또는 그의 단편을 포함하는 조성물은 연속 주입에 의해, 또는 예를 들어 1일, 1주, 또는 1주에 1-7회의 간격의 투여에 의해 제공될 수 있다. 투여는 정맥내로, 피하로, 국소로, 경구로, 비강으로, 직장으로, 근육내, 뇌내로 또는 흡입에 의해 제공될 수 있다. 구체적 투여 프로토콜은 유의한 바람직하지 않은 부작용을 피하는 최대 용량 또는 투여 빈도를 수반하는 것이다.
본 개시내용의 면역접합체에 대해, 환자에게 투여되는 투여량은 환자 체중 기준으로 0.0001 mg/kg 내지 100 mg/kg일 수 있다. 투여량은 환자 체중 기준으로 0.0001 mg/kg 내지 20 mg/kg, 0.0001 mg/kg 내지 10 mg/kg, 0.0001 mg/kg 내지 5 mg/kg, 0.0001 내지 2 mg/kg, 0.0001 내지 1 mg/kg, 0.0001 mg/kg 내지 0.75 mg/kg, 0.0001 mg/kg 내지 0.5 mg/kg, 0.0001 mg/kg 내지 0.25 mg/kg, 0.0001 내지 0.15 mg/kg, 0.0001 내지 0.10 mg/kg, 0.001 내지 0.5 mg/kg, 0.01 내지 0.25 mg/kg 또는 0.01 내지 0.10 mg/kg일 수 있다. 항체 또는 그의 단편의 투여량은 킬로그램 (㎏) 단위의 환자의 체중에 mg/kg 단위의 투여될 용량을 곱하는 것을 사용하여 계산될 수 있다.
면역접합체의 투여가 반복될 수 있고, 투여는 적어도 1일, 2일, 3일, 5일, 10일, 15일, 30일, 45일, 2개월, 75일, 3개월, 또는 적어도 6개월 간격일 수 있다. 구체적 측면에서, 본 개시내용의 면역접합체의 투여는 3주마다 반복된다.
특정한 환자에 대한 유효량은 치료될 상태, 환자의 전반적인 건강, 투여의 방법, 경로 및 용량, 및 부작용의 중증도와 같은 인자에 따라 달라질 수 있다 (예를 들어, 문헌 [Maynard et al., A Handbook of SOPs for Good Clinical Practice, Interpharm Press, Boca Raton, Fla., 1996; Dent, Good Laboratory and Good Clinical Practice, Urch Publ., London, UK, 2001] 참조).
투여 경로는, 예를 들어 국소 또는 피부 적용, 정맥내, 복강내, 뇌내, 근육내, 안내, 동맥내, 뇌척수내, 병변내 주사 또는 주입, 또는 지속 방출 시스템 또는 이식물에 의한 것일 수 있다 (예를 들어, 문헌 [Sidman et al., Biopolymers 22:547-556, 1983; Langer et al., J. Biomed. Mater. Res. 15:167-277, 1981; Langer, Chem. Tech. 12:98-105, 1982; Epstein et al., Proc. Natl. Acad. Sci. USA 82:3688-3692, 1985; Hwang et al., Proc. Natl. Acad. Sci. USA 77:4030-4034, 1980]; 미국 특허 번호 6,350,466 및 6,316,024 참조). 필요한 경우에, 조성물은 또한 가용화제, 또는 주사 부위에서의 통증을 완화하는 리도카인과 같은 국부 마취제, 또는 둘 다를 포함할 수 있다. 추가로, 폐 투여는, 예를 들어 흡입기 또는 네뷸라이저의 사용, 및 에어로졸화제를 사용한 제제화에 의해 사용될 수 있다. 예를 들어, 미국 특허 번호 6,019,968, 5,985,320, 5,985,309, 5,934,272, 5,874,064, 5,855,913, 5,290,540, 및 4,880,078; 및 PCT 공개 번호 WO 92/19244, WO 97/32572, WO 97/44013, WO 98/31346, 및 WO 99/66903을 참조하며, 이들은 각각 그 전문이 본원에 참조로 포함된다.
본 개시내용의 조성물은 또한 관련 기술분야에 공지된 다양한 방법 중 1종 이상을 사용하여 1종 이상의 투여 경로를 통해 투여될 수 있다. 통상의 기술자가 인지하는 바와 같이, 투여 경로 및/또는 방식은 목적하는 결과에 따라 달라질 것이다. 면역접합체에 대한 선택된 투여 경로는 정맥내, 근육내, 피내, 복강내, 피하, 척수 또는 다른 비경구 투여 경로, 예를 들어 주사 또는 주입에 의한 것을 포함한다. 비경구 투여는 경장 및 국소 투여 이외에 통상적으로 주사에 의한 투여 방식을 나타내고, 정맥내, 근육내, 동맥내, 척수강내, 피막내, 안와내, 심장내, 피내, 복강내, 경기관, 피하, 각피하, 관절내, 피막하, 지주막하, 척수내, 경막외 및 흉골내 주사 및 주입을 비제한적으로 포함한다. 대안적으로, 본 개시내용의 조성물은 비-비경구 경로, 예컨대 국소, 표피 또는 점막 투여 경로를 통해, 예를 들어 비강내로, 경구로, 질로, 직장으로, 설하로 또는 국소로 투여될 수 있다. 한 측면에서, 본 개시내용의 면역접합체는 주입에 의해 투여된다. 또 다른 측면에서, 면역접합체는 피하로 투여된다.
본 개시내용의 면역접합체가 제어 방출 또는 지속 방출 시스템으로 투여되는 경우에, 펌프가 제어 또는 지속 방출을 달성하는데 사용될 수 있다 (문헌 [Langer, 상기 문헌; Sefton, CRC Crit. Ref Biomed. Eng. 14:20, 1987; Buchwald et al., Surgery 88:507, 1980; Saudek et al., N. Engl. J. Med. 321:574, 1989] 참조). 중합체 물질이 면역접합체의 요법의 제어 또는 지속 방출을 달성하기 위해 사용될 수 있다 (예를 들어, 문헌 [Medical Applications of Controlled Release, Langer and Wise (eds.), CRC Pres., Boca Raton, Fla., 1974; Controlled Drug Bioavailability, Drug Product Design and Performance, Smolen and Ball (eds.), Wiley, New York, 1984; Ranger and Peppas, J. Macromol. Sci. Rev. Macromol. Chem. 23:61, 1983] 참조; 또한 문헌 [Levy et al., Science 228:190, 1985; During et al., Ann. Neurol. 25:351, 1989; Howard et al., J. Neurosurg. 7 1:105, 1989] 참조; 미국 특허 번호 5,679,377; 미국 특허 번호 5,916,597; 미국 특허 번호 5,912,015; 미국 특허 번호 5,989,463; 미국 특허 번호 5,128,326; PCT 공개 번호 WO 99/15154; 및 PCT 공개 번호 WO 99/20253). 지속 방출 제제에 사용된 중합체의 예는 폴리(2-히드록시 에틸 메타크릴레이트), 폴리(메틸 메타크릴레이트), 폴리(아크릴산), 폴리(에틸렌-코-비닐 아세테이트), 폴리(메타크릴산), 폴리글리콜리드 (PLG), 폴리무수물, 폴리(N-비닐 피롤리돈), 폴리(비닐 알콜), 폴리아크릴아미드, 폴리(에틸렌 글리콜), 폴리락티드 (PLA), 폴리(락티드-코-글리콜리드) (PLGA) 및 폴리오르토에스테르를 포함하나 이에 제한되지는 않는다. 한 측면에서, 지속 방출 제제에 사용된 중합체는 불활성이고, 걸러 낼 수 있는 불순물이 없으며, 보관 시에 안정하고, 멸균성이고, 생분해성이다. 제어 또는 지속 방출 시스템은 예방적 또는 치료적 표적의 근위에 놓일 수 있고, 따라서 단지 전신 용량의 분획만이 요구된다 (예를 들어, 문헌 [Goodson, in Medical Applications of Controlled Release, 상기 문헌, vol. 2, pp. 115-138, 1984] 참조).
제어 방출 시스템은 문헌 [Langer, Science 249:1527-1533, 1990]의 검토에서 논의된다. 관련 기술분야의 통상의 기술자에게 공지된 임의의 기술이 본 개시내용의 1종 이상의 면역접합체를 포함하는 지속 방출 제제를 생산하는데 사용될 수 있다. 예를 들어, 미국 특허 번호 4,526,938, PCT 공개 WO 91/05548, PCT 공개 WO 96/20698, 문헌 [Ning et al., Radiotherapy & Oncology 39:179-189, 1996; Song et al., PDA Journal of Pharmaceutical Science & Technology 50:372-397, 1995; Cleek et al., Pro. Int'l. Symp. Control. Rel. Bioact. Mater. 24:853-854, 1997; 및 Lam et al., Proc. Int'l. Symp. Control Rel. Bioact. Mater. 24:759-760, 1997]을 참조하며, 이들 각각은 그 전문이 본원에 참조로 포함된다.
본 개시내용의 면역접합체가 국소로 투여되는 경우에, 이들은 연고, 크림, 경피 패치, 로션, 겔, 샴푸, 스프레이, 에어로졸, 용액, 에멀젼 형태, 또는 관련 기술분야의 통상의 기술자에게 널리 공지된 다른 형태로 제제화될 수 있다. 예를 들어, 문헌 [Remington's Pharmaceutical Sciences and Introduction to Pharmaceutical Dosage Forms, 19th ed., Mack Pub. Co., Easton, Pa. (1995)]을 참조한다. 비-분무가능한 국소 투여 형태의 경우, 국소 적용에 상용성인 담체 또는 1종 이상의 부형제를 포함하며 일부 경우에 물보다 큰 동적 점도를 갖는 점성 내지 반-고체 또는 고체 형태가 전형적으로 사용된다. 적합한 제제는, 원하는 경우에 멸균되거나, 예를 들어 삼투압과 같은 다양한 특성에 영향을 미치기 위한 보조제 (예를 들어, 보존제, 안정화제, 습윤제, 완충제 또는 염)와 혼합되는 용액, 현탁액, 에멀젼, 크림, 연고, 분말, 도찰제, 살브 등을 비제한적으로 포함한다. 다른 적합한 국소 투여 형태는 활성 성분이 일부 경우에 고체 또는 액체 불활성 담체와 조합되어 가압 휘발성 물질 (예를 들어, 기체상 추진제, 예컨대 프레온)과의 혼합물 중에 또는 압착병 내에 패키징되는 분무가능한 에어로졸 제제를 포함한다. 보습제 또는 함습제가 또한 원하는 경우에 제약 조성물 및 투여 형태에 첨가될 수 있다. 이러한 추가의 성분의 예는 관련 기술분야에 널리 공지되어 있다.
면역접합체를 포함하는 조성물이 비강내로 투여되는 경우에, 이는 에어로졸 형태, 스프레이, 미스트 내에 또는 점적제의 형태로 제제화될 수 있다. 특히, 본 개시내용에 따라 사용하기 위한 예방제 또는 치료제는 적합한 추진제 (예를 들어, 디클로로디플루오로메탄, 트리클로로플루오로메탄, 디클로로테트라플루오로에탄, 이산화탄소 또는 다른 적합한 기체)를 사용하여, 가압 팩 또는 네뷸라이저로부터 에어로졸 스프레이 제공물의 형태로 편리하게 전달될 수 있다. 가압 에어로졸의 경우에, 투여 단위는 계량된 양을 전달하기 위해 밸브를 제공함으로써 결정될 수 있다. 흡입기 또는 취입기에 사용하기 위한 캡슐 및 카트리지 (예를 들어, 젤라틴으로 구성됨)는 화합물의 분말 혼합물 및 적합한 분말 기재, 예컨대 락토스 또는 전분을 함유하여 제제화될 수 있다.
제2 치료제, 예를 들어 시토카인, 스테로이드, 화학요법제, 항생제 또는 방사선과의 공-투여 또는 치료 방법이 관련 기술분야에 공지되어 있다 (예를 들어, 문헌 [Hardman et al., (eds.) (2001) Goodman and Gilman's The Pharmacological Basis of Therapeutics, 10.sup.th ed., McGraw-Hill, New York, N.Y.; Poole and Peterson (eds.) (2001) Pharmacotherapeutics for Advanced Practice:A Practical Approach, Lippincott, Williams & Wilkins, Phila., Pa.; Chabner and Longo (eds.) (2001) Cancer Chemotherapy and Biotherapy, Lippincott, Williams & Wilkins, Phila., Pa.] 참조). 유효량의 치료제는 증상을 적어도 10%; 적어도 20%; 적어도 약 30%; 적어도 40% 또는 적어도 50% 감소시킬 수 있다.
면역접합체와 조합되어 투여될 수 있는 추가의 요법 (예를 들어, 예방제 또는 치료제)은 본 개시내용의 면역접합체와 5분 미만의 간격, 30분 미만의 간격, 1시간 간격, 약 1시간 간격, 약 1 내지 약 2시간 간격, 약 2시간 내지 약 3시간 간격, 약 3시간 내지 약 4시간 간격, 약 4시간 내지 약 5시간 간격, 약 5시간 내지 약 6시간 간격, 약 6시간 내지 약 7시간 간격, 약 7시간 내지 약 8시간 간격, 약 8시간 내지 약 9시간 간격, 약 9시간 내지 약 10시간 간격, 약 10시간 내지 약 11시간 간격, 약 11시간 내지 약 12시간 간격, 약 12시간 내지 18시간 간격, 18시간 내지 24시간 간격, 24시간 내지 36시간 간격, 36시간 내지 48시간 간격, 48시간 내지 52시간 간격, 52시간 내지 60시간 간격, 60시간 내지 72시간 간격, 72시간 내지 84시간 간격, 84시간 내지 96시간 간격, 또는 96시간 내지 120시간 간격으로 투여될 수 있다. 2종 이상의 요법은 1회 동일한 환자 방문에서 투여될 수 있다.
특정 측면에서, 면역접합체는 생체내에서 적절한 분포를 보장하도록 제제화될 수 있다. 예를 들어, 혈액-뇌 장벽 (BBB)은 많은 고도로 친수성인 화합물을 배제시킨다. 본 개시내용의 치료 화합물이 (원하는 경우에) BBB를 횡단하도록 보장하기 위해, 이들은, 예를 들어 리포솜 내에 제제화될 수 있다. 리포솜을 제조하는 방법에 대해, 예를 들어 미국 특허 번호 4,522,811; 5,374,548; 및 5,399,331을 참조한다. 리포솜은 특정 세포 또는 기관 내로 선택적으로 수송되어 표적화 약물 전달을 증진시키는 1개 이상의 모이어티를 포함할 수 있다 (예를 들어, 문헌 [Ranade, (1989) J. Clin. Pharmacol. 29:685] 참조). 예시적인 표적화 모이어티는 폴레이트 또는 비오틴 (예를 들어 미국 특허 번호 5,416,016 (Low et al.)); 만노시드 (Umezawa et al., (1988) Biochem. Biophys. Res. Commun. 153:1038); 항체 (Bloeman et al., (1995) FEBS Lett. 357:140; Owais et al., (1995) Antimicrob. Agents Chemother. 39:180); 계면활성제 단백질 A 수용체 (Briscoe et al., (1995) Am. J. Physiol. 1233:134); p 120 (Schreier et al., (1994) J. Biol. Chem. 269:9090)을 포함하며; 또한 문헌 [K. Keinanen; M. L. Laukkanen (1994) FEBS Lett. 346:123; J. J. Killion; I. J. Fidler (1994) Immunomethods 4:273]을 참조한다.
본 개시내용은 면역접합체를 포함하는 제약 조성물을 이를 필요로 하는 대상체에게 단독으로 또는 다른 요법과 조합하여 투여하기 위한 프로토콜을 제공한다. 조합 요법 (예를 들어, 예방제 또는 치료제)은 대상체에게 병용으로 또는 순차적으로 투여될 수 있다. 조합 요법의 요법 (예를 들어, 예방제 또는 치료제)은 또한 주기적으로 투여될 수 있다. 주기 요법은 소정의 기간 동안의 제1 요법 (예를 들어, 제1 예방제 또는 치료제)의 투여, 이어서 소정의 기간 동안의 제2 요법 (예를 들어, 제2 예방제 또는 치료제)의 투여, 및 상기 순차적인 투여의 반복, 즉 요법 (예를 들어, 작용제) 중 하나에 대한 저항성의 발생을 감소시키고/거나, 요법 (예를 들어, 작용제) 중 하나의 부작용을 피하거나는 감소시키고/거나, 요법의 효능을 개선시키기 위한 주기를 수반한다.
본 개시내용의 조합 요법의 요법 (예를 들어, 예방제 또는 치료제)은 대상체에게 공동으로 투여될 수 있다.
용어 "공동으로"는 요법 (예를 들어, 예방제 또는 치료제)을 정확히 동시에 투여하는 것에 제한되지는 않으며, 오히려 항체 또는 그의 단편을 포함하는 제약 조성물이 대상체에게 순서대로, 및 면역접합체가 다른 요법(들)과 함께 이들이 달리 투여된 경우보다 증가된 이익을 제공하도록 작용할 수 있는 시간 간격으로 투여되는 것을 의미한다. 예를 들어, 각각의 요법은 동시에 또는 상이한 시점에 임의의 순서로 순차적으로 대상체에게 투여될 수 있지만; 동시에 투여되지 않는 경우에도, 이들은 목적하는 치료 또는 예방 효과를 제공하기 위해 충분히 가까운 시간 내로 투여되어야 한다. 각각의 요법은 대상체에게 임의의 적절한 형태로 및 임의의 적합한 경로에 의해 개별적으로 투여될 수 있다. 다양한 측면에서, 요법 (예를 들어, 예방제 또는 치료제)은 대상체에게 15분 미만, 30분 미만, 1시간 미만의 간격, 약 1시간 간격, 약 1시간 내지 약 2시간 간격, 약 2시간 내지 약 3시간 간격, 약 3시간 내지 약 4시간 간격, 약 4시간 내지 약 5시간 간격, 약 5시간 내지 약 6시간 간격, 약 6시간 내지 약 7시간 간격, 약 7시간 내지 약 8시간 간격, 약 8시간 내지 약 9시간 간격, 약 9시간 내지 약 10시간 간격, 약 10시간 내지 약 11시간 간격, 약 11시간 내지 약 12시간 간격, 24시간 간격, 48시간 간격, 72시간 간격, 또는 1주 간격으로 투여된다. 다른 측면에서, 2종 이상의 요법 (예를 들어, 예방제 또는 치료제)은 동일한 환자 방문에서 투여될 수 있다.
조합 요법의 예방제 또는 치료제는 대상체에게 동일한 제약 조성물로 투여될 수 있다. 대안적으로, 조합 요법의 예방제 또는 치료제는 대상체에게 개별 제약 조성물로 공동으로 투여될 수 있다. 예방제 또는 치료제는 대상체에게 동일한 또는 상이한 투여 경로에 의해 투여될 수 있다.
실시예
실시예 1: 파지-디스플레이 기술에 의한 CDH6 항체의 확인
HuCAL 플라티눔(PLATINUM)® 패닝
항체를 CDH6-ECD에 결합한 클론의 선택에 의해 확인하였다. 항체 변이체의 공급원으로서 상업적으로 입수가능한 파지 디스플레이 라이브러리인 모르포시스(Morphosys) HuCAL 플라티눔® 라이브러리를 사용하였다. 파지미드 라이브러리는 HuCAL® 개념을 기반으로 하고 (Knappik et al., J Mol Biol. 2000: 296(1):57-86; Prassler et al., J Mol Biol. 2011: 413(1):261-78 2011), Fab를 파지 표면 상에 디스플레이하기 위한 시스디스플레이(CysDisplay)™ 기술을 사용한다 (Rothe et al., J Mol Biol. 2008: 376(4):1182-200). 항-CDH6 항체의 단리를 위해, 고체 상 및 용액 패닝 접근법을 사용하여 표준 패닝 전략을 수행하였다.
항원 (재조합 단백질 및 세포주) 및 패닝과 스크리닝에 사용된 항체 시약의 개관
CDH6 항원의 다양한 형태에 대해 패닝을 수행하였다. 패닝 및 후속 스크리닝과 특징화를 위한 항원으로 사용된 재조합 CDH6 단백질 및 CDH6-발현 세포주의 개관이 표 1에 제공되어 있다.
인간, 마우스 및 래트 CDH6 세포외 도메인은 진뱅크(GenBank) 또는 유니프롯(Uniprot) 데이터베이스로부터의 아미노산 서열을 기반으로 하여 합성된 유전자였다 (표 1 참조). 시노몰구스 CDH6 cDNA 주형은 다양한 시노 조직으로부터의 mRNA를 사용하여 생성된 아미노산 서열 및 상동성 정보를 기반으로 하여 합성된 유전자였다 (예를 들어 지아젠 래보러토리즈(Zyagen Laboratories), 캘리포니아주 샌디에고). 모든 합성된 DNA 단편을, 정제를 가능하게 하는 친화성 태그가 열거되어 있는 적절한 발현 벡터 또는 포유동물 세포 표면 발현을 위한 지아젠 래보러토리즈 발현 벡터 내로 클로닝하였다.
인간 CDH6을 외인성 발현하는 안정한 CHO 세포주의 생성을 위해 TREX 발현 시스템 (인비트로젠, 캘리포니아주 칼스배드)을 제조업체의 지침에 따라 사용하였다. 간략하게, CHO-TREx 세포 (인비트로젠, R718-07, 캘리포니아주 칼스배드)를 10% FBS (인비트로젠, 10082-147, 캘리포니아주 칼스배드) 및 10 μg/ml 블라스티시딘 (인비트로젠 A11139-02, 캘리포니아주 칼스배드)이 함유된 DMEM 배지 (인비트로젠 11995-085)에서 성장시켰다. 6 웰 플레이트에서 세포가 90% 전면생장률에 도달하였을 때 형질감염을 수행하였다. 선형화된 CDH6 플라스미드 DNA 4 μg을 멸균 에펜도르프 튜브에서 100 μl DMEM 배지 (FBS 부재)와 혼합하고, 리포펙타민(Lipofectamine)® 2000 (인비트로젠 11668-019, 캘리포니아주 칼스배드) 12 μl를 또 다른 멸균 에펜도르프 튜브에서 100 μl DMEM 배지와 혼합하였다. DNA 및 리포펙타민®을 함께 혼합하고, 15분 동안 실온에서 인큐베이션하였다. CHO-TREx 세포의 배양 배지를 각 웰에 대해 FBS가 없는 1 ml DMEM 배지로 교체하고; 상기 DNA/리포펙타민® 혼합물의 절반을 각 웰에 첨가하고, 2시간 동안 인큐베이션하였다. 이어서, 2 ml 성장 배지 (FBS 존재)를 각 웰에 첨가하고, 세포를 밤새 인큐베이션하였다. 형질감염된 세포를 다음 날 6 웰 플레이트로부터 T175 플라스크로 분할하고, 10% FBS, 10 μg/ml 블라스티시딘, 800-1000 μg/ml 제네티신 (인비트로젠 10131-027)이 함유된 선택 DMEM 배지에서 성장시켰다. 선택 배지에서 20-24시간 동안 최종 1 μg/ml 테트라시클린에 의해 CDH6 발현을 유도하였다. 양성 세포를 최종 5 μg/ml 항-CDH6 1차 모노클로날 항체 (미네소타주 미네아폴리스 소재 알앤디 시스템즈(R&D Systems)로부터의 MAB2715)로 표지하고, 이어서 PE 접합된 항-마우스 2차 항체 (cat #12-4010-87, 이바이오사이언스(eBioscience), 캘리포니아주 샌디에고)로 표지하고, FACS에 의해 분류하였다.
마우스, 래트 및 시노몰구스 기원으로부터의 CDH6의 외인성 발현을 특색으로 하는 안정한 CHO 세포주를, 각각의 cDNA가 포유동물 발현 벡터 (pcDNA6.1, 인비트로젠, 캘리포니아주 칼스배드) 내로 클로닝된 CHO-K1 세포 (인비트로젠, 캘리포니아주 칼스배드)의 형질감염에 의해 생성하였다. 6 웰 플레이트에서 세포가 90% 전면생장률에 도달하였을 때 형질감염을 수행하였다. 선형화된 CDH6 플라스미드 DNA 4 μg을 멸균 에펜도르프 튜브에서 FBS 없이 100 μl DMEM 배지와 혼합하고, 리포펙타민® 2000 (인비트로젠 11668-019, 캘리포니아주 칼스배드) 12 μl를 또 다른 멸균 에펜도르프 튜브에서 100 μl DMEM 배지와 혼합하였다. DNA 및 리포펙타민® 용액을 함께 혼합하고, 15분 동안 실온에서 인큐베이션하였다. CHO 세포의 배양 배지를 각 웰에 대해 FBS가 없는 1 ml DMEM 배지로 교체하고; 상기 DNA/리포펙타민® 혼합물의 절반을 각 웰에 첨가하고, 2시간 동안 인큐베이션하였다. 이어서, 2 ml 성장 배지 (FBS 존재)를 각 웰에 첨가하고, 세포를 밤새 인큐베이션하였다. 형질감염된 세포를 다음 날 6 웰 플레이트로부터 T175 플라스크로 분할하고, 10% FBS, 10 μg/ml 블라스티시딘, 800-1000 μg/ml 제네티신 (인비트로젠 10131-027 캘리포니아주 칼스배드)이 함유된 선택 DMEM 배지에서 성장시켰다. 선택 배지에서 20-24시간 동안 최종 1 μg/ml 테트라시클린에 의해 CDH6 발현을 유도하였다. 양성 세포를 최종 5 μg/ml 항-CDH6 1차 모노클로날 항체 (미네소타주 미네아폴리스 소재 알앤디 시스템즈로부터의 MAB2715)로 표지하고, 이어서 PE 접합된 항-마우스 2차 항체 (cat #12-4010-87, 이바이오사이언스, 캘리포니아주 샌디에고)로 표지하고, FACS에 의해 분류하였다. 대안적인 상업적으로 입수가능한 항-CDH6 항체는 2B6 항체 (#GWB-E8FDF3 - 젠웨이(Genway), 캘리포니아주 샌디에고)이다. 이러한 항체를 또한 세포에서의 CDH6 발현을 확인하는데 사용하였다.
<표 1>
패닝 및 스크리닝을 위한 항원, 항체 및 세포주 시약의 개관
재조합 단백질 발현 및 정제:
His6-SUMO-CDH6 aa54-260-APP-Avi
단백질을 2xYT + 50 μg/ml 카나마이신 및 30 μg/ml 클로람페니콜을 사용하여 pET 벡터로 로제타(Rosetta)® 2 De3 pLysS 세포 (밀리포어, 매사추세츠주 빌러리카)에서 발현시켰다. 세포를 30℃에서 0.1 mM IPTG에 의해 유도하였다. 펠릿을 400 ml 20mM 트리스-Cl, pH 8.0, 150 mM NaCl, 3mM CaCl2, 20 mM 이미다졸 중에 재현탁시키고, + PI 탭 (50 ml당 1개) + 1mg/ml 리소자임을 새로 첨가하고, 덩어리를 부수기 위해 균질화하고, 미세유동화기에 3회 통과시켰다. 30분 동안 20K x g에서 원심분리 후에 상청액의 가용성 분획을 2 ml Ni-NTA (퀴아젠 슈퍼플로우 수지, 퀴아젠, 네덜란드 벤로) 상에서의 정제에 사용하였다. 용해물을 2.0 ml/분으로 칼럼 상에 통과시키고, 10 CV (칼럼 부피) 20mM 트리스-Cl, pH 8.0, 150 mM NaCl, 3mM CaCl2, 20 mM 이미다졸로 세척하고, 단백질을 20mM 트리스-Cl, pH 8.0, 500 mM NaCl, 3mM CaCl2, 250 mM 이미다졸을 사용하여 4 x 3.0 ml 용리 분획으로 용리시켰다. 분획을 풀링하였다. SUMO 프로테아제 (인비트로젠) 200 유닛에 ~15ml 단백질 (~20mg)을 첨가하고, 10K 컷오프를 갖는 스네이크스킨(Snakeskin)® 주름형 튜빙 (써모 사이언티픽(Thermo Scientific), 일리노이주 록포드)을 사용하여 4℃에서 밤새 20 mM 트리스-Cl, pH 8.0, 150 mM NaCl, 3 mM CaCl2 5L 내로 투석하였다. 투석된 물질을 수집하고, 2.0 ml Ni-NTA 수지 상에서 재정제하여 비절단 단백질 및 SUMO 프로테아제를 제거하였다. 절단된 단백질을 통과물로부터 수집하고, 분획을 세척하였다. 역 Ni-NTA로부터의 통과물 및 세척물을 5 ml 부피로 농축시키고, NaCl을 최종 농도 500mM로 첨가하고, S200 칼럼, 완충제 = 20mM 트리스-Cl, pH 8.0, 150 mM NaCl, 3mM CaCl2 상에 주입하였다.
hCDH6aa54-615 APPavi, hCDH6aa54-615 Fc, 시노CDH6 FL, 마우스CDH6 FL, 래트CDH6 FL
단백질을 이전에 현탁 배양에 적합화시킨 HEK293 (ATCC CRL-1573) 유래된 세포주에서 발현시키고, 노파르티스(Novartis) 독점적 무혈청 배지에서 성장시켰다. 소규모 발현 검증은 리포펙션을 기반으로 하여 일시적 6-웰-플레이트 형질감염 검정에서 수행하였다. 일시적 형질감염을 통한 대규모 단백질 생산은 웨이브(Wave)™ 생물반응기 시스템 (지이 헬스케어, 펜실베니아주 피츠버그)에서 10-20 L 규모로 수행하였다. DNA 폴리에틸렌이민 (폴리사이언시스(Polysciences), 펜실베니아주 워링톤)을 플라스미드 담체로서 1:3 (w:w)의 비로 사용하였다. 세포 배양 상청액을 형질감염 7-10일 후에 수거하고, 정제 전에 교차-유동 여과 및 투석여과에 의해 농축시켰다.
정제를 위해, 일시적 형질감염으로부터의 정화 및 여과된 조건화 상청액을 20 ml 친화성 칼럼 상에 5ml/분으로 통과시켰다. 20 CV PBS, 1% TX100, 0.3% t-n-부틸포스페이트 완충제, 20CV PBS로 세척하였다. 100 mM 시트르산나트륨, 150 mM NaCl pH3.0을 사용하여 용리시키고, 단백질을 2 ml 분획으로 수집하였다. 풀링된 분획을 중화시키고, 이어서 5L 50 mM 트리스, 150 mM NaCl pH 7.4, 3 mM CaCl2를 사용하여 4℃에서 밤새 투석하였다. 이어서, 샘플을 투석 완충제로 평형화시킨 슈퍼덱스® S200 16/60 칼럼 (지이 헬스케어, 펜실베니아주 피츠버그) 상에서의 크기 배제 크로마토그래피에 적용하였다.
다양한 패닝 전략의 개관이 표 2에 나타나 있다.
<표 2>
패닝 전략의 개관
단백질의 비오티닐화
Avi-태그부착된 단백질을, Avi-태그부착된 단백질 상의 단백질 비오티닐화를 위해 제조업체 (아비디티(Avidity), 콜로라도주 오로라) 표준 프로토콜에 따라 BirA 효소를 사용하여 비오티닐화하였다. 비오티닐화 후에 단백질을 50 mM 트리스, 150 mM NaCl pH 7.4, 3 mM CaCl2 내로의 슈퍼덱스® S200 16/60 칼럼 상에서의 크기 배제 크로마토그래피에 적용하였다.
비-Avi-태그부착 단백질을 비오티닐화 전에 슬라이드-에이-라이저(Slide-A-Lyzer) 소형 투석 장치, 10000 MWCO (#69576 써모 사이언티픽, 일리노이주 록포드)를 사용하여 실온에서 교반 하에 1x PBS 5L에 대하여 2시간 동안 2회 투석하였다. EZ-연결 술포-NHS-LC-비오틴 (#21327 써모 사이언티픽, 일리노이주 록포드)을 제조업체의 프로토콜에 따라 재구성하였다. 단백질을 실온에서 30분 동안 20배 몰 과량의 비오틴 시약과 함께 인큐베이션하였다. 비-반응 비오틴 시약을 제거하기 위해 샘플을 다시 실온에서 교반 하에 1x PBS 5L에 대하여 2시간 동안 2회 투석하였다. 단백질 농도를 나노드롭(Nanodrop)® 장치 (써모 사이언티픽, 일리노이주 록포드)에 의해 결정하였다. 샘플을 사용시까지 4℃에서 보관하였다.
고체 상 패닝
고체 상 패닝에서 Fab 단편 디스플레이 파지를 직접 고정화에 의해 표면 지지체에 결합된 항원과 함께 인큐베이션하였다.
항원 선택 과정 전에, 코팅 체크 ELISA를 수행하여 최적 항원 코팅 농도를 결정하였다. 이러한 목적을 위해 24 내지 0.19 μg/mL에 걸친 hCDH6aa54-615 APPavi의 2배 희석물 시리즈 (표 1 참조)를 생성하였다. 개별 희석물을 사용하여 직접 고정화를 통해 96-웰 맥시소르프(Maxisorp)™ 플레이트 (#442404 눈크(Nunc), 뉴욕주 로체스터) 상의 웰을 코팅하였다. 코팅 후에 플레이트를 300 μL PBS로 3회 세척하고, 이후 실온에서 2시간 동안 1x 차단 완충제 (2.5% 밀크, 2.5% BSA, 0.05% 트윈 20)에 의해 차단시켰다.
결합된 항원을 검출하기 위해, 항-APP 항체 (사내 생성됨)를 1 μM, 0.18 μM 또는 0.04 μM에 첨가하였다. 2차 검출은 1:5000 희석물로의 AP-표지된 항-마우스 IgG-F(ab)2 특이적 항체 (Ref. 115-056-006 잭슨-이뮤노리서치(Jackson-Immunoresearch), 펜실베니아주 웨스트 그로브)를 사용하여 수행하였다. 신호 포화가 관찰된 항원 농도를 SP 패닝을 위한 코팅 농도로서 선택하였다.
팬코드 1038.1-8
SP 패닝을 위해 1038.1-8 (표 2 참조) hCDH6aa54-615 APPavi (표 1 참조, 서열식별번호: 4)를 직접 고정화를 통해 96-웰 맥시소르프™ 플레이트 (눈크) 상에 코팅하였다. 2개의 웰을 파지 서브풀 조합당 300 μL 항원 용액 (PBS 중 10 μg/mL)으로 코팅하였다. 코팅을 1 mM CaCl2의 존재 하에 (패닝 1038.1-4) 또는 부재 하에 (패닝 1038.4-8) 밤새 4℃에서 수행하였다. 이후, 웰을 400 μL PBS로 2회 세척하고, 400 rpm으로 실온에서 2시간 동안 400 μL 차단 완충제에 의해 차단시켰다. 차단 후에 세포를 400 μL PBS로 2회 세척하였다.
결합 전에 파지 서브풀을 차단시키고, 실온에서 1시간 동안 10 μg/mL로 비관련 FLT2-APPavi를 사용하여 고갈시켜 비특이적 태그 결합제를 제거하였다. 차단 및 고갈된 파지 풀 300 μL를 코팅된 웰마다 옮기고, 실온에서 2시간 동안 400 rpm으로 인큐베이션하였다. 비-특이적 결합된 파지를 하기 표 3에 열거된 세척 단계에 의해 제거하였다.
<표 3>
특이적으로 결합된 파지를 실온에서 10분 동안 300 μL 용리 완충제로 용리시켰다. 용리된 파지를 사용하여 이. 콜라이 TG1F+ (스트라타진(Stratagene)/애질런트(Agilent), 캘리포니아주 산타 클라라) 14 mL를 OD600= 0.6-0.8의 광학 밀도로 재감염시켰다. 이. 콜라이 TG1F+ 및 파지 용리액의 혼합물을 파지 감염을 위해 37℃에서의 수조에서 45분 동안 인큐베이션하였다. 박테리아 펠릿을 2xYT 배지 중에 재현탁시키고, 34 μg/mL 클로람페니콜이 보충된 LB 한천 플레이트 상에 플레이팅하고, 37℃에서 밤새 인큐베이션하였다. 콜로니를 플레이트에서 긁어내고, 풍부화된 클론의 폴리클로날 증폭 및 파지 생산에 사용하였다. 정제된 파지를 사용하여 다음 패닝 라운드를 시작하였다. 패닝의 제2 라운드는 보다 엄격한 세척 조건을 제외하고는 제1 라운드의 프로토콜에 따라 수행하였다. 제3 패닝 라운드는 ~104 cfu/mL의 낮은 제2 라운드 산출 역가로 인해 생략하였다.
팬코드 1038.9-16
SP 패닝 1038.9-16 (표 2)에 대해, His6-SUMO-CDH6 aa54-260-APP-Avi (표 1 참조)를 팬코드 1038.1-8에 대해 기재된 바와 같이 직접 고정화를 통해 96-웰 맥시소르프™ 플레이트 (눈크) 상에 코팅하였다. 패닝 1038.13-16에 대해, 도구 항체 MAB2715의 100 nM 용액 300 μL를 웰마다 첨가하고, 실온에서 1시간 동안 인큐베이션하여 MAB2715와 동일한 에피토프를 인식하는 결합제의 선택을 피하였다. 항원 코팅 및 제3 패닝 라운드 이외에 패닝을 팬코드 1038.1-8에 대해 기재된 바와 같이 수행하였다.
팬코드 1038.26-28
SP 패닝 1038.26-28 (표 2 참조)에 대해, hCDH6aa54-615-Fc (표 1 참조, 서열식별번호: 6)를 팬코드 1038.1-8에 대해 기재된 바와 같이 직접 고정화를 통해 96-웰 맥시소르프™ 플레이트 (눈크) 상에 코팅하였다. 항원 코팅 및 세척 엄격도 이외에 패닝을 팬코드 1038.1-8에 대해 기재된 바와 같이 수행하였다.
액체 상 패닝
용액 패닝 동안, Fab 단편 디스플레이 파지 및 비오티닐화 항원을 파지에 의한 항원의 접근가능성을 증가시킬 것으로 예상되는 용액 중에서 인큐베이션하였다. 항원 비오티닐화를 상기 기재된 바와 같이 수행하였다.
팬코드 1038.17-22
각각의 파지 라이브러리 서브풀을 터닝 휠 상에서 실온에서 2시간 동안 동등 부피의 2x 케미블로커에 의해 차단시켰다. APPavi 및 비오틴 태그에 대한 항체의 선택을 피하기 위해, 비관련 FLT3-APPAvi 및 비오티닐화 항-시클로스포린 msIgG를 차단 단계에서 각각 7.5 및 18 μg/mL로 첨가하였다. 스트렙타비딘-비드에 결합하는 파지 입자의 제거를 위해, 차단된 스트렙타비딘 비드 (# 112.06D 인비트로젠, 캘리포니아주 칼스배드)를 사용하여 차단된 파지 입자의 예비-흡착을 수행하였다. 차단 및 예비-흡착된 파지를 터닝 휠 상에서 실온에서 1시간 동안 비오티닐화 hCDH6aa54-615 APPavi (표 1 참조, 서열식별번호: 4)와 함께 인큐베이션하였다. 파지-항원 복합체를 세척된 비드로 포획하거나 (팬코드 1038.17-19) 또는 비-2B6 에피토프에 대한 결합제를 풍부화시키기 위해 비오티닐화 항-CDH6 항체 2B6 (젠웨이, 캘리포니아주 샌디에고)으로 예비코팅된 비드로 포획하였다 (팬코드 1038.20-22). 비-특이적 파지 입자를 여러 세척 단계에 의해 제거하였다 (하기 참조). 특이적으로 결합된 파지 입자의 용리, 감염 및 증폭을 팬코드 1038.1-8에 대해 기재된 바와 같이 수행하였다.
<표 4>
팬코드 1038.17-25에 대한 세척 조건
팬코드 1038.23-25
패닝은 항원으로서 비오티닐화 hCDH6aa54-615-Fc (표 1 참조, 서열식별번호: 6)를 사용한다는 것을 제외하고는 팬코드 1038.1-8에 대해 기재된 바와 같이 수행하였다.
서브클로닝
FAB-FH 발현을 위한 전환
가용성 Fab 단편의 신속한 발현을 용이하게 하기 위해, 선택된 HuCAL® 플라티눔 파지 입자의 Fab 코딩 삽입물을 pMORPH®30 디스플레이 벡터로부터 pMORPH®11_FH 발현 벡터 내로 서브클로닝하였다. 최종 패닝 라운드 산출 파지로 감염된 이. 콜라이를 함유하는 글리세롤 스톡을 사용하여, pMORPH®30 DNA 정제를 위한 배양물을 뉴클레오본드(Nucleobond)® 엑스트라 미디 플러스 키트를 사용하여 제조업체 매뉴얼 (#740.412.50 마슈레이 나겔(Macherey nagel), 펜실베니아주 베들레헴)에 따라 접종하였다. 각각의 pMORPH®30 DNA 산출 풀 5 μg을 EcoRI/XbaI/BmtI를 통해 삼중 소화시켰다 (모든 제한 효소는 매사추세츠주 입스위치 소재 뉴잉글랜드 바이오랩스(New England Biolabs)로부터 구입하였음). 생성된 EcoRI/XbaI Fab 코딩 1485 bp 단편을 위자드(Wizard) SV 겔/PCR 클린-업 키트를 사용하여 제조업체 매뉴얼 (A9282, 프로메가(Promega), 위스콘신주 매디슨)에 따라 겔 정제하고, EcoRI/XbaI 컷 pMORPH®11_FH 벡터 백본 내로 라이게이션하였다.
IgG 발현을 위한 전환
전장 IgG를 발현시키기 위해, VH 및 VL의 가변 도메인 단편을 RapCLONE® 프로토콜 (모르포시스, 독일 마르틴스리드/플라네그)에 따라 Fab 발현 벡터로부터 pMORPH®4_hIgG1f 벡터 내로 서브클로닝하였다. RapCLONE®은 다량의 Fab 발현 벡터에서 IgG 발현 벡터로의 배치 전환을 위한 2-단계 클로닝 방법이다. 제1 클로닝 단계에서, 진핵 발현 카세트를 BsiWI/MfeI (κ 풀의 경우) 또는 HpaI/MfeI (λ 풀의 경우) 소화 및 이후 라이게이션을 통해 pMORPH®11 발현 벡터 내로 도입하였다. 이에 이어서 제2 클로닝 단계에서, 발현 카세트를 함유하는 Fab 풀을 EcoRV/BlpI (κ 및 λ 풀 둘 다)를 사용하여 소화시키고, 이후 포유동물 세포에서의 발현을 위해 pMORPH®4_IgG1f 수용자 벡터 내로 클로닝하였다.
Fab 단편의 발현 및 정제
이. 콜라이 TG1F의 형질전환
전기천공-적격 이. 콜라이 TG1F- 분취물 50 μL를 얼음 상에서 해동시키고, 50 pg/μl pMORPH®11_FH 플라스미드 DNA와 혼합하고, 예비-냉각된 전기천공 큐벳 내로 옮겼다. 세포를 전기천공하고 (바이오-라드 진 펄서; 설정: 1.75 kV, 200 Ω 및 25 μF (바이오-라드, 캘리포니아주 허큘레스)), 950 μl 예비-가온된 SOB 배지 내로 옮기고, 220 rpm으로 진탕하면서 37℃에서 1시간 동안 인큐베이션하였다. 적절한 부피의 형질전환 샘플을 34 μg/mL 클로람페니콜이 보충된 LB 한천 플레이트 상에 플레이팅하고, 37℃에서 밤새 인큐베이션하여 단일 콜로니를 수득하였다.
마스터 플레이트의 생성
클로람페니콜 저항성 단일 클론을 34 μg/ml의 클로람페니콜 및 1% 글루코스가 보충된 60 μl 2xYT 배지로 예비-충전된 멸균 384-웰 마이크로타이터 플레이트 (눈크)의 웰 내로 골라넣고, 37℃에서 밤새 성장시켰다. 다음날 아침에, 60% 글리세롤 및 1% 글루코스를 함유하는 20 μl 멸균 2xYT 배지를 마스터 플레이트의 각 웰 내에 첨가하였다. 플레이트를 알루미늄 호일로 밀봉하고, -8℃에서 보관하였다.
ELISA 스크리닝을 위한 Fab-함유 박테리아 용해물의 생성
마스터 플레이트의 각 웰의 5 μl를 34 μg/ml의 클로람페니콜 및 0.1 % 글루코스가 보충된 웰당 40 μl 2xYT 배지로 예비-충전된 멸균 384-웰 마이크로타이터 플레이트로 옮겼다. 플레이트를 배양물이 다소 혼탁해질 때까지 480 rpm 및 80% 습도에서 37℃에서 인큐베이션하였다. 34 μg/ml 클로람페니콜 및 5 mM IPTG가 보충된 2xYT 배지 10 μl를 Fab 단편 발현의 유도를 위해 웰마다 첨가하였다. 플레이트를 기체-투과성 테이프로 밀봉하고, 500 rpm 및 80% 습도에서 22℃에서 밤새 인큐베이션하였다. 다음 날 15 μl BEL 용해 완충제 (2.5 mg/ml 리소자임 (#10837059001, 로슈(Roche), 뉴저지주 너틀리), 4 mM EDTA, 10 U/μl 벤조나제 (# 1.01654.0001 머크, 뉴저지주 화이트 하우스 스테이션))를 각 웰에 첨가하고, 플레이트를 500 rpm 및 80% 습도에서 1시간 동안 인큐베이션하였다. 생성된 Fab 용해물을 웰당 15 μL의 2x 케미블로커를 첨가함으로써 차단시키고, 이어서 22℃, 500 rpm 및 80% 습도에서 30분 동안 인큐베이션하였다.
FACS 스크리닝을 위한 Fab-함유 박테리아 용해물의 생성
압축 플레이트의 각 웰의 5 μl를 34 μg/ml의 클로람페니콜이 보충된 웰당 100 μl 2xYT 배지로 예비-충전된 멸균 96-웰 둥근 바닥 마이크로타이터 플레이트로 옮겼다. 플레이트를 배양물이 다소 혼탁해질 때까지 500 rpm 및 80% 습도에서 37℃에서 인큐베이션하였다. 34 μg/ml 클로람페니콜 및 5 mM IPTG가 보충된 2xYT 배지 10 μl를 Fab 단편 발현의 유도를 위해 웰마다 첨가하였다. 플레이트를 기체-투과성 테이프로 밀봉하고, 500 rpm 및 80% 습도에서 22℃에서 밤새 인큐베이션하였다. 다음 날 플레이트를 1200g에서 10분 동안 원심분리하고, 상청액을 폐기하였다. 박테리아 펠릿을 용해 단계를 용이하게 하기 위해 -20℃에서 밤새 동결시켰다. 해동된 펠릿을 BEL 용해 완충제 200 μL 중에 재현탁시키고 (상기 참조), 22℃ 및 250 rpm에서 1시간 동안 인큐베이션하였다. 생성된 Fab 용해물을 1200 g에 10분 동안 원심분리하여 세포 파편을 제거하였다. Fab 함유 상청액을 스크리닝 목적에 사용하였다.
이. 콜라이에서의 His6-태그부착된 Fab 단편의 발현 및 정제
pMORPH®11 Fab FH DNA를 함유하는 이. 콜라이 TG1F- 형질전환체를 34 μg/mL 클로람페니콜 및 1% 글루코스가 보충된 2xYT 상에서 선발하였다. 개별 클론을 고르고, 34 μg/mL 클로람페니콜 및 1% 글루코스가 보충된 3 mL 2xYT 시드 배양물 내로 옮기고, 37℃, 220 rpm에서 4시간 동안 인큐베이션하였다. 시드 배양물을 사용하여, 50 mL 본배양물에 34 μg/mL 클로람페니콜 및 1% 글루코스가 보충된 2xYT를 접종하고, OD600이 0.6의 값에 도달할 때까지 30℃에서 250 mL 진탕 플라스크에서 인큐베이션하였다. Fab 발현은 IPTG를 최종 농도 0.75 mM로 첨가함으로써 유도하고, 배양물을 25℃ 및 220 rpm에서 밤새 추가로 인큐베이션하였다. 다음 날 세포를 수거하고, 세포 펠릿을 -20℃에서 밤새 동결시켰다. 세포 펠릿을 요동 테이블 상에서 실온에서 1시간 동안 용해 완충제 (25 mM 트리스/pH=8, 500 mM NaCl, 2 mM MgCl2, 10 U/μl 벤조나제 (# 1.01654.0001 머크), 0.1% 리소자임 (# 10837059001, 로슈), EDTA 1 정제 부재 완전 프로테아제 억제제/완충제 50 mL (#11873580001 로슈)) 중에 재현탁시키고 인큐베이션함으로써 파괴하였다. 용해물을 15000 g에서의 30분 동안 원심분리 및 200 nm 세공 크기의 필터를 통한 상청액의 여과에 의해 정화시켰다. His6-태그부착된 Fab 단편을 고정화된 금속 이온 친화성 크로마토그래피 (Ni-NTA 슈퍼플로우(Superflow)® 비드, #30430 퀴아젠, 네덜란드 벤로)를 통해 단리하고, 이미다졸을 사용하여 용리시켰다. 1x PBS로의 완충제 교환을 PD-10 칼럼 (# 17-0851-01 지이 헬스케어, 펜실베니아주 피츠버그)을 사용하여 수행하였다. 샘플을 멸균-여과하고, 단백질 농도를 UV-분광광도측정법에 의해 결정하였다. 샘플의 순도를 변성, 환원 15% SDS-PAGE에서 분석하였다. 샘플의 정체를 MS에 의해 확인하였다.
IgG의 발현 및 정제
스크리닝 규모에서의 IgG의 발현
진핵 HEK293c18 세포 (ATCC CRL-10852)를, 특이성 및/또는 기능적 검정에서의 후속 사용을 위한 전장 IgG를 함유하는 조건화 세포 배양 상청액의 생성을 위해 96-웰 발현 시스템에서 사용하였다. 진핵 HEK293 c18 세포를 형질감염 배지 (2% L-글루타민 (#25030-024 깁코(Gibco), 뉴욕주 그랜드 아일랜드), 10% FCS (#3302 팬 바이오테크(PAN Biotech), 독일 아이덴바흐) 및 1% 페니실린/스트렙토마이신 (#15140-122 깁코, 뉴욕주 그랜드 아일랜드)이 보충된 D-MEM) 중에 재현탁시키고, 전 날 웰당 50 μl 중 대략 4x104개 세포의 밀도로 96-웰 F형-바닥 플레이트에 시딩하였다. 형질감염 마스터 믹스를 웰당 0.6 μl 리포펙타민® 2000 (#11668 인비트로젠, 캘리포니아주 칼스배드)과 25 μL 옵티-멤(Opti-MEM)® I 배지 (#31985-047 인비트로젠, 캘리포니아주 칼스배드)와의 혼합에 의해 제조하였다. 마스터 믹스를 실온에서 15분 동안 인큐베이션하였다. 새로운 플레이트 상에서 20 μL의 옵티-멤® I 배지를 300 ng DNA를 함유하는 웰에 첨가하고, 서서히 혼합하였다. 그 후에, DNA를 예비-인큐베이션된 리포펙타민® 2000과 합하고, 서서히 혼합하고, 실온에서 20분 동안 인큐베이션하였다. 이어서, 예비-인큐베이션된 리포펙타민® 2000/DNA 복합체 100μl를 세포가 함유된 플레이트의 각 웰로 옮기고, 서서히 혼합하였다. 플레이트를 일시적 발현을 위해 37℃ 및 6% CO2에서 40시간 동안 인큐베이션하였다. 배양물 상청액을 96-웰 V형-바닥 플레이트로 옮기고, 원심분리에 의해 정화시켰다. 생성된 IgG-함유 상청액은 공지된 표준물을 참조하여 IgG 단백질 농도의 평가를 위해 항-Fd 포획 ELISA에 의해 시험하고, 나중의 사용을 위해 -80℃에서 보관하였다.
항-Fd ELISA에 의한 IgG 발현 체크
스크리닝 규모 유래된 상청액에서 IgG 수준을 평가하기 위해 맥시소르프® 96-웰 플레이트 (#437111 눈크)를 PBS 중에 1:1000 희석된 50 μl/웰 Fd-단편-특이적 양 항-인간 IgG (#PC075 더 바인딩 사이트(The Binding Site), 캘리포니아주 샌디에고)로 코팅하였다. 코팅 후에, 플레이트를 TBST로 2회 세척하고, TBST 중 5% 탈지유 분말에 의해 1시간 동안 차단시켰다. 한편 IgG 함유 상청액을 TBST 중 2.5% 탈지유 분말 중에 1:50 희석하였다. TBST를 사용하여 ELISA 플레이트를 3회 세척한 후에, 희석된 상청액 100 μL를 웰마다 옮기고, 플레이트를 실온에서 1시간 동안 인큐베이션하였다. 이후, 플레이트를 TBST로 5회 세척하고, 포획된 IgG를 1시간 동안 TBST 중 0.5% 탈지유 분말 중 50 μl F(ab')2-특이적 염소 항-인간 IgG (#109-055-097 디아노바(Dianova), 독일 함부르크) (1:5000 희석됨)와 함께 인큐베이션함으로써 검출하였다. 플레이트를 TBST로 5회 세척한 후에 아토포스(AttoPhos) 형광 기질 (#1484281 로슈, 뉴저지주 너틀리)을 제조업체의 지침에 따라 첨가하고, ELISA 판독기를 사용하여 535 nm에서의 형광 방출과 430 nm에서의 여기를 기록하였다.
마이크로규모에서의 IgG의 발현 및 정제
CAP-T® 세포 (세벡 파마슈티칼스(CEVEC Pharmaceuticals), 독일 쾰른)를, 유전자 전달 비히클로서 40 kDa 선형 PEI (PEI Max (#24765-2 폴리사이언시스, 펜실베니아주 워링톤))를 사용하여 프리스타일293(FreeStyle293)® 발현 배지 (#12338 인비트로젠, 캘리포니아주 칼스배드)에서 pMORPH®4 IgG 발현 플라스미드로 일시적으로 감염시켰다.
형질감염 1일 전에, 세포를 지수 성장을 보장하기 위해 ~0.8E+06개 세포/ml로 희석하였다. 형질감염 날에 세포를 9 mL 예비-가온된 프리스타일® (인비트로젠, 캘리포니아주 칼스배드) 배지 중에 ~0.5x107개 세포/ml로 희석하고, 125ml 진탕 플라스크 내로 옮겼다. 30 μg DNA를 500 μl 옵티멤 중에 희석하였다. 1200 μg PEI Max (#24765-2 폴리사이언시스, 펜실베니아주 워링톤)를 8.80 mL 옵티멤 배지 중에 희석하였다. DNA 용액을 세포에 적가하고, 서서히 혼합하였다. 그 후, 500 μL PEI Max 용액을 세포에 첨가하고, 서서히 혼합하였다.
세포를 100 rpm에서 교반하면서 37℃, 6% CO2, 85% 습도에서 인큐베이션하였다. 4시간 후에, 4 mM L-글루타민 및 5μg/ml 블라스티시딘 (#R21001 인비트로젠, 캘리포니아주 칼스배드)이 보충된 10 mL 예비-가온된 PEM을 첨가하였다. 동시에, 400 μL 발프로산 (#P4543-25G 시그마 알드리치, 미주리주 세인트 루이스)을 최종 농도 4 mM로 첨가하였다. 세포를 100 rpm에서 교반하면서 37℃, 6% CO2, 85% 습도에서 6일 동안 인큐베이션하였다.
4℃에서 1200 g에서 5분 동안 원심분리 후에 상청액을 멸균-여과하고 (0.2 μm 세공 크기), 액체 취급 스테이션을 사용하여 단백질 A 친화성 크로마토그래피 (맙셀렉트® SURE, 지이 헬스케어, 펜실베니아주 피츠버그)에 적용하였다. 1x PBS로 완충제 교환을 수행하고, 샘플을 멸균 여과하였다 (0.2 μm 세공 크기). 단백질 농도를 UV 분광광도측정법에 의해 결정하고, IgG의 순도를 SDS-PAGE에서 변성, 환원 조건 하에 분석하였다.
Fab-함유 미가공 박테리아 용해물의 스크리닝
ELISA 스크리닝
ELISA 스크리닝을 사용하여, 단일 Fab 클론을 표적 항원에의 결합에 대해 패닝 산출물로부터 확인하였다. Fab 단편을 Fab 함유 조 이. 콜라이 용해물을 사용하여 시험하였다.
직접적으로 코팅된 항원에 대한 ELISA 스크리닝
맥시소르프® (#442404 눈크, 뉴욕주 로체스터) 384-웰 플레이트는 PBS 중 3 μg/ml의 농도에서의 huCDH6 단백질로 4℃에서 밤새 코팅하였다. 세척 후에 플레이트를 1xPBST 중 5% 탈지유에 의해 2시간 동안 차단시켰다. Fab-함유 이. 콜라이 용해물을 첨가하고, 실온에서 1시간 동안 결합되도록 하였다.
결합된 Fab 단편을 검출하기 위해 플레이트를 TBST로 5x 세척하고, AP-항 인간 IgG F(ab')2 (#109-055-097 잭슨 이뮤노리서치, 펜실베니아주 웨스트 그로브)를 1/2500 희석물로 첨가하였다. 실온에서 1시간 후에 플레이트를 TBST로 5x 세척하고, 아토포스 기질을 제조업체의 명세에 따라 첨가하였다. 플레이트를 기질 첨가 5분 후에 ELISA 판독기에서 판독하였다.
뉴트라비딘(NeutrAvidin)® 플레이트를 사용하는 비오티닐화 항원에 대한 ELISA 스크리닝
맥시소르프® (#442404 눈크, 뉴욕주 로체스터) 384-웰 플레이트를 PBS 중 10 μg/ml의 농도로의 뉴트라비딘® (#31000 써모 사이언티픽, 일리노이주 록포드)으로 4℃에서 밤새 코팅하였다. 세척 후에, 플레이트를 1x 케미블로커에 의해 2시간 동안 차단시켰다. Fab-함유 이. 콜라이 용해물을 새로운 384-웰 마이크로타이터 플레이트 내로 분배하고, 비오티닐화 CDH6 항원을 2.5 μg/mL의 농도로 첨가하였다. 이어서, Fab-항원 복합체를 뉴트라비딘® 코팅된 플레이트로 옮기고, 실온에서 1시간 동안 포획되도록 하고, 상기 기재된 바와 같이 검출하였다.
FACS 스크리닝
FACS 스크리닝에서, CDH6 항원을 발현한 세포 표면에 결합하는 Fab 단편을 패닝 산출물로부터 확인하였다. 1x105개 세포/웰을 U형 바닥 96 웰 플레이트 내로 옮기고, Fab-함유 박테리아 용해물 40 μl/웰과 혼합하였다. 플레이트를 1시간 동안 4℃에서 진탕하면서 인큐베이션하였다. 인큐베이션 후에, 100 μl/웰 빙냉 FACS 완충제 (PBS 중 3% FCS, 0.02% NaN3, 2 mM EDTA)를 첨가하고, 세포를 250 g에서 5분 동안 4℃에서 스핀 다운시키고, 180 μl/웰 빙냉 FACS 완충제로 2회 세척하였다. 각각의 세척 단계 후에, 세포를 원심분리하고, 주의깊게 재현탁시켰다. 마지막 세척 단계 후에 세포를 1/200 희석된 2차 검출 항체 (PE-접합된 염소 항-인간 IgG (#109-116-088, 잭슨 이뮤노리서치, 펜실베니아주 웨스트 그로브)) 50 μL 중에 재현탁시켰다. 4℃에서 1시간 인큐베이션 후에 세포를 다시 180 μL/웰 빙냉 FACS 완충제 중에서 2회 세척하였다. 최종적으로, 세포 펠릿을 0.4% 파라포름알데히드를 갖는 120 μl/웰 FACS 완충제 중에 재현탁시키고, HTS 플레이트 판독기가 구비된 FACS칼리버(FACSCalibur)® (비디 바이오사이언시스(BD Biosciences), 캘리포니아주 산호세)에서 분석하였다.
클론 서열분석
Fab 클론 서열분석
확인된 세포 결합 Fab 히트를 VL 및 VH 서열분석에 적용하였다. 34 μg/mL 클로람페니콜 및 1% 글루코스가 보충된 1.2 mL 2xYT에 압축 플레이트로부터 유래하는 박테리아 글리세롤 스톡 5 μL를 접종하고, 37℃ 및 500 rpm에서 밤새 96-웰 심부 웰 마이크로타이터 플레이트에서 성장시켰다. 다음 날 박테리아를 수거하고, pMORPH®11 FH 플라스미드를 96-웰 DNA 정제 키트 (뉴클레오스핀(Nucleospin)® 96 플라스미드 정제 키트 #740625.24, 마슈레이 나겔, 펜실베니아주 베들레헴)를 사용하여 제조업체 프로토콜에 따라 정제하였다. VL 및 VH를 서열분석하기 위해 각각 프라이머 M13rev (5' CAGGAAACAGCTATGAC 3' (서열식별번호: 10)) 및 HuCAL_VH (5' GATAAGCATGCGTAGGAGAAA 3 (서열식별번호: 11))를 사용하였다.
IgG 클론 서열분석
고유한 세포 결합 Fab 클론을 폴리클로날 방식으로 IgG1 포맷으로 전환시키고, 이후 VL 및 VH 서열분석에 의해 검색하였다. 100 μg/mL Amp 및 1% 글루코스가 보충된 1.5 mL 2xYT 배지에 단일 클론을 접종하고, 37℃, 450 rpm 및 80% 습도에서 밤새 96-웰 심부 웰 마이크로타이터 플레이트에서 성장시켰다. 다음 날 박테리아를 수거하고, IgG 코딩 pMORPH®4 플라스미드를 96-웰 DNA 정제 키트 (뉴클레오스핀® 96 플라스미드 정제 키트 #740625.24, 마슈레이 나겔, 펜실베니아주 베들레헴)를 사용하여 제조업체의 프로토콜에 따라 정제하였다. VL 및 VH를 서열분석하기 위해 각각 프라이머 (T7 5' TAATACGACTCACTATAGGG 3' (서열식별번호: 12)) 및 CMV HC (5' CTCTAGCGCCACCATGAAACA 3' (서열식별번호: 13))를 사용하였다.
시험관내 검정
옥텟(Octet)® QK를 사용하는 친화도 평가
동역학적 파라미터를 결정하는 것에 의한 친화도 평가를 생물-층 간섭측정법 기술을 통해 수행하였다. 모든 Fab 샘플을 스트렙타비딘 침지 및 판독 바이오센서 (#18-0009 포르테바이오(ForteBio), 캘리포니아주 멘로 파크)를 사용하여 측정하였다. 플레이트를 옥텟® QK 기기 (포르테바이오, 캘리포니아주 멘로 파크)에 배치하고, 챔버에서 27℃로 평형화되도록 하였다. 300초 동안 150 nM의 비오티닐화 CDH6 단백질을 함유하는 웰에 센서를 배치하여 실행을 개시하였다. 그 후에 센서를 250 nM Fab 샘플을 함유하는 웰에 배치하였다. Fab 회합 및 해리를, 모두 컴퓨터 제어 하에 시간에 따른 층 두께 (나노미터, nm)에서의 변화를 측정함으로써 600초 동안 각각 기록하였다. 옥텟® 사용자 소프트웨어 버전 3.0을 사용하여 데이터를 자동으로 처리하였다.
항체 세포 내재화 성향의 평가
표적 매개된 세포내이입에 의한 IgG의 세포 내재화를 하기 기재된 바와 같이 VTI 어레이스캔(ArrayScan)® HC 판독기 (써모피셔(ThermoFischer), 매사추세츠주 월섬)를 사용하여 현미경검사에 의해 평가하였다. 근원 분석 프로토콜은 스팟 검출기 V4 알고리즘 (써모사이언티픽 셀로믹스(ThermoScientific Cellomics)®, 써모 사이언티픽, 일리노이주 록포드)이었다. 간략하게, 이러한 분석 프로토콜은 면역형광 염색 후에 IgG 함유 리소솜 소포와 같은 세포내 점상 대상을 확인시켜 주는 신속하고 일반적인 스팟 분석을 제공한다. 이어서, 총 점상 형광 강도를 분석된 세포의 수에 걸쳐 평균내었다.
OVCAR3 세포 (ATCC HTB-161)를 T150 조직 배양 플라스크에서 대략 90% 전면생장률로 성장시켰다. 배지를 폐기하고, 세포를 10 mL PBS로 플러싱하였다. 세포를 37℃에서 5분 동안 4 mL 세포 해리 완충제와 함께 인큐베이션하여 떼어냈다. 떼어낸 세포를 성장 배지 중에 철저히 재현탁시키고, 50 mL 튜브로 옮겼다. 바이-셀(Vi-Cell)® 분석기 (베크만 쿨터, 캘리포니아주 쿨터) 상에서 세포를 계수한 후에, 현탁액을 성장 배지 중에 105개 세포/mL로 조정하였다. 세포 현탁액 100 μL를 투명한 바닥을 갖는 96-웰 마이크로타이터 플레이트의 웰마다 시딩하였다. 플레이트를 5% CO2에서 37℃에서 24시간 동안 인큐베이션하여 세포가 부착되고 증대되도록 하였다.
IgG 함유 세포 배양 상청액을 기재된 바와 같이 생성하고, 1:4 내지 1:128의 범위의 희석물을 포함하는 4배 희석물 시리즈로 PBS 중에 희석하였다. 각각의 샘플 희석물 100 μl를 세포 함유 마이크로타이터 플레이트의 웰마다 분배하고, IgG 내재화를 허용하기 위해 37℃에서 2시간 동안 인큐베이션하였다. 이후, 세포를 웰당 100 μl 1x 셀픽스(CellFix)® 시약 (#340181 비디 바이오사이언시스, 캘리포니아주 산호세)을 첨가함으로써 고정시켰다. 10분 후에 플레이트를 PBS로 2회 세척하고, 이어서 세포를 웰당 100 μl 0.1% 트리톤 X-100을 첨가함으로써 투과화시켰다. 10분 후에 플레이트를 PBS로 다시 2회 세척하고, 실온에서 2시간 동안 100 μl 1x 오디세이 차단 완충제 (#927-40000 리코르(LiCor), 네브라스카주 링컨)에 의해 차단시켰다. 내재화된 IgG를 검출하기 위해 훽스트(Hoechst) 핵 염색 시약 (#B2261 시그마, 미주리주 세인트 루이스)의 1:10000 희석물이 보충된 2차 항체 알렉사 플루오르® 488 염소 항-인간 IgG (#11001 몰레큘라 프로브스(Molecular Probes), 뉴욕주 그랜드 아일랜드)의 1:000 희석물 100 μl를 웰마다 첨가하고, 암소에서 실온에서 1시간 동안 인큐베이션하였다. 이어서, 세포를 최종 흡인 없이 PBS로 2회 세척하였다. 이어서, 플레이트를 셀로믹스® VTI 어레이스캔 HC 판독기 (써모피셔, 매사추세츠주 월섬) 내로 로딩하고, 분석하였다. IgG 내재화의 정도를 세포당 평균 스팟 강도 (MSAI)에 의해 평가하였다.
항-인간 FAB-DM1 접합체를 사용하는 대용 ADC 검정
수용체 결합 후에 내재화하여 세포독성 페이로드를 전달하는 CDH6 항체의 능력을 시험하기 위해, 항-인간 Fab-DM1 시약 (아피니퓨어(AffiniPure)®, SMCC-DM1이 접합된 Fab 단편 염소 항-인간 IgG H+L)을 정제된 IgG와 고정된 1:2 비로 혼합함으로써 대용 ADC 검정을 수행하였다. 이들 세포가 CDH6의 높은 발현을 보여줌에 따라 암 세포주 OVCAR3 (맥코이 + 20% FCS 중에서 배양된 난소 장액성 암종)에 대해 세포독성 잠재력을 시험하였다.
배양 중의 세포를 계수하고, 배지 중에 1x105개 세포/ml의 농도로 희석하였다. 1000개 세포/웰을 384-웰 플레이트 (코닝 코스타(Corning Costar)#3707, 코닝, 매사추세츠주 툭스베리)로 옮겼다. 1차 항체/Fab-DM1 용액은 1.4ml 매트릭스 튜브 (써모, # 3790, 일리노이주 록포드)에서 666nM Fab-DM1을 세포 배양 배지 중에 희석된 333nM 1차 인간 IgG와 합함으로써 제조하고, 30분 동안 37℃에서 인큐베이션하였다. 10-포인트, 1:3 연속 희석물을 384-웰 심부-웰 플레이트 (브랜드테크 사이언티픽 인크(Brandtech Scientific Inc) #701355, 코네티컷주 에섹스)에서 제조하고, 25μl를 검정 플레이트마다 옮겨 (삼중) 각각 66nM 및 33nM의 FAB-DM1/인간 IgG의 최고 시작 농도를 생성하였다. 대조군의 경우, 단지 세포만을 갖는 웰 (=100% 생존율 대조군) 및 단지 Fab-DM1과 함께 인큐베이션한 세포만을 갖는 웰 (2차 시약의 비특이적 사멸을 체크하기 위함)을 제조하였다. 플레이트를 37℃ 및 5% CO2에서 120시간 동안 인큐베이션하였다. 1차 항체/Fab-DM1 복합체의 세포 활성을 셀타이터-글로(CellTiter-Glo)® 시약 (#G7571 프로메가, 위스콘신주 매디슨)을 사용하여 제조업체의 지침에 따라 결정하였다. 생존율을 단지 세포만의 대조군에 대해 정규화하였다.
항체 스크리닝 개요
HuCAL® 플라티눔 파지미드 라이브러리를 인간 CDH6-ECD 항원에 대한 특이적 Fab 단편을 선택하는데 사용하였다. 재조합 인간 APPAvi 및 Fc 융합체, 뿐만 아니라 말단절단된 도메인 APPAvi 융합 단백질을 패닝에 사용하였다. HuCAL® 플라티눔 항체-파지 입자를 상이한 서브풀 조합으로 총 8개의 상이한 패닝 전략에 적용하여 28개 패닝 산출 풀이 생성되었다. 요약하면, 8개의 전략 중 6개가 생산적이었고, 771개의 ELISA 양성 스크리닝 히트가 생성되었다.
인간 CDH6을 발현하는 CHO 세포, 뿐만 아니라 OVCAR3 세포에 대한 FACS 스크리닝으로 271개의 확인된 세포 결합 히트가 생성되었다. 이들 세포 결합 히트를 통합하기 위해 VL 및 VH 서열분석을 수행하여 53개의 고유한 Fab 항체 클론의 확인이 이루어졌다.
기능적 특징화를 가능하게 하기 위해, Fab 항체 클론을 IgG 포맷으로 전환시켜, 47개의 고유한 IgG 클론을 생성하였으며, 이 중 44개는 스크리닝 규모에서 성공적으로 발현되었다. 정제된 고유한 IgG를 FACS에 의한 인간/시노/래트/마우스 교차-반응성, 옥텟에 의한 친화도 순위화, 세포 내재화 검정, 뿐만 아니라 항-인간 IgG Fab-SMCC-DM1 2차 시약을 사용하는 대용 ADC 검정을 포함한 일련의 특징화 검정에 적용하였다. 이들 검정을 기반으로 하여, 인간 IgG를 규모확대 생산, 이후 ADC 링커/페이로드에 대한 직접 접합, 및 시험관내 및 생체내 실험에서의 ADC로서의 시험을 위해 선택하였다.
추가의 심도있는 특징화를 위해 선택된 항-CDH6 IgG에 대한 서열 정보가 표 5에 제시되어 있다.
<표 5>
심도있는 특징화를 위해 선택된 IgG의 서열 정보.
실시예 2: 부위-지정 돌연변이유발에 의한 항체 조작
번역후 변형 및 배선화를 위한 잠재적 부위의 제거를 위한 부위-지정 돌연변이유발을 퀵체인지(QuikChange)® 부위-지정 돌연변이유발 키트 (애질런트 테크놀로지스(Agilent Technologies), 캘리포니아주 산타 클라라)를 사용하여 항-CDH6 항체의 하위세트에 대해 수행하였다. NOV690 서열을 D53S 및 VL (3-J-배선)로 변화시켜 항체 NOV1126이 생성되었다. NOV695 서열을 N31Q 및 S49Y로 변화시켜 항체 NOV1127이 생성되었다. NOV0720 서열을 N31Q, N95Q 및 VL (3-J-배선)로 변화시켜 항체 NOV1132가 생성되었다. 조작된 항체에 대한 서열 정보가 표 6에 기재되어 있다.
<표 6>
조작된 항체의 서열 정보
실시예 3: 인간, 시노몰구스, 래트 및 마우스 기원으로부터의 CDH6의 발현을 특색으로 하는 세포에 대한 항-CDH6 IgG 결합의 결정
상이한 종으로부터의 CDH6의 발현을 특색으로 하는 세포에 대한 항-CDH6 결합을 결정하기 위해, CDH6을 내인성으로 발현하는 OVCAR3 세포에 대해서 뿐만 아니라 인간, 시노몰구스, 래트 및 마우스 기원으로부터의 CDH6을 발현하도록 조작된 CHO 세포에 대해서도 FACS (형광 활성화 세포 분류) 분석을 수행하였다.
OVCAR3 (난소 장액성 암종)을 ATCC (#HTB-161)로부터 입수하였다. CDH6 발현을 특색으로 하는 CHO 세포주의 생성은 상기 실시예 1에 기재되어 있다.
세포 현탁액은 배양 중의 세포를 아큐타제(Accutase)® 세포 해리 시약 (#A1110501 깁코, 뉴욕주 그랜드 아일랜드)을 사용하여 제조업체의 지침에 따라 처리하고, 이어서 세포를 FACS 완충제 (PBS/1%BSA, 깁코) 중에서 세척함으로써 제조하였다. 세포를 FACS 완충제 중에 1x106개 세포/ml로 재현탁시키고, 96-웰 둥근 바닥 플레이트 (코닝 #CLS3360) 내로 100μl/웰로 분취하였다. 1차 항체의 8-포인트, 1:5 연속 희석물을 제조하여 10 μg/ml의 최고 최종 시작 농도를 생성하고, 100μl/웰을 세포 현탁액에 첨가하였다. 음성 대조군 항체로서 인간 이소형 대조군 IgG (알앤디 시스템즈 #1-001-A, 미네소타주 미네아폴리스)를 사용하였다. 세포를 얼음 상에서 30분 동안 1차 항체 용액과 함께 인큐베이션하고, 이어서 냉 FACS 완충제 200μl 중에서 3회 세척하였다. 세포를 1/500 희석물로 사용된 PE-접합된 항-인간 인간 Fc (잭슨 이뮤노 리서치 #109-116-098, 펜실베니아주 웨스트 그로브) 100μl 중에 재현탁시키고, 세포를 얼음 상에서 30분 동안 인큐베이션하였다. 200μl 냉 FACS 완충제 중에서의 3회 세척 후에, 세포를 BD FACS 칸토(Canto) II® (비디 바이오사이언시스, 캘리포니아주 산호세) 상에서 분석하였다. 샘플당 신호의 기하평균을 플루우조(FlowJo)® 소프트웨어를 사용하여 결정하였으며, EC50을 농도에 대한 신호의 기하평균 플롯팅 및 팁코 스팟파이어(Tibco Spotfire)® (팁코(Tibco), 매사추세츠주 보스톤)를 사용하는 비-선형 회귀 곡선 피팅에 의해 결정하였다. 모든 20종의 분석된 항-CDH6 항체는 OVCAR3 세포 (도 1) 및 CDH6을 발현하는 CHO 세포에 대한 용량-의존성, 표적-특이적 결합을 디스플레이하였다. CDH6을 발현하지 않는 야생형 CHO 세포에 대해서는 어떤 반응성도 관찰되지 않았다. EC50 값이 표 7에 요약되어 있다.
<표 7>
OVCAR3 세포, 및 CDH6 발현에 대해 음성인 CHO 세포 (CHO-TREX) 또는 인간, 시노몰구스, 래트 또는 마우스 CDH6을 발현하도록 조작된 CHO 세포에 대한 CDH6 IgG의 세포 결합 친화도 (EC50 [nM]).
실시예 4: 비아코어 측정에 의한, 인간, 시노몰구스, 래트 및 마우스로부터의 재조합 CDH6에 대한 결합 친화도 및 교차-반응성의 결정
인간, 시노몰구스, 래트 및 마우스 기원으로부터의 재조합 CDH6 단백질에 대한 항체의 친화도를 CM5 센서 칩이 있는 비아코어® T100 기기 (지이 헬스케어, 펜실베니아주 피츠버그) 상에서 SPR 기술을 사용하여 결정하였다.
간략하게, 0.5% 소 알부민 분획 V (7.5% 용액) (깁코 15260-037)가 보충된 HBS-P (0.01 M HEPES, pH 7.4, 0.15 M NaCl, 0.005% 계면활성제 P20)를 모든 실험을 위한 실행 완충제로서 사용하였다. 고정화 수준 및 분석물 상호작용을 반응 단위 (RU)에 의해 측정하였다. 파일럿 실험을 수행하여 항-인간 Fc 항체 (잭슨 이뮤노리서치 109-006-098, 펜실베니아주 웨스트 그로브)의 고정화 및 시험 항체의 포획의 실행가능성을 시험 및 확인하였다.
동역학적 측정을 위해, 항체를 고정화된 항-인간 Fc 항체를 통해 센서 칩 표면에 포획하고 유리 용액에서 결합하는 CDH6 단백질의 능력을 결정하는 실험을 수행하였다. 간략하게, pH 5에서의 30 μg/ml의 항-인간 Fc 항체를 CM5 센서 칩 상에서 아민 커플링을 통해 12 μl/분의 유량으로 모든 4개의 유동 셀 상에 7500 RU에 도달하도록 고정화시켰다. 이어서, 5-10μg/ml의 시험 항체를 유동 셀 2, 3 및 4에 12초 동안 10 μl/분으로 주입하였다. 이후, 0.78-50 nM의 CDH6 수용체 세포외 도메인 (ECD)을 2배 시리즈로 희석하고, 참조 (유동 셀 1) 및 시험 유동 셀 2, 3 및 4에 걸쳐 100초 동안 80 μl/분의 유량으로 주입하였다. 시험된 ECD의 표가 하기에 열거되어 있다. 결합의 해리가 10분 동안 이어졌다. 각각의 주입 사이클 후에, 칩 표면을 30초 동안 60 μl/분으로 10 mM 글리신 pH 2.0을 사용하여 재생시켰다. 모든 실험을 25℃에서 수행하고, 비아코어® T100 평가 소프트웨어 버전 2.0.3을 사용하는 단순한 1:1 상호작용 모델에 의해 반응 데이터를 전반적으로 피팅하여 회합률 (ka), 해리율 (kd) 및 친화도 (KD)의 추정치를 수득하였다. 결과는 이러한 패널에서 검정된 대부분의 CDH6 항체가 인간, 시노몰구스, 래트 및 마우스에 대해 교차-반응성이라는 것을 시사한다 (표 8).
<표 8>
비아코어 측정에 의한 인간, 시노몰구스, 래트 및 마우스로부터의 재조합 CDH6에 대한 항-CDH6 IgG의 결합 친화도.
실시예 5: 비아코어에 의한 에피토프 비닝
CDH6 항체의 에피토프 맵핑을 CM5 (s) 센서 칩이 있는 비아코어® A-100 기기 (지이 헬스케어, 펜실베니아주 피츠버그) 상에서 표면 플라즈몬 공명 (SPR) 기술을 사용하여 결정하였다. 간략하게, HBS-EP 완충제 (10 mM HEPES pH 7.4, 150 mM NaCl, 3.4 mM; 0.25% BSA 및 10mM 칼슘이 보충된 EDTA, 0.005% 계면활성제 P20)를 모든 실험을 위한 실행 완충제로서 사용하였다. 고정화 수준 및 분석물 상호작용을 반응 단위 (RU)에 의해 측정하였다. 파일럿 실험을 수행하여 항-인간 Fc 항체 (카탈로그 번호 잭슨 이뮤노리서치 109-006-098, 펜실베니아주 웨스트 그로브)의 고정화 및 시험 항체의 포획의 실행가능성을 시험 및 확인하였다.
에피토프 맵핑을 위해, 항-인간 Fc 항체를 10,000RU로 모든 4개의 유동 셀의 스팟 1,2 및 4,5 상에 고정화시켰다. 스팟 5를 참조로서 사용하였다. 이어서, 각각의 1차 시험 CDH6 항체를 300 RU보다 더 큰 반응 수준으로 바이오센서 칩 상의 고정화된 항-인간 Fc를 통해 포획하고, 이어서 20 RU 초과의 결합 수준으로 스팟 1 및 5에 CDH6 단백질을 주입하였다. 스팟 2 및 4를 추가의 참조 표면으로서 사용하였다. 각각의 유동 셀의 모든 5개 스팟을 1 mg/ml의 인간 IgG의 2회 주입에 의해 차단시켜 항-인간 Fc의 임의의 유리 결합 부위를 차단시켰다. 이어서, 각각의 2차 시험 항체를 스팟 1,2 또는 4,5에 걸쳐 주입하여 1차 시험 항체 및 CDH6 단백질의 복합체에 대한 결합도를 평가하였다. 인간 IgG 이소형 대조군을 1차 및 2차 시험 음성 대조군 항체로서 사용하였다. 항체 쌍의 모든 가능한 조합이 평가될 때까지 항체를 모든 4개의 유동 셀에서 병행하여 시험하였다. 각각의 1차 및 2차 항체 결합 사이클 후의 모든 유동 셀 표면의 재생을 10mM 글리신 pH2.0의 주입에 의해 수행하였다.
결과를 비아코어® 4000 평가 소프트웨어에서 에피토프 맵핑 모듈을 사용하여 평가하고, 행에는 1차 항체 및 열에는 2차 항체가 있는 매트릭스로서 제시하였다 (도 2). 1차 시험 항체 및 CDH6 단백질의 복합체에 대한 2차 시험 항체의 추가의 결합은 양성 반응 값에 의해 나타내어진다. 음성 쌍이 표시되고, 인간 IgG 이소형 대조군과 비교된다. 이어서, 매트릭스를 사용하여, 어느 항체가 어느 에피토프를 인식하는지를, 이들이 다른 항체와 어떻게 쌍을 형성할 수 있는지에 기반하여 맵핑한다. 항체 NOV0710 및 NOV0712는 서로 경쟁하며, 이는 이들이 항원 상의 중복 에피토프를 인식한다는 것을 나타낸다. 항체 NOV1127은 그것이 1차 및 2차 결합 항체로서 사용되었을 때 양성 결합을 나타내며, 이는 그것이 CDH6 상의 다중 부위에 결합할 수 있다는 것 또는 초기 결합이 입체형태를 변경하고 추가의 결합 부위를 개방시킨 다는 것을 시사한다. NOV0719, NOV0692, NOV1126 및 NOV1132는 모두 이러한 분석을 기반으로 하여 특유의 에피토프에 결합하는 것으로 보인다.
실시예 6: 수소-중수소 교환/질량 분광측정법에 의한 에피토프 맵핑
질량 분광측정법 (MS)과 조합된 수소-중수소 교환 (HDx) (Woods et al., J. Cell Biochem. 2001 S37: 89-98)을 사용하여, 카드헤린 도메인 (EC) 1 내지 5를 포함하는 hCDH6 전장 (aa54-615)의 전장 세포외 도메인 (ECD) 상의 항체 NOV1127, NOV0719, NOV0710 및 NOV0712의 결합 부위를 맵핑하였다 (표 9). 추가로, HDx를 또한 항체 NOV0710 및 NOV0712를 사용하여 EC 1 내지 3으로 이루어진 말단절단된 ECD에 대해 수행하였다 (표 10). HDx에서 단백질의 교환가능한 아미드 수소는 중수소에 의해 대체된다. 이러한 과정은 단백질 구조/역학 및 용매 접근가능성에 감수성이고, 따라서 리간드 결합 시에 중수소 흡수의 감소를 겪는 위치를 보고할 수 있다. 이들 실험의 목표는 잠재적 에피토프를 확인하고 본 발명자들의 치료 항체에 결합되었을 때의 hCDH6의 역학을 이해하는 것이었다. 중수소 흡수의 변화는 직접 결합 및 알로스테릭 사건 둘 다에 감수성이라는 것을 주목하는 것이 중요하며; 에피토프를 정확히 결정하기 위해서 HDx를 직교 기술 (예를 들어 X선 결정학)과 조합하여야 한다.
자동화 HDx/MS 실험을 문헌에 기재된 것과 유사한 방법을 사용하여 수행하였다 (Chalmers et al., Anal Chem. 2006: 78(4):1005-14). 리프(LEAP) 오토샘플러, 나노액퀴티(nanoACQUITY) UPLC 시스템 및 시냅트(Synapt) G2 질량 분광계를 포함하는 워터스(Waters) HDx-MS® 플랫폼 상에서 수행하였다 (워터스 코포레이션(Waters Corp), 매사추세츠주 밀포드). 전장 hCDH6(54-615)의 단백질 백본을 중수소로 표지하는데 사용된 중수소 완충제는 50 mM D-트리스, 150 mM NaCl pH 7.4 + 3 mM CaCl2였으며; 용액 중 중수소의 전체 백분율은 89.5%였다. hCDH6 항체의 부재 하의 hCDH6(54-615) 중수소 표지 실험을 위해, 600 pmol의 hCDH6(54-615) 부피 5 μl를 냉각된 튜브에서 중수소 완충제 45 μl를 사용하여 희석하고, 4℃에서 회전기기 상에서 25분 동안 인큐베이션하였다. 이어서, 표지 반응을 얼음 상에서 75 μl의 냉각된 켄칭 완충제에 의해 3분 동안 켄칭시키고, 이어서 자동화 펩신 소화 및 펩티드 분석을 위해 LC-MS 시스템 상에 주입하였다. 결합된 hCDH6 항체의 존재 하의 hCDH6(54-615) 중수소 표지 실험을 위해, 먼저 600 pmol의 hCDH6 항체를 써모 프로테인 G 플러스(Thermo Protein G Plus) 비드 상에 고정화시키고, 디숙신이미딜 수베레이트 (DSS)를 사용하여 가교시켰다. 표지 실험을 수행하기 위해, 항체 비드 (600 pmol 항체를 함유함)를 4℃에서 30분 동안 600 pmol hCDH6(54-615)과 함께 인큐베이션하였다. 30분 후에 비드를 200 μl의 트리스 완충제 (50mM 트리스, 150 mM NaCl pH 7.4 +3 mM CaCl2)에 의해 세척하였다. 이어서, 200 μl의 냉각된 중수소 완충제 (80.6% 중수소)를 첨가하고, 복합체를 4℃에서 25분 동안 인큐베이션하였다. 25분 후에, 표지 반응을 얼음 상에서 125 μl의 냉각된 켄칭 완충제에 의해 2.5분 동안 켄칭하였다. 원심분리에서 30초 동안 샘플을 스핀시킨 후에, 켄칭된 용액을 자동화 펩신 소화 및 펩티드 분석을 위해 LC-MS 시스템 상에 주입하였다. 유사한 실험을 또한, 단지 EC 3 내지 5만을 함유하는 hCDH6(267-615) 구축물에 대해 수행하였다. 이들 실험에서 완충제는 25 mM HEPES pH = 7.4, 150 mM NaCl, 3 mM CaCl2 및 94.2% 중수소를 함유하는 완충제의 중수소 버전이었다.
모든 측정을 최소 3개의 분석 삼중물을 사용하여 수행하였다. 모든 중수소 교환 실험을 0.5 M TCEP 및 3 M 우레아 (pH = 2.5)를 사용하여 켄칭시켰다. 켄칭 후에, 교환된 항원을 12℃에서 포로스자임(Poroszyme)® 고정화 펩신 칼럼 (2.1 x 30 mm)을 사용하여 온-라인 펩신 소화에 적용한 후에, 워터스 뱅가드(Waters Vanguard)® HSS T3 (워터스 코포레이션, 매사추세츠주 밀포드) 포획 칼럼 상에 포획하였다. 펩티드를 포획 칼럼으로부터 용리시키고, 워터스 CSH C18 1 x 100 mm 칼럼 (1℃에서 유지함) 상에서 2 내지 35% B의 이원 8분 구배를 사용하여 40 μl/분의 유량으로 분리하였다 (이동상 A는 99.9% 물 및 0.1% 포름산이었고; 이동상 B는 99.9% 아세토니트릴 및 0.1% 포름산이었음).
hCDH6(54-615) 단백질에 대해, 서열의 73%를 중수소 교환 실험에 의해 모니터링하였으며; 이들 펩티드의 완전한 목록은 표 9에 보여진다. 도 3은 hCDH6(54-615) 단백질의 교환 상태 특징의 개요를 제공한다. 중수소의 보다 많은 흡수를 갖는 영역, 예컨대 EC3은 보다 많이 용매 노출되거나 또는 보다 약한 수소 결합 네트워크를 가지며, 보다 적은 중수소 흡수를 나타내는 영역은, 예컨대 영역 EC4 및 EC5이다. 전체적으로, EC1 내지 EC3 도메인은 EC4 또는 EC5 도메인보다 중수소의 보다 많은 교환 상태를 갖는 것으로 관찰된다. 예를 들어, EC2 펩티드 181-199 VTATDADDPTYGNSAKVVY (서열식별번호: 336) 평균 중수소 흡수는 3.61 Da 이고; 대조적으로 EC5 펩티드 506-525 IQTLHAVDKDDPYSGHQFSF (서열식별번호: 337) 평균 중수소 흡수는 단지 1.18 Da이다. 제한된 중수소 흡수는 항체 결합 시에 -0.5 Da 이하의 이동을 겪는 보호 영역을 결정시켜 줄 수 있으며, 이는 동적 범위에서의 제한으로 인해 보다 어렵다.
EC1 (54-159)의 경우, 모든 4종의 항체의 결합 시에 중수소 흡수의 감소를 나타내는 유일한 펩티드는 61-70 FLLEEYTGSD (서열식별번호: 338)이다. EC1에서 항체 NOV1127 및 NOV0719는 구조 내 유의한 탈안정화를 0.5 Da 이상의 중수소 흡수의 변화로 입증한다. NOV0710 및 NOV0712의 결합은 EC1 도메인에서의 항원의 중수소 교환 거동을 유의하게 변경시키지 않는다.
EC2 (160-268)의 경우, 영역 195-202 AKVVYSIL (서열식별번호: 339)은 모든 항체에 의한 중수소 흡수의 경계선 감소를 갖는다. 보다 흥미롭게도, 영역 203-221 QGQPYFSVESETGIIKTAL (서열식별번호: 340)은 NOV0719에 의해 실질적으로 탈안정화되고 (즉, 중수소 흡수의 증가를 겪음), NOV0712에 의해 다소 덜 탈안정화된다. NOV1127이나 NOV0710 모두 이러한 영역을 +0.5 Da 이상의 중수소 혼입의 변화로 탈안정화시키지는 않는다. 마지막으로, 영역 225-237 DRENREQYQVVIQ (서열식별번호: 341)는 단지 hCDH6 항원에 대한 NOV0719 결합 시에만 탈안정화된다.
EC3 (269-383)의 경우, 영역 275-302 FKTPESSPPGTPIGRIKASDADVGENAE (서열식별번호: 342)는 NOV0719에 의해 탈안정화된다. 영역 303-315 IEYSITDGEGLDM (서열식별번호: 343)은 모든 항체에 의한 중수소 흡수의 감소를 나타내지만, NOV0710 및 NOV0712에 의한 감소가 다른 2종의 항체에 비해 더 현저하다. 영역 316-330 FDVITDQETQEGIIT (서열식별번호: 344)는 모든 항체에 대해 중수소 흡수의 감소를 나타내지만, NOV0719에 의한 감소가 가장 작다. 대조적으로, 영역 331-336 VKKLLD (서열식별번호: 345)는 NOV0719에 의해 탈안정화되고, NOV0712에 의해 보다 덜 탈안정화된다. 영역 337-358 FEKKKVYTLKVEASNPYVEPRF (서열식별번호: 346)는 NOV0719를 제외한 모든 항체에 의해 중수소 흡수의 감소를 나타내고; 대조적으로 NOV0719는 영역 337-345 FEKKKVYTL (서열식별번호: 347)을 탈안정화시키는 유일한 항체이다. 마지막으로, NOV0719는 또한 영역 370-376 VRIVVED (서열식별번호: 348)를 탈안정화시키는 것으로 관찰된다.
EC4 (384-486) 및 EC5 (487-608)의 경우, 항원의 중수소 교환 상태 (도 3)는 전체적으로 비교적 낮으며, 이는 중수소 흡수의 감소의 검출을 어렵게 만든다. EC4의 경우 영역 407-427 AQDPDAARNPVKYSVDRHTDM (서열식별번호: 349)은 NOV0710에 의한 중수소 흡수의 경계선 감소를 나타낸다. 임의의 4종의 항체와 결합 시에 중수소 흡수를 감소시키는 EC4에서의 다른 영역은 존재하지 않는다. 전체적으로, 이들 관찰은 EC4가 에피토프에 관여되지 않는다는 것을 강하게 시사한다. 대조적으로, 459-488 IATEINNPKQSSRVPLYIKVLDVNDNAPEF (서열식별번호: 350)는 NOV0719, NOV1127, NOV0712 및 NOV0710에 의한 다양한 탈안정화 정도를 나타낸다 (탈안정화가 감소하는 순서로 열거됨).
EC5의 경우, 단지 영역 564-586 YLLPVVISDNDYPVQSSTGTVTV (서열식별번호: 351)만이, hCDH6이 NOV0710 또는 NOV0712와 복합체를 이룰 때 중수소 흡수의 경계선 감소를 나타낸다. 5종의 세포외 도메인 중에서 단지 EC5만이, hCDH6이 임의의 4종의 연구된 항체와 상호작용할 때 어떤 탈안정화 영역도 나타내지 않는다는 것을 주목해야 한다.
단지 EC3-EC5로만 이루어진 hCDH6 구축물인 hCDH6(267-615)에 대해 HDx 실험을 또한 수행하였다. 이들 실험에서 항체 NOV0710 및 NOV0712를 연구하였다. 이러한 구축물은 결정학 실험에 사용된 것과 동일한 구축물이다. 이들 실험에서의 서열 커버리지는 86%였다. 표 10은 모든 펩티드의 종합 목록을 제공한다.
이러한 구축물을 사용한 경우, EC3에서 중수소 흡수의 감소는 NOV0710 및 NOV0712 둘 다에 대해 영역 295-315 ADVGENAEIEYSITDGEGLDM (서열식별번호: 352)에서 관찰되고; 이것은 영역 303-315 IEYSITDGEGLDM (서열식별번호: 343)이 모든 항체에 의한 중수소 흡수의 감소를 나타내지만, NOV0712 및 NOV0710에 의해서는 보다 많은 감소를 나타내는 전장 항원과 매우 유사한 거동이다. 흥미롭게도, 2종의 구축물 사이에 차등적 거동을 나타내는 EC3에서의 영역이 존재한다. 예를 들어, hCDH6(54-615)에서, NOV0719는 많은 영역에서 유의한 탈안정화를 야기하였지만 다른 항체는 그렇지 않았다. 대조적으로, hCDH6(267-615) 단백질을 사용한 경우, NOV0710 및 NOV0712는 둘 다 전장 hCDH6(54-615) 단백질에서 NOV0719에 의해 탈안정화되는 것으로 관찰된 영역과 매우 유사한 영역에서 유의한 탈안정화를 나타낸다.
EC4에서 탈안정화에 대한 유사한 경향이 관찰된다. hCDH6(54-615)에서 NOV0719는 이러한 도메인의 C-말단 측부 상에서 가장 유의한 탈안정화를 야기하였고, NOV0710 및 NOV0712는 보다 작은 효과를 나타냈다. 대조적으로, hCDH6(267-615) 구축물을 사용할 때 NOV0710 및 NOV712는 둘 다 EC4의 C-말단 측부를 유의하게 탈안정화시킨다. 탈안정화 거동의 차이에 대한 기원은 잘 이해되지 않는다. EC4 도메인에서 어떤 구축물도 NOV0710 또는 NOV0712에 의한 중수소 흡수의 실질적 감소를 갖지 않는다는 것을 주목하는 것이 중요하며, 이는 이러한 영역이 NOV0710 또는 NOV0712 결합에 관여되지 않는다는 것을 시사한다.
EC5에서 본 발명자들은 hCDH6(54-615) 구축물과 hCDH6(267-615) 구축물 사이에 가장 일치하는 중수소 교환 거동을 관찰한다. 어떤 구축물도 NOV0712 또는 NOV0710에 의한 유의한 탈안정화를 나타내지 않았다. 또한, hCD6 (54-615)에서 중수소 흡수의 약한 및 종종 유의하지 않은 감소 (-0.5 Da 이상)가 hCDH6(267-615)에서는 보다 현저하게 된다. EC5에서 NOV0710 또는 NOV0712 결합 시에 중수소 흡수의 가장 유의한 감소를 나타내는 hCDH6(267-615) 영역은 영역 492-505 YETFVCEKAKADQL (서열식별번호: 353), 551-563 TRKNGYNRHEMST (서열식별번호: 354) 및 572-586 DNDYPVQSSTGTVTV (서열식별번호: 355)를 포함한다. 572-586 DNDYPVQSSTGTVTV(서열식별번호: 355)의 실질적 보호는 NOV0710 및 NOV0712 둘 다에 대한 X선 결정학으로부터의 에피토프 데이터와 매우 일치한다. 아미노산 N573, D574 및 Y575는 큰 접근가능한 표면적을 나타내고, 항체와의 복합체의 형성 시에 모든 3개가 실질적으로 매립된다. V577이 또한 둘 다의 구조에서 매립되며, 추가의 일치를 나타낸다. 결정학 데이터는 492-505 YETFVCEKAKADQL (서열식별번호: 353)이 매립되는 것을 나타내지 않으므로 이러한 보호는 자연에서 알로스테릭인 것으로 보이며, 551-563 TRKNGYNRHEMST (서열식별번호: 354)의 보호는 특히 R552가 실질적으로 매립되는 NOV0712 복합체에서 유의성이 있는 것으로 보인다.
마지막으로, 2종의 항체와의 복합체 형성 시에 중수소 흡수의 유의한 (-0.5 Da 이하) 감소를 나타내는 EC5 도메인에서의 다른 영역이 존재한다는 것을 주목하는 것이 중요하다. NOV0710의 경우 이들 영역은 510-525 HAVDKDDPYSGHQFSF (서열식별번호: 356), 542-550 NKDNTAGIL (서열식별번호: 357) 및 591-604 CDHHGNMQSCHAEA (서열식별번호: 358)를 포함한다. NOV0712의 경우 이들 영역은 512-522 VDKDDPYSGHQ (서열식별번호: 359), 542-550 NKDNTAGIL (서열식별번호: 357) 및 591-604 CDHHGNMQSCHAEA (서열식별번호: 358)를 포함한다.
전체적으로, HDx 데이터는, 확인된 CDH6-결합 항체에 의한 CDH6의 결합으로 CDH6 단백질의 여러 영역에서 보다 많은 중수소 교환 경향을 가지게 되며, 가능하게는 CDH6 항체의 결합에 의해 유발된 입체형태적 변화를 반영한다는 것을 시사한다. CDH6 항체의 존재 하의 중수소 교환로부터 보호된 것으로 확인된 영역은 NOV0710 및 NOV0712에 대한 X선 결정학 데이터와 일치하며 이들 항체의 에피토프의 일부를 형성한다.
<표 9>
카드헤린 1-5 도메인을 함유하는 hCDH6(54-615)에 대한 NOV1127, NOV0719, NOV0710 및 NOV0712의 효과.
<표 10>
카드헤린 3-5 도메인을 함유하는 hCDH6(267-615)에 대한 NOV0710 및 NOV0712의 효과.
실시예 7: X선 결정학에 의한 에피토프 맵핑
NOV0712 또는 NOV0710의 Fab 단편 (서열식별번호: 444-447, 표 12 및 13)에 결합된 인간 CDH6 단편 (세포외 도메인 5 또는 EC5, 서열식별번호: 443, 표 11)의 결정 구조를 결정하였다. 하기 상술된 바와 같이, CDH6 EC5를 포유동물 세포에서 NOV0712 또는 NOV0710 Fab와 공동-발현시켜 정제된 복합체를 생산하였다. 이어서, 단백질 결정학을 사용하여, NOV712 또는 NOV710 Fab에 결합된 CDH6 EC5에 대한 원자 해상도 데이터를 생성하고 에피토프를 규정하였다.
에피토프 맵핑을 위한 단백질 생산
결정학을 위해 생산된 CDH6 EC5, NOV0712 Fab 및 NOV0710 Fab의 서열이 상기 표 9에 제시되어 있다. CDH6 EC5 구축물은 인간 CDH6 (유니프롯 식별자 P55285, 서열식별번호: 533, 표 11)과 관련하여 밑줄표시된 것으로 제시되어 있으며 하기 표 11에서 서열식별번호: 534로서 제시되어 있는 잔기 490 내지 608을 포함한다. 마우스 IgG 카파 경쇄로부터의 N-말단 신호 서열은 CDH6 EC5의 분비된 발현에 사용되고 발현 동안에 절단되어 CDH6 EC5의 무손상 N-말단을 남긴다. CDH6 EC5의 C-말단은 β 아밀로이드 (APP 태그, 아미노산: EFRHDS (서열식별번호: 531))로부터 유래된 정제 태그와 융합되며, 이에 앞서 정제 후 태그의 절단 및 제거를 용이하게 하기 위한 프리시전(PreScission)® 프로테아제 (지이 헬스케어, 뉴저지주 피스카타웨이) 인식 부위 (아미노산: LEVLFQGP (서열식별번호: 532))가 존재한다. NOV0712 및 NOV0710 Fab에 대해, 중쇄 및 경쇄의 서열이 제시된다 (서열식별번호: 535-538).
<표 11>
구조 생물학 연구에 사용된 단백질
CDH6 EC5를 결정학을 위한 복합체를 생산하기 위해 엑스피293(Expi293)® 세포에서 NOV0712 또는 NOV0710 Fab와 공동-발현시켰다. 상세히, CDH6 EC5를 코딩하는 플라스미드 0.5 mg을 NOV0712 또는 NOV0710 Fab 플라스미드 0.5 mg과 혼합하고, 옵티멤 I 배지 (라이프 테크놀로지스(Life Technologies), 뉴욕주 그랜드 아일랜드) 50ml 내로 희석하고, 30분 동안 동일한 배지 50ml에서 PEI (폴리사이언시스, 펜실베니아주 워링톤) 2.5mg과 인큐베이션하였다. 이어서, 혼합물을 형질감염을 위해 8%의 CO2 하에 37℃에서 1백만개 세포/ml로 엑스피293® 발현 배지 (라이프 테크놀로지스, 뉴욕주 그랜드 아일랜드) 내 현탁액 중에서 성장하는 엑스피293® 세포 1L 내에 첨가하였다. 72시간 후에, CDH6 EC5-Fab 복합체를 함유하는 배지를 원심분리에 의해 수거하고, CaCl2에 의해 3mM로 보충하였다. 이어서, 항-APP 태그 IgG (사내 생성)와 접합된 세파로스(Sepharose)® 4B 비드 (지이 헬스케어, 펜실베니아주 피츠버그) 10ml를 배지 내에 첨가하고, 밤새 4℃에서 계속 교반하였다. 다음 날 비드를 중력 칼럼 내로 패킹하고, 25mM Hepes pH 7.4 + 150mM NaCl (HBS), 및 3mM CaCl2로 세척하였다. 표적 복합체를 3 칼럼 부피 (CV)의 100 mM 글리신 pH 2.5, 150mM NaCl, 3mM CaCl2에 의해 1/10 (v/v)의 1 M 트리스 pH 8.5 내로 용리시켰다. 복합체를 밤새 4℃에서 1/100 (w/w) GST-태그부착된 프리시전® 프로테아제 (지이 헬스케어, 펜실베니아주 피츠버그)와 함께 인큐베이션하여 APP 태그를 절단하였다. 다음 날, 혼합물을 1ml GS트랩(GSTrap)® HP 칼럼 (지이 헬스케어, 펜실베니아주 피츠버그)에 통과시킴으로써 프리시전® 프로테아제를 제거하였다. 이어서, 통과물을 밤새 37℃에서 1/10 (w/w)의 PNGaseF (사내 정제됨)와 함께 인큐베이션하여 N-연결된 글리코실화를 제거하였다. 탈글리코실화 후에, 혼합물을 농축시키고, HBS + 3 mM CaCl2 중에서 평형화시킨 하이로드(HiLoad) 16/600 슈퍼덱스® 200 pg (지이 헬스케어, 펜실베니아주 피츠버그) 상에 로딩하였다. 정제된 CDH6-Fab 복합체를 함유하는 피크 분획을 결정화를 위해 SDS-PAGE 및 LCMS에 의해 분석하고, 풀링하고, 농축시켰다.
결정화 및 구조 결정
CDH6 EC5/NOV0712 및 CDH6 EC5/NOV0710 복합체를 각각 9.3 mg/ml 및 14.7 mg/ml로 농축시키고, 5분 동안 20,000 g에서 원심분리하고, 결정화를 위해 스크리닝하였다. 데이터 수집을 위한 결정을 20℃에서 현적 증기 확산에 의해 성장시켰다. CDH6 EC5/NOV0712 복합체의 결정은 복합체 1.2 μl를 20% (v/v) PEG3350 및 0.2 M 시트르산수소이암모늄을 함유하는 저장 용액 1 μl와 혼합하고, 동일한 저장 용액 450 μl + 물 50 μl에 대하여 방울을 평형화시킴으로써 성장시켰다. CDH6 EC5/NOV0710 복합체의 결정은 복합체 0.8 μl를 0.7 M 시트르산삼나트륨 및 0.1 M 트리스·HCl pH 8.5를 함유하는 저장 용액 1 μl와 혼합하고, 동일한 저장 용액 425 μl + 물 75 μl에 대하여 방울을 평형화시킴으로써 성장시켰다. 데이터 수집 전에, 결정을 75%의 저장 용액 + 25% 글리세롤로 옮기고, 액체 질소에서 급속 냉각시켰다.
회절 데이터를 어드밴스드 포톤 소스(Advanced Photon Source) (아르곤 국립 연구소(Argonne National Laboratory), 미국)에서 빔라인 17-ID에서 수집하였다. 데이터를 오토프록(Autoproc) (글로벌 페이징, 리미티드(Global Phasing, LTD))을 사용하여 처리하고 척도화하였다. CDH6 EC5/NOV0712의 데이터는 셀 치수 a=78.83 Å, b=88.86 Å, c=186.73 Å, 알파=90°, 베타=90°, 감마=90°를 갖는 공간군 P212121에서 2.3 Å로 처리하였다. CDH6 EC5/NOV0710 복합체의 데이터는 셀 치수 a=101.48 Å, b=101.48 Å, c=240.96 Å, 알파=90°, 베타=90°, 감마=90°를 갖는 공간군 P43212에서 3.5 Å로 처리하였다. 복합체의 구조는 검색 모델로서의 사내 Fab 구조에 의한 페이저(Phaser) (McCoy et al., (2007) J. Appl. Cryst. 40:658-674)를 사용하는 분자 대체에 의해 해결하였다. CDH6 EC5의 구조는 CCP4 프로그램 모음 (Winn et al., (2011) Acta. Cryst. D67:235-242)에서 부카니어(Buccaneer) (K. Cowtan (2006) Acta Cryst. D62:1002-1011)를 사용하는 스크래치로부터 구축하였다. 최종 모델은 COOT (Emsley & Cowtan (2004) Acta Cryst. D60:2126-2132)에서 구축하고, 부스터(Buster) (글로벌 페이징, 리미티드, 영국 캠브리지)에 의해 정밀화하였다. CDH6 EC5/NOV0712 복합체의 경우, Rwork 및 Rfree 값은 각각 21.2% 및 25.2%이고; 결합 길이 및 결합 각의 루트-평균-제곱 (r.m.s) 편차 값은 각각 0.010Å 및 1.24°이다. CDH6 EC5/NOV0710 복합체의 경우, Rwork 및 Rfree 값은 각각 18.2% 및 24.6%이고; 결합 길이 및 결합 각의 r.m.s 편차 값은 각각 0.010Å 및 1.28°이다.
NOV0712 또는 NOV0710 Fab와 접촉하는 CDH6 EC5의 잔기, 상호작용의 유형 및 매립된 표면적이 모두 PISA에 의해 확인되고 (Krissinel et al., (2007) J Mol Biol. 372:774-97), 표 12 및 13에 열거되어 있다. 비대칭 유닛 (결정 내 가장 작은 고유한 유닛)에 1개 초과 카피의 복합체를 함유하는 구조의 경우, 단지 모든 카피에 공통인 상기 항체-접촉 잔기만이 에피토프 잔기로서 열거되어 있다.
CDH6 상의 NOV0712 및 NOV0710의 에피토프
전체 구조
이번에 CDH6 EC5의 보고된 결정 구조는 존재하지 않는다. CDH6 EC5의 전체 폴딩은 CDH2 (N-카드헤린) EC5의 경우와 유사하며, 둘 다 하나의 3-가닥 β 시트가 또 다른 4-가닥 β 시트에 대하여 스태킹된 채로 구성되어 있다. 2개 구조의 오버레이는 2.4Å의 작은 루트-평균-제곱 거리 (RMSD) 및 N- 및 C-말단의 유사한 배향을 생성한다. 따라서, CDH6 EC5/Fab 복합체 구조를 EC5 도메인의 중첩을 기반으로 하여 CDH2의 전장 ECD 구조 상에 오버레이하여, NOV0712 및 NOV0710 Fab의 배향을 추정할 수 있다. 도 4에 제시된 바와 같이, NOV0712 및 NOV0710은 CDH6 EC5의 동일한 측부에 결합하고, 유의하게는 중복된 에피토프를 공유한다. NOV0712는 NOV0710과 비교하여 EC4-EC5 링커로부터 다소 멀리 이동하여 결합한다.
NOV0712의 에피토프
CDH6 EC5/NOV0712 복합체의 결정 구조를 사용하여 CDH6 상의 NOV0712 에피토프를 확인한다. NOV0712 Fab에 의한 CDH6 EC5 상의 상호작용 표면은 여러 연속 및 불연속 (즉, 비인접) 서열: 즉 표 12에 상술된 바와 같은 잔기 503, 520-527, 529, 532-534, 538-543, 550, 552, 569 및 571-577에 의해 형성된다. 이들 잔기는 NOV0712 Fab에 의해 인식되는 3차원 입체형태적 에피토프를 형성한다 (도 5 상부 패널). 결정학에 의해 규정된 이러한 에피토프는 수소 중수소 교환 질량 분광측정법 (HDx-MS)에 의해 규정된 것과 양호하게 일치하며, 여기서 잔기 572-586은 NOV0712에 의해 실질적으로 보호된다. 흥미롭게도, CDH6 EC5의 잠재적 Ca2+-결합 루프 (잔기 571-579)는 그 중 대다수 (10개 잔기 중 8개)가 NOV0712와 접촉하며, 특히 Tyr575 및 Asn573은 항체에 의해 거의 완전히 매립된다 (표 12 및 도 5 하부 패널). CDH2 구조와 비교하여, CDH6 EC5의 이러한 Ca2+-결합 루프는 NOV0712 결합에 의해 유도된 "외향" 입체형태로 존재한다 (예를 들어 CDH6의 Asp572는 CDH2에서의 그의 상응하는 잔기 Asp525와 비교하여 외향으로 뒤집혀 있음).
<표 12>
CDH6 상의 NOV0712 에피토프. 결정 구조에서 NOV0712와 접촉하는 CDH6의 모든 잔기가 PISA에 의해 확인되고, 열거되고, NOV0712에 의한 그의 매립된 표면적에 의해 분류되어 있다. 또한 적용가능한 경우에 상호작용의 유형이 열거되어 있다.
1측쇄 원자에 의해 결합된 수소; 2주쇄 원자에 의해 결합된 수소
NOV0710의 에피토프
CDH6 EC5/NOV0710 복합체의 결정 구조를 사용하여 CDH6 상의 NOV0710 에피토프를 확인한다. NOV0712 Fab에 의한 CDH6 EC5 상의 상호작용 표면은 여러 연속 및 불연속 (즉, 비인접) 서열: 즉 표 13에 상술된 바와 같은 잔기 520-527, 529, 541, 543, 569 및 571-579에 의해 형성된다. 이들 잔기는 NOV0710 Fab에 의해 인식되는 3차원 입체형태적 에피토프를 형성한다 (도 6 상부 패널). 결정학에 의해 규정된 이러한 에피토프는 수소 중수소 교환 질량 분광측정법 (HDx-MS)에 의해 규정된 것과 양호하게 일치하며, 여기서 잔기 572-586은 NOV0710에 의해 실질적으로 보호된다. 흥미롭게도, CDH6 EC5/NOV0712 복합체 구조에서와 유사하게, CDH6 EC5의 잠재적 Ca2+-결합 루프에서의 모든 잔기 (잔기 571-579)는 NOV0710과 접촉하며; 그 중에서 Tyr575, Asn573 및 Asp574는 항체에 의해 거의 완전히 매립된다 (표 13 및 도 6 하부 패널). CDH2 구조와 비교하여, CDH6 EC5의 이러한 Ca2+-결합 루프는 NOV0710 결합에 의해 유도된 "외향" 입체형태로 존재한다 (예를 들어 CDH6의 Asp572는 CDH2에서의 그의 상응하는 잔기 Asp525와 비교하여 외향으로 뒤집혀 있음).
<표 13>
CDH6 상의 NOV0710 에피토프. 결정 구조에서 NOV0710과 접촉하는 CDH6의 모든 잔기가 PISA에 의해 확인되고, 열거되고, NOV0710에 의한 그의 매립된 표면적에 의해 분류되어 있다. 또한 적용가능한 경우에 상호작용의 유형이 열거되어 있다.
1주쇄 원자에 의해 결합된 수소; 2측쇄 원자에 의해 결합된 수소
실시예 8: 공-결정 구조를 검증하고 야생형 및 돌연변이체 CDH6 단백질에 대한 항-CDH6 항체의 결합을 결정하기 위한 ELISA 검정.
CDH6 단백질/NOV0710 또는 /NOV0712 결정 구조의 분석은 높은 매립된 표면 값을 갖는 여러 아미노산 잔기 (Asn573, Asp574 및 Tyr575)를 강조하였으며, 이는 이들이 CDH6 단백질과 항체의 상호작용을 매개하는데 중요할 수 있다는 것을 시사한다. 본 발명자들은 이들 잔기를 알라닌으로 대체한 재조합 돌연변이체 CDH6 단백질을 생산하고 (표 11, 서열식별번호: 539, 540 및 541), ELISA를 수행하였다. 96-웰 맥시소르프 플레이트 (눈크)를 밤새 4℃에서 1 ug/ml, 100ul/웰의 적절한 재조합 인간 단백질 (CDH6 wt, CDH6-N573A, CDH6-D574A 또는 CDH6-Y575A)로 코팅하였다. 이어서, 모든 웰을 PBS/ 0.1% 트윈-20으로 3회 세척하고, PBS/ 1% BSA/ 0.1% 트윈-20으로 1시간 동안 차단시키고, PBS/ 0.1% 트윈-20으로 3회 세척하였다. 항-CDH6 항체의 연속 희석물을 관련 웰에 첨가하고 (100 nM 시작 농도; 5배 연속 희석물), 플레이트를 실온에서 2시간 동안 인큐베이션하였다. 플레이트를 PBS/ 0.1% 트윈-20으로 3회 세척한 후에, PBS/ 1% BSA/ 0.1% 트윈-20 중에 1/10000으로 희석된 염소 항-인간 퍼옥시다제 연결된 검출 항체 (피어스(Pierce), #31412, 써모, 일리노이주 록포드)를 첨가하였다. 플레이트를 실온에서 1시간 동안 인큐베이션한 후에, PBS/ 0.1% 트윈-20으로 3회 세척하였다. 100 μl TMB (3,3', 5,5' 테트라메틸 벤지딘) 기질 용액 (BioFx)을 모든 웰에 6분 동안 첨가한 후에, 50 μl 2.5% H2SO4로 반응을 정지시켰다. 각각의 재조합 단백질에 대한 CDH6 항체 결합의 정도를 스펙트라맥스(SpectraMax) 플레이트 판독기 (몰레큘라 디바이시스(Molecular Devices))를 사용하여 OD450을 측정함으로써 결정하였다. 용량 반응 곡선을 그래프패드 프리즘(Graphpad Prism)을 사용하여 분석하였다.
도 7에 제시된 바와 같이, D574A 돌연변이는 NOV0710 및 NOV712 둘 다의 결합을 제거하였으며, 이는 이러한 잔기가 두 항체의 공유된 에피토프의 본질적 성분이라는 것을 확인시켜 준다. 흥미롭게도, N573A 돌연변이체는 NOV712에 대한 결합을 유지하지만 NOV0710에 대한 결합은 유지하지 않고, 반면에 Y575A 돌연변이체는 NOV0710에 대한 결합을 유지하지만 NOV0712에 대한 결합은 유지하지 않는다. 주의목할 만하게, 이들 돌연변이 중 어떤 것도 특유의 에피토프에 결합하는 항-CDH6 항체 NOV0720의 결합에는 영향을 미치지 않았으며 (NOV0720은 도 2에서의 NOV1132의 비-배선화 버전임) - 이는 돌연변이체가 단백질의 전체 아키텍처를 변경하지는 않았다는 것을 나타낸다. 이들 데이터는 추가로 공-결정 구조를 검증하고, 2종의 항체가 중복되는, 그러나 특유의 에피토프를 특색으로 한다는 것을 확인시켜 준다.
실시예 9: 항체 약물 접합체의 제조
1-단계 공정에 의한 DM4 접합체의 제조
항-CDH6 항체, 예를 들어 NOV7012를 약 10.0 mg/mL의 농도로 DM4 (항체의 양에 비해 6.8배 몰 과량)와 혼합하고, 이어서, 술포-SPDB (항체의 양에 비해 약 5.2배 과량)와 혼합하였다. 20℃에서 10% DMA를 함유하는 20 mM EPPS [4-(2-히드록시에틸)-1-피페라진프로판술폰산] 완충제 (pH 8.1) 중에서 대략 16시간 동안 반응을 수행하였다. 1 M 아세트산을 첨가하여 pH를 5.0으로 조정함으로써 반응을 켄칭시켰다. pH 조정 후에, 반응 혼합물을 다층 (0.45/0.22 μm) PVDF 필터를 통해 여과하고, 정제하고, 접선 유동 여과를 사용하여 10 mM 숙시네이트 완충제 (pH 4.5) 내로 투석여과하였다. 접선 유동 여과를 위한 기기 파라미터의 예가 하기 표 14에 열거되어 있다.
<표 14>
접선 유동 여과를 위한 기기 파라미터
상기에 기재된 방법으로부터 수득된 접합체를 다음에 의해 분석하였다: 세포독성제 로딩 (항체에 대한 메이탄시노이드 비, MAR)에 대해 UV 분광분석법; 접합체 단량체의 결정에 대해 SEC-HPLC; 및 유리 메이탄시노이드 백분율에 대해 역상 HPLC 또는 소수성 차폐 상 (Hisep)-HPLC.
SPDB 또는 술포-SPDB 링커 및 DM4 세포독성제를 갖는 ADC의 제조
항-CDH6 항체, 예를 들어 항체 NOV0712를 25℃에서 ~10% DMA를 함유하는 50 mM EPPS 완충제 (pH 8.1) 중에서 DM4 (6.8 및 10.2배 몰 과량)와 합하였다. 이러한 혼합물에 N-숙신이미딜 4-(2-피리딜디티오)부타노에이트 (SPDB, 각각 4.0 및 6.0배 몰 과량)를 첨가하여 최종 항체 농도가 4 mg/mL이고 최종 DMA 함량이 10%이도록 하였다. 반응을 50 EPPS 완충제 (pH 8.1) 중에서 25℃에서 ~16시간 동안 진행되도록 하였다. 접합 반응 혼합물을 10 mM 숙시네이트, 250 mM 글리신, 0.5% 수크로스, 0.01% 트윈 20, pH 5.5로 평형화 및 용리시키는 세파덱스™ G25 칼럼을 사용하여 정제하였다. 유사한 절차를 사용하여 항-CDH6-술포-SPDB-DM4 접합체를 생성하였다.
상기 방법들은 SPDB-DM4 또는 술포-SPDB-DM4와 항체의 접합에 유용하다. 하기 표 15는 CDH6 ADC의 예를 제공한다.
<표 15>
DM4-접합된 항체의 특성
계내 공정에 의한 SMCC-DM1 접합체의 제조
항-CDH6 항체는 또한 하기 절차에 따라 계내 공정을 사용하여 SMCC-DM1과 접합시킬 수 있다. CDH6 항체를 술포숙신이미딜 4-(N-말레이미도메틸) 시클로헥산-1-카르복실레이트 (술포-SMCC) 링커를 사용하여 DM1에 접합하였다. DM1 및 술포-SMCC 이종이관능성 링커의 원액을 DMA 중에서 제조하였다. 1.3:1 몰 당량의 DM1 대 링커 비 및 1.95 mM의 DM1의 최종 농도로, 술포-SMCC 및 DM1 티올을 함께 혼합하여, 40% v/v의 수성 50 mM 숙시네이트 완충제, 2 mM EDTA, pH 5.0을 함유하는 DMA 중에서 25℃에서 10분 동안 반응시켰다. 이어서, 항체를 상기 계내 생성된 SMCC-DM1의 분취물과 반응시켜 최종 접합 조건이 50 mM EPPS, pH 8.0, 10% DMA (v/v) 중 2.5 mg/mL의 Ab 및 대략 6.5의 SMCC:Ab의 몰비를 포함하도록 하였다. 25℃에서의 대략 18시간 후에, 접합 반응 혼합물을 10 mM 숙시네이트, 250 mM 글리신, 0.5% 수크로스, 0.01% 트윈 20, pH 5.5로 평형화시킨 세파덱스® G25 칼럼을 사용하여 정제하였다.
실시예 10: 인간, 시노몰구스, 래트 또는 마우스 기원으로부터의 CDH6을 발현하는 CHO 세포주의 패널에서 항-CDH6 항체 및 ADC 결합의 검증.
상이한 종으로부터의 CDH6의 발현을 특색으로 하는 세포에 대한 비접합 항-CDH6 항체 및 항-CDH6-ADC 결합을 검증하기 위해, 인간, 시노몰구스, 래트 및 마우스 기원으로부터의 CDH6을 발현하도록 조작된 CHO 세포에 대해 FACS (형광 활성화 세포 분류) 분석을 수행하였다. CDH6 발현을 특색으로 하는 CHO 세포주의 생성은 실시예 1에 기재되어 있고; FACS 방법은 실시예 3에 기재되어 있다.
도 8에 제시된 바와 같이, 비-접합 CDH6 항체 NOV0712는 인간, 시노몰구스, 래트 및 마우스 기원으로부터의 CDH6에 대해 필적하는 세포 결합을 특색으로 한다. NOV0712-술포-SPDB-DM4 항체 약물 접합체는 비-접합 NOV0712에 대한 필적하는 결합 프로파일을 특색으로 한다. 이러한 결과는 링커 및 페이로드의 부가가 항-CDH6 항체의 결합 친화도 또는 특이성을 방해하지 않는다는 것을 나타낸다.
실시예 11: 난소암 세포주의 패널에서 CDH6 발현 및 CDH6-표적화 항체 및 ADC의 세포 결합의 평가.
CDH6-표적화 ADC의 세포 활성의 평가를 위한 준비로, 난소암 세포주의 패널에서 CDH6 세포 표면 발현 수준을 FACS에 의해 평가하였다. 특히, 배양 중의 세포를 아큐타제® 세포 해리 시약 (깁코, #A1110501, 뉴욕주 그랜드 아일랜드)으로 제조업체의 지침에 따라 처리하고, 이어서 세포를 FACS 완충제 (RPMI/1%BSA, 깁코, 뉴욕주 그랜드 아일랜드) 중에서 세척함으로써 세포 현탁액을 제조하였다. 세포를 FACS 완충제 중에 1x106개 세포/ml로 재현탁시키고, 96-웰 둥근 바닥 플레이트 (코닝 #CLS3360, 매사추세츠주 툭스베리) 내로 100 μl/웰로 분취하였다. 1차 항체 (항-CDH6-PE (알앤디 시스템즈, #FAB2715P, 미네소타주 미네아폴리스)) 및 대조군 마우스 IgG1 PE 접합체 (알앤디 시스템즈, #IC002P, 미네소타주 미네아폴리스)를 FACS 완충제 중에 5 μg/ml로 희석하였다. 200μl 냉 FACS 완충제 중에서의 3회 세척 후에, 세포를 BD FACS 칸토 II® (비디 바이오사이언시스, 캘리포니아주 산호세) 상에서 분석하였다. 샘플당 신호의 기하평균을 플루우조® 소프트웨어를 사용하여 결정하였다.
OVCAR3은 높은 수준의 CDH6을 내인성으로 발현한다. CDH6 발현이 억제된 동질유전자 세포주를 생성하기 위해, OVCAR3에 CDH6 표적화 shRNA를 전달하는 렌티바이러스 벡터를 제조업체의 지침 (미션 shRNA 박테리아 글리세롤 스톡, #TRCN0000054117, 시그마, 미주리주 세인트 루이스)에 따라 형질도입하였다. OVCAR8 세포는 CDH6을 내인성으로 발현하지 않는다. CDH6 발현을 특색으로 하는 동질유전자 세포주를 생성하기 위해, OVCAR8 세포에 인간 CDH6 cDNA의 발현을 조종하는 렌티바이러스 구축물을 형질도입하였다. 특히, CDH6에 대한 cDNA를 진코포에이아(Genecopoeia) (#Z2028, 진코포에이아, 매사추세츠주 록빌)로부터 구입하고, 게이트웨이(Gateway)® 클로닝을 사용하여 제조업체의 지침 (인비트로젠, 캘리포니아주 칼스배드)에 따라 pLenti6.3 내로 클로닝하였다.
FACS 분석은 야생형 OVCAR3에서 높은 수준의 내인성 CDH6 발현 및 OVCAR3 shRNACDH6 세포주에서 CDH6의 억제를 확인시켜 주었다. OVCAR8 세포는 CDH6-음성인 것으로 확인되었고, 한편 높은 수준의 외인성 CDH6 발현이 OVCAR8 조작된 CDH6+ 세포에서 관찰되었다 (도 9).
FACS 분석은 추가로 생성된 CDH6-표적화 항체 및 ADC의 특이적 세포 결합을 확인시켜 주었다. 도 10에 제시된 바와 같이, 비-접합 CDH6 항체 (NOV0712)는 동일한 항체 및 술포-SPDB-DM4 링커/페이로드를 사용하는 술포-SPDB-DM4 접합체의 경우와 필적하는 CDH6-특이적 결합 패턴을 특색으로 한다. 비-결합 비접합 IgG 또는 ADC를 대조군으로서 포함시켰으며, 이는 예상된 바와 같이 세포 결합을 유발하지는 않는 것으로 밝혀졌다. 다시, 링커 및 페이로드의 부가는 항-CDH6 항체의 친화도 또는 특이성을 방해하지 않는 것으로 나타났다.
실시예 12: CDH6-표적화 ADC의 시험관내 세포 활성
선택 항-CDH6 항체의 SMCC-DM1 접합체의 시험관내 세포 활성을 OVCAR3 (RPMI + 20% FCS에서 배양된 난소 장액성 암종), JHOS4 (1:1 F12:DMEM+10%FBS), KNS42 (EMEM+10%FBS), NCIH661 (RPMI+10%FBS), SNU8 (RPMI+10%FBS) 및 OVCAR8 (RPMI+10%FBS)을 포함하는 세포주의 패널에 결정하였다. OVCAR3 및 NCIH661 세포주를 ATCC (#HTB-161 및 #HTB-183, ATCC, 버지니아주 마나사스)로부터 입수하였다. JHOS4를 리켄(RIKEN) (#RCB1678, 리켄 셀 뱅크(RIKEN Cell Bank), 리켄, 일본 츠쿠바)으로부터 입수하였다. KNS42를 보건 과학 연구 자원 은행 HSRRB (#IFO50356, HSRRB, 일본 오사카)로부터 입수하였다. SNU8을 한국 세포주 은행 KCLB (#00008, KCLB, 한국 서울)로부터 입수하였다. OVCAR8을 NCI/DCTD 종양/세포주 저장소 (NCI, 매사추세츠주 프레드릭)로부터 입수하였다.
배양 중의 세포를 계수하고, 배지 중에 1x105개 세포/ml의 농도로 희석하였다. 1000개 세포/웰을 384-웰 플레이트 (코닝 코스타#3707, 코닝, 매사추세츠주 툭스베리)로 옮겼다. ADC 원액을 1.4ml 매트릭스 튜브 (써모, # 3790, 일리노이주 록포드)에서 제조하였다. 10-포인트, 1:3 연속 희석물을 384-웰 심부-웰 플레이트 (브랜드테크 사이언티픽 인크 #701355, 코네티컷주 에섹스)에서 제조하고, 25μl를 검정 플레이트마다 옮겨 (삼중) 33nM의 ADC의 최고 시작 농도를 생성하였다. 대조군의 경우, 단지 세포만을 갖는 웰 (=100% 생존율 대조군) 및 SMCC-DM1 접합된 비-표적화 항체와 인큐베이션된 세포를 갖는 웰 (비-표적 조종된 활성을 체크하기 위함)을 제조하였다. 플레이트를 37℃ 및 5% CO2에서 120시간 동안 인큐베이션하였다. 1차 항체/Fab-DM1 복합체의 세포 활성을 셀타이터-글로® 시약 (프로메가 #G7571, 위스콘신주 매디슨)을 사용하여 제조업체의 지침에 따라 결정하였다. 생존율을 단지 세포만의 대조군에 대해 정규화하고, 데이터를 팁코 스팟파이어 (팁코 소프트웨어 인크(Tibco Software Inc), 캘리포니아주 팔로 알토)를 사용하여 플롯팅하였다.
CDH6-표적화 ADC의 하위세트는 CDH6 발현이 결핍된 세포 (OVCAR8)에 대해 불활성인 농도 (1.22 nM)에서 CDH6-양성 세포의 증식을 억제할 수 있으며, 이는 CDH6-ADC의 표적-의존성 시험관내 세포 활성을 나타낸다 (표 16).
<표 16>
항-CDH6 SMCC-DM1 접합체의 시험관내 세포 활성. 표는 1.22nM ADC에서의 퍼센트 억제를 제시한다.
실시예 13: 난소암 세포주에 대한 SMCC-DM1 및 SPDB-DM4 접합체로서의 항-CDH6 ADC의 시험관내 세포 활성
SMCC-DM1 및 SPDB-DM4 접합체로서의 항-CDH6 ADC의 시험관내 세포 활성을 난소암 세포주의 패널에서 평가하였다 (도 11 및 표 17). 항-CDH6 ADC의 표적-의존성 및 비활성을 평가하기 위해, 동질유전자 세포주 쌍을 실시예 11에 기재된 바와 같이 OVCAR3 및 OVCAR8 세포주에 대해 생성하였다.
SMCC-DM1 및 SPDB-DM4 포맷 둘 다의 CDH6-표적화 ADC는 비-발현 동질유전자 세포주 쌍 및 이소형 대조군 ADC와 비교하여 CDH6 발현 세포주에서의 증식의 억제에 의해 나타난 바와 같은 표적 및 용량-의존성 세포 활성을 나타냈다 (도 11 및 표 17). 세포 활성 검정을 실시예 12에 개시된 바와 같이 수행하고 분석하였다.
<표 17>
난소암 세포주에서 SMCC-DM1 및 SPDB-DM4 접합체로서의 항-CDH6 ADC의 시험관내 세포 활성.
실시예 14: 비-접합 형태 또는 술포-SPDB-DM4 항체 약물 접합체로서의 CDH6-표적화 항체 또는 대조군 IgG의 시험관내 세포 활성.
비-접합 IgG 또는 술포-SPDB-DM4 접합체로서의 CDH6 항체 NOV0712의 세포 활성을 난소암 세포주의 패널에서 평가하였다 (실시예 11 참조). 비-접합 형태 또는 술포-SPDB-DM4 접합체로서의 비-표적화 이소형 대조군 항체를 추가로 포함시켰다. 도 12에 제시된 바와 같이, CDH6-표적화 항체 약물 접합체 NOV0712-술포-SPDB-DM4는 비-발현 세포주 (OVCAR8)와 비교하여 CDH6 발현 세포주 (OVCAR3 및 OVCAR8-CDH6+)에서의 증식의 억제에 의해 나타난 바와 같은 표적 및 용량-의존성 세포 활성을 나타냈다. 비-접합 NOV0712 항체 또는 비-표적화 IgG 대조군에서는 어떤 세포 활성도 관찰되지 않았다. 비-특이적 세포독성을 유발하는 것으로 이해된 농도에서의 미미한 활성이 IgG-술포-SPDB-DM4 접합체에서 관찰되었다. 세포 활성 검정을 실시예 9에 개시된 바와 같이 수행하고 분석하였다.
실시예 15: 난소암의 이종이식편 마우스 모델에서 SMCC-DM1 접합체로서의 항-CDH6 항체의 생체내 효능.
선택 항-CDH6 ADC의 항종양 활성을 OVCAR-3 난소 이종이식편 모델에서 평가하였다. 암컷 NOD-scid-감마 마우스에 포스페이트 완충제 용액 (PBS) 중 50% 매트리겔(Matrigel)™ (비디 바이오사이언시스)을 함유하는 10x106개 OVCAR-3 세포를 우측 측복부 상에 피하로 이식하였다. 현탁액 중 세포를 함유하는 총 주입 부피는 200 μl였다. 이식 29일 후에 165 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 7개 군 중 1개에 무작위로 배정한 후에 (n = 4/군), 마우스에게 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1-SMCC-DM1 (10 mg/kg), 또는 5종의 항-CDH6-SMCC-DM1 중 1종 (10 mg/kg)을 투여하였다. 종양 부피 및 체중을 매주 2회 측정하였다. 각각의 항-CDH6-SMCC-DM1 ADC는 대조군 부문과 비교하였을 때 10mg/kg의 단일 용량 후에 종양 성장에 대한 억제 효과를 도출하였다 (도 13).
제2 선택 항-CDH6 ADC의 항종양 활성을 OVCAR-3 난소 이종이식편 모델에서 평가하였다. 암컷 NOD-scid-감마 마우스에 PBS 중 50% 매트리겔™ (비디 바이오사이언시스, 캘리포니아주 산 호세)을 함유하는 10x106개 OVCAR-3 세포를 우측 측복부 상에 피하로 이식하였다. 현탁액 중 세포를 함유하는 총 주입 부피는 200 μl였다. 이식 35일 후에 166 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 7개 군 중 1개에 무작위로 배정한 후에 (n = 4/군), 마우스에게 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1-SMCC-DM1 (10 mg/kg), 또는 5종의 항-CDH6-SMCC-DM1 중 1종 (10 mg/kg) + 이전 OVCAR3 분류에서 투여된 NOV0712-SMCC-DM1을 투여하였다. 종양 부피 및 체중을 매주 2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 5종의 항-CDH6-SMCC-DM1 중에서, 단지 NOV0690만이 NOV0712-SMCC-DM1의 경우와 필적하는 반응을 도출하였다 (도 14).
실시예 16: 난소암의 이종이식편 마우스 모델에서 SPDB-DM4 접합체로서의 항-CDH6 항체의 생체내 효능
선택 항-CDH6 ADC의 항종양 활성을 OVCAR-3 난소 이종이식편 모델에서 평가하였다. 암컷 NOD-scid-감마 마우스에 PBS 중 50% 매트리겔™ (비디 바이오사이언시스, 캘리포니아주 산 호세)을 함유하는 10x106개 OVCAR-3 세포를 우측 측복부 상에 피하로 이식하였다. 현탁액 중 세포를 함유하는 총 주입 부피는 200 μl였다. 이식 21일 후에 174 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 9개 군 중 1개에 무작위로 배정한 후에 (n = 5/군), 마우스에게 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1-SPDB-DM4 (5 mg/kg), 비-표적 이소형 대조군 hIgG1-SMCC-DM1 (5 mg/kg), NOV0712-SMCC-DM1 (5 mg/kg), 또는 7종의 항-CDH6-SPDB-DM4 중 1종 (5 mg/kg)을 투여하였다. 종양 부피 및 체중을 매주 1-2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 이러한 투여량에서 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 모든 항-CDH6-SPDB-DM4 작용제 중에서, NOV0712-SPDB-DM4는 투여 70일 후에 퇴행이 있는 최대 항종양 효과를 도출하였다. NOV0710-SPDB-DM4는 이러한 연구에서 두번째로 가장 활성인 항-CDH6-SPDB-DM4였다 (도 15).
실시예 17: 난소암의 이종이식편 마우스 모델에서 SMCC-DM1 또는 SPDB-DM4 접합체로서의 항-CDH6 항체의 생체내 효능
상이한 링커-페이로드 포맷의 선택 항-CDH6 ADC NOV0712의 항종양 활성을 OVCAR-3 난소 이종이식편 모델에서 평가하였다. 암컷 NOD-scid-감마 마우스에 PBS 중 50% 매트리겔™ (비디 바이오사이언시스, 캘리포니아주 산 호세)을 함유하는 10x106개 OVCAR-3 세포를 우측 측복부 상에 피하로 이식하였다. 현탁액 중 세포를 함유하는 총 주입 부피는 200 μl였다. 이식 21일 후에 174 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 5개 군 중 1개에 무작위로 배정한 후에 (n = 5/군), 마우스에게 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1-SPDB-DM4 (5 mg/kg), 비-표적 이소형 대조군 hIgG1-SMCC-DM1 (5 mg/kg), NOV0712-SMCC-DM1 (5 mg/kg), 또는 NOV0712-SPDB-DM4 (5 mg/kg)를 투여하였다. 종양 부피 및 체중을 매주 1-2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았다 (데이터는 제시되지 않음). NOV0712의 L/P 포맷은 둘 다 항종양 효과를 도출하였지만, SPDB-DM4 포맷이 훨씬 더 강력하여 투여후 70일을 넘어서 동안 지속적인 퇴행을 유발하였다 (도 16). 약동학적 샘플링은 2종의 작용제가 유사한 PK 프로파일을 갖는다는 것을 나타낸다 (데이터는 제시되지 않음).
실시예 18: 난소암의 환자-유래된 원발성 종양 이종이식편 마우스 모델에서 상이한 링커/페이로드 포맷을 갖는 항-CDH6 ADC의 생체내 효능
항-CDH6 ADC NOV0712의 상이한 링커 페이로드 포맷의 항종양 활성을 마우스 내로 이종이식된 CDH6 발현 원발성 (환자 유래된) 난소 종양에서 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 무흉선 누드 마우스에 우측 측복부 상에 피하로 대략 27 mm3 크기의 종양 단편을 이식하였다. 이식 48일 후에 178 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 8개 군 중 1개에 무작위화한 후에 (n = 5/군), 마우스에게 14일마다 (Q14D) 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1 링커 페이로드 포맷 (-SMCC-DM1, -SPDB-DM4, -술포-SPDB-DM4), NOV0712 링커 페이로드 포맷 (-SMCC-DM1, -SPDB-DM4, -술포-SPDB-DM4) 또는 네이키드 항체로서의 NOV0712를 투여하였다. 모든 군에 5 mg/kg을 투여하였다. 종양 부피 및 체중을 매주 1-2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았다 (데이터는 제시되지 않음). 네이키드 항체로서의 NOV0712는 종양 성장 시에 억제 효과를 나타내지 않았다. NOV0712-SMCC-DM1 처리는 이러한 모델에서 퇴행을 유발하지 않았다. NOV0712의 각각의 절단가능한 포맷은 퇴행을 유발하였다. 5mg/kg Q14D (14일마다 단일 용량)의 NOV0712-술포-SPDB-DM4는 제1 투여후 150일에 걸쳐 지속되는 지속적인 종양 퇴행을 갖는 가장 강력한 치료 부문이었다 (도 17).
실시예 19: 난소암의 이종이식편 마우스 모델에서의 상이한 링커/페이로드 포맷을 갖는 항-CDH6 ADC의 생체내 효능
선택 항-CDH6 ADC의 항종양 활성을 OVCAR-3 난소 이종이식편 모델에서 평가하였다. 암컷 NOD-scid-감마 마우스에 PBS 중 50% 매트리겔™ (비디 바이오사이언시스, 캘리포니아주 산 호세)을 함유하는 10x106개 OVCAR-3 세포를 우측 측복부 상에 피하로 이식하였다. 현탁액 중 세포를 함유하는 총 주입 부피는 200 μl였다. 이식 20일 후에 172 mm3의 평균 종양 부피를 갖는 마우스를 연구에 등록시켰다. 8개 군 중 1개에 무작위화한 후에 (n = 5/군), 마우스에게 단일 i.v. 용량의 PBS (10 ml/kg), 비-표적 이소형 대조군 hIgG1-SPDB-DM4 (5 mg/kg), 비-표적 이소형 대조군 hIgG1-술포-SPDB-DM4 (5 mg/kg), NOV0712-SPDB-DM4 (1.25, 2.5 및 5 mg/kg), 또는 NOV0712-술포-SPDB-DM4 (1.25, 2.5 및 5 mg/kg)를 투여하였다. 종양 부피 및 체중을 연구의 지속기간 내내 매주 1-2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). NOV0712-술포-SPDB-DM4는 2.5 mg/kg 및 5 mg/kg 둘 다의 용량 수준에서 NOV0712-SPDB보다 더 효과적이었다. 단일 2.5 mg/kg 용량의 NOV0712-술포-SPDB-DM4는 퇴행을 유발하였다. 단일 5 mg/kg 용량의 각각의 포맷은 퇴행을 유발하였지만, 술포-SPDB-DM4 용량의 효과는 -SPDB-DM4 용량의 경우보다 더 오래 지속되었다. 전체적인 이들 데이터는 술포-SPDB-DM4 포맷이 평가된 패널 중에서 ADC를 사용하여 CDH6을 표적화하는데 가장 활성인 포맷이라는 것을 제시한다 (도 18).
실시예 20: 난소암의 환자-유래된 원발성 종양 이종이식편 마우스 모델에서 상이한 링커/페이로드 포맷을 갖는 항-CDH6 ADC의 생체내 효능
항-CDH6 ADC NOV0712의 상이한 링커 페이로드 포맷의 항종양 활성을 마우스 내로 이종이식된 CDH6 발현 원발성 (환자 유래된) 난소 종양에서 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 무흉선 누드 마우스에 우측 측복부 상에 피하로 대략 27 mm3 크기의 종양 단편을 이식하였다. 원발성 종양의 성장 동역학에서의 가변적 잠복기로 인해, 마우스를 회전 방식으로 연구 내에 진입시켰다. 종양은 147 mm3 내지 244 mm3의 적절한 부피에 도달하였을 때 군에 배정하였다 (등록 시에 평균 크기는 180 mm3였음). 최초 5마리의 마우스를 군 1 내지 5에 배정하고, 다음 5마리의 마우스를 군 5 내지 1에 배정하였으며, 5개 군 (n=5/군)이 채워질 때까지 계속 그렇게 하였다. 등록 시에 각각 마우스에게 2주마다 (Q14D) i.v. 용량의 PBS(10 ml/kg), 비-표적 이소형 대조군 hIgG1-SMCC-DM1 (5 mg/kg), NOV0712-SMCC-DM1 (5 mg/kg), 비-표적 이소형 대조군 hIgG1-술포-SPDB-DM4 (2.5 mg/kg), 또는 NOV0712-술포-SPDB-DM4 (2.5 mg/kg)를 투여하였다. 종양 부피 및 체중을 매주 2회 측정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). NOV0712-SMCC-DM1 5mg/kg Q14D는 이러한 모델에서 종양 성장을 감소시킬 수 있다. NOV0712-술포-SPDB-DM4 2.5mg/kg Q14D는 이러한 모델에서 42일 동안 종양 성장을 중지시키는데 효과적이다 (도 19).
실시예 21: 난소암의 환자-유래된 원발성 종양 이종이식편 마우스 모델의 패널에서 항-CDH6 ADC의 생체내 효능.
항-CDH6 ADC (NOV0712-술포-SPDB-DM4)의 항종양 활성을 28개의 난소 원발성 종양 이종이식편 (PTX) 모델의 패널에서 1x1x1 설계 (1PTX x 1마리 동물 x 1가지 처리)로 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 암컷 누드 마우스에 12 게이지 투관침을 사용하여 3 x 3 x 3 mm 종양 단편을 액와 영역에 피하로 이식하였다. 종양이 200-250 mm3로 측정되었을 때, 마우스를 연구 내로 등록시키고, 처리를 시작하였다. 등록 시에 각각 마우스에게 2주마다 (Q14D) i.v. 용량의 NOV0712-술포-SPDB-DM4 (5 mg/kg)를 투여하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 종양 부피 및 체중을 매주 2회 측정하였다. 항종양 활성의 가시화를 위해, 시간에 따른 퍼센트 종양 부피 변화를 팁코 스팟파이어 (팁코 소프트웨어, 캘리포니아주 팔로 알토)를 사용하여 플롯팅하였다.
PTX 샘플에서의 CDH6 발현은 비처리 PTX 종양으로부터의 샘플에 대해 면역조직화학 (IHC)을 사용하여 평가하였다. 특히, 희생 시에, 종양을 즉시 절제하고, 조직학 카세트 내로 수집하고, 24시간 동안 10% 완충 포르말린 중에 고정시켰다. 이어서, 카세트를 70% EtOH 내로 옮기고, 상용 조직학적 절차를 사용하여 처리하고 파라핀 중에 포매시켰다. 실험용 FFPE 블록을 슬라이드 상의 전체 절편으로서 3.5μm로 절단하였다. 토끼 폴리클로날 항 인간 CDH6 항체를 시그마 알드리치로부터 입수하고 (Cat # HPA007047, 시그마 알드리치, 미주리주 세인트 루이스), 1차 면역조직화학 (IHC) 항체로서 사용하였다. 항체는 벤타나 디스커버리 XT 바이오마커 플랫폼(Ventana DISCOVERY XT Biomarker Platform) (애리조나주 투손) 상에서 벤타나 비오틴-무함유 DAB 검출 시스템을 사용하여 검출하였다.
최적화된 프로토콜은 벤타나 세포 컨디셔닝 #1 항원 검색 시약 (Cat # 950-124)에 대한 표준 노출을 포함하였다. 1차 항체를 다코 시토메이션(DAKO Cytomation) 항체 희석제 (Cat # S0809) 중에 1:200의 농도로 희석하고, 100 μl 부피로 적용하고, 37℃에서 60분 동안 인큐베이션하였다. 이후 벤타나 옴니맵(Ventana OmniMap) 예비희석된 HRP-접합된 항-토끼 2차 항체 (Cat #760-4311)와의 인큐베이션을 4분 동안 수행하였다. 이어서, 2차 항체를 크로모맵(ChromoMap) DAB 키트 (Cat # 760-159)를 사용하여 검출하고, 슬라이드를 벤타나 헤마톡실린 (Cat # 760-2021)으로 4분 동안 대조염색하고, 이어서 벤타나 블루잉 시약 (Cat # 760-2037)으로 4분 동안 대조염색하였다. 이어서, 슬라이드를 증가하는 농도의 에탄올 (95-100%) 중에서, 이어서 크실렌 중에서 탈수시키고, 이어서 커퍼슬립으로 덮었다. 커버슬립으로 덮은 슬라이드를 광 현미경검사에 의해 평가하고, 라이카(Leica)/아페리오 스캔스코프(Aperio ScanScope) 슬라이드 스캐너 (캘리포니아주 비스타)에 의해 스캐닝하였다. 염색된 슬라이드의 스캐닝된 이미지를 통합된 라이카 이슬라이드 매니저/아페리오 스펙트럼 (캘리포니아주 비스타)으로부터 오프닝하는 인디카 랩스 할로(Indica Labs HALO) (뉴멕시코주 코랄레스)에서 착수하였다. 디지털 영상을 관찰하고, 주석달고, 종양 검출을 위한 분류자 모듈 (CDH6 루즈 종양 2, CDH6 Neg 종양 2 & CDH6 종양 & CDH6 종양2)의 존재 또는 분류자 모듈의 부재 하에 면적 정량화 알고리즘 (면적 정량화 v1.0)을 사용하여 분석하였다. 영상 분석 알고리즘을 사용하여 임의의 강도를 검출하였다.
도 20에 예시된 바와 같이, 항-CDH6 ADC의 효능을 PTX 모델에서, 특히 IHC에 의한 20 퍼센트 초과의 CDH6 양성 종양 면적을 특색으로 하는 종양 모델 중에서 관찰하였다. 특히 주목할 만하게, 11개/28개 (39%) PTX 모델은 150일을 초과하여 지속되는 완전한 종양 퇴행을 나타내면서 반응하였다.
실시예 22: 신암의 환자-유래된 원발성 종양 이종이식편 마우스 모델에서 2.5 mg/kg i.v. Q14D 또는 5 mg/kg i.v. Q14D로 투여된 항-CDH6 ADC의 생체내 효능.
항-CDH6 ADC (5mg/kg Q14D 또는 2.5 mg/kg Q14D로 투여된 NOV0712-술포-SPDB-DM4)의 항종양 활성을 신장 원발성 종양 이종이식편 (PTX) 모델에서 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 암컷 누드 마우스에 12 게이지 투관침을 사용하여 3 x 3 x 3 mm 종양 단편을 액와 영역에 피하로 이식하였다. 평균 종양 부피가 195 mm3 (범위 115-282 mm3)였을 때, 종양 단편 이식 후 제20일에 동물을 처리군 내로 무작위화하였다. 처리를 제20일에 개시하였다. 항종양 활성을 종양 세포 이식 후 제53일에, 처리의 개시 33일 후에, 모든 동물이 연구에서 남아있는 마지막 날에 결정하였다. 비-표적화 ADC를 CDH6-특이적 항종양 활성을 결정하기 위한 대조군으로서 포함시켰다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 난소암 모델에서와 같이, 항-CDH6 ADC의 효능이 IHC에 의한 20 퍼센트 초과의 CDH6 양성 종양 면적을 특색으로 하는 신장 PTX 모델에서 관찰되었다. 도 21에 제시된 바와 같이, 항-CDH6 ADC 처리는 신장 PTX 모델에서 원발성 인간 종양에 대하여 효과적이었다.
실시예 23: 신암의 환자-유래된 원발성 종양 이종이식편 마우스 모델에서 2.5 mg/kg Q14D i.v. 또는 5 mg/kg i.v. Q14D로 투여된 항-CDH6 ADC의 생체내 효능.
항-CDH6 ADC (NOV0712-술포-SPDB-DM4)의 항종양 활성을 신장 원발성 종양 이종이식편 (PTX) 모델에서 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 암컷 누드 마우스에 12 게이지 투관침을 사용하여 3 x 3 x 3 mm 종양 단편을 액와 영역에 피하로 이식하였다. 평균 종양 부피가 160mm3 (범위 113-245 mm3)였을 때, 종양 단편 이식 후 제14일에 동물을 처리군 내로 무작위화하였다. 처리를 제14일에 개시하였다. 항종양 활성을 종양 세포 이식 후 제50일에 결정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 난소암 모델에서와 같이, 항-CDH6 ADC의 효능이 IHC에 의한 20 퍼센트 초과의 CDH6 양성 종양 면적을 특색으로 하는 신장 PTX 모델에서 관찰되었다. 도 22에 제시된 바와 같이, 항-CDH6 ADC 처리는 신장 PTX 모델에서 원발성 인간 종양에 대하여 효과적이었다.
실시예 24: 신암의 환자-유래된 원발성 종양 이종이식편 마우스 모델에서 2.5 mg/kg Q14D 또는 5 mg/kg Q14D로 투여된 항-CDH6 ADC의 생체내 효능.
항-CDH6 ADC (NOV0712-술포-SPDB-DM4)의 항종양 활성을 신장 원발성 종양 이종이식편 (PTX) 모델에서 평가하였다. 인간 원발성 이종이식편 모델은 nu/nu 누드 마우스 내 피하로의 원발성 인간 종양 조직의 직접 이식에 의해 확립하였다. 생성된 이종이식편을 이러한 연구에 사용하기 전에 4 내지 10회 연속으로 계대배양하였다. 암컷 누드 마우스에 12 게이지 투관침을 사용하여 3 x 3 x 3 mm 종양 단편을 액와 영역에 피하로 이식하였다. 평균 종양 부피가 176 mm3 (범위 123-267 mm3)였을 때, 종양 단편 이식 후 제19일에 동물을 처리군 내로 무작위화하였다. 처리를 제19일에 개시하였다. 항종양 활성을 종양 세포 이식 후 제49일에 결정하였다. 어떤 마우스도 체중의 감소를 나타내지 않았으며, 이는 ADC가 잘 허용되었다는 것을 나타낸다 (데이터는 제시되지 않음). 난소암 모델에서와 같이, 항-CDH6 ADC의 효능이 IHC에 의한 20 퍼센트 초과의 CDH6 양성 종양 면적을 특색으로 하는 신장 PTX 모델에서 관찰되었다. 도 23에 제시된 바와 같이, 항-CDH6 ADC 처리는 5 mg/kg 내지 2.5 mg/kg 투여량 둘 다에서 신장 PTX 모델에서 원발성 인간 종양에 대하여 효과적이었다.
실시예 25: 조합 요법에서의 항-CDH6 ADC
항 CDH6 ADC를 소분자 억제제 또는 다른 항체와 조합할 수 있다. 챌리스(Chalice) 소프트웨어 (잘리쿠스(Zalicus), 매사추세츠주 캠브리지) 또는 콤보익스플로러(ComboExplorer) 어플리케이션 (노파르티스, 스위스 바젤)을 사용하여, 조합의 반응을 약물 자체의 용량에 의한 상가작용에 대해 폭넓게 사용되는 로에베(Loewe) 모델에 대하여 단일 작용제와 비교한다 (Lehar et al. Nat. Biotechnol. (2009) 27: 659-666; Zimmermann et al., Drug Discov. Today (2007) 12: 34-42). 상가작용과 비교하여 초과 억제를 전체 용량-매트릭스 차트로서 플롯팅하여, 상승작용이 일어나는 약물 농도를 가시화할 수 있다. 표 18은 여러 항-CDH6 ADC/화합물 조합을 제시한다.
셀타이터 글로® 발광 검정 (프로메가, 위스콘신주 매디슨)을 사용하여 세포 ATP 함량을 측정함으로써 세포 생존율을 결정하였다. 약물 첨가 1일 전에, 250-500개 세포를 384-웰 플레이트 (그라이너(Greiner), 노스캐롤라이나주 먼로) 내로 20 μl 성장 배지에 플레이팅하였다. 이어서, 세포를 120시간 동안 다양한 농도의 단일 작용제로서의 항-CDH6 ADC (표 18), 단일 작용제 화합물, 또는 항-CDH6 ADC/화합물 조합물과 함께 인큐베이션한 후에, 셀타이터 글로® 시약을 각 웰에 첨가하고, 엔비전(Envision)® 플레이트 판독기 (퍼킨 엘머(Perkin Elmer), 매사추세츠주 월섬) 상에서 발광을 기록하였다. 발광 값을 사용하여 DMSO-처리된 세포 (0% 억제)에 비해 세포 생존율 억제를 계산하였다.
<표 18>
항-CDH6 ADC 조합
도 24는 소분자 억제제와 조합된 항-CDH6 ADC의 예이다. SNU-8 난소암 세포를 실시예 12에 기재된 바와 같이 성장시키고, 항-CDH6 ADC (NOV0712-술포-SPDB-DM4) 및 Bcl2/Bcl-Xl 억제제 (ABT-263), Bcl-Xl 억제제 (WEHI-539), IAP 억제제 (NVP-LCL161) 또는 MEK 억제제 (트라메티닙)의 적정으로 처리하였다. 강력한 상승작용은 Bcl2/Bcl-Xl, Bcl-Xl, MEK 및 IAP 억제제와 조합된 항-CDH6 ADC (NOV0712-술포-SPDB-DM4)에서 관찰된다. ADC의 자기-자기 조합, 예를 들어 (CDH6-ADC + CDH6-ADC), 또는 소분자의 자기-자기 조합, 예를 들어 (NVP-LCL161 + NVP-LCL161)을 실제 상승작용으로부터 용량 상가작용을 구별하기 위해 포함시켰으며, 이들 점수는 도 24에서 보고되어 있다. 도 24에서 나타난 바와 같이, CDH6-ADC + 소분자 조합은 자기-자기 조합과 비교하여 유의하게 더 높은 로에베 상승작용 점수를 특색으로 한다.
이러한 데이터는 ADC와 항유사분열 페이로드 (예를 들어, 술포-SPDB-DM4, SPDB-DM4, SMCC-DM1)와의 조합 또는 아폽토시스촉진 소분자 억제제 (예를 들어, ABT-261, ABT-199, WEHI-539 또는 IAP 억제제, 예컨대 NVP-LCL161) 또는 MAPK 경로 억제제 (즉, 트라메티닙)를 갖는 다른 페이로드와의 조합이 항유사분열/세포독성 ADC 페이로드 단독에 의해 유도된 초기 G2/M 정지 또는 손상 후 보다 효과적인 아폽토시스 실행을 유발한다는 것을 나타낸다.
추가로, CDH6-ADC 조합은 생체내 조합 치료에 의해 추가로 평가할 수 있다. CDH6-ADC는 PD/PD-L1 경로를 표적화하는 면역조정제, 예컨대 키트루다(Keytruda)® (펨브롤리주맙) 또는 옵디보(Opdivo)® (니볼루맙)와 상승작용할 수 있다. 요약하면, 본원에 개시된 항-CDH6 ADC는 다른 분자와 조합되어 사용될 때 상승작용적 효과를 발휘하여 치료에 대한 보다 많은 옵션을 제시할 수 있다.
실시예 26: 종양 적응증에서의 CDH6 발현
CDH6은 난소암, 신암, 연부조직암, CNS암, 갑상선암 및 담관암종을 포함한 여러 종양 적응증에서 과다발현된다. CDH6 mRNA 발현을 아피메트릭스(Affymetrix) 진칩(GeneChip)® 플랫폼 (캘리포니아주 산타 클라라)을 사용하여 평가하였다. 특히, 인간 종양 및 정상 조직 샘플에 대한 데이터를 포함하는 노파르티스-내부 mRNA 발현 데이터베이스를 CDH6 프로브세트 214803_at를 사용하여 검색하고, 데이터를 팁코 스팟파이어 (캘리포니아주 팔로 알토)를 사용하여 플롯팅하였다. 그래프 상의 각각의 포인트는 개별 종양 또는 정상 조직 샘플을 나타낸다. 도 25에 예시된 바와 같이, CDH6은 제한된 정상 조직 발현을 특색으로 하고, 난소암, 신암, CNS암, 연부조직암, 갑상선암 및 담관암종에서 유의하게 과다발현된다.
CDH6 발현을 실시예 21에 기재된 프로토콜을 사용하여 면역조직화학에 의해 인간 원발성 종양 샘플의 수집물에서 추가로 평가하였다. 도 26에 제시된 바와 같이, CDH6 단백질 발현을 대다수의 난소암 및 신암 샘플에서 관찰하였다. 또한, CDH6 발현을 자궁내막암, 뿐만 아니라 담관암종에서 빈번하게 검출하였다. 흥미롭게도, 담관암종 내의 CDH6 발현은 간내 하위유형 중에서 풍부화되어 있었다.
본원에 기재된 실시예 및 측면은 단지 예시적 목적을 위한 것이고, 그에 비추어 다양한 변형 또는 변화가 관련 기술분야의 통상의 기술자에게 시사될 것이며, 본 출원의 취지 및 범위 및 첨부된 청구범위의 범주 내에 포함되어야 하는 것으로 이해된다.
SEQUENCE LISTING
<110> NOVARTIS AG
<120> ANTI-CDH6 ANTIBODY DRUG CONJUGATES
<130> PAT056392-WO-PCT
<140>
<141>
<150> 62/036,382
<151> 2014-08-12
<160> 541
<170> PatentIn version 3.5
<210> 1
<211> 2988
<212> DNA
<213> Homo sapiens
<400> 1
acttcattca cttgcaaatc agtgtgtgcc cacaagagcc agctctcccg agcccgtaac 60
cttcgcatcc caagagctgc agtttcagcc gcgacagcaa gaacggcaga gccggcgacc 120
gcggcggcgg cggcggcgga ggcaggagca gcctgggcgg gtcgcagggt ctccgcgggc 180
gcaggaaggc gagcagagat atcctctgag agccaagcaa agaacattaa ggaaggaagg 240
aggaatgagg ctggatacgg tgcagtgaaa aaggcacttc caagagtggg gcactcacta 300
cgcacagact cgacggtgcc atcagcatga gaacttaccg ctacttcttg ctgctctttt 360
gggtgggcca gccctaccca actctctcaa ctccactatc aaagaggact agtggtttcc 420
cagcaaagaa aagggccctg gagctctctg gaaacagcaa aaatgagctg aaccgttcaa 480
aaaggagctg gatgtggaat cagttctttc tcctggagga atacacagga tccgattatc 540
agtatgtggg caagttacat tcagaccagg atagaggaga tggatcactt aaatatatcc 600
tttcaggaga tggagcagga gatctcttca ttattaatga aaacacaggc gacatacagg 660
ccaccaagag gctggacagg gaagaaaaac ccgtttacat ccttcgagct caagctataa 720
acagaaggac agggagaccc gtggagcccg agtctgaatt catcatcaag atccatgaca 780
tcaatgacaa tgaaccaata ttcaccaagg aggtttacac agccactgtc cctgaaatgt 840
ctgatgtcgg tacatttgtt gtccaagtca ctgcgacgga tgcagatgat ccaacatatg 900
ggaacagtgc taaagttgtc tacagtattc tacagggaca gccctatttt tcagttgaat 960
cagaaacagg tattatcaag acagctttgc tcaacatgga tcgagaaaac agggagcagt 1020
accaagtggt gattcaagcc aaggatatgg gcggccagat gggaggatta tctgggacca 1080
ccaccgtgaa catcacactg actgatgtca acgacaaccc tccccgattc ccccagagta 1140
cataccagtt taaaactcct gaatcttctc caccggggac accaattggc agaatcaaag 1200
ccagcgatgc tgatgtggga gaaaatgctg aaattgagta cagcatcaca gacggtgagg 1260
ggctggatat gtttgatgtc atcaccgacc aggaaaccca ggaagggatt ataactgtca 1320
aaaagctctt ggactttgaa aagaagaaag tgtataccct taaagtggaa gcctccaatc 1380
cttatgttga gccacgattt ctctacttgg ggcctttcaa agattcagcc acggttagaa 1440
ttgtggtgga ggatgtagat gagccacctg tcttcagcaa actggcctac atcttacaaa 1500
taagagaaga tgctcagata aacaccacaa taggctccgt cacagcccaa gatccagatg 1560
ctgccaggaa tcctgtcaag tactctgtag atcgacacac agatatggac agaatattca 1620
acattgattc tggaaatggt tcgattttta catcgaaact tcttgaccga gaaacactgc 1680
tatggcacaa cattacagtg atagcaacag agatcaataa tccaaagcaa agtagtcgag 1740
tacctctata tattaaagtt ctagatgtca atgacaacgc cccagaattt gctgagttct 1800
atgaaacttt tgtctgtgaa aaagcaaagg cagatcagtt gattcagacc ttgcatgctg 1860
ttgacaagga tgacccttat agtgggcacc aattttcgtt ttccttggcc cctgaagcag 1920
ccagtggctc aaactttacc attcaagaca acaaagacaa cacggcggga atcttaactc 1980
ggaaaaatgg ctataataga cacgagatga gcacctatct cttgcctgtg gtcatttcag 2040
acaacgacta cccagttcaa agcagcactg ggacagtgac tgtccgggtc tgtgcatgtg 2100
accaccacgg gaacatgcaa tcctgccacg cggaggcgct catccacccc acgggactga 2160
gcacgggggc tctggttgcc atccttctgt gcatcgtgat cctactagtg acagtggtgc 2220
tgtttgcagc tctgaggcgg cagcgaaaaa aagagccttt gatcatttcc aaagaggaca 2280
tcagagataa cattgtcagt tacaacgacg aaggtggtgg agaggaggac acccaggctt 2340
ttgatatcgg caccctgagg aatcctgaag ccatagagga caacaaatta cgaagggaca 2400
ttgtgcccga agcccttttc ctaccccgac ggactccaac agctcgcgac aacaccgatg 2460
tcagagattt cattaaccaa aggttaaagg aaaatgacac ggaccccact gccccgccat 2520
acgactcctt ggccacttac gcctatgaag gcactggctc cgtggcggat tccctgagct 2580
cgctggagtc agtgaccacg gatgcagatc aagactatga ttaccttagt gactggggac 2640
ctcgattcaa aaagcttgca gatatgtatg gaggagtgga cagtgacaaa gactcctaat 2700
ctgttgcctt tttcattttc caatacgaca ctgaaatatg tgaagtggct atttctttat 2760
atttatccac tactccgtga aggcttctct gttctacccg ttccaaaagc caatggctgc 2820
agtccgtgtg gatccaatgt tagagacttt tttctagtac acttttatga gcttccaagg 2880
ggcaaatttt tattttttag tgcatccagt taaccaagtc agcccaacag gcaggtgccg 2940
gaggggagga cagggaacag tatttccact tgttctcagg gcagcgtg 2988
<210> 2
<211> 790
<212> PRT
<213> Homo sapiens
<400> 2
Met Arg Thr Tyr Arg Tyr Phe Leu Leu Leu Phe Trp Val Gly Gln Pro
1 5 10 15
Tyr Pro Thr Leu Ser Thr Pro Leu Ser Lys Arg Thr Ser Gly Phe Pro
20 25 30
Ala Lys Lys Arg Ala Leu Glu Leu Ser Gly Asn Ser Lys Asn Glu Leu
35 40 45
Asn Arg Ser Lys Arg Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu
50 55 60
Glu Tyr Thr Gly Ser Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp
65 70 75 80
Gln Asp Arg Gly Asp Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly
85 90 95
Ala Gly Asp Leu Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
100 105 110
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
115 120 125
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
130 135 140
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
145 150 155 160
Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr
165 170 175
Phe Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly
180 185 190
Asn Ser Ala Lys Val Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe
195 200 205
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met
210 215 220
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp
225 230 235 240
Met Gly Gly Gln Met Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile
245 250 255
Thr Leu Thr Asp Val Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr
260 265 270
Tyr Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly
275 280 285
Arg Ile Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu
290 295 300
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr
305 310 315 320
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
325 330 335
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
340 345 350
Tyr Val Glu Pro Arg Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala
355 360 365
Thr Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser
370 375 380
Lys Leu Ala Tyr Ile Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr
385 390 395 400
Thr Ile Gly Ser Val Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro
405 410 415
Val Lys Tyr Ser Val Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn
420 425 430
Ile Asp Ser Gly Asn Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg
435 440 445
Glu Thr Leu Leu Trp His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn
450 455 460
Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp
465 470 475 480
Val Asn Asp Asn Ala Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val
485 490 495
Cys Glu Lys Ala Lys Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val
500 505 510
Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala
515 520 525
Pro Glu Ala Ala Ser Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp
530 535 540
Asn Thr Ala Gly Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu
545 550 555 560
Met Ser Thr Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro
565 570 575
Val Gln Ser Ser Thr Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp
580 585 590
His His Gly Asn Met Gln Ser Cys His Ala Glu Ala Leu Ile His Pro
595 600 605
Thr Gly Leu Ser Thr Gly Ala Leu Val Ala Ile Leu Leu Cys Ile Val
610 615 620
Ile Leu Leu Val Thr Val Val Leu Phe Ala Ala Leu Arg Arg Gln Arg
625 630 635 640
Lys Lys Glu Pro Leu Ile Ile Ser Lys Glu Asp Ile Arg Asp Asn Ile
645 650 655
Val Ser Tyr Asn Asp Glu Gly Gly Gly Glu Glu Asp Thr Gln Ala Phe
660 665 670
Asp Ile Gly Thr Leu Arg Asn Pro Glu Ala Ile Glu Asp Asn Lys Leu
675 680 685
Arg Arg Asp Ile Val Pro Glu Ala Leu Phe Leu Pro Arg Arg Thr Pro
690 695 700
Thr Ala Arg Asp Asn Thr Asp Val Arg Asp Phe Ile Asn Gln Arg Leu
705 710 715 720
Lys Glu Asn Asp Thr Asp Pro Thr Ala Pro Pro Tyr Asp Ser Leu Ala
725 730 735
Thr Tyr Ala Tyr Glu Gly Thr Gly Ser Val Ala Asp Ser Leu Ser Ser
740 745 750
Leu Glu Ser Val Thr Thr Asp Ala Asp Gln Asp Tyr Asp Tyr Leu Ser
755 760 765
Asp Trp Gly Pro Arg Phe Lys Lys Leu Ala Asp Met Tyr Gly Gly Val
770 775 780
Asp Ser Asp Lys Asp Ser
785 790
<210> 3
<211> 233
<212> PRT
<213> Homo sapiens
<400> 3
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Glu Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Gly
195 200 205
Gly Gly Gly Ser Glu Phe Arg His Asp Ser Gly Leu Asn Asp Ile Phe
210 215 220
Glu Ala Gln Lys Ile Glu Trp His Glu
225 230
<210> 4
<211> 562
<212> PRT
<213> Homo sapiens
<400> 4
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Glu Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala
<210> 5
<211> 585
<212> PRT
<213> Homo sapiens
<220>
<221> MOD_RES
<222> (580)..(580)
<223> Lys(BIOTIN)
<400> 5
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Glu Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala Gly Ser Glu Phe Arg His Asp Ser Gly Leu Asn Asp Ile Phe
565 570 575
Glu Ala Gln Lys Ile Glu Trp His Glu
580 585
<210> 6
<211> 791
<212> PRT
<213> Homo sapiens
<400> 6
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Glu Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala Gly Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
565 570 575
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
580 585 590
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
595 600 605
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
610 615 620
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
625 630 635 640
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
645 650 655
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
660 665 670
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
675 680 685
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
690 695 700
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
705 710 715 720
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
725 730 735
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
740 745 750
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
755 760 765
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
770 775 780
Leu Ser Leu Ser Pro Gly Lys
785 790
<210> 7
<211> 585
<212> PRT
<213> Macaca fascicularis
<400> 7
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Glu Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro His Val Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu Arg Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala Gly Ser Glu Phe Arg His Asp Ser Gly Leu Asn Asp Ile Phe
565 570 575
Glu Ala Gln Lys Ile Glu Trp His Glu
580 585
<210> 8
<211> 585
<212> PRT
<213> Mus musculus
<400> 8
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Val Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Asp Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ala Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly His Glu Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro His Val Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Asp Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Arg Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Ala Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu Arg Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Ser Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala Gly Ser Glu Phe Arg His Asp Ser Gly Leu Asn Asp Ile Phe
565 570 575
Glu Ala Gln Lys Ile Glu Trp His Glu
580 585
<210> 9
<211> 585
<212> PRT
<213> Rattus norvegicus
<400> 9
Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu Glu Tyr Thr Gly Ser
1 5 10 15
Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp
20 25 30
Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu Phe
35 40 45
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
50 55 60
Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala Gln Ala Ile Asn Arg
65 70 75 80
Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu Phe Ile Ile Lys Ile
85 90 95
His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys Asp Val Tyr Thr
100 105 110
Ala Thr Val Pro Glu Met Ala Asp Val Gly Thr Phe Val Val Gln Val
115 120 125
Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val
130 135 140
Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu
145 150 155 160
Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met Asp Arg Glu Asn Arg
165 170 175
Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp Met Gly Gly Gln Met
180 185 190
Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile Thr Leu Thr Asp Val
195 200 205
Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr Tyr Gln Phe Lys Thr
210 215 220
Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser
225 230 235 240
Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp
245 250 255
Gly Glu Gly His Asp Met Phe Asp Val Ile Thr Asp Gln Glu Thr Gln
260 265 270
Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Arg
275 280 285
Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro His Ile Glu Pro Arg
290 295 300
Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val Arg Ile Val
305 310 315 320
Val Asp Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr Ile
325 330 335
Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr Thr Ile Gly Ser Val
340 345 350
Ala Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val
355 360 365
Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn
370 375 380
Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu Leu Trp
385 390 395 400
His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser
405 410 415
Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala
420 425 430
Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys
435 440 445
Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro
450 455 460
Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser
465 470 475 480
Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile
485 490 495
Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu
500 505 510
Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr
515 520 525
Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp His His Gly Asn Met
530 535 540
Gln Ser Cys His Ala Glu Ala Leu Ile His Pro Thr Gly Leu Ser Thr
545 550 555 560
Gly Ala Gly Ser Glu Phe Arg His Asp Ser Gly Leu Asn Asp Ile Phe
565 570 575
Glu Ala Gln Lys Ile Glu Trp His Glu
580 585
<210> 10
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 10
caggaaacag ctatgac 17
<210> 11
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 11
gataagcatg cgtaggagaa a 21
<210> 12
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 12
taatacgact cactataggg 20
<210> 13
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 13
ctctagcgcc accatgaaac a 21
<210> 14
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 14
Ser Gly Ser Ser Ser Asn Ile Gly Ser Gln Tyr Val Tyr
1 5 10
<210> 15
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 15
Tyr Asn Ser Glu Arg Pro Ser
1 5
<210> 16
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 16
Gln Thr Trp Asp Ala Ser Ser Gln Ser Phe Val
1 5 10
<210> 17
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 17
Ser Tyr Ala Ile Ser
1 5
<210> 18
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 18
Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 19
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 19
Lys Phe Pro Gly Arg Gly Pro Phe Ala Tyr
1 5 10
<210> 20
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 20
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Lys Phe Pro Gly Arg Gly Pro Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 21
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 21
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Gln
20 25 30
Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Tyr Asn Ser Glu Arg Pro Ser Gly Met Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Thr Trp Asp Ala Ser Ser
85 90 95
Gln Ser Phe Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 22
<211> 357
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 22
caggtgcaat tggtgcagag cggtgccgaa gtgaaaaaac cgggcagcag cgtgaaagtt 60
agctgcaaag catccggagg gacgttttct tcttacgcta tctcttgggt gcgccaggcc 120
ccgggccagg gcctcgagtg gatgggcggt atcatcccga tcttcggcac tgcgaactac 180
gcccagaaat ttcagggccg ggtgaccatt accgccgatg aaagcaccag caccgcctat 240
atggaactga gcagcctgcg cagcgaagat acggccgtgt attattgcgc gcgtaaattc 300
ccgggtcgtg gtccgttcgc ttactggggc caaggcaccc tggtgactgt tagctca 357
<210> 23
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 23
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tctcagtacg tgtactggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac tacaactctg aacgcccgag cggcatgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag acttgggacg cttcttctca gtctttcgtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 24
<211> 449
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 24
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Lys Phe Pro Gly Arg Gly Pro Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 25
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 25
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Gln
20 25 30
Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Tyr Asn Ser Glu Arg Pro Ser Gly Met Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Thr Trp Asp Ala Ser Ser
85 90 95
Gln Ser Phe Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 26
<211> 1347
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 26
caggtgcaat tggtgcagag cggtgccgaa gtgaaaaaac cgggcagcag cgtgaaagtt 60
agctgcaaag catccggagg gacgttttct tcttacgcta tctcttgggt gcgccaggcc 120
ccgggccagg gcctcgagtg gatgggcggt atcatcccga tcttcggcac tgcgaactac 180
gcccagaaat ttcagggccg ggtgaccatt accgccgatg aaagcaccag caccgcctat 240
atggaactga gcagcctgcg cagcgaagat acggccgtgt attattgcgc gcgtaaattc 300
ccgggtcgtg gtccgttcgc ttactggggc caaggcaccc tggtgactgt tagctcagcc 360
tccaccaagg gtccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaactcct ggggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccggg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatcccg ggaggagatg 1080
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtaaa 1347
<210> 27
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 27
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tctcagtacg tgtactggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac tacaactctg aacgcccgag cggcatgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag acttgggacg cttcttctca gtctttcgtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 28
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 28
Thr Gly Thr Ser Ser Asp Val Gly Ala Tyr Asn Tyr Val Ser
1 5 10
<210> 29
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 29
Gly Val Ser Lys Arg Pro Ser
1 5
<210> 30
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 30
Gln Ser Tyr Asp His Leu Leu His Val Val
1 5 10
<210> 31
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 31
Thr Tyr Gly Ile His
1 5
<210> 32
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 32
Tyr Ile His Tyr Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 33
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 33
His Ala Tyr Gly Tyr Met Asp Phe
1 5
<210> 34
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 34
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr
20 25 30
Gly Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile His Tyr Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Ala Tyr Gly Tyr Met Asp Phe Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 35
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 35
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Ala Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Gly Val Ser Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp His Leu
85 90 95
Leu His Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 36
<211> 351
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 36
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt cacctttaac acttacggta tccattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttcctac atccattact ctggttcttc tacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcatgct 300
tacggttaca tggatttctg gggccaaggc accctggtga ctgttagctc a 351
<210> 37
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 37
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc gcttacaact acgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc tacggtgttt ctaaacgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc cagtcttacg accatctgct gcatgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 38
<211> 447
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 38
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Thr Tyr
20 25 30
Gly Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile His Tyr Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Ala Tyr Gly Tyr Met Asp Phe Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 39
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 39
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Ala Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Gly Val Ser Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp His Leu
85 90 95
Leu His Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 40
<211> 1341
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 40
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt cacctttaac acttacggta tccattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttcctac atccattact ctggttcttc tacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcatgct 300
tacggttaca tggatttctg gggccaaggc accctggtga ctgttagctc agcctccacc 360
aagggtccat cggtcttccc cctggcaccc tcctccaaga gcacctctgg gggcacagcg 420
gccctgggct gcctggtcaa ggactacttc cccgaaccgg tgacggtgtc gtggaactca 480
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 540
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 600
aacgtgaatc acaagcccag caacaccaag gtggacaaga gagttgagcc caaatcttgt 660
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 720
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 780
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 840
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 900
cgggtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 960
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1020
gggcagcccc gagaaccaca ggtgtacacc ctgcccccat cccgggagga gatgaccaag 1080
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1140
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1200
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcagcagggg 1260
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 1320
ctctccctgt ctccgggtaa a 1341
<210> 41
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 41
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc gcttacaact acgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc tacggtgttt ctaaacgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc cagtcttacg accatctgct gcatgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 42
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 42
Ser Gly Ser Ser Ser Asn Ile Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 43
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 43
Arg Asp Asn Gln Arg Pro Ser
1 5
<210> 44
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 44
Ala Ala Trp Thr Ser Gly Ser Ile Gly Trp Val
1 5 10
<210> 45
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 45
Ser Tyr Ala Met Thr
1 5
<210> 46
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 46
Gly Ile Ser Gly Gly Gly Ser Asn Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 47
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 47
Gly Gly Gly Gln Tyr Phe Asp Tyr
1 5
<210> 48
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 48
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Gly Gly Gly Ser Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Gln Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 49
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 49
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Tyr Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asp Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Thr Ser Gly Ser
85 90 95
Ile Gly Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 50
<211> 351
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 50
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgcta tgacttgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccggt atctctggtg gtggttctaa cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtggtggt 300
ggtcagtact tcgattactg gggccaaggc accctggtga ctgttagctc a 351
<210> 51
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 51
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tacaactacg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac cgtgacaacc agcgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgcgct gcttggactt ctggttctat cggttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 52
<211> 447
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 52
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Gly Gly Gly Ser Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Gln Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 53
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 53
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Tyr Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asp Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Thr Ser Gly Ser
85 90 95
Ile Gly Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 54
<211> 1341
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 54
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgcta tgacttgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccggt atctctggtg gtggttctaa cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtggtggt 300
ggtcagtact tcgattactg gggccaaggc accctggtga ctgttagctc agcctccacc 360
aagggtccat cggtcttccc cctggcaccc tcctccaaga gcacctctgg gggcacagcg 420
gccctgggct gcctggtcaa ggactacttc cccgaaccgg tgacggtgtc gtggaactca 480
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 540
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 600
aacgtgaatc acaagcccag caacaccaag gtggacaaga gagttgagcc caaatcttgt 660
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 720
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 780
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 840
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 900
cgggtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 960
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1020
gggcagcccc gagaaccaca ggtgtacacc ctgcccccat cccgggagga gatgaccaag 1080
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1140
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1200
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcagcagggg 1260
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 1320
ctctccctgt ctccgggtaa a 1341
<210> 55
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 55
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tacaactacg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac cgtgacaacc agcgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgcgct gcttggactt ctggttctat cggttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 56
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 56
Arg Ala Ser Gln Thr Ile Asn Ser Tyr Leu Asn
1 5 10
<210> 57
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 57
Arg Ala Ser Asn Leu Gln Ser
1 5
<210> 58
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 58
Gln Gln Gly Asp Ser Ser Trp Thr
1 5
<210> 59
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 59
Ser Tyr Ala Ile Ser
1 5
<210> 60
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 60
Phe Ile Lys Ser Asn Ala Asp Gly Tyr Thr Thr Asn Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 61
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 61
Ile Arg Tyr Phe Arg Asn Trp Asp Tyr
1 5
<210> 62
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 62
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Lys Ser Asn Ala Asp Gly Tyr Thr Thr Asn Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ile Arg Tyr Phe Arg Asn Trp Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 63
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 63
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Thr Ile Asn Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Asp Ser Ser Trp Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 64
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 64
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct tcttacgcta tctcttgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggcttc atcaaatcta acgctgacgg ttacactact 180
aactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
atccgttact tccgtaactg ggattactgg ggccaaggca ccctggtgac tgttagctca 360
<210> 65
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 65
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gactattaac tcttacctga actggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctaccgt gcttctaacc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccagcag ggtgactctt cttggacctt tggccagggc 300
acgaaagttg aaattaaa 318
<210> 66
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 66
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Lys Ser Asn Ala Asp Gly Tyr Thr Thr Asn Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ile Arg Tyr Phe Arg Asn Trp Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 67
<211> 213
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Thr Ile Asn Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Asp Ser Ser Trp Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 68
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 68
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct tcttacgcta tctcttgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggcttc atcaaatcta acgctgacgg ttacactact 180
aactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
atccgttact tccgtaactg ggattactgg ggccaaggca ccctggtgac tgttagctca 360
gcctccacca agggtccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1080
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 69
<211> 639
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 69
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gactattaac tcttacctga actggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctaccgt gcttctaacc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccagcag ggtgactctt cttggacctt tggccagggc 300
acgaaagttg aaattaaacg tacggtggcc gctcccagcg tgttcatctt cccccccagc 360
gacgagcagc tgaagagcgg caccgccagc gtggtgtgcc tgctgaacaa cttctacccc 420
cgggaggcca aggtgcagtg gaaggtggac aacgccctgc agagcggcaa cagccaggaa 480
agcgtcaccg agcaggacag caaggactcc acctacagcc tgagcagcac cctgaccctg 540
agcaaggccg actacgagaa gcacaaggtg tacgcctgcg aggtgaccca ccagggcctg 600
tccagccccg tgaccaagag cttcaaccgg ggcgagtgt 639
<210> 70
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 70
Ser Gly Ser Ser Ser Asn Ile Gly Ser Tyr Tyr Val Ser
1 5 10
<210> 71
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 71
Tyr Asn Thr Lys Arg Pro Ser
1 5
<210> 72
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 72
Gln Ser Trp Asp Lys Leu Gly Lys Gly Tyr Val
1 5 10
<210> 73
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 73
Gly Asn Ser Ala Ala Trp Asn
1 5
<210> 74
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 74
Ile Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 75
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 75
Ser Ser Tyr Ser Gly Gly Phe Asp Tyr
1 5
<210> 76
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 76
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Gly Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Ile Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Ser Ser Tyr Ser Gly Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 77
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 77
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Tyr
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Tyr Asn Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Lys Leu Gly
85 90 95
Lys Gly Tyr Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 78
<211> 363
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 78
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc ggtaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggcatcatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgttcttctt actctggtgg tttcgattac tggggccaag gcaccctggt gactgttagc 360
tca 363
<210> 79
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 79
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tcttactacg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac tacaacacta aacgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag tcttgggaca aactgggtaa aggttacgtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 80
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 80
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Gly Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Ile Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Ser Ser Tyr Ser Gly Gly Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 81
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 81
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Tyr
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Tyr Asn Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Lys Leu Gly
85 90 95
Lys Gly Tyr Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 82
<211> 1353
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 82
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc ggtaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggcatcatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgttcttctt actctggtgg tttcgattac tggggccaag gcaccctggt gactgttagc 360
tcagcctcca ccaagggtcc atcggtcttc cccctggcac cctcctccaa gagcacctct 420
gggggcacag cggccctggg ctgcctggtc aaggactact tccccgaacc ggtgacggtg 480
tcgtggaact caggcgccct gaccagcggc gtgcacacct tcccggctgt cctacagtcc 540
tcaggactct actccctcag cagcgtggtg accgtgccct ccagcagctt gggcacccag 600
acctacatct gcaacgtgaa tcacaagccc agcaacacca aggtggacaa gagagttgag 660
cccaaatctt gtgacaaaac tcacacatgc ccaccgtgcc cagcacctga actcctgggg 720
ggaccgtcag tcttcctctt ccccccaaaa cccaaggaca ccctcatgat ctcccggacc 780
cctgaggtca catgcgtggt ggtggacgtg agccacgaag accctgaggt caagttcaac 840
tggtacgtgg acggcgtgga ggtgcataat gccaagacaa agccgcggga ggagcagtac 900
aacagcacgt accgggtggt cagcgtcctc accgtcctgc accaggactg gctgaatggc 960
aaggagtaca agtgcaaggt ctccaacaaa gccctcccag cccccatcga gaaaaccatc 1020
tccaaagcca aagggcagcc ccgagaacca caggtgtaca ccctgccccc atcccgggag 1080
gagatgacca agaaccaggt cagcctgacc tgcctggtca aaggcttcta tcccagcgac 1140
atcgccgtgg agtgggagag caatgggcag ccggagaaca actacaagac cacgcctccc 1200
gtgctggact ccgacggctc cttcttcctc tacagcaagc tcaccgtgga caagagcagg 1260
tggcagcagg ggaacgtctt ctcatgctcc gtgatgcatg aggctctgca caaccactac 1320
acgcagaaga gcctctccct gtctccgggt aaa 1353
<210> 83
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 83
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tcttactacg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac tacaacacta aacgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag tcttgggaca aactgggtaa aggttacgtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 84
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 84
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Phe Val Ser
1 5 10
<210> 85
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 85
Asp Asn Ser Asn Arg Pro Ser
1 5
<210> 86
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 86
Ser Ser Tyr Asp Ser Phe Asp His Ser Trp Val
1 5 10
<210> 87
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 87
Ser Phe Ala Met Asn
1 5
<210> 88
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 88
Val Ile Ser Ser Ser Gly Ser Asn Thr Asn Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 89
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 89
Pro Ser Tyr Phe Gln Ala Met Asp Tyr
1 5
<210> 90
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 90
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Ser Ser Gly Ser Asn Thr Asn Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Ser Tyr Phe Gln Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 91
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 91
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Phe Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Asp Ser Phe Asp
85 90 95
His Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 92
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 92
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tctttcgcta tgaactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atctcttctt ctggttctaa caccaactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtccgtct 300
tacttccagg ctatggatta ctggggccaa ggcaccctgg tgactgttag ctca 354
<210> 93
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 93
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tctaacttcg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac gacaactcta accgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgctct tcttacgact ctttcgacca ttcttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 94
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 94
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Ser Ser Gly Ser Asn Thr Asn Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Ser Tyr Phe Gln Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 95
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 95
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Phe Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Asp Ser Phe Asp
85 90 95
His Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 96
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 96
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tctttcgcta tgaactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atctcttctt ctggttctaa caccaactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtccgtct 300
tacttccagg ctatggatta ctggggccaa ggcaccctgg tgactgttag ctcagcctcc 360
accaagggtc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agagagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgggtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga ggagatgacc 1080
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg taaa 1344
<210> 97
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 97
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt tctaacttcg tgtcttggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac gacaactcta accgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgctct tcttacgact ctttcgacca ttcttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 98
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 98
Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala His
1 5 10
<210> 99
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 99
Tyr Asp His Glu Arg Pro Ser
1 5
<210> 100
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 100
Tyr Ser Arg Ala Ser Ser Asn Leu Val
1 5
<210> 101
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 101
Asp His Ala Ile Asp
1 5
<210> 102
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 102
Val Ile Ala Gly Asp Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 103
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 103
Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val
1 5 10
<210> 104
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 104
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp His
20 25 30
Ala Ile Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ala Gly Asp Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 105
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 105
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asp His Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Tyr Ser Arg Ala Ser Ser Asn Leu Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 106
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 106
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct gaccatgcta tcgactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atcgctggtg acggttctat cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgacact 300
ggtgtttacc gtgaatacat ggatgtttgg ggccaaggca ccctggtgac tgttagctca 360
<210> 107
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 107
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgatgctat cggtactaaa ttcgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacgac catgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ctactctcgt gcttcttcta acctggtgtt tggcggcggc 300
acgaagttaa ccgtccta 318
<210> 108
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 108
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp His
20 25 30
Ala Ile Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ala Gly Asp Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 109
<211> 212
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 109
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asp His Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Tyr Ser Arg Ala Ser Ser Asn Leu Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala
100 105 110
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
115 120 125
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
130 135 140
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
145 150 155 160
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
165 170 175
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
180 185 190
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
195 200 205
Thr Glu Cys Ser
210
<210> 110
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 110
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct gaccatgcta tcgactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atcgctggtg acggttctat cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgacact 300
ggtgtttacc gtgaatacat ggatgtttgg ggccaaggca ccctggtgac tgttagctca 360
gcctccacca agggtccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1080
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 111
<211> 636
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 111
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgatgctat cggtactaaa ttcgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacgac catgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ctactctcgt gcttcttcta acctggtgtt tggcggcggc 300
acgaagttaa ccgtcctagg tcagcccaag gctgccccct cggtcactct gttcccgccc 360
tcctctgagg agcttcaagc caacaaggcc acactggtgt gtctcataag tgacttctac 420
ccgggagccg tgacagtggc ctggaaggca gatagcagcc ccgtcaaggc gggagtggag 480
accaccacac cctccaaaca aagcaacaac aagtacgcgg ccagcagcta tctgagcctg 540
acgcctgagc agtggaagtc ccacagaagc tacagctgcc aggtcacgca tgaagggagc 600
accgtggaga agacagtggc ccctacagaa tgttca 636
<210> 112
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 112
Thr Gly Thr Ser Ser Asp Val Gly Arg Tyr Asn Phe Val Ser
1 5 10
<210> 113
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 113
Arg Val Ser Asn Arg Pro Ser
1 5
<210> 114
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 114
Gln Ser Trp Thr Thr Tyr Ser Asn Val Val
1 5 10
<210> 115
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 115
Ser Tyr Ala Leu Asn
1 5
<210> 116
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 116
Arg Ile Lys Ser Lys Thr Tyr Gly Gly Ser Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 117
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 117
Asp Arg Gly Gly Tyr Val Gly Phe Asp Ser
1 5 10
<210> 118
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 118
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Leu Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Tyr Gly Gly Ser Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Gly Tyr Val Gly Phe Asp Ser Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 119
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 119
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Arg Tyr
20 25 30
Asn Phe Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Arg Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Thr Thr Tyr
85 90 95
Ser Asn Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 120
<211> 363
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 120
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct tcttacgctc tgaactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aaacttacgg tggttctact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
gaccgtggtg gttacgttgg tttcgattct tggggccaag gcaccctggt gactgttagc 360
tca 363
<210> 121
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 121
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc cgttacaact tcgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc taccgtgttt ctaaccgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc cagtcttgga ctacttactc taacgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 122
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 122
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Leu Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Tyr Gly Gly Ser Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Asp Arg Gly Gly Tyr Val Gly Phe Asp Ser Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 123
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 123
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Arg Tyr
20 25 30
Asn Phe Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Arg Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Thr Thr Tyr
85 90 95
Ser Asn Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 124
<211> 1353
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 124
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct tcttacgctc tgaactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aaacttacgg tggttctact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
gaccgtggtg gttacgttgg tttcgattct tggggccaag gcaccctggt gactgttagc 360
tcagcctcca ccaagggtcc atcggtcttc cccctggcac cctcctccaa gagcacctct 420
gggggcacag cggccctggg ctgcctggtc aaggactact tccccgaacc ggtgacggtg 480
tcgtggaact caggcgccct gaccagcggc gtgcacacct tcccggctgt cctacagtcc 540
tcaggactct actccctcag cagcgtggtg accgtgccct ccagcagctt gggcacccag 600
acctacatct gcaacgtgaa tcacaagccc agcaacacca aggtggacaa gagagttgag 660
cccaaatctt gtgacaaaac tcacacatgc ccaccgtgcc cagcacctga actcctgggg 720
ggaccgtcag tcttcctctt ccccccaaaa cccaaggaca ccctcatgat ctcccggacc 780
cctgaggtca catgcgtggt ggtggacgtg agccacgaag accctgaggt caagttcaac 840
tggtacgtgg acggcgtgga ggtgcataat gccaagacaa agccgcggga ggagcagtac 900
aacagcacgt accgggtggt cagcgtcctc accgtcctgc accaggactg gctgaatggc 960
aaggagtaca agtgcaaggt ctccaacaaa gccctcccag cccccatcga gaaaaccatc 1020
tccaaagcca aagggcagcc ccgagaacca caggtgtaca ccctgccccc atcccgggag 1080
gagatgacca agaaccaggt cagcctgacc tgcctggtca aaggcttcta tcccagcgac 1140
atcgccgtgg agtgggagag caatgggcag ccggagaaca actacaagac cacgcctccc 1200
gtgctggact ccgacggctc cttcttcctc tacagcaagc tcaccgtgga caagagcagg 1260
tggcagcagg ggaacgtctt ctcatgctcc gtgatgcatg aggctctgca caaccactac 1320
acgcagaaga gcctctccct gtctccgggt aaa 1353
<210> 125
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 125
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc cgttacaact tcgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc taccgtgttt ctaaccgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc cagtcttgga ctacttactc taacgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 126
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 126
Ser Gly Asp Ser Ile Gly Ser Lys Tyr Ala Gln
1 5 10
<210> 127
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 127
Tyr Asn Ser Glu Arg Pro Ser
1 5
<210> 128
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 128
Gln Ser Trp Asp Gly Gln Ser Thr Ile Arg Val
1 5 10
<210> 129
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 129
Arg Tyr Trp Met Asp
1 5
<210> 130
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 130
Arg Ile Lys Ser Lys Ala Asn Gly Gly Ile Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 131
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 131
Gly Met Thr Phe Leu Gly Ile
1 5
<210> 132
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 132
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Trp Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Ala Asn Gly Gly Ile Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Gly Met Thr Phe Leu Gly Ile Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 133
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 133
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Ser Ile Gly Ser Lys Tyr Ala
20 25 30
Gln Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asn Ser Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Gly Gln Ser Thr Ile
85 90 95
Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 134
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 134
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct cgttactgga tggactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aagctaacgg tggtatcact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
ggtatgactt tcctgggtat ctggggccaa ggcaccctgg tgactgttag ctca 354
<210> 135
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 135
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgattctat cggttctaaa tacgctcagt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacaac tctgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gacggtcagt ctactatccg tgtgtttggc 300
ggcggcacga agttaaccgt ccta 324
<210> 136
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 136
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Trp Met Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Ala Asn Gly Gly Ile Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Gly Met Thr Phe Leu Gly Ile Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 137
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 137
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Ser Ile Gly Ser Lys Tyr Ala
20 25 30
Gln Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asn Ser Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Gly Gln Ser Thr Ile
85 90 95
Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys Ser
210
<210> 138
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 138
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt caccttttct cgttactgga tggactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aagctaacgg tggtatcact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
ggtatgactt tcctgggtat ctggggccaa ggcaccctgg tgactgttag ctcagcctcc 360
accaagggtc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agagagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgggtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga ggagatgacc 1080
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg taaa 1344
<210> 139
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 139
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgattctat cggttctaaa tacgctcagt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacaac tctgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gacggtcagt ctactatccg tgtgtttggc 300
ggcggcacga agttaaccgt cctaggtcag cccaaggctg ccccctcggt cactctgttc 360
ccgccctcct ctgaggagct tcaagccaac aaggccacac tggtgtgtct cataagtgac 420
ttctacccgg gagccgtgac agtggcctgg aaggcagata gcagccccgt caaggcggga 480
gtggagacca ccacaccctc caaacaaagc aacaacaagt acgcggccag cagctatctg 540
agcctgacgc ctgagcagtg gaagtcccac agaagctaca gctgccaggt cacgcatgaa 600
gggagcaccg tggagaagac agtggcccct acagaatgtt ca 642
<210> 140
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 140
Arg Ala Ser Gln Ser Ile Ser Phe Tyr Leu Ala
1 5 10
<210> 141
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 141
Gly Ala Ser Thr Leu Gln Ser
1 5
<210> 142
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 142
His Gln Tyr Ser Tyr Trp Leu Arg Thr
1 5
<210> 143
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 143
Ser Tyr Ala Leu His
1 5
<210> 144
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 144
Tyr Ile Phe Tyr Asp Ser Ser Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 145
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 145
Phe Leu Tyr Ser Ala Tyr Gly Val Ala Asn
1 5 10
<210> 146
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 146
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Leu His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Phe Tyr Asp Ser Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Leu Tyr Ser Ala Tyr Gly Val Ala Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 147
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 147
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Phe Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys His Gln Tyr Ser Tyr Trp Leu Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 148
<211> 357
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 148
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgctc tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttcctac atcttctacg actcttcttc tacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtttcctg 300
tactctgctt acggtgttgc taactggggc caaggcaccc tggtgactgt tagctca 357
<210> 149
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 149
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gtctatttct ttctacctgg cttggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctacggt gcttctactc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccatcag tactcttact ggctgcgtac ctttggccag 300
ggcacgaaag ttgaaattaa a 321
<210> 150
<211> 449
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 150
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Leu His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Phe Tyr Asp Ser Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Leu Tyr Ser Ala Tyr Gly Val Ala Asn Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 151
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 151
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Phe Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys His Gln Tyr Ser Tyr Trp Leu Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 152
<211> 1347
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 152
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgctc tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttcctac atcttctacg actcttcttc tacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtttcctg 300
tactctgctt acggtgttgc taactggggc caaggcaccc tggtgactgt tagctcagcc 360
tccaccaagg gtccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaactcct ggggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccggg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatcccg ggaggagatg 1080
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtaaa 1347
<210> 153
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 153
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gtctatttct ttctacctgg cttggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctacggt gcttctactc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccatcag tactcttact ggctgcgtac ctttggccag 300
ggcacgaaag ttgaaattaa acgtacggtg gccgctccca gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gaaagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 154
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 154
Arg Ala Ser Gln Gly Ile Phe Thr Tyr Leu Asn
1 5 10
<210> 155
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 155
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 156
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 156
Gln Gln Tyr Tyr Ser Thr Ser Leu Thr
1 5
<210> 157
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 157
Ser Asn Ser Ala Ala Trp Asn
1 5
<210> 158
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 158
Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 159
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 159
Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr
1 5 10
<210> 160
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 160
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 161
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 161
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Phe Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Thr Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 162
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 162
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaacgtt cttaccgtga ctacttcgat tactggggcc aaggcaccct ggtgactgtt 360
agctca 366
<210> 163
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 163
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gggtattttc acttacctga actggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctctgct gcttctactc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccagcag tactactcta cttctctgac ctttggccag 300
ggcacgaaag ttgaaattaa a 321
<210> 164
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 164
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 165
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 165
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Phe Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Thr Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 166
<211> 1356
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 166
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaacgtt cttaccgtga ctacttcgat tactggggcc aaggcaccct ggtgactgtt 360
agctcagcct ccaccaaggg tccatcggtc ttccccctgg caccctcctc caagagcacc 420
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga accggtgacg 480
gtgtcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 540
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 600
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 660
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 720
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 780
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 840
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 900
tacaacagca cgtaccgggt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 960
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1020
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1080
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1140
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1200
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 1260
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1320
tacacgcaga agagcctctc cctgtctccg ggtaaa 1356
<210> 167
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 167
gatatccaga tgacccagag cccgagcagc ctgagcgcca gcgtgggcga tcgcgtgacc 60
attacctgca gagccagcca gggtattttc acttacctga actggtacca gcagaaaccg 120
ggcaaagcgc cgaaactatt aatctctgct gcttctactc tgcaaagcgg cgtgccgagc 180
cgctttagcg gcagcggatc cggcaccgat ttcaccctga ccattagctc tctgcaaccg 240
gaagactttg cgacctatta ttgccagcag tactactcta cttctctgac ctttggccag 300
ggcacgaaag ttgaaattaa acgtacggtg gccgctccca gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gaaagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac cggggcgagt gt 642
<210> 168
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 168
Ser Gly Asp Asn Ile Arg Lys Tyr Val Val His
1 5 10
<210> 169
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 169
Arg Asp Asn Asn Arg Pro Ser
1 5
<210> 170
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 170
Gln Ser Trp Asp Ser Phe Leu Ala Val Val
1 5 10
<210> 171
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 171
Ser Tyr Ala Met His
1 5
<210> 172
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 172
Phe Ile Ser Ser Leu Gly Ser Tyr Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 173
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 173
Glu Thr Ala Gly Tyr Gly Tyr Ala Phe Asp Pro
1 5 10
<210> 174
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 174
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Phe Ile Ser Ser Leu Gly Ser Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Gly Tyr Gly Tyr Ala Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 175
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 175
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Arg Lys Tyr Val Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Arg Asp Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Ser Phe Leu Ala Val
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 176
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 176
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgcta tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccttc atctcttctc tgggttctta cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgaaact 300
gctggttacg gttacgcttt cgatccgtgg ggccaaggca ccctggtgac tgttagctca 360
<210> 177
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 177
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacat ccgtaaatac gttgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctaccgtgac aacaaccgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gactctttcc tggctgttgt gtttggcggc 300
ggcacgaagt taaccgtcct a 321
<210> 178
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 178
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Phe Ile Ser Ser Leu Gly Ser Tyr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Gly Tyr Gly Tyr Ala Phe Asp Pro Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 179
<211> 213
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 179
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Arg Lys Tyr Val Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Arg Asp Asn Asn Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Ser Phe Leu Ala Val
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Cys Ser
210
<210> 180
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 180
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct tcttacgcta tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccttc atctcttctc tgggttctta cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgaaact 300
gctggttacg gttacgcttt cgatccgtgg ggccaaggca ccctggtgac tgttagctca 360
gcctccacca agggtccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1080
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 181
<211> 639
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 181
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacat ccgtaaatac gttgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctaccgtgac aacaaccgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gactctttcc tggctgttgt gtttggcggc 300
ggcacgaagt taaccgtcct aggtcagccc aaggctgccc cctcggtcac tctgttcccg 360
ccctcctctg aggagcttca agccaacaag gccacactgg tgtgtctcat aagtgacttc 420
tacccgggag ccgtgacagt ggcctggaag gcagatagca gccccgtcaa ggcgggagtg 480
gagaccacca caccctccaa acaaagcaac aacaagtacg cggccagcag ctatctgagc 540
ctgacgcctg agcagtggaa gtcccacaga agctacagct gccaggtcac gcatgaaggg 600
agcaccgtgg agaagacagt ggcccctaca gaatgttca 639
<210> 182
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 182
Ser Gly Ser Ser Ser Asn Ile Gly Leu Asp Tyr Val Asn
1 5 10
<210> 183
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 183
Arg Asn Lys Gln Arg Pro Ser
1 5
<210> 184
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 184
Gln Ala Trp Ala Gly Arg Thr Asn Tyr Val Val
1 5 10
<210> 185
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 185
Asn Tyr Trp Ile Gly
1 5
<210> 186
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 186
Phe Ile Asp Pro Gly Val Ser Tyr Thr Arg Tyr Ser Pro Ser Phe Gln
1 5 10 15
Gly
<210> 187
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 187
Val Leu Ala His Ser Thr Glu Tyr Asn Trp Pro Ala Phe
1 5 10
<210> 188
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 188
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Asp Pro Gly Val Ser Tyr Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Val Leu Ala His Ser Thr Glu Tyr Asn Trp Pro Ala Phe Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 189
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 189
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Leu Asp
20 25 30
Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asn Lys Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Ala Gly Arg Thr
85 90 95
Asn Tyr Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 190
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 190
caggtgcaat tggtgcagag cggtgcggaa gtgaaaaaac cgggcgaaag cctgaaaatt 60
agctgcaaag gctccggata tagcttcact aactactgga tcggttgggt gcgccagatg 120
ccgggcaaag gtctcgagtg gatgggcttc atcgacccgg gtgttagcta cacccgttat 180
agcccgagct ttcagggcca ggtgaccatt agcgcggata aaagcatcag caccgcgtat 240
ctgcaatgga gcagcctgaa agcgagcgat accgcgatgt attattgcgc gcgtgttctg 300
gctcattcta ctgaatacaa ctggccggct ttctggggcc aaggcaccct ggtgactgtt 360
agctca 366
<210> 191
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 191
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt ctggactacg tgaactggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac cgtaacaaac agcgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag gcttgggctg gtcgtactaa ctacgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 192
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 192
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Phe Ile Asp Pro Gly Val Ser Tyr Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Val Leu Ala His Ser Thr Glu Tyr Asn Trp Pro Ala Phe Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 193
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 193
Asp Ile Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Leu Asp
20 25 30
Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Asn Lys Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln
65 70 75 80
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Ala Gly Arg Thr
85 90 95
Asn Tyr Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 194
<211> 1356
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 194
caggtgcaat tggtgcagag cggtgcggaa gtgaaaaaac cgggcgaaag cctgaaaatt 60
agctgcaaag gctccggata tagcttcact aactactgga tcggttgggt gcgccagatg 120
ccgggcaaag gtctcgagtg gatgggcttc atcgacccgg gtgttagcta cacccgttat 180
agcccgagct ttcagggcca ggtgaccatt agcgcggata aaagcatcag caccgcgtat 240
ctgcaatgga gcagcctgaa agcgagcgat accgcgatgt attattgcgc gcgtgttctg 300
gctcattcta ctgaatacaa ctggccggct ttctggggcc aaggcaccct ggtgactgtt 360
agctcagcct ccaccaaggg tccatcggtc ttccccctgg caccctcctc caagagcacc 420
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga accggtgacg 480
gtgtcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 540
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 600
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 660
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 720
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 780
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 840
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 900
tacaacagca cgtaccgggt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 960
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1020
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1080
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1140
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1200
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 1260
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1320
tacacgcaga agagcctctc cctgtctccg ggtaaa 1356
<210> 195
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 195
gatatcgtgc tgacccagcc gccgagcgtg agcggtgcac cgggccagcg cgtgaccatt 60
agctgtagcg gcagcagcag caacattggt ctggactacg tgaactggta ccagcagctg 120
ccgggcacgg cgccgaaact gctgatctac cgtaacaaac agcgcccgag cggcgtgccg 180
gatcgcttta gcggatccaa aagcggcacc agcgccagcc tggcgattac cggcctgcaa 240
gcagaagacg aagcggatta ttactgccag gcttgggctg gtcgtactaa ctacgttgtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 196
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 196
Thr Gly Thr Ser Ser Asp Val Gly Ser Tyr Asn Tyr Val Ser
1 5 10
<210> 197
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 197
Tyr Val Ser Asn Arg Pro Ser
1 5
<210> 198
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 198
Ala Ser Tyr Thr His Gln Gly Ser Trp Val
1 5 10
<210> 199
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 199
Thr Tyr Tyr Met His
1 5
<210> 200
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 200
Val Ile Ser Ser Asp Gly Ser Phe Thr Phe Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 201
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 201
His Gly Tyr Gly Ala Phe Asp Tyr
1 5
<210> 202
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 202
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Ser Asp Gly Ser Phe Thr Phe Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Gly Tyr Gly Ala Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 203
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 203
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Ser Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Tyr Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Tyr Thr His Gln
85 90 95
Gly Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 204
<211> 351
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 204
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct acttactaca tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atctcttctg acggttcttt caccttctat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcatggt 300
tacggtgctt tcgattactg gggccaaggc accctggtga ctgttagctc a 351
<210> 205
<211> 330
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 205
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc tcttacaact acgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc tactacgttt ctaaccgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc gcttcttaca ctcatcaggg ttcttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta 330
<210> 206
<211> 447
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 206
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Ser Asp Gly Ser Phe Thr Phe Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Gly Tyr Gly Ala Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 207
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 207
Asp Ile Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Ser Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Tyr Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Tyr Thr His Gln
85 90 95
Gly Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 208
<211> 1341
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 208
caggtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct acttactaca tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atctcttctg acggttcttt caccttctat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtcatggt 300
tacggtgctt tcgattactg gggccaaggc accctggtga ctgttagctc agcctccacc 360
aagggtccat cggtcttccc cctggcaccc tcctccaaga gcacctctgg gggcacagcg 420
gccctgggct gcctggtcaa ggactacttc cccgaaccgg tgacggtgtc gtggaactca 480
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 540
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 600
aacgtgaatc acaagcccag caacaccaag gtggacaaga gagttgagcc caaatcttgt 660
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 720
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 780
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 840
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 900
cgggtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 960
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1020
gggcagcccc gagaaccaca ggtgtacacc ctgcccccat cccgggagga gatgaccaag 1080
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1140
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1200
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcagcagggg 1260
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 1320
ctctccctgt ctccgggtaa a 1341
<210> 209
<211> 648
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 209
gatatcgcgc tgacccagcc ggcgagcgtg agcggtagcc cgggccagag cattaccatt 60
agctgcaccg gcaccagcag cgatgtgggc tcttacaact acgtgtcttg gtaccagcag 120
catccgggca aggcgccgaa actgatgatc tactacgttt ctaaccgtcc gagcggcgtg 180
agcaaccgtt ttagcggatc caaaagcggc aacaccgcga gcctgaccat tagcggcctg 240
caagcggaag acgaagcgga ttattactgc gcttcttaca ctcatcaggg ttcttgggtg 300
tttggcggcg gcacgaagtt aaccgtccta ggtcagccca aggctgcccc ctcggtcact 360
ctgttcccgc cctcctctga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 420
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 480
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 540
tatctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 600
catgaaggga gcaccgtgga gaagacagtg gcccctacag aatgttca 648
<210> 210
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 210
Arg Ala Ser Gln Ser Ile Ser Leu Trp Leu Asn
1 5 10
<210> 211
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 211
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 212
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 212
Gln Gln Tyr Tyr Thr Ser Pro Tyr Thr
1 5
<210> 213
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 213
Ser Tyr Ala Met Ser
1 5
<210> 214
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 214
Val Ile Arg Ser Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 215
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 215
Gly Gly Gly Tyr Phe Asp Tyr
1 5
<210> 216
<211> 116
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 216
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Arg Ser Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 217
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 217
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Leu Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Thr Ser Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 218
<211> 348
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 218
caggtgcagc tgctggaatc aggcggcgga ctggtgcaac ctggcggatc cctgaggctg 60
agctgcgctg ctagtggctt caccttctct agctacgcta tgagctgggt ccgccaggcc 120
cctggtaaag gcctcgagtg ggtgtcagtg attagatcta gcggctctag cacctactac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga actccctgag ggccgaggac accgccgtct actactgcgc tagaggcgga 300
ggctacttcg actactgggg tcaaggcacc ctggtcaccg tgtctagc 348
<210> 219
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 219
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctattagc ctgtggctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gcctctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctatctctag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tactacacta gcccctacac cttcggtcag 300
ggcactaagg tcgagattaa g 321
<210> 220
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 220
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Arg Ser Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 221
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 221
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Leu Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Thr Ser Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 222
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 222
caggtgcagc tgctggaatc aggcggcgga ctggtgcaac ctggcggatc cctgaggctg 60
agctgcgctg ctagtggctt caccttctct agctacgcta tgagctgggt ccgccaggcc 120
cctggtaaag gcctcgagtg ggtgtcagtg attagatcta gcggctctag cacctactac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga actccctgag ggccgaggac accgccgtct actactgcgc tagaggcgga 300
ggctacttcg actactgggg tcaaggcacc ctggtcaccg tgtctagcgc tagcactaag 360
ggcccaagtg tgtttcccct ggcccccagc agcaagtcta cttccggcgg aactgctgcc 420
ctgggttgcc tggtgaagga ctacttcccc gagcccgtga cagtgtcctg gaactctggg 480
gctctgactt ccggcgtgca caccttcccc gccgtgctgc agagcagcgg cctgtacagc 540
ctgagcagcg tggtgacagt gccctccagc tctctgggaa cccagaccta tatctgcaac 600
gtgaaccaca agcccagcaa caccaaggtg gacaagagag tggagcccaa gagctgcgac 660
aagacccaca cctgcccccc ctgcccagct ccagaactgc tgggagggcc ttccgtgttc 720
ctgttccccc ccaagcccaa ggacaccctg atgatcagca ggacccccga ggtgacctgc 780
gtggtggtgg acgtgtccca cgaggaccca gaggtgaagt tcaactggta cgtggacggc 840
gtggaggtgc acaacgccaa gaccaagccc agagaggagc agtacaacag cacctacagg 900
gtggtgtccg tgctgaccgt gctgcaccag gactggctga acggcaaaga atacaagtgc 960
aaagtctcca acaaggccct gccagcccca atcgaaaaga caatcagcaa ggccaagggc 1020
cagccacggg agccccaggt gtacaccctg ccccccagcc gggaggagat gaccaagaac 1080
caggtgtccc tgacctgtct ggtgaagggc ttctacccca gcgatatcgc cgtggagtgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc ccccagtgct ggacagcgac 1200
ggcagcttct tcctgtacag caagctgacc gtggacaagt ccaggtggca gcagggcaac 1260
gtgttcagct gcagcgtgat gcacgaggcc ctgcacaacc actacaccca gaagtccctg 1320
agcctgagcc ccggcaag 1338
<210> 223
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 223
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctattagc ctgtggctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gcctctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctatctctag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tactacacta gcccctacac cttcggtcag 300
ggcactaagg tcgagattaa gcgtacggtg gccgctccca gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcataag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac aggggcgagt gc 642
<210> 224
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 224
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 225
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 225
Ala Val Ser Thr Leu Gln Ser
1 5
<210> 226
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 226
Gln Gln Ser Gly Thr Phe Pro Pro Thr Thr
1 5 10
<210> 227
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 227
Ser His Gly Met His
1 5
<210> 228
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 228
Val Ile Ser Gly Ser Gly Ser Asn Thr Gly Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 229
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 229
Gln Trp Gly Ser Tyr Ala Phe Asp Ser
1 5
<210> 230
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 230
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Gly Ser Gly Ser Asn Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gln Trp Gly Ser Tyr Ala Phe Asp Ser Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 231
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 231
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Val Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Gly Thr Phe Pro Pro
85 90 95
Thr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 232
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 232
caggtgcagc tgctggaatc aggcggcgga ctggtgcagc ctggcggatc cctgaggctg 60
agctgcgctg ctagtggctt cacctttagc tctcacggaa tgcactgggt ccgccaggcc 120
cctggtaaag gcctcgagtg ggtgtcagtg attagcggta gcggctctaa caccggctac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga actccctgag ggccgaggac accgccgtct actactgcgc tagacagtgg 300
ggctcctacg ccttcgatag ctggggtcaa ggcaccctgg tcaccgtgtc tagc 354
<210> 233
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 233
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctatctct agctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gtgtctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctattagtag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tcaggcacct tcccccctac taccttcggt 300
cagggcacta aggtcgagat taag 324
<210> 234
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 234
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Gly Ser Gly Ser Asn Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gln Trp Gly Ser Tyr Ala Phe Asp Ser Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 235
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 235
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Val Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Gly Thr Phe Pro Pro
85 90 95
Thr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 236
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 236
caggtgcagc tgctggaatc aggcggcgga ctggtgcagc ctggcggatc cctgaggctg 60
agctgcgctg ctagtggctt cacctttagc tctcacggaa tgcactgggt ccgccaggcc 120
cctggtaaag gcctcgagtg ggtgtcagtg attagcggta gcggctctaa caccggctac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga actccctgag ggccgaggac accgccgtct actactgcgc tagacagtgg 300
ggctcctacg ccttcgatag ctggggtcaa ggcaccctgg tcaccgtgtc tagcgctagc 360
actaagggcc caagtgtgtt tcccctggcc cccagcagca agtctacttc cggcggaact 420
gctgccctgg gttgcctggt gaaggactac ttccccgagc ccgtgacagt gtcctggaac 480
tctggggctc tgacttccgg cgtgcacacc ttccccgccg tgctgcagag cagcggcctg 540
tacagcctga gcagcgtggt gacagtgccc tccagctctc tgggaaccca gacctatatc 600
tgcaacgtga accacaagcc cagcaacacc aaggtggaca agagagtgga gcccaagagc 660
tgcgacaaga cccacacctg ccccccctgc ccagctccag aactgctggg agggccttcc 720
gtgttcctgt tcccccccaa gcccaaggac accctgatga tcagcaggac ccccgaggtg 780
acctgcgtgg tggtggacgt gtcccacgag gacccagagg tgaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcacaa cgccaagacc aagcccagag aggagcagta caacagcacc 900
tacagggtgg tgtccgtgct gaccgtgctg caccaggact ggctgaacgg caaagaatac 960
aagtgcaaag tctccaacaa ggccctgcca gccccaatcg aaaagacaat cagcaaggcc 1020
aagggccagc cacgggagcc ccaggtgtac accctgcccc ccagccggga ggagatgacc 1080
aagaaccagg tgtccctgac ctgtctggtg aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcccgagaac aactacaaga ccaccccccc agtgctggac 1200
agcgacggca gcttcttcct gtacagcaag ctgaccgtgg acaagtccag gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg caag 1344
<210> 237
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 237
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctatctct agctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gtgtctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctattagtag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tcaggcacct tcccccctac taccttcggt 300
cagggcacta aggtcgagat taagcgtacg gtggccgctc ccagcgtgtt catcttcccc 360
cccagcgacg agcagctgaa gagcggcacc gccagcgtgg tgtgcctgct gaacaacttc 420
tacccccggg aggccaaggt gcagtggaag gtggacaacg ccctgcagag cggcaacagc 480
caggagagcg tcaccgagca ggacagcaag gactccacct acagcctgag cagcaccctg 540
accctgagca aggccgacta cgagaagcat aaggtgtacg cctgcgaggt gacccaccag 600
ggcctgtcca gccccgtgac caagagcttc aacaggggcg agtgc 645
<210> 238
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 238
Ser Gly Asp Asn Leu Arg Ser Tyr Tyr Val His
1 5 10
<210> 239
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 239
Gly Asn Asn Lys Arg Pro Ser
1 5
<210> 240
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 240
Gly Val Tyr Thr Leu Ser Ser Val Val
1 5
<210> 241
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 241
Ser Asn Ser Ala Ala Trp Asn
1 5
<210> 242
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 242
Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 243
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 243
Gly Leu Val Gly Arg Tyr Gly Gln Pro Tyr His Phe Asp Val
1 5 10
<210> 244
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 244
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Gly Leu Val Gly Arg Tyr Gly Gln Pro Tyr His
100 105 110
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 245
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 245
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Leu Arg Ser Tyr Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asn Asn Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gly Val Tyr Thr Leu Ser Ser Val Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 246
<211> 378
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 246
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtggtctgg ttggtcgtta cggtcagccg taccatttcg atgtttgggg ccaaggcacc 360
ctggtgactg ttagctca 378
<210> 247
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 247
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacct gcgttcttac tacgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacggtaac aacaaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg cggtgtttac actctgtctt ctgttgtgtt tggcggcggc 300
acgaagttaa ccgtccta 318
<210> 248
<211> 456
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 248
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Gly Leu Val Gly Arg Tyr Gly Gln Pro Tyr His
100 105 110
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 249
<211> 212
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 249
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Leu Arg Ser Tyr Tyr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Asn Asn Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gly Val Tyr Thr Leu Ser Ser Val Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala
100 105 110
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
115 120 125
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
130 135 140
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
145 150 155 160
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
165 170 175
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
180 185 190
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
195 200 205
Thr Glu Cys Ser
210
<210> 250
<211> 1368
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 250
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctgcttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtggtctgg ttggtcgtta cggtcagccg taccatttcg atgtttgggg ccaaggcacc 360
ctggtgactg ttagctcagc ctccaccaag ggtccatcgg tcttccccct ggcaccctcc 420
tccaagagca cctctggggg cacagcggcc ctgggctgcc tggtcaagga ctacttcccc 480
gaaccggtga cggtgtcgtg gaactcaggc gccctgacca gcggcgtgca caccttcccg 540
gctgtcctac agtcctcagg actctactcc ctcagcagcg tggtgaccgt gccctccagc 600
agcttgggca cccagaccta catctgcaac gtgaatcaca agcccagcaa caccaaggtg 660
gacaagagag ttgagcccaa atcttgtgac aaaactcaca catgcccacc gtgcccagca 720
cctgaactcc tggggggacc gtcagtcttc ctcttccccc caaaacccaa ggacaccctc 780
atgatctccc ggacccctga ggtcacatgc gtggtggtgg acgtgagcca cgaagaccct 840
gaggtcaagt tcaactggta cgtggacggc gtggaggtgc ataatgccaa gacaaagccg 900
cgggaggagc agtacaacag cacgtaccgg gtggtcagcg tcctcaccgt cctgcaccag 960
gactggctga atggcaagga gtacaagtgc aaggtctcca acaaagccct cccagccccc 1020
atcgagaaaa ccatctccaa agccaaaggg cagccccgag aaccacaggt gtacaccctg 1080
cccccatccc gggaggagat gaccaagaac caggtcagcc tgacctgcct ggtcaaaggc 1140
ttctatccca gcgacatcgc cgtggagtgg gagagcaatg ggcagccgga gaacaactac 1200
aagaccacgc ctcccgtgct ggactccgac ggctccttct tcctctacag caagctcacc 1260
gtggacaaga gcaggtggca gcaggggaac gtcttctcat gctccgtgat gcatgaggct 1320
ctgcacaacc actacacgca gaagagcctc tccctgtctc cgggtaaa 1368
<210> 251
<211> 636
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 251
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacct gcgttcttac tacgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacggtaac aacaaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg cggtgtttac actctgtctt ctgttgtgtt tggcggcggc 300
acgaagttaa ccgtcctagg tcagcccaag gctgccccct cggtcactct gttcccgccc 360
tcctctgagg agcttcaagc caacaaggcc acactggtgt gtctcataag tgacttctac 420
ccgggagccg tgacagtggc ctggaaggca gatagcagcc ccgtcaaggc gggagtggag 480
accaccacac cctccaaaca aagcaacaac aagtacgcgg ccagcagcta tctgagcctg 540
acgcctgagc agtggaagtc ccacagaagc tacagctgcc aggtcacgca tgaagggagc 600
accgtggaga agacagtggc ccctacagaa tgttca 636
<210> 252
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 252
Ser Gly Asp Lys Ile Pro Thr Tyr Thr Val His
1 5 10
<210> 253
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 253
Asp Asp Asn Lys Arg Pro Ser
1 5
<210> 254
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 254
Gln Ser Thr Ala Ser Gly Thr Val Val
1 5
<210> 255
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 255
Ser Tyr Ala Leu His
1 5
<210> 256
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 256
Arg Ile Lys Ser Lys Thr Asn Gly Gly Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 257
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 257
Val Asp Ala Thr Tyr Ser Tyr Ser Gly Tyr Tyr Tyr Pro Met Asp Tyr
1 5 10 15
<210> 258
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 258
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Ser Tyr
20 25 30
Ala Leu His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asn Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Val Asp Ala Thr Tyr Ser Tyr Ser Gly Tyr Tyr Tyr
100 105 110
Pro Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 259
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 259
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Lys Ile Pro Thr Tyr Thr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Asp Asn Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Thr Ala Ser Gly Thr Val Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 260
<211> 381
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 260
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt cacctttaac tcttacgctc tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aaactaacgg tggtactact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
gttgacgcta cttactctta ctctggttac tactacccga tggattactg gggccaaggc 360
accctggtga ctgttagctc a 381
<210> 261
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 261
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataaaat cccgacttac actgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacgac aacaaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtctact gcttctggta ctgttgtgtt tggcggcggc 300
acgaagttaa ccgtccta 318
<210> 262
<211> 457
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 262
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Ser Tyr
20 25 30
Ala Leu His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asn Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Val Asp Ala Thr Tyr Ser Tyr Ser Gly Tyr Tyr Tyr
100 105 110
Pro Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 263
<211> 212
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 263
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Lys Ile Pro Thr Tyr Thr Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Asp Asn Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Thr Ala Ser Gly Thr Val Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala
100 105 110
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
115 120 125
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
130 135 140
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
145 150 155 160
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
165 170 175
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
180 185 190
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
195 200 205
Thr Glu Cys Ser
210
<210> 264
<211> 1371
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 264
caggtgcaat tggtggaaag cggcggtggc ctggtgaaac caggcggcag cctgcgcctg 60
agctgcgccg cctccggatt cacctttaac tcttacgctc tgcattgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtgggccgt atcaaatcta aaactaacgg tggtactact 180
gactatgccg ccccagtgaa aggccgcttt accattagcc gcgatgattc gaaaaacacc 240
ctgtatctgc aaatgaacag cctgaaaacc gaagatacgg ccgtgtatta ttgcgcgcgt 300
gttgacgcta cttactctta ctctggttac tactacccga tggattactg gggccaaggc 360
accctggtga ctgttagctc agcctccacc aagggtccat cggtcttccc cctggcaccc 420
tcctccaaga gcacctctgg gggcacagcg gccctgggct gcctggtcaa ggactacttc 480
cccgaaccgg tgacggtgtc gtggaactca ggcgccctga ccagcggcgt gcacaccttc 540
ccggctgtcc tacagtcctc aggactctac tccctcagca gcgtggtgac cgtgccctcc 600
agcagcttgg gcacccagac ctacatctgc aacgtgaatc acaagcccag caacaccaag 660
gtggacaaga gagttgagcc caaatcttgt gacaaaactc acacatgccc accgtgccca 720
gcacctgaac tcctgggggg accgtcagtc ttcctcttcc ccccaaaacc caaggacacc 780
ctcatgatct cccggacccc tgaggtcaca tgcgtggtgg tggacgtgag ccacgaagac 840
cctgaggtca agttcaactg gtacgtggac ggcgtggagg tgcataatgc caagacaaag 900
ccgcgggagg agcagtacaa cagcacgtac cgggtggtca gcgtcctcac cgtcctgcac 960
caggactggc tgaatggcaa ggagtacaag tgcaaggtct ccaacaaagc cctcccagcc 1020
cccatcgaga aaaccatctc caaagccaaa gggcagcccc gagaaccaca ggtgtacacc 1080
ctgcccccat cccgggagga gatgaccaag aaccaggtca gcctgacctg cctggtcaaa 1140
ggcttctatc ccagcgacat cgccgtggag tgggagagca atgggcagcc ggagaacaac 1200
tacaagacca cgcctcccgt gctggactcc gacggctcct tcttcctcta cagcaagctc 1260
accgtggaca agagcaggtg gcagcagggg aacgtcttct catgctccgt gatgcatgag 1320
gctctgcaca accactacac gcagaagagc ctctccctgt ctccgggtaa a 1371
<210> 265
<211> 636
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 265
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataaaat cccgacttac actgttcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacgac aacaaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtctact gcttctggta ctgttgtgtt tggcggcggc 300
acgaagttaa ccgtcctagg tcagcccaag gctgccccct cggtcactct gttcccgccc 360
tcctctgagg agcttcaagc caacaaggcc acactggtgt gtctcataag tgacttctac 420
ccgggagccg tgacagtggc ctggaaggca gatagcagcc ccgtcaaggc gggagtggag 480
accaccacac cctccaaaca aagcaacaac aagtacgcgg ccagcagcta tctgagcctg 540
acgcctgagc agtggaagtc ccacagaagc tacagctgcc aggtcacgca tgaagggagc 600
accgtggaga agacagtggc ccctacagaa tgttca 636
<210> 266
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 266
Arg Ala Ser Gln Ser Ile Val Ser Tyr Leu Asn
1 5 10
<210> 267
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 267
Asp Ala Ser Ser Leu Gln Ser
1 5
<210> 268
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 268
Gln Gln Ser Gly Ser His Ser Ile Thr
1 5
<210> 269
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 269
Ser His Trp Val His
1 5
<210> 270
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 270
Val Ile Ser Tyr Met Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 271
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 271
Gly Ser Tyr Asp Met Ala Phe Asp Val
1 5
<210> 272
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 272
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Trp Val His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Tyr Met Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Tyr Asp Met Ala Phe Asp Val Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 273
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 273
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Val Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Gly Ser His Ser Ile
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 274
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 274
caggtgcagc tgctggaatc aggcggcgga ctggtgcagc ctggcggtag cctgagactg 60
agctgcgctg ctagtggctt cacctttagc tctcactggg tgcactgggt cagacaggcc 120
cctggtaaag gcctggagtg ggtgtcagtg attagctata tgggctctag cacctactac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga atagcctgag agccgaggac accgccgtct actactgcgc tagaggctcc 300
tacgatatgg ccttcgacgt gtggggtcag ggcaccctgg tcaccgtgtc tagc 354
<210> 275
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 275
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctatcgtc agctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgac gcctctagcc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctattagtag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tcaggctctc actctatcac cttcggtcag 300
ggcactaagg tcgagattaa g 321
<210> 276
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 276
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Trp Val His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Tyr Met Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ser Tyr Asp Met Ala Phe Asp Val Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 277
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 277
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Val Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Gly Ser His Ser Ile
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 278
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 278
caggtgcagc tgctggaatc aggcggcgga ctggtgcagc ctggcggtag cctgagactg 60
agctgcgctg ctagtggctt cacctttagc tctcactggg tgcactgggt cagacaggcc 120
cctggtaaag gcctggagtg ggtgtcagtg attagctata tgggctctag cacctactac 180
gccgatagcg tgaagggccg gttcactatc tctagggata actctaagaa caccctgtac 240
ctgcagatga atagcctgag agccgaggac accgccgtct actactgcgc tagaggctcc 300
tacgatatgg ccttcgacgt gtggggtcag ggcaccctgg tcaccgtgtc tagcgctagc 360
actaagggcc caagtgtgtt tcccctggcc cccagcagca agtctacttc cggcggaact 420
gctgccctgg gttgcctggt gaaggactac ttccccgagc ccgtgacagt gtcctggaac 480
tctggggctc tgacttccgg cgtgcacacc ttccccgccg tgctgcagag cagcggcctg 540
tacagcctga gcagcgtggt gacagtgccc tccagctctc tgggaaccca gacctatatc 600
tgcaacgtga accacaagcc cagcaacacc aaggtggaca agagagtgga gcccaagagc 660
tgcgacaaga cccacacctg ccccccctgc ccagctccag aactgctggg agggccttcc 720
gtgttcctgt tcccccccaa gcccaaggac accctgatga tcagcaggac ccccgaggtg 780
acctgcgtgg tggtggacgt gtcccacgag gacccagagg tgaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcacaa cgccaagacc aagcccagag aggagcagta caacagcacc 900
tacagggtgg tgtccgtgct gaccgtgctg caccaggact ggctgaacgg caaagaatac 960
aagtgcaaag tctccaacaa ggccctgcca gccccaatcg aaaagacaat cagcaaggcc 1020
aagggccagc cacgggagcc ccaggtgtac accctgcccc ccagccggga ggagatgacc 1080
aagaaccagg tgtccctgac ctgtctggtg aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcccgagaac aactacaaga ccaccccccc agtgctggac 1200
agcgacggca gcttcttcct gtacagcaag ctgaccgtgg acaagtccag gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg caag 1344
<210> 279
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 279
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gtctatcgtc agctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgac gcctctagcc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctattagtag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tcaggctctc actctatcac cttcggtcag 300
ggcactaagg tcgagattaa gcgtacggtg gccgctccca gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcataag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac aggggcgagt gc 642
<210> 280
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 280
Ser Gly Asp Asn Ile Gly Ser Met Thr Ala His
1 5 10
<210> 281
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 281
Asp Lys Asn Glu Arg Pro Ser
1 5
<210> 282
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 282
Gln Ser Trp Asp Asp Ser Tyr Asn Ser Val Val
1 5 10
<210> 283
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 283
Ser Asn Ser Ala Gly Trp Asn
1 5
<210> 284
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 284
Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 285
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 285
Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe Asp Tyr
1 5 10
<210> 286
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 286
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Gly Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 287
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 287
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Met Thr Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Lys Asn Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Asp Ser Tyr Asn Ser
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 288
<211> 375
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 288
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctggttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaaaaat acactgtttc tttctacgac ttcttcgatt actggggcca aggcaccctg 360
gtgactgtta gctca 375
<210> 289
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 289
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacat cggttctatg actgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacaaa aacgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gacgactctt acaactctgt tgtgtttggc 300
ggcggcacga agttaaccgt ccta 324
<210> 290
<211> 455
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 290
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Asn
20 25 30
Ser Ala Gly Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 291
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 291
Asp Ile Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Met Thr Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Lys Asn Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Asp Ser Tyr Asn Ser
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys Ser
210
<210> 292
<211> 1365
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 292
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctaactctg ctggttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaaaaat acactgtttc tttctacgac ttcttcgatt actggggcca aggcaccctg 360
gtgactgtta gctcagcctc caccaagggt ccatcggtct tccccctggc accctcctcc 420
aagagcacct ctgggggcac agcggccctg ggctgcctgg tcaaggacta cttccccgaa 480
ccggtgacgg tgtcgtggaa ctcaggcgcc ctgaccagcg gcgtgcacac cttcccggct 540
gtcctacagt cctcaggact ctactccctc agcagcgtgg tgaccgtgcc ctccagcagc 600
ttgggcaccc agacctacat ctgcaacgtg aatcacaagc ccagcaacac caaggtggac 660
aagagagttg agcccaaatc ttgtgacaaa actcacacat gcccaccgtg cccagcacct 720
gaactcctgg ggggaccgtc agtcttcctc ttccccccaa aacccaagga caccctcatg 780
atctcccgga cccctgaggt cacatgcgtg gtggtggacg tgagccacga agaccctgag 840
gtcaagttca actggtacgt ggacggcgtg gaggtgcata atgccaagac aaagccgcgg 900
gaggagcagt acaacagcac gtaccgggtg gtcagcgtcc tcaccgtcct gcaccaggac 960
tggctgaatg gcaaggagta caagtgcaag gtctccaaca aagccctccc agcccccatc 1020
gagaaaacca tctccaaagc caaagggcag ccccgagaac cacaggtgta caccctgccc 1080
ccatcccggg aggagatgac caagaaccag gtcagcctga cctgcctggt caaaggcttc 1140
tatcccagcg acatcgccgt ggagtgggag agcaatgggc agccggagaa caactacaag 1200
accacgcctc ccgtgctgga ctccgacggc tccttcttcc tctacagcaa gctcaccgtg 1260
gacaagagca ggtggcagca ggggaacgtc ttctcatgct ccgtgatgca tgaggctctg 1320
cacaaccact acacgcagaa gagcctctcc ctgtctccgg gtaaa 1365
<210> 293
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 293
gatatcgaac tgacccagcc gccgagcgtg agcgtgagcc cgggccagac cgcgagcatt 60
acctgtagcg gcgataacat cggttctatg actgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacaaa aacgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagcggcac ccaggcggaa 240
gacgaagcgg attattactg ccagtcttgg gacgactctt acaactctgt tgtgtttggc 300
ggcggcacga agttaaccgt cctaggtcag cccaaggctg ccccctcggt cactctgttc 360
ccgccctcct ctgaggagct tcaagccaac aaggccacac tggtgtgtct cataagtgac 420
ttctacccgg gagccgtgac agtggcctgg aaggcagata gcagccccgt caaggcggga 480
gtggagacca ccacaccctc caaacaaagc aacaacaagt acgcggccag cagctatctg 540
agcctgacgc ctgagcagtg gaagtcccac agaagctaca gctgccaggt cacgcatgaa 600
gggagcaccg tggagaagac agtggcccct acagaatgtt ca 642
<210> 294
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 294
Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala His
1 5 10
<210> 295
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 295
Tyr Asp His Glu Arg Pro Ser
1 5
<210> 296
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 296
Tyr Ser Arg Ala Ser Ser Asn Leu Val
1 5
<210> 297
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 297
Asp His Ala Ile Asp
1 5
<210> 298
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 298
Val Ile Ala Gly Ser Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 299
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 299
Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val
1 5 10
<210> 300
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 300
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp His
20 25 30
Ala Ile Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ala Gly Ser Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 301
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 301
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asp His Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Tyr Ser Arg Ala Ser Ser Asn Leu Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 302
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 302
gaagtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct gaccatgcta tcgactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atcgctggta gcggttctat cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgacact 300
ggtgtttacc gtgaatacat ggatgtttgg ggccaaggca ccctggtgac tgttagctca 360
<210> 303
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 303
agctatgaac tgacccagcc gctgagcgtg agcgtggcgc tgggccagac cgcgcgcatt 60
acctgtagcg gcgatgctat cggtactaaa ttcgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacgac catgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagccgcgc gcaggcgggc 240
gacgaagcgg attattactg ctactctcgt gcttcttcta acctggtgtt tggcggcggc 300
acgaagttaa ccgtccta 318
<210> 304
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 304
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp His
20 25 30
Ala Ile Asp Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ala Gly Ser Gly Ser Ile Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Val Tyr Arg Glu Tyr Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 305
<211> 212
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 305
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Ala Ile Gly Thr Lys Phe Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asp His Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Tyr Ser Arg Ala Ser Ser Asn Leu Val
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala Ala
100 105 110
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
115 120 125
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
130 135 140
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
145 150 155 160
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
165 170 175
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
180 185 190
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
195 200 205
Thr Glu Cys Ser
210
<210> 306
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 306
gaagtgcaat tgctggaaag cggcggtggc ctggtgcagc cgggtggcag cctgcgtctg 60
agctgcgcgg cgtccggatt caccttttct gaccatgcta tcgactgggt gcgccaggcc 120
ccgggcaaag gtctcgagtg ggtttccgtt atcgctggta gcggttctat cacctactat 180
gcggatagcg tgaaaggccg ctttaccatc agccgcgata attcgaaaaa caccctgtat 240
ctgcaaatga acagcctgcg tgcggaagat acggccgtgt attattgcgc gcgtgacact 300
ggtgtttacc gtgaatacat ggatgtttgg ggccaaggca ccctggtgac tgttagctca 360
gcctccacca agggtccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1080
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 307
<211> 636
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 307
agctatgaac tgacccagcc gctgagcgtg agcgtggcgc tgggccagac cgcgcgcatt 60
acctgtagcg gcgatgctat cggtactaaa ttcgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctactacgac catgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagccgcgc gcaggcgggc 240
gacgaagcgg attattactg ctactctcgt gcttcttcta acctggtgtt tggcggcggc 300
acgaagttaa ccgtcctagg tcagcccaag gctgccccct cggtcactct gttcccgccc 360
tcctctgagg agcttcaagc caacaaggcc acactggtgt gtctcataag tgacttctac 420
ccgggagccg tgacagtggc ctggaaggca gatagcagcc ccgtcaaggc gggagtggag 480
accaccacac cctccaaaca aagcaacaac aagtacgcgg ccagcagcta tctgagcctg 540
acgcctgagc agtggaagtc ccacagaagc tacagctgcc aggtcacgca tgaagggagc 600
accgtggaga agacagtggc ccctacagaa tgttca 636
<210> 308
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 308
Arg Ala Ser Gln Gly Ile Phe Thr Tyr Leu Asn
1 5 10
<210> 309
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 309
Ala Ala Ser Thr Leu Gln Ser
1 5
<210> 310
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 310
Gln Gln Tyr Tyr Ser Thr Ser Leu Thr
1 5
<210> 311
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 311
Ser Gln Ser Ala Ala Trp Asn
1 5
<210> 312
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 312
Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 313
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 313
Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr
1 5 10
<210> 314
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 314
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Gln
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 315
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 315
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Phe Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Thr Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 316
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 316
caggtgcagc tgcagcagtc aggccctggc ctggtcaagc ctagtcagac cctgagcctg 60
acctgcgcta ttagcggcga tagtgtgtct agtcagtcag ccgcctggaa ctggattaga 120
cagtcaccct ctaggggcct ggagtggctg ggtagaatct actataggtc taagtggtat 180
aacgactacg ccgtcagcgt gaagtctagg atcactatta accccgacac ctctaagaat 240
cagtttagcc tgcagctgaa tagcgtgacc cccgaggaca ccgccgtcta ctactgcgct 300
agagagcggt cctatagaga ctacttcgac tactggggtc agggcaccct ggtcaccgtg 360
tctagc 366
<210> 317
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 317
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gggaatcttc acctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gcctctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctatctctag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tactactcta ctagcctgac cttcggtcag 300
ggcactaagg tcgagattaa g 321
<210> 318
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 318
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Gln
20 25 30
Ser Ala Ala Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Arg Ser Tyr Arg Asp Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 319
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 319
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Phe Thr Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Thr Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 320
<211> 1356
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 320
caggtgcagc tgcagcagtc aggccctggc ctggtcaagc ctagtcagac cctgagcctg 60
acctgcgcta ttagcggcga tagtgtgtct agtcagtcag ccgcctggaa ctggattaga 120
cagtcaccct ctaggggcct ggagtggctg ggtagaatct actataggtc taagtggtat 180
aacgactacg ccgtcagcgt gaagtctagg atcactatta accccgacac ctctaagaat 240
cagtttagcc tgcagctgaa tagcgtgacc cccgaggaca ccgccgtcta ctactgcgct 300
agagagcggt cctatagaga ctacttcgac tactggggtc agggcaccct ggtcaccgtg 360
tctagcgcta gcactaaggg cccaagtgtg tttcccctgg cccccagcag caagtctact 420
tccggcggaa ctgctgccct gggttgcctg gtgaaggact acttccccga gcccgtgaca 480
gtgtcctgga actctggggc tctgacttcc ggcgtgcaca ccttccccgc cgtgctgcag 540
agcagcggcc tgtacagcct gagcagcgtg gtgacagtgc cctccagctc tctgggaacc 600
cagacctata tctgcaacgt gaaccacaag cccagcaaca ccaaggtgga caagagagtg 660
gagcccaaga gctgcgacaa gacccacacc tgccccccct gcccagctcc agaactgctg 720
ggagggcctt ccgtgttcct gttccccccc aagcccaagg acaccctgat gatcagcagg 780
acccccgagg tgacctgcgt ggtggtggac gtgtcccacg aggacccaga ggtgaagttc 840
aactggtacg tggacggcgt ggaggtgcac aacgccaaga ccaagcccag agaggagcag 900
tacaacagca cctacagggt ggtgtccgtg ctgaccgtgc tgcaccagga ctggctgaac 960
ggcaaagaat acaagtgcaa agtctccaac aaggccctgc cagccccaat cgaaaagaca 1020
atcagcaagg ccaagggcca gccacgggag ccccaggtgt acaccctgcc ccccagccgg 1080
gaggagatga ccaagaacca ggtgtccctg acctgtctgg tgaagggctt ctaccccagc 1140
gatatcgccg tggagtggga gagcaacggc cagcccgaga acaactacaa gaccaccccc 1200
ccagtgctgg acagcgacgg cagcttcttc ctgtacagca agctgaccgt ggacaagtcc 1260
aggtggcagc agggcaacgt gttcagctgc agcgtgatgc acgaggccct gcacaaccac 1320
tacacccaga agtccctgag cctgagcccc ggcaag 1356
<210> 321
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 321
gatattcaga tgactcagtc acctagtagc ctgagcgcta gtgtgggcga tagagtgact 60
atcacctgta gagcctctca gggaatcttc acctacctga actggtatca gcagaagccc 120
ggtaaagccc ctaagctgct gatctacgcc gcctctaccc tgcagtcagg cgtgccctct 180
aggtttagcg gtagcggtag tggcaccgac ttcaccctga ctatctctag cctgcagccc 240
gaggacttcg ctacctacta ctgtcagcag tactactcta ctagcctgac cttcggtcag 300
ggcactaagg tcgagattaa gcgtacggtg gccgctccca gcgtgttcat cttccccccc 360
agcgacgagc agctgaagag cggcaccgcc agcgtggtgt gcctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaacgccc tgcagagcgg caacagccag 480
gagagcgtca ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcataag gtgtacgcct gcgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac aggggcgagt gc 642
<210> 322
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 322
Ser Gly Asp Asn Ile Gly Ser Met Thr Ala His
1 5 10
<210> 323
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 323
Asp Lys Asn Glu Arg Pro Ser
1 5
<210> 324
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 324
Gln Ser Trp Asp Asp Ser Tyr Thr Ser Val Val
1 5 10
<210> 325
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 325
Ser Gln Ser Ala Gly Trp Asn
1 5
<210> 326
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 326
Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala Val Ser Val
1 5 10 15
Lys Ser
<210> 327
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 327
Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe Asp Tyr
1 5 10
<210> 328
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 328
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Gln
20 25 30
Ser Ala Gly Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 329
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 329
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Met Thr Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Lys Asn Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Asp Ser Tyr Thr Ser
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 330
<211> 375
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 330
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctcagtctg ctggttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaaaaat acactgtttc tttctacgac ttcttcgatt actggggcca aggcaccctg 360
gtgactgtta gctca 375
<210> 331
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 331
agctatgaac tgacccagcc gctgagcgtg agcgtggcgc tgggccagac cgcgcgcatt 60
acctgtagcg gcgataacat cggttctatg actgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacaaa aacgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagccgcgc gcaggcgggc 240
gacgaagcgg attattactg ccagtcttgg gacgactctt acacctctgt tgtgtttggc 300
ggcggcacga agttaaccgt ccta 324
<210> 332
<211> 455
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 332
Gln Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Val Ser Ser Gln
20 25 30
Ser Ala Gly Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Ile Tyr Tyr Arg Ser Lys Trp Tyr Asn Asp Tyr Ala
50 55 60
Val Ser Val Lys Ser Arg Ile Thr Ile Asn Pro Asp Thr Ser Lys Asn
65 70 75 80
Gln Phe Ser Leu Gln Leu Asn Ser Val Thr Pro Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Ala Arg Glu Lys Tyr Thr Val Ser Phe Tyr Asp Phe Phe
100 105 110
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 333
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 333
Ser Tyr Glu Leu Thr Gln Pro Leu Ser Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asp Asn Ile Gly Ser Met Thr Ala
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Asp Lys Asn Glu Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Ala Gln Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Trp Asp Asp Ser Tyr Thr Ser
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys Ser
210
<210> 334
<211> 1365
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 334
caggtgcaat tgcagcagag cggtccgggc ctggtgaaac cgagccagac cctgagcctg 60
acctgcgcga tttccggaga tagcgtgagc tctcagtctg ctggttggaa ctggattcgt 120
cagagcccga gccgtggcct cgagtggctg ggccgtatct actaccgtag caaatggtac 180
aacgactatg ccgtgagcgt gaaaagccgc attaccatta acccggatac ttcgaaaaac 240
cagtttagcc tgcaactgaa cagcgtgacc ccggaagata cggccgtgta ttattgcgcg 300
cgtgaaaaat acactgtttc tttctacgac ttcttcgatt actggggcca aggcaccctg 360
gtgactgtta gctcagcctc caccaagggt ccatcggtct tccccctggc accctcctcc 420
aagagcacct ctgggggcac agcggccctg ggctgcctgg tcaaggacta cttccccgaa 480
ccggtgacgg tgtcgtggaa ctcaggcgcc ctgaccagcg gcgtgcacac cttcccggct 540
gtcctacagt cctcaggact ctactccctc agcagcgtgg tgaccgtgcc ctccagcagc 600
ttgggcaccc agacctacat ctgcaacgtg aatcacaagc ccagcaacac caaggtggac 660
aagagagttg agcccaaatc ttgtgacaaa actcacacat gcccaccgtg cccagcacct 720
gaactcctgg ggggaccgtc agtcttcctc ttccccccaa aacccaagga caccctcatg 780
atctcccgga cccctgaggt cacatgcgtg gtggtggacg tgagccacga agaccctgag 840
gtcaagttca actggtacgt ggacggcgtg gaggtgcata atgccaagac aaagccgcgg 900
gaggagcagt acaacagcac gtaccgggtg gtcagcgtcc tcaccgtcct gcaccaggac 960
tggctgaatg gcaaggagta caagtgcaag gtctccaaca aagccctccc agcccccatc 1020
gagaaaacca tctccaaagc caaagggcag ccccgagaac cacaggtgta caccctgccc 1080
ccatcccggg aggagatgac caagaaccag gtcagcctga cctgcctggt caaaggcttc 1140
tatcccagcg acatcgccgt ggagtgggag agcaatgggc agccggagaa caactacaag 1200
accacgcctc ccgtgctgga ctccgacggc tccttcttcc tctacagcaa gctcaccgtg 1260
gacaagagca ggtggcagca ggggaacgtc ttctcatgct ccgtgatgca tgaggctctg 1320
cacaaccact acacgcagaa gagcctctcc ctgtctccgg gtaaa 1365
<210> 335
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 335
agctatgaac tgacccagcc gctgagcgtg agcgtggcgc tgggccagac cgcgcgcatt 60
acctgtagcg gcgataacat cggttctatg actgctcatt ggtaccagca gaaaccgggc 120
caggcgccgg tgctggtgat ctacgacaaa aacgaacgtc cgagcggcat cccggaacgt 180
tttagcggat ccaacagcgg caacaccgcg accctgacca ttagccgcgc gcaggcgggc 240
gacgaagcgg attattactg ccagtcttgg gacgactctt acacctctgt tgtgtttggc 300
ggcggcacga agttaaccgt cctaggtcag cccaaggctg ccccctcggt cactctgttc 360
ccgccctcct ctgaggagct tcaagccaac aaggccacac tggtgtgtct cataagtgac 420
ttctacccgg gagccgtgac agtggcctgg aaggcagata gcagccccgt caaggcggga 480
gtggagacca ccacaccctc caaacaaagc aacaacaagt acgcggccag cagctatctg 540
agcctgacgc ctgagcagtg gaagtcccac agaagctaca gctgccaggt cacgcatgaa 600
gggagcaccg tggagaagac agtggcccct acagaatgtt ca 642
<210> 336
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 336
Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys
1 5 10 15
Val Val Tyr
<210> 337
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 337
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
1 5 10 15
Gln Phe Ser Phe
20
<210> 338
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 338
Phe Leu Leu Glu Glu Tyr Thr Gly Ser Asp
1 5 10
<210> 339
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 339
Ala Lys Val Val Tyr Ser Ile Leu
1 5
<210> 340
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 340
Gln Gly Gln Pro Tyr Phe Ser Val Glu Ser Glu Thr Gly Ile Ile Lys
1 5 10 15
Thr Ala Leu
<210> 341
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 341
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln
1 5 10
<210> 342
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 342
Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile
1 5 10 15
Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu
20 25
<210> 343
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 343
Ile Glu Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met
1 5 10
<210> 344
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 344
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr
1 5 10 15
<210> 345
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 345
Val Lys Lys Leu Leu Asp
1 5
<210> 346
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 346
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
1 5 10 15
Tyr Val Glu Pro Arg Phe
20
<210> 347
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 347
Phe Glu Lys Lys Lys Val Tyr Thr Leu
1 5
<210> 348
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 348
Val Arg Ile Val Val Glu Asp
1 5
<210> 349
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 349
Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr Ser Val Asp
1 5 10 15
Arg His Thr Asp Met
20
<210> 350
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 350
Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10 15
Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
20 25 30
<210> 351
<211> 23
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 351
Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser
1 5 10 15
Ser Thr Gly Thr Val Thr Val
20
<210> 352
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 352
Ala Asp Val Gly Glu Asn Ala Glu Ile Glu Tyr Ser Ile Thr Asp Gly
1 5 10 15
Glu Gly Leu Asp Met
20
<210> 353
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 353
Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys Ala Asp Gln Leu
1 5 10
<210> 354
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 354
Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr
1 5 10
<210> 355
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 355
Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr Gly Thr Val Thr Val
1 5 10 15
<210> 356
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 356
His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe
1 5 10 15
<210> 357
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 357
Asn Lys Asp Asn Thr Ala Gly Ile Leu
1 5
<210> 358
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 358
Cys Asp His His Gly Asn Met Gln Ser Cys His Ala Glu Ala
1 5 10
<210> 359
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 359
Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln
1 5 10
<210> 360
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 360
Leu Glu Glu Tyr Thr Gly Ser Asp
1 5
<210> 361
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 361
Tyr Gln Tyr Val Gly Lys Leu
1 5
<210> 362
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 362
Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp Gly
1 5 10 15
Ser Leu
<210> 363
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 363
Tyr Gln Tyr Val Gly Lys Leu His Ser Asp Gln Asp Arg Gly Asp Gly
1 5 10 15
Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu
20 25 30
<210> 364
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 364
Tyr Ile Leu Ser Gly Asp Gly Ala Gly Asp Leu
1 5 10
<210> 365
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 365
Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
1 5 10
<210> 366
<211> 24
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 366
Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu
1 5 10 15
Asp Arg Glu Glu Lys Pro Val Tyr
20
<210> 367
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 367
Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu
1 5 10 15
Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu
20 25
<210> 368
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 368
Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu
1 5 10 15
Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
20 25
<210> 369
<211> 23
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 369
Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp
1 5 10 15
Arg Glu Glu Lys Pro Val Tyr
20
<210> 370
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 370
Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp Arg Glu Glu Lys
1 5 10 15
Pro Val Tyr
<210> 371
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 371
Asn Thr Gly Asp Ile Gln Ala Thr Lys Arg Leu Asp Arg Glu Glu Lys
1 5 10 15
Pro Val Tyr Ile Leu
20
<210> 372
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 372
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr
1 5 10
<210> 373
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 373
Ile Leu Arg Ala Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu
1 5 10 15
Pro Glu Ser Glu
20
<210> 374
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 374
Ile Leu Arg Ala Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu
1 5 10 15
Pro Glu Ser Glu Phe
20
<210> 375
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 375
Arg Ala Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu
1 5 10 15
Ser Glu
<210> 376
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 376
Arg Ala Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu
1 5 10 15
Ser Glu Phe
<210> 377
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 377
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
1 5 10 15
<210> 378
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 378
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
1 5 10 15
Phe
<210> 379
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 379
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe
1 5 10 15
<210> 380
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 380
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
1 5 10 15
Lys Glu
<210> 381
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 381
Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe
1 5 10
<210> 382
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 382
Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
1 5 10 15
<210> 383
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 383
Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr Lys
1 5 10 15
<210> 384
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 384
Thr Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly
1 5 10 15
Thr
<210> 385
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 385
Thr Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly
1 5 10 15
Thr Phe
<210> 386
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 386
Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr Phe
1 5 10 15
<210> 387
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 387
Phe Val Val Gln Val Thr Ala Thr
1 5
<210> 388
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 388
Val Val Gln Val Thr Ala Thr
1 5
<210> 389
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 389
Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn
1 5 10 15
Ser
<210> 390
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 390
Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly Asn
1 5 10 15
Ser Ala Lys Val Val Tyr
20
<210> 391
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 391
Asp Ala Asp Asp Pro Thr Tyr Gly Asn Ser Ala Lys Val Val Tyr
1 5 10 15
<210> 392
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 392
Ser Ile Leu Gln Gly Gln Pro Tyr Phe
1 5
<210> 393
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 393
Gln Gly Gln Pro Tyr Phe Ser Val Glu
1 5
<210> 394
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 394
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu
1 5 10
<210> 395
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 395
Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu
1 5 10
<210> 396
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 396
Thr Gly Ile Ile Lys Thr Ala Leu Leu
1 5
<210> 397
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 397
Leu Asn Met Asp Arg Glu Asn Arg Glu Gln Tyr
1 5 10
<210> 398
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 398
Leu Asn Met Asp Arg Glu Asn Arg Glu Gln Tyr Gln
1 5 10
<210> 399
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 399
Asp Arg Glu Asn Arg Glu Gln Tyr Gln
1 5
<210> 400
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 400
Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile
1 5 10 15
Lys Ala Ser Asp
20
<210> 401
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 401
Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile
1 5 10 15
Lys Ala Ser Asp Ala Asp Val Gly Glu Asn
20 25
<210> 402
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 402
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met
1 5 10
<210> 403
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 403
Ser Ile Thr Asp Gly Glu Gly Leu Asp Met
1 5 10
<210> 404
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 404
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu
1 5 10
<210> 405
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 405
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val
1 5 10 15
Lys Lys Leu Leu Asp
20
<210> 406
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 406
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val
1 5 10 15
Lys Lys Leu Leu Asp Phe
20
<210> 407
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 407
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val
1 5 10 15
Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys Val Tyr Thr Leu
20 25 30
<210> 408
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 408
Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys
1 5 10 15
Lys Leu Leu Asp
20
<210> 409
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 409
Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys
1 5 10 15
Lys Leu Leu Asp Phe
20
<210> 410
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 410
Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys
1 5 10 15
Leu Leu Asp Phe
20
<210> 411
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 411
Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10
<210> 412
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 412
Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe
1 5 10
<210> 413
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 413
Val Lys Lys Leu Leu Asp Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys
1 5 10 15
Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg Phe
20 25
<210> 414
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 414
Glu Lys Lys Lys Val Tyr Thr Leu
1 5
<210> 415
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 415
Lys Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg Phe
1 5 10
<210> 416
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 416
Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys
1 5 10 15
Leu
<210> 417
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 417
Asp Val Asp Glu Pro Pro Val Phe
1 5
<210> 418
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 418
Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu
1 5 10
<210> 419
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 419
Val Asp Glu Pro Pro Val Phe
1 5
<210> 420
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 420
Val Asp Glu Pro Pro Val Phe Ser Lys Leu
1 5 10
<210> 421
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 421
Ala Arg Asn Pro Val Lys Tyr Ser Val Asp Arg His Thr Asp Met
1 5 10 15
<210> 422
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 422
Ser Val Asp Arg His Thr Asp Met
1 5
<210> 423
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 423
Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn Gly Ser Ile Phe
1 5 10
<210> 424
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 424
Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu
1 5 10
<210> 425
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 425
Leu Asp Arg Glu Thr Leu Leu
1 5
<210> 426
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 426
Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10 15
Tyr
<210> 427
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 427
Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10
<210> 428
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 428
Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10 15
<210> 429
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 429
Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10
<210> 430
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 430
Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10
<210> 431
<211> 27
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 431
Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys
1 5 10 15
Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
20 25
<210> 432
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 432
Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10
<210> 433
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 433
Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 434
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 434
Ile Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 435
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 435
Val Cys Glu Lys Ala Lys Ala Asp Gln Leu
1 5 10
<210> 436
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 436
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
1 5 10 15
Gln
<210> 437
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 437
His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln
1 5 10
<210> 438
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 438
His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser
1 5 10 15
<210> 439
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 439
Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe
1 5 10
<210> 440
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 440
Arg Val Cys Ala Cys Asp His His Gly Asn Met Gln Ser Cys His Ala
1 5 10 15
<210> 441
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 441
Cys Asp His His Gly Asn Met Gln Ser Cys His Ala
1 5 10
<210> 442
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 442
Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro
1 5 10
<210> 443
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 443
Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile
1 5 10 15
Lys Ala Ser Asp
20
<210> 444
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 444
Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly Arg Ile
1 5 10 15
Lys Ala Ser Asp Ala
20
<210> 445
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 445
Pro Pro Gly Thr Pro Ile Gly Arg Ile Lys Ala Ser Asp
1 5 10
<210> 446
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 446
Ala Asp Val Gly Glu Asn Ala Glu
1 5
<210> 447
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 447
Ile Glu Tyr Ser Ile Thr Asp Gly Glu Gly
1 5 10
<210> 448
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 448
Ser Ile Thr Asp Gly Glu Gly
1 5
<210> 449
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 449
Thr Asp Gly Glu Gly Leu Asp Met
1 5
<210> 450
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 450
Phe Asp Val Ile Thr Asp Gln Glu
1 5
<210> 451
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 451
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu
1 5 10
<210> 452
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 452
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val
1 5 10 15
Lys Lys Leu Leu Asp
20
<210> 453
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 453
Phe Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val
1 5 10 15
Lys Lys Leu Leu Asp Phe
20
<210> 454
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 454
Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr
1 5 10
<210> 455
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 455
Asp Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys
1 5 10 15
Lys Leu Leu Asp
20
<210> 456
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 456
Val Ile Thr Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys
1 5 10 15
Leu Leu Asp
<210> 457
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 457
Thr Asp Gln Glu Thr Gln Glu
1 5
<210> 458
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 458
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10 15
<210> 459
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 459
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10 15
Phe
<210> 460
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 460
Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10
<210> 461
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 461
Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10
<210> 462
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 462
Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
1 5 10
<210> 463
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 463
Gly Ile Ile Thr Val Lys Lys Leu Leu Asp Phe
1 5 10
<210> 464
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 464
Ile Ile Thr Val Lys Lys Leu Leu Asp Phe
1 5 10
<210> 465
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 465
Ile Thr Val Lys Lys Leu Leu Asp
1 5
<210> 466
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 466
Ile Thr Val Lys Lys Leu Leu Asp Phe
1 5
<210> 467
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 467
Thr Val Lys Lys Leu Leu Asp
1 5
<210> 468
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 468
Thr Val Lys Lys Leu Leu Asp Phe
1 5
<210> 469
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 469
Val Lys Lys Leu Leu Asp Phe
1 5
<210> 470
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 470
Phe Glu Lys Lys Lys Val Tyr Thr
1 5
<210> 471
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 471
Glu Lys Lys Lys Val Tyr Thr Leu
1 5
<210> 472
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 472
Lys Val Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg Phe
1 5 10
<210> 473
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 473
Glu Ala Ser Asn Pro Tyr Val Glu Pro Arg Phe
1 5 10
<210> 474
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 474
Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr
1 5 10
<210> 475
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 475
Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala Thr Val
1 5 10
<210> 476
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 476
Leu Gly Pro Phe Lys Asp Ser Ala Thr
1 5
<210> 477
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 477
Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe
1 5 10
<210> 478
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 478
Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser Lys
1 5 10 15
Leu
<210> 479
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 479
Val Val Glu Asp Val Asp Glu Pro Pro Val Phe
1 5 10
<210> 480
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 480
Asp Val Asp Glu Pro Pro Val Phe
1 5
<210> 481
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 481
Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu
1 5 10
<210> 482
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 482
Asp Val Asp Glu Pro Pro Val Phe Ser Lys Leu Ala Tyr
1 5 10
<210> 483
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 483
Val Asp Glu Pro Pro Val Phe
1 5
<210> 484
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 484
Val Asp Glu Pro Pro Val Phe Ser Lys Leu
1 5 10
<210> 485
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 485
Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro Val Lys Tyr
1 5 10
<210> 486
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 486
Ala Arg Asn Pro Val Lys Tyr Ser Val Asp Arg His Thr Asp Met
1 5 10 15
<210> 487
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 487
Val Asp Arg His Thr Asp Met
1 5
<210> 488
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 488
Asp Arg Ile Phe Asn Ile Asp Ser Gly Asn Gly Ser Ile Phe
1 5 10
<210> 489
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 489
Thr Ser Lys Leu Leu Asp Arg Glu Thr Leu
1 5 10
<210> 490
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 490
Leu Asp Arg Glu Thr Leu Leu
1 5
<210> 491
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 491
Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10 15
<210> 492
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 492
Ile Ala Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10 15
Tyr
<210> 493
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 493
Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10 15
<210> 494
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 494
Thr Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile
1 5 10 15
Lys Val
<210> 495
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 495
Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10
<210> 496
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 496
Glu Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10
<210> 497
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 497
Ile Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr
1 5 10
<210> 498
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 498
Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu
1 5 10
<210> 499
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 499
Asn Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile
1 5 10
<210> 500
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 500
Tyr Ile Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 501
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 501
Ile Lys Val Leu Asp Val Asn Asp
1 5
<210> 502
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 502
Ile Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 503
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 503
Lys Val Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 504
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 504
Leu Asp Val Asn Asp Asn Ala Pro Glu Phe
1 5 10
<210> 505
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 505
Phe Val Cys Glu Lys Ala Lys Ala Asp Gln Leu
1 5 10
<210> 506
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 506
Val Cys Glu Lys Ala Lys Ala Asp Gln Leu
1 5 10
<210> 507
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 507
Lys Ala Lys Ala Asp Gln Leu
1 5
<210> 508
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 508
Ile Gln Thr Leu His Ala Val Asp
1 5
<210> 509
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 509
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
1 5 10 15
Gln
<210> 510
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 510
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
1 5 10 15
Gln Phe Ser
<210> 511
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 511
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
1 5 10 15
Gln Phe Ser Phe
20
<210> 512
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 512
His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln
1 5 10
<210> 513
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 513
His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser
1 5 10 15
<210> 514
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 514
Val Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe
1 5 10
<210> 515
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 515
Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr
1 5 10 15
<210> 516
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 516
Arg Lys Asn Gly Tyr Asn Arg His Glu Met Ser Thr
1 5 10
<210> 517
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 517
Tyr Leu Leu Pro Val Val Ile Ser
1 5
<210> 518
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 518
Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr
1 5 10
<210> 519
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 519
Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln
1 5 10 15
<210> 520
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 520
Pro Val Val Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr Gly
1 5 10 15
Thr Val Thr Val
20
<210> 521
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 521
Arg Val Cys Ala Cys Asp His His Gly Asn Met Gln Ser Cys
1 5 10
<210> 522
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 522
Arg Val Cys Ala Cys Asp His His Gly Asn Met Gln Ser Cys His Ala
1 5 10 15
<210> 523
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 523
Cys Asp His His Gly Asn Met Gln Ser Cys
1 5 10
<210> 524
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 524
Cys Asp His His Gly Asn Met Gln Ser Cys His Ala
1 5 10
<210> 525
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 525
Asp His His Gly Asn Met Gln Ser Cys His Ala
1 5 10
<210> 526
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 526
Leu Ile His Pro Thr Gly Leu Ser Thr Gly Ala Gly Ser Glu
1 5 10
<210> 527
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 527
Phe Arg His Asp Ser Gly Leu
1 5
<210> 528
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 528
Phe Arg His Asp Ser Gly Leu Asn Asp
1 5
<210> 529
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 529
Glu Ala Gln Lys Ile Glu Trp His
1 5
<210> 530
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 530
Glu Ala Gln Lys Ile Glu Trp His Glu
1 5
<210> 531
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 531
Glu Phe Arg His Asp Ser
1 5
<210> 532
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 532
Leu Glu Val Leu Phe Gln Gly Pro
1 5
<210> 533
<211> 790
<212> PRT
<213> Homo sapiens
<400> 533
Met Arg Thr Tyr Arg Tyr Phe Leu Leu Leu Phe Trp Val Gly Gln Pro
1 5 10 15
Tyr Pro Thr Leu Ser Thr Pro Leu Ser Lys Arg Thr Ser Gly Phe Pro
20 25 30
Ala Lys Lys Arg Ala Leu Glu Leu Ser Gly Asn Ser Lys Asn Glu Leu
35 40 45
Asn Arg Ser Lys Arg Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu
50 55 60
Glu Tyr Thr Gly Ser Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp
65 70 75 80
Gln Asp Arg Gly Asp Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly
85 90 95
Ala Gly Asp Leu Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
100 105 110
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
115 120 125
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
130 135 140
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
145 150 155 160
Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr
165 170 175
Phe Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly
180 185 190
Asn Ser Ala Lys Val Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe
195 200 205
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met
210 215 220
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp
225 230 235 240
Met Gly Gly Gln Met Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile
245 250 255
Thr Leu Thr Asp Val Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr
260 265 270
Tyr Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly
275 280 285
Arg Ile Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu
290 295 300
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr
305 310 315 320
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
325 330 335
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
340 345 350
Tyr Val Glu Pro Arg Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala
355 360 365
Thr Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser
370 375 380
Lys Leu Ala Tyr Ile Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr
385 390 395 400
Thr Ile Gly Ser Val Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro
405 410 415
Val Lys Tyr Ser Val Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn
420 425 430
Ile Asp Ser Gly Asn Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg
435 440 445
Glu Thr Leu Leu Trp His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn
450 455 460
Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp
465 470 475 480
Val Asn Asp Asn Ala Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val
485 490 495
Cys Glu Lys Ala Lys Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val
500 505 510
Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala
515 520 525
Pro Glu Ala Ala Ser Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp
530 535 540
Asn Thr Ala Gly Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu
545 550 555 560
Met Ser Thr Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro
565 570 575
Val Gln Ser Ser Thr Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp
580 585 590
His His Gly Asn Met Gln Ser Cys His Ala Glu Ala Leu Ile His Pro
595 600 605
Thr Gly Leu Ser Thr Gly Ala Leu Val Ala Ile Leu Leu Cys Ile Val
610 615 620
Ile Leu Leu Val Thr Val Val Leu Phe Ala Ala Leu Arg Arg Gln Arg
625 630 635 640
Lys Lys Glu Pro Leu Ile Ile Ser Lys Glu Asp Ile Arg Asp Asn Ile
645 650 655
Val Ser Tyr Asn Asp Glu Gly Gly Gly Glu Glu Asp Thr Gln Ala Phe
660 665 670
Asp Ile Gly Thr Leu Arg Asn Pro Glu Ala Ile Glu Asp Asn Lys Leu
675 680 685
Arg Arg Asp Ile Val Pro Glu Ala Leu Phe Leu Pro Arg Arg Thr Pro
690 695 700
Thr Ala Arg Asp Asn Thr Asp Val Arg Asp Phe Ile Asn Gln Arg Leu
705 710 715 720
Lys Glu Asn Asp Thr Asp Pro Thr Ala Pro Pro Tyr Asp Ser Leu Ala
725 730 735
Thr Tyr Ala Tyr Glu Gly Thr Gly Ser Val Ala Asp Ser Leu Ser Ser
740 745 750
Leu Glu Ser Val Thr Thr Asp Ala Asp Gln Asp Tyr Asp Tyr Leu Ser
755 760 765
Asp Trp Gly Pro Arg Phe Lys Lys Leu Ala Asp Met Tyr Gly Gly Val
770 775 780
Asp Ser Asp Lys Asp Ser
785 790
<210> 534
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 534
Glu Phe Tyr Glu Thr Phe Val Cys Glu Lys Ala Lys Ala Asp Gln Leu
1 5 10 15
Ile Gln Thr Leu His Ala Val Asp Lys Asp Asp Pro Tyr Ser Gly His
20 25 30
Gln Phe Ser Phe Ser Leu Ala Pro Glu Ala Ala Ser Gly Ser Asn Phe
35 40 45
Thr Ile Gln Asp Asn Lys Asp Asn Thr Ala Gly Ile Leu Thr Arg Lys
50 55 60
Asn Gly Tyr Asn Arg His Glu Met Ser Thr Tyr Leu Leu Pro Val Val
65 70 75 80
Ile Ser Asp Asn Asp Tyr Pro Val Gln Ser Ser Thr Gly Thr Val Thr
85 90 95
Val Arg Val Cys Ala Cys Asp His His Gly Asn Met Gln Ser Cys His
100 105 110
Ala Glu Ala Leu Ile His Pro
115
<210> 535
<211> 225
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 535
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Ser Gly Ser Gly Ser Asn Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gln Trp Gly Ser Tyr Ala Phe Asp Ser Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His
225
<210> 536
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 536
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Val Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Gly Thr Phe Pro Pro
85 90 95
Thr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 537
<211> 223
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 537
Gln Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Arg Ser Ser Gly Ser Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Gly Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
<210> 538
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 538
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Leu Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Thr Ser Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 539
<211> 790
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 539
Met Arg Thr Tyr Arg Tyr Phe Leu Leu Leu Phe Trp Val Gly Gln Pro
1 5 10 15
Tyr Pro Thr Leu Ser Thr Pro Leu Ser Lys Arg Thr Ser Gly Phe Pro
20 25 30
Ala Lys Lys Arg Ala Leu Glu Leu Ser Gly Asn Ser Lys Asn Glu Leu
35 40 45
Asn Arg Ser Lys Arg Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu
50 55 60
Glu Tyr Thr Gly Ser Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp
65 70 75 80
Gln Asp Arg Gly Asp Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly
85 90 95
Ala Gly Asp Leu Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
100 105 110
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
115 120 125
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
130 135 140
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
145 150 155 160
Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr
165 170 175
Phe Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly
180 185 190
Asn Ser Ala Lys Val Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe
195 200 205
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met
210 215 220
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp
225 230 235 240
Met Gly Gly Gln Met Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile
245 250 255
Thr Leu Thr Asp Val Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr
260 265 270
Tyr Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly
275 280 285
Arg Ile Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu
290 295 300
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr
305 310 315 320
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
325 330 335
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
340 345 350
Tyr Val Glu Pro Arg Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala
355 360 365
Thr Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser
370 375 380
Lys Leu Ala Tyr Ile Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr
385 390 395 400
Thr Ile Gly Ser Val Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro
405 410 415
Val Lys Tyr Ser Val Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn
420 425 430
Ile Asp Ser Gly Asn Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg
435 440 445
Glu Thr Leu Leu Trp His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn
450 455 460
Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp
465 470 475 480
Val Asn Asp Asn Ala Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val
485 490 495
Cys Glu Lys Ala Lys Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val
500 505 510
Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala
515 520 525
Pro Glu Ala Ala Ser Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp
530 535 540
Asn Thr Ala Gly Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu
545 550 555 560
Met Ser Thr Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro
565 570 575
Val Gln Ser Ser Thr Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp
580 585 590
His His Gly Asn Met Gln Ser Cys His Ala Glu Ala Leu Ile His Pro
595 600 605
Thr Gly Leu Ser Thr Gly Ala Leu Val Ala Ile Leu Leu Cys Ile Val
610 615 620
Ile Leu Leu Val Thr Val Val Leu Phe Ala Ala Leu Arg Arg Gln Arg
625 630 635 640
Lys Lys Glu Pro Leu Ile Ile Ser Lys Glu Asp Ile Arg Asp Asn Ile
645 650 655
Val Ser Tyr Asn Asp Glu Gly Gly Gly Glu Glu Asp Thr Gln Ala Phe
660 665 670
Asp Ile Gly Thr Leu Arg Asn Pro Glu Ala Ile Glu Asp Asn Lys Leu
675 680 685
Arg Arg Asp Ile Val Pro Glu Ala Leu Phe Leu Pro Arg Arg Thr Pro
690 695 700
Thr Ala Arg Asp Asn Thr Asp Val Arg Asp Phe Ile Asn Gln Arg Leu
705 710 715 720
Lys Glu Asn Asp Thr Asp Pro Thr Ala Pro Pro Tyr Asp Ser Leu Ala
725 730 735
Thr Tyr Ala Tyr Glu Gly Thr Gly Ser Val Ala Asp Ser Leu Ser Ser
740 745 750
Leu Glu Ser Val Thr Thr Asp Ala Asp Gln Asp Tyr Asp Tyr Leu Ser
755 760 765
Asp Trp Gly Pro Arg Phe Lys Lys Leu Ala Asp Met Tyr Gly Gly Val
770 775 780
Asp Ser Asp Lys Asp Ser
785 790
<210> 540
<211> 790
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 540
Met Arg Thr Tyr Arg Tyr Phe Leu Leu Leu Phe Trp Val Gly Gln Pro
1 5 10 15
Tyr Pro Thr Leu Ser Thr Pro Leu Ser Lys Arg Thr Ser Gly Phe Pro
20 25 30
Ala Lys Lys Arg Ala Leu Glu Leu Ser Gly Asn Ser Lys Asn Glu Leu
35 40 45
Asn Arg Ser Lys Arg Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu
50 55 60
Glu Tyr Thr Gly Ser Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp
65 70 75 80
Gln Asp Arg Gly Asp Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly
85 90 95
Ala Gly Asp Leu Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
100 105 110
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
115 120 125
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
130 135 140
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
145 150 155 160
Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr
165 170 175
Phe Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly
180 185 190
Asn Ser Ala Lys Val Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe
195 200 205
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met
210 215 220
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp
225 230 235 240
Met Gly Gly Gln Met Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile
245 250 255
Thr Leu Thr Asp Val Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr
260 265 270
Tyr Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly
275 280 285
Arg Ile Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu
290 295 300
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr
305 310 315 320
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
325 330 335
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
340 345 350
Tyr Val Glu Pro Arg Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala
355 360 365
Thr Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser
370 375 380
Lys Leu Ala Tyr Ile Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr
385 390 395 400
Thr Ile Gly Ser Val Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro
405 410 415
Val Lys Tyr Ser Val Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn
420 425 430
Ile Asp Ser Gly Asn Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg
435 440 445
Glu Thr Leu Leu Trp His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn
450 455 460
Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp
465 470 475 480
Val Asn Asp Asn Ala Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val
485 490 495
Cys Glu Lys Ala Lys Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val
500 505 510
Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala
515 520 525
Pro Glu Ala Ala Ser Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp
530 535 540
Asn Thr Ala Gly Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu
545 550 555 560
Met Ser Thr Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro
565 570 575
Val Gln Ser Ser Thr Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp
580 585 590
His His Gly Asn Met Gln Ser Cys His Ala Glu Ala Leu Ile His Pro
595 600 605
Thr Gly Leu Ser Thr Gly Ala Leu Val Ala Ile Leu Leu Cys Ile Val
610 615 620
Ile Leu Leu Val Thr Val Val Leu Phe Ala Ala Leu Arg Arg Gln Arg
625 630 635 640
Lys Lys Glu Pro Leu Ile Ile Ser Lys Glu Asp Ile Arg Asp Asn Ile
645 650 655
Val Ser Tyr Asn Asp Glu Gly Gly Gly Glu Glu Asp Thr Gln Ala Phe
660 665 670
Asp Ile Gly Thr Leu Arg Asn Pro Glu Ala Ile Glu Asp Asn Lys Leu
675 680 685
Arg Arg Asp Ile Val Pro Glu Ala Leu Phe Leu Pro Arg Arg Thr Pro
690 695 700
Thr Ala Arg Asp Asn Thr Asp Val Arg Asp Phe Ile Asn Gln Arg Leu
705 710 715 720
Lys Glu Asn Asp Thr Asp Pro Thr Ala Pro Pro Tyr Asp Ser Leu Ala
725 730 735
Thr Tyr Ala Tyr Glu Gly Thr Gly Ser Val Ala Asp Ser Leu Ser Ser
740 745 750
Leu Glu Ser Val Thr Thr Asp Ala Asp Gln Asp Tyr Asp Tyr Leu Ser
755 760 765
Asp Trp Gly Pro Arg Phe Lys Lys Leu Ala Asp Met Tyr Gly Gly Val
770 775 780
Asp Ser Asp Lys Asp Ser
785 790
<210> 541
<211> 790
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 541
Met Arg Thr Tyr Arg Tyr Phe Leu Leu Leu Phe Trp Val Gly Gln Pro
1 5 10 15
Tyr Pro Thr Leu Ser Thr Pro Leu Ser Lys Arg Thr Ser Gly Phe Pro
20 25 30
Ala Lys Lys Arg Ala Leu Glu Leu Ser Gly Asn Ser Lys Asn Glu Leu
35 40 45
Asn Arg Ser Lys Arg Ser Trp Met Trp Asn Gln Phe Phe Leu Leu Glu
50 55 60
Glu Tyr Thr Gly Ser Asp Tyr Gln Tyr Val Gly Lys Leu His Ser Asp
65 70 75 80
Gln Asp Arg Gly Asp Gly Ser Leu Lys Tyr Ile Leu Ser Gly Asp Gly
85 90 95
Ala Gly Asp Leu Phe Ile Ile Asn Glu Asn Thr Gly Asp Ile Gln Ala
100 105 110
Thr Lys Arg Leu Asp Arg Glu Glu Lys Pro Val Tyr Ile Leu Arg Ala
115 120 125
Gln Ala Ile Asn Arg Arg Thr Gly Arg Pro Val Glu Pro Glu Ser Glu
130 135 140
Phe Ile Ile Lys Ile His Asp Ile Asn Asp Asn Glu Pro Ile Phe Thr
145 150 155 160
Lys Glu Val Tyr Thr Ala Thr Val Pro Glu Met Ser Asp Val Gly Thr
165 170 175
Phe Val Val Gln Val Thr Ala Thr Asp Ala Asp Asp Pro Thr Tyr Gly
180 185 190
Asn Ser Ala Lys Val Val Tyr Ser Ile Leu Gln Gly Gln Pro Tyr Phe
195 200 205
Ser Val Glu Ser Glu Thr Gly Ile Ile Lys Thr Ala Leu Leu Asn Met
210 215 220
Asp Arg Glu Asn Arg Glu Gln Tyr Gln Val Val Ile Gln Ala Lys Asp
225 230 235 240
Met Gly Gly Gln Met Gly Gly Leu Ser Gly Thr Thr Thr Val Asn Ile
245 250 255
Thr Leu Thr Asp Val Asn Asp Asn Pro Pro Arg Phe Pro Gln Ser Thr
260 265 270
Tyr Gln Phe Lys Thr Pro Glu Ser Ser Pro Pro Gly Thr Pro Ile Gly
275 280 285
Arg Ile Lys Ala Ser Asp Ala Asp Val Gly Glu Asn Ala Glu Ile Glu
290 295 300
Tyr Ser Ile Thr Asp Gly Glu Gly Leu Asp Met Phe Asp Val Ile Thr
305 310 315 320
Asp Gln Glu Thr Gln Glu Gly Ile Ile Thr Val Lys Lys Leu Leu Asp
325 330 335
Phe Glu Lys Lys Lys Val Tyr Thr Leu Lys Val Glu Ala Ser Asn Pro
340 345 350
Tyr Val Glu Pro Arg Phe Leu Tyr Leu Gly Pro Phe Lys Asp Ser Ala
355 360 365
Thr Val Arg Ile Val Val Glu Asp Val Asp Glu Pro Pro Val Phe Ser
370 375 380
Lys Leu Ala Tyr Ile Leu Gln Ile Arg Glu Asp Ala Gln Ile Asn Thr
385 390 395 400
Thr Ile Gly Ser Val Thr Ala Gln Asp Pro Asp Ala Ala Arg Asn Pro
405 410 415
Val Lys Tyr Ser Val Asp Arg His Thr Asp Met Asp Arg Ile Phe Asn
420 425 430
Ile Asp Ser Gly Asn Gly Ser Ile Phe Thr Ser Lys Leu Leu Asp Arg
435 440 445
Glu Thr Leu Leu Trp His Asn Ile Thr Val Ile Ala Thr Glu Ile Asn
450 455 460
Asn Pro Lys Gln Ser Ser Arg Val Pro Leu Tyr Ile Lys Val Leu Asp
465 470 475 480
Val Asn Asp Asn Ala Pro Glu Phe Ala Glu Phe Tyr Glu Thr Phe Val
485 490 495
Cys Glu Lys Ala Lys Ala Asp Gln Leu Ile Gln Thr Leu His Ala Val
500 505 510
Asp Lys Asp Asp Pro Tyr Ser Gly His Gln Phe Ser Phe Ser Leu Ala
515 520 525
Pro Glu Ala Ala Ser Gly Ser Asn Phe Thr Ile Gln Asp Asn Lys Asp
530 535 540
Asn Thr Ala Gly Ile Leu Thr Arg Lys Asn Gly Tyr Asn Arg His Glu
545 550 555 560
Met Ser Thr Tyr Leu Leu Pro Val Val Ile Ser Asp Asn Asp Tyr Pro
565 570 575
Val Gln Ser Ser Thr Gly Thr Val Thr Val Arg Val Cys Ala Cys Asp
580 585 590
His His Gly Asn Met Gln Ser Cys His Ala Glu Ala Leu Ile His Pro
595 600 605
Thr Gly Leu Ser Thr Gly Ala Leu Val Ala Ile Leu Leu Cys Ile Val
610 615 620
Ile Leu Leu Val Thr Val Val Leu Phe Ala Ala Leu Arg Arg Gln Arg
625 630 635 640
Lys Lys Glu Pro Leu Ile Ile Ser Lys Glu Asp Ile Arg Asp Asn Ile
645 650 655
Val Ser Tyr Asn Asp Glu Gly Gly Gly Glu Glu Asp Thr Gln Ala Phe
660 665 670
Asp Ile Gly Thr Leu Arg Asn Pro Glu Ala Ile Glu Asp Asn Lys Leu
675 680 685
Arg Arg Asp Ile Val Pro Glu Ala Leu Phe Leu Pro Arg Arg Thr Pro
690 695 700
Thr Ala Arg Asp Asn Thr Asp Val Arg Asp Phe Ile Asn Gln Arg Leu
705 710 715 720
Lys Glu Asn Asp Thr Asp Pro Thr Ala Pro Pro Tyr Asp Ser Leu Ala
725 730 735
Thr Tyr Ala Tyr Glu Gly Thr Gly Ser Val Ala Asp Ser Leu Ser Ser
740 745 750
Leu Glu Ser Val Thr Thr Asp Ala Asp Gln Asp Tyr Asp Tyr Leu Ser
755 760 765
Asp Trp Gly Pro Arg Phe Lys Lys Leu Ala Asp Met Tyr Gly Gly Val
770 775 780
Asp Ser Asp Lys Asp Ser
785 790
Claims (61)
- 하기 화학식의 항체 약물 접합체 또는 그의 제약상 허용되는 염.
Ab-(L-(D)m)n
여기서
Ab는 인간 CDH6의 에피토프에 특이적으로 결합하는 항체 또는 그의 항원 결합 단편이고;
L은 링커이고;
D는 약물 모이어티이고;
m은 1 내지 8의 정수이고;
n은 1 내지 10의 정수이다. - 제1항에 있어서, 상기 n이 3 또는 4인 항체 약물 접합체.
- 제1항 또는 제2항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 서열식별번호: 4의 아미노산 서열을 포함하는 CDH6의 세포외 도메인에 특이적으로 결합하는 것인 항체 약물 접합체.
- 제1항 또는 제2항에 있어서, 상기 항체 또는 항원 결합 단편이 서열식별번호: 534의 아미노산 서열을 포함하는 인간 CDH6의 에피토프에 특이적으로 결합하는 것인 항체 약물 접합체.
- 제1항 내지 제4항 중 어느 한 항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역. - 제1항 내지 제5항 중 어느 한 항에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 약물 접합체.
- 제1항 내지 제6항 중 어느 한 항에 있어서, CDR 내의 1 또는 2개의 아미노산이 변형, 결실 또는 치환된 것인 항체 약물 접합체.
- 제1항 내지 제4항 중 어느 한 항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL). - 제1항 내지 제4항 중 어느 한 항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 234를 포함하는 중쇄, 및 서열식별번호: 235를 포함하는 경쇄;
(ii) 서열식별번호: 220을 포함하는 중쇄, 및 서열식별번호: 221을 포함하는 경쇄;
(iii) 서열식별번호: 276을 포함하는 중쇄, 및 서열식별번호: 277을 포함하는 경쇄;
(iv) 서열식별번호: 318을 포함하는 중쇄, 및 서열식별번호: 319를 포함하는 경쇄;
(v) 서열식별번호: 24를 포함하는 중쇄, 및 서열식별번호: 25를 포함하는 경쇄;
(vi) 서열식별번호: 38을 포함하는 중쇄, 및 서열식별번호: 39를 포함하는 경쇄;
(vii) 서열식별번호: 52를 포함하는 중쇄, 및 서열식별번호: 53을 포함하는 경쇄;
(viii) 서열식별번호: 66을 포함하는 중쇄, 및 서열식별번호: 67을 포함하는 경쇄;
(ix) 서열식별번호: 80을 포함하는 중쇄, 및 서열식별번호: 81을 포함하는 경쇄;
(x) 서열식별번호: 94를 포함하는 중쇄, 및 서열식별번호: 95를 포함하는 경쇄;
(xi) 서열식별번호: 108을 포함하는 중쇄, 및 서열식별번호: 109를 포함하는 경쇄;
(xii) 서열식별번호: 122를 포함하는 중쇄, 및 서열식별번호: 123을 포함하는 경쇄;
(xiii) 서열식별번호: 136을 포함하는 중쇄, 및 서열식별번호: 137을 포함하는 경쇄;
(xiv) 서열식별번호: 150을 포함하는 중쇄, 및 서열식별번호: 151을 포함하는 경쇄;
(xv) 서열식별번호: 164를 포함하는 중쇄, 및 서열식별번호: 165를 포함하는 경쇄;
(xvi) 서열식별번호: 178을 포함하는 중쇄, 및 서열식별번호: 179를 포함하는 경쇄;
(xvii) 서열식별번호: 192를 포함하는 중쇄, 및 서열식별번호: 193을 포함하는 경쇄;
(xviii) 서열식별번호: 206을 포함하는 중쇄, 및 서열식별번호: 207을 포함하는 경쇄;
(xix) 서열식별번호: 248을 포함하는 중쇄, 및 서열식별번호: 249를 포함하는 경쇄;
(xx) 서열식별번호: 262를 포함하는 중쇄, 및 서열식별번호: 263을 포함하는 경쇄;
(xxi) 서열식별번호: 290을 포함하는 중쇄, 및 서열식별번호: 291을 포함하는 경쇄;
(xxii) 서열식별번호: 304를 포함하는 중쇄, 및 서열식별번호: 305를 포함하는 경쇄; 또는
(xxiii) 서열식별번호: 332를 포함하는 중쇄, 및 서열식별번호: 333을 포함하는 경쇄. - 제1항 내지 제9항 중 어느 한 항에 있어서, 가변 경쇄 또는 가변 중쇄 영역에 걸쳐 적어도 90, 91, 92, 93, 94, 95, 96, 97, 98 또는 99% 동일성을 보유하는 항체 약물 접합체.
- 제1항 내지 제10항 중 어느 한 항에 있어서, 항체가 모노클로날 항체, 키메라 항체, 인간화 항체, 인간 조작된 항체, 인간 항체, 단일 쇄 항체(scFv) 또는 항체 단편인 항체 약물 접합체.
- 제1항 내지 제11항 중 어느 한 항에 있어서, 상기 링커 (L)가 절단가능한 링커, 비-절단가능한 링커, 친수성 링커, 사전하전된 링커 및 디카르복실산 기반 링커로 이루어진 군으로부터 선택된 것인 항체 약물 접합체.
- 제12항에 있어서, 링커가 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB), N-숙신이미딜-3-(2-피리딜디티오)프로피오네이트 (SPDP), N-숙신이미딜 4-(2-피리딜디티오)펜타노에이트 (SPP), N-숙신이미딜 4-(2-피리딜디티오)부타노에이트 (SPDB), N-숙신이미딜 아이오도아세테이트 (SIA), N-숙신이미딜(4-아이오도아세틸)아미노벤조에이트 (SIAB), 말레이미드 PEG NHS, N-숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (SMCC), N-술포숙신이미딜 4-(말레이미도메틸) 시클로헥산카르복실레이트 (술포-SMCC) 또는 2,5-디옥소피롤리딘-1-일 17-(2,5-디옥소-2,5-디히드로-1H-피롤-1-일)-5,8,11,14-테트라옥소-4,7,10,13-테트라아자헵타데칸-1-오에이트 (CX1-1)로 이루어진 군으로부터 선택된 가교 시약으로부터 유래된 것인 항체 약물 접합체.
- 제13항에 있어서, 상기 링커가 N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)로부터 유래된 것인 항체 약물 접합체.
- 제1항 내지 제14항 중 어느 한 항에 있어서, 상기 약물 모이어티 (D)가 메이탄시노이드, V-ATPase 억제제, 아폽토시스촉진제, Bcl2 억제제, MCL1 억제제, HSP90 억제제, IAP 억제제, mTor 억제제, 미세관 안정화제, 미세관 탈안정화제, 아우리스타틴, 돌라스타틴, MetAP (메티오닌 아미노펩티다제), 단백질 CRM1의 핵 유출의 억제제, DPPIV 억제제, 프로테아솜 억제제, 미토콘드리아에서의 포스포릴 전달 반응의 억제제, 단백질 합성 억제제, 키나제 억제제, CDK2 억제제, CDK9 억제제, 키네신 억제제, HDAC 억제제, DNA 손상 작용제, DNA 알킬화제, DNA 삽입제, DNA 작은 홈 결합제 및 DHFR 억제제로 이루어진 군으로부터 선택된 것인 항체 약물 접합체.
- 제15항에 있어서, 약물 모이어티가 메이탄시노이드인 항체 약물 접합체.
- 제16항에 있어서, 메이탄시노이드가 N(2')-데아세틸-N2-(4-메르캅토-4-메틸-1-옥소펜틸)-메이탄신 (DM4) 또는 N(2')-데아세틸-N(2')-(3-메르캅토-1-옥소프로필)-메이탄신 (DM1)인 항체 약물 접합체.
- 제1항 내지 제17항 중 어느 한 항에 있어서, 또 다른 치료제와 조합된 항체 약물 접합체.
- 제1항 내지 제18항 중 어느 한 항에 있어서, 표 18에 열거된 치료제와 조합된 항체 약물 접합체.
- 제1항 내지 제19항 중 어느 한 항에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
- 제1항 내지 제20항 중 어느 한 항에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
- 제22항에 있어서, 상기 항체 또는 항원 결합 단편이 (서열식별번호: 4)에서의 인간 CDH6의 에피토프에 특이적으로 결합하는 것인 항체 약물 접합체.
- 제22항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 (서열식별번호: 534)에서의 인간 CDH6에 특이적으로 결합하는 것인 항체 약물 접합체.
- 제22항에 있어서, 상기 Ab가 다음을 포함하는 항체 또는 그의 항원 결합 단편인 항체 약물 접합체:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역. - 제22항 내지 제25항 중 어느 한 항에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 약물 접합체.
- 제22항 내지 제26항 중 어느 한 항에 있어서, CDR 내의 1 또는 2개의 아미노산이 변형, 결실 또는 치환된 것인 항체 약물 접합체.
- 제22항에 있어서, 상기 항체 또는 그의 항원 결합 단편이 다음을 포함하는 것인 항체 약물 접합체:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL). - 제22항 내지 제28항 중 어느 한 항에 있어서, 가변 경쇄 또는 가변 중쇄 영역에 걸쳐 적어도 90, 91, 92, 93, 94, 95, 96, 97, 98 또는 99% 동일성을 보유하는 항체 약물 접합체.
- 제22항 내지 제29항 중 어느 한 항에 있어서, 항체가 모노클로날 항체, 키메라 항체, 인간화 항체, 인간 조작된 항체, 인간 항체, 단일 쇄 항체(scFv) 또는 항체 단편인 항체 약물 접합체.
- 제22항 내지 제29항 중 어느 한 항에 있어서, 상기 n이 2 내지 8의 정수인 항체 약물 접합체.
- 제22항 내지 제29항 중 어느 한 항에 있어서, 상기 n이 3 내지 4의 정수인 항체 약물 접합체.
- 제22항 내지 제32항 중 어느 한 항에 있어서, 또 다른 치료제와 조합된 항체 약물 접합체.
- 제22항 내지 제32항 중 어느 한 항에 있어서, 표 18에 열거된 치료제와 조합된 항체 약물 접합체.
- 제22항 내지 제34항 중 어느 한 항에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
- 제22항 내지 제35항 중 어느 한 항에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
- 제1항 내지 제32항 중 어느 한 항의 항체 약물 접합체 및 제약상 허용되는 담체를 포함하는 제약 조성물.
- 제37항에 있어서, 동결건조물로서 제조되는 제약 조성물.
- CDH6 양성 암의 치료를 필요로 하는 환자에게 제1항 내지 제32항 중 어느 한 항의 항체 약물 접합체, 또는 제37항 또는 제38항의 제약 조성물을 투여하는 것을 포함하는, 상기 환자에서 CDH6 양성 암을 치료하는 방법.
- 제39항에 있어서, 상기 암이 난소암, 신암, 간암, 연부조직암, CNS암, 갑상선암 및 담관암종으로 이루어진 군으로부터 선택된 것인 방법.
- 제39항에 있어서, 항체 약물 접합체 또는 제약 조성물이 또 다른 치료제와 조합되어 투여되는 것인 방법.
- 제39항에 있어서, 항체 약물 접합체 또는 제약 조성물이 표 18에 열거된 치료제와 조합되어 투여되는 것인 방법.
- 제39항에 있어서, 항체 약물 접합체 또는 제약 조성물이 BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합되어 투여되는 것인 방법.
- 제39항에 있어서, 항체 약물 접합체 또는 제약 조성물이 면역 조정 분자와 조합되어 투여되는 것인 방법.
- 제1항 내지 제32항 중 어느 한 항에 있어서, 의약으로서 사용하기 위한 항체 약물 접합체.
- 제1항 내지 제32항, 제37항 및 제38항 중 어느 한 항에 있어서, CDH6 양성 암의 치료에 사용하기 위한 항체 약물 접합체 또는 제약 조성물.
- 제45항에 있어서, 또 다른 치료제와 조합되어 투여되는 항체 약물 접합체.
- 제45항에 있어서, 표 18에 열거된 치료제와 조합되어 투여되는 항체 약물 접합체.
- 제45항에 있어서, BCL2 억제제, BCL-XL 억제제, BCL2/BCL-XL 억제제, IAP 억제제 또는 MEK 억제제와 조합된 항체 약물 접합체.
- 제45항에 있어서, 면역 조정 분자와 조합된 항체 약물 접합체.
- 제1항 내지 제30항 중 어느 한 항의 항체 또는 항원 결합 단편을 코딩하는 핵산.
- 제51항의 핵산을 포함하는 벡터.
- 제52항에 따른 벡터를 포함하는 숙주 세포.
- 제53항의 숙주 세포를 배양하는 단계 및 항체를 배양물로부터 회수하는 단계를 포함하는, 항체 또는 항원 결합 단편을 생산하는 방법.
- (a) N-숙신이미딜-4-(2-피리딜디티오)2-술포-부타노에이트 (술포-SPDB)를 약물 모이어티 DM-4에 화학적으로 연결시키는 단계;
(b) 상기 링커-약물을 제55항의 세포 배양물로부터 회수된 항체에 접합시키는 단계; 및
(c) 항체 약물 접합체를 정제하는 단계
를 포함하는, 항-CDH6 항체 약물 접합체를 생산하는 방법. - UV 분광광도계로 측정된 평균 항체에 대한 메이탄시노이드의 비 (MAR)가 약 3.5인, 제55항에 따라 제조된 항체 약물 접합체.
- 다음을 포함하는 항체 또는 그의 항원 결합 단편:
(i) (a) 서열식별번호: 224의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 225의 LCDR2, (c) 서열식별번호: 226의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 227의 HCDR1, (e) 서열식별번호: 228의 HCDR2, 및 (f) 서열식별번호: 229의 HCDR3을 포함하는 중쇄 가변 영역;
(ii) (a) 서열식별번호: 210의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 211의 LCDR2, (c) 서열식별번호: 212의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 213의 HCDR1, (e) 서열식별번호: 214의 HCDR2, 및 (f) 서열식별번호: 215의 HCDR3을 포함하는 중쇄 가변 영역;
(iii) (a) 서열식별번호: 266의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 267의 LCDR2, (c) 서열식별번호: 268의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 269의 HCDR1, (e) 서열식별번호: 270의 HCDR2, 및 (f) 서열식별번호: 271의 HCDR3을 포함하는 중쇄 가변 영역;
(iv) (a) 서열식별번호: 308의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 309의 LCDR2, (c) 서열식별번호: 310의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 311의 HCDR1, (e) 서열식별번호: 312의 HCDR2, 및 (f) 서열식별번호: 313의 HCDR3을 포함하는 중쇄 가변 영역;
(v) (a) 서열식별번호: 14의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 15의 LCDR2, (c) 서열식별번호: 16의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 17의 HCDR1, (e) 서열식별번호: 18의 HCDR2, 및 (f) 서열식별번호: 19의 HCDR3을 포함하는 중쇄 가변 영역;
(vi) (a) 서열식별번호: 28의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 29의 LCDR2, (c) 서열식별번호: 30의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 31의 HCDR1, (e) 서열식별번호: 32의 HCDR2, 및 (f) 서열식별번호: 33의 HCDR3을 포함하는 중쇄 가변 영역;
(vii) (a) 서열식별번호: 42의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 43의 LCDR2, (c) 서열식별번호: 44의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 45의 HCDR1, (e) 서열식별번호: 46의 HCDR2, 및 (f) 서열식별번호: 47의 HCDR3을 포함하는 중쇄 가변 영역;
(viii) (a) 서열식별번호: 56의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 57의 LCDR2, (c) 서열식별번호: 58의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 59의 HCDR1, (e) 서열식별번호: 60의 HCDR2, 및 (f) 서열식별번호: 61의 HCDR3을 포함하는 중쇄 가변 영역;
(ix) (a) 서열식별번호: 70의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 71의 LCDR2, (c) 서열식별번호: 72의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 73의 HCDR1, (e) 서열식별번호: 74의 HCDR2, 및 (f) 서열식별번호: 75의 HCDR3을 포함하는 중쇄 가변 영역;
(x) (a) 서열식별번호: 84의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 85의 LCDR2, (c) 서열식별번호: 86의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 87의 HCDR1, (e) 서열식별번호: 88의 HCDR2, 및 (f) 서열식별번호: 89의 HCDR3을 포함하는 중쇄 가변 영역;
(xi) (a) 서열식별번호: 98의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 99의 LCDR2, (c) 서열식별번호: 100의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 101의 HCDR1, (e) 서열식별번호: 102의 HCDR2, 및 (f) 서열식별번호: 103의 HCDR3을 포함하는 중쇄 가변 영역;
(xii) (a) 서열식별번호: 112의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 113의 LCDR2, (c) 서열식별번호: 114의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 115의 HCDR1, (e) 서열식별번호: 116의 HCDR2, 및 (f) 서열식별번호: 117의 HCDR3을 포함하는 중쇄 가변 영역;
(xiii) (a) 서열식별번호: 126의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 127의 LCDR2, (c) 서열식별번호: 128의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 129의 HCDR1, (e) 서열식별번호: 130의 HCDR2, 및 (f) 서열식별번호: 131의 HCDR3을 포함하는 중쇄 가변 영역;
(xiv) (a) 서열식별번호: 140의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 141의 LCDR2, (c) 서열식별번호: 142의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 143의 HCDR1, (e) 서열식별번호: 144의 HCDR2, 및 (f) 서열식별번호: 145의 HCDR3을 포함하는 중쇄 가변 영역;
(xv) (a) 서열식별번호: 154의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 155의 LCDR2, (c) 서열식별번호: 156의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 157의 HCDR1, (e) 서열식별번호: 158의 HCDR2, 및 (f) 서열식별번호: 159의 HCDR3을 포함하는 중쇄 가변 영역;
(xvi) (a) 서열식별번호: 168의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 169의 LCDR2, (c) 서열식별번호: 170의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 171의 HCDR1, (e) 서열식별번호: 172의 HCDR2, 및 (f) 서열식별번호: 173의 HCDR3을 포함하는 중쇄 가변 영역;
(xvii) (a) 서열식별번호: 182의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 183의 LCDR2, (c) 서열식별번호: 184의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 185의 HCDR1, (e) 서열식별번호: 186의 HCDR2, 및 (f) 서열식별번호: 187의 HCDR3을 포함하는 중쇄 가변 영역;
(xviii) (a) 서열식별번호: 196의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 197의 LCDR2, (c) 서열식별번호: 198의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 199의 HCDR1, (e) 서열식별번호: 200의 HCDR2, 및 (f) 서열식별번호: 201의 HCDR3을 포함하는 중쇄 가변 영역;
(xix) (a) 서열식별번호: 238의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 239의 LCDR2, (c) 서열식별번호: 240의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 241의 HCDR1, (e) 서열식별번호: 242의 HCDR2, 및 (f) 서열식별번호: 243의 HCDR3을 포함하는 중쇄 가변 영역;
(xx) (a) 서열식별번호: 252의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 253의 LCDR2, (c) 서열식별번호: 254의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 255의 HCDR1, (e) 서열식별번호: 256의 HCDR2, 및 (f) 서열식별번호: 257의 HCDR3을 포함하는 중쇄 가변 영역;
(xxi) (a) 서열식별번호: 280의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 281의 LCDR2, (c) 서열식별번호: 282의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 283의 HCDR1, (e) 서열식별번호: 284의 HCDR2, 및 (f) 서열식별번호: 285의 HCDR3을 포함하는 중쇄 가변 영역;
(xxii) (a) 서열식별번호: 294의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 295의 LCDR2, (c) 서열식별번호: 296의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 297의 HCDR1, (e) 서열식별번호: 298의 HCDR2, 및 (f) 서열식별번호: 299의 HCDR3을 포함하는 중쇄 가변 영역; 또는
(xxiii) (a) 서열식별번호: 322의 LCDR1 (CDR-상보성 결정 영역), (b) 서열식별번호: 323의 LCDR2, (c) 서열식별번호: 324의 LCDR3을 포함하는 경쇄 가변 영역; 및 (d) 서열식별번호: 325의 HCDR1, (e) 서열식별번호: 326의 HCDR2, 및 (f) 서열식별번호: 327의 HCDR3을 포함하는 중쇄 가변 영역. - 제57항에 있어서, CDR 내의 적어도 1개의 아미노산이 표 5 또는 6 내의 또 다른 항-CDH6 항체의 상응하는 CDR의 상응하는 잔기에 의해 치환된 것인 항체 또는 그의 항원 결합 단편.
- 제57항에 있어서, 다음을 포함하는 항체 또는 그의 항원 결합 단편:
(i) 서열식별번호: 230을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 231을 포함하는 경쇄 가변 영역 (vL);
(ii) 서열식별번호: 216을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 217을 포함하는 경쇄 가변 영역 (vL);
(iii) 서열식별번호: 272를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 273을 포함하는 경쇄 가변 영역 (vL);
(iv) 서열식별번호: 314를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 315를 포함하는 경쇄 가변 영역 (vL);
(v) 서열식별번호: 20을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 21을 포함하는 경쇄 가변 영역 (vL);
(vi) 서열식별번호: 34를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 35를 포함하는 경쇄 가변 영역 (vL);
(vii) 서열식별번호: 48을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 49를 포함하는 경쇄 가변 영역 (vL);
(viii) 서열식별번호: 62를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 63을 포함하는 경쇄 가변 영역 (vL);
(ix) 서열식별번호: 76을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 77을 포함하는 경쇄 가변 영역 (vL);
(x) 서열식별번호: 90을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 91을 포함하는 경쇄 가변 영역 (vL);
(xi) 서열식별번호: 104를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 105를 포함하는 경쇄 가변 영역 (vL);
(xii) 서열식별번호: 118을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 119를 포함하는 경쇄 가변 영역 (vL);
(xiii) 서열식별번호: 132를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 133을 포함하는 경쇄 가변 영역 (vL);
(xiv) 서열식별번호: 146을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 147을 포함하는 경쇄 가변 영역 (vL);
(xv) 서열식별번호: 160을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 161을 포함하는 경쇄 가변 영역 (vL);
(xvi) 서열식별번호: 174를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 175를 포함하는 경쇄 가변 영역 (vL);
(xvii) 서열식별번호: 188을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 189를 포함하는 경쇄 가변 영역 (vL);
(xviii) 서열식별번호: 202를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 203을 포함하는 경쇄 가변 영역 (vL);
(xix) 서열식별번호: 244를 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 245를 포함하는 경쇄 가변 영역 (vL);
(xx) 서열식별번호: 258을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 259를 포함하는 경쇄 가변 영역 (vL);
(xxi) 서열식별번호: 286을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 287을 포함하는 경쇄 가변 영역 (vL);
(xxii) 서열식별번호: 300을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 301을 포함하는 경쇄 가변 영역 (vL); 또는
(xxiii) 서열식별번호: 328을 포함하는 중쇄 가변 영역 (vH), 및 서열식별번호: 329를 포함하는 경쇄 가변 영역 (vL). - 표지되어 있는 제57항 또는 제59항의 항체 또는 그의 항원 결합 단편을 포함하는 진단 시약.
- 제60항에 있어서, 표지가 방사성표지, 형광단, 발색단, 영상화제 및 금속 이온으로 이루어진 군으로부터 선택된 것인 진단 시약.
Applications Claiming Priority (3)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201462036382P | 2014-08-12 | 2014-08-12 | |
| US62/036,382 | 2014-08-12 | ||
| PCT/IB2015/056032 WO2016024195A1 (en) | 2014-08-12 | 2015-08-07 | Anti-cdh6 antibody drug conjugates |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| KR20170040249A true KR20170040249A (ko) | 2017-04-12 |
Family
ID=54150477
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020177003507A KR20170040249A (ko) | 2014-08-12 | 2015-08-07 | 항-cdh6 항체 약물 접합체 |
Country Status (27)
| Country | Link |
|---|---|
| US (2) | US10238748B2 (ko) |
| EP (1) | EP3180360A1 (ko) |
| JP (1) | JP2017524364A (ko) |
| KR (1) | KR20170040249A (ko) |
| CN (1) | CN106659790A (ko) |
| AP (1) | AP2017009719A0 (ko) |
| AR (1) | AR104664A1 (ko) |
| AU (1) | AU2015302959B2 (ko) |
| BR (1) | BR112017001588A2 (ko) |
| CA (1) | CA2954599A1 (ko) |
| CL (1) | CL2017000170A1 (ko) |
| CO (1) | CO2017001191A2 (ko) |
| CU (1) | CU20170014A7 (ko) |
| DO (1) | DOP2017000045A (ko) |
| EA (1) | EA201790334A1 (ko) |
| EC (1) | ECSP17011657A (ko) |
| IL (1) | IL250189A0 (ko) |
| MA (1) | MA40513A (ko) |
| MX (1) | MX2017001946A (ko) |
| NI (1) | NI201700016A (ko) |
| PE (1) | PE20170903A1 (ko) |
| PH (1) | PH12017500002A1 (ko) |
| SG (1) | SG11201700254QA (ko) |
| TN (1) | TN2016000577A1 (ko) |
| TW (1) | TW201613643A (ko) |
| UY (1) | UY36266A (ko) |
| WO (1) | WO2016024195A1 (ko) |
Cited By (1)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20200006061A (ko) * | 2017-05-15 | 2020-01-17 | 다이이찌 산쿄 가부시키가이샤 | 항 cdh6 항체 및 항 cdh6 항체-약물 콘주게이트 |
Families Citing this family (19)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US10441654B2 (en) | 2014-01-24 | 2019-10-15 | Children's Hospital Of Eastern Ontario Research Institute Inc. | SMC combination therapy for the treatment of cancer |
| AU2015302959B2 (en) * | 2014-08-12 | 2018-09-20 | Novartis Ag | Anti-CDH6 antibody drug conjugates |
| CN105603086B (zh) * | 2016-02-01 | 2019-03-05 | 中南大学 | 基于cdh6的eb病毒相关性鼻咽癌诊断试剂盒 |
| EP3442584B1 (en) | 2016-03-15 | 2021-07-28 | Seagen Inc. | Combinations of pbd-based antibody drug conjugates with bcl-2 inhibitors |
| EP3360898A1 (en) | 2017-02-14 | 2018-08-15 | Boehringer Ingelheim International GmbH | Bispecific anti-tnf-related apoptosis-inducing ligand receptor 2 and anti-cadherin 17 binding molecules for the treatment of cancer |
| WO2018185618A1 (en) * | 2017-04-03 | 2018-10-11 | Novartis Ag | Anti-cdh6 antibody drug conjugates and anti-gitr antibody combinations and methods of treatment |
| IL301636B2 (en) | 2017-09-29 | 2025-02-01 | Daiichi Sankyo Co Ltd | Conjugation of an antibody with a pyrrolobenzodiazepine derivative |
| WO2019195770A1 (en) | 2018-04-06 | 2019-10-10 | Atyr Pharma, Inc. | Compositions and methods comprising anti-nrp2 antibodies |
| TW202415410A (zh) | 2018-07-31 | 2024-04-16 | 日商第一三共股份有限公司 | 抗體-藥物結合物之用途 |
| JP7458981B2 (ja) * | 2018-08-06 | 2024-04-01 | 第一三共株式会社 | 抗体-薬物コンジュゲートとチューブリン阻害剤の組み合わせ |
| CA3119956A1 (en) | 2018-11-14 | 2020-05-22 | Daiichi Sankyo Company, Limited | Anti-cdh6 antibody-pyrrolobenzodiazepine derivative conjugate |
| CN112206327A (zh) * | 2019-07-12 | 2021-01-12 | 上海药明生物技术有限公司 | 一种抗体偶联药物的制备及其高通量筛选方法 |
| CA3156803A1 (en) | 2019-10-03 | 2021-04-08 | Atyr Pharma, Inc. | Compositions and methods comprising anti-nrp2 antibodies |
| AU2021322183A1 (en) * | 2020-08-04 | 2023-03-02 | Exelixis, Inc. | PD-L1 binding agents and uses thereof |
| CN114276432B (zh) * | 2020-09-27 | 2024-07-23 | 鲁南制药集团股份有限公司 | 一种重组人胰岛素c肽的纯化方法 |
| CN118317795A (zh) | 2021-09-15 | 2024-07-09 | 第一三共株式会社 | 用在治疗化疗耐药性癌症的方法中的抗体-药物偶联物 |
| WO2023102875A1 (en) * | 2021-12-10 | 2023-06-15 | Multitude Therapeutics Inc. | Anti-cdh6 antibody drug conjugate |
| AU2023274540A1 (en) | 2022-05-24 | 2024-12-12 | Daiichi Sankyo Company, Limited | Dosage regimen of an anti-cdh6 antibody-drug conjugate |
| WO2024165049A1 (zh) * | 2023-02-10 | 2024-08-15 | 山东先声生物制药有限公司 | 抗cdh6的抗体及其用途 |
Family Cites Families (121)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US633410A (en) | 1898-09-22 | 1899-09-19 | George A Ames | Ice-cutter. |
| US2429001A (en) | 1946-12-28 | 1947-10-14 | Axel H Stone | Artificial hand |
| US2779780A (en) | 1955-03-01 | 1957-01-29 | Du Pont | 1, 4-diamino-2, 3-dicyano-1, 4-bis (substituted mercapto) butadienes and their preparation |
| US3720760A (en) | 1968-09-06 | 1973-03-13 | Pharmacia Ab | Method for determining the presence of reagin-immunoglobulins(reagin-ig)directed against certain allergens,in aqueous samples |
| US3896111A (en) | 1973-02-20 | 1975-07-22 | Research Corp | Ansa macrolides |
| US4151042A (en) | 1977-03-31 | 1979-04-24 | Takeda Chemical Industries, Ltd. | Method for producing maytansinol and its derivatives |
| US4137230A (en) | 1977-11-14 | 1979-01-30 | Takeda Chemical Industries, Ltd. | Method for the production of maytansinoids |
| US4307016A (en) | 1978-03-24 | 1981-12-22 | Takeda Chemical Industries, Ltd. | Demethyl maytansinoids |
| US4265814A (en) | 1978-03-24 | 1981-05-05 | Takeda Chemical Industries | Matansinol 3-n-hexadecanoate |
| JPS5562090A (en) | 1978-10-27 | 1980-05-10 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS5566585A (en) | 1978-11-14 | 1980-05-20 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4256746A (en) | 1978-11-14 | 1981-03-17 | Takeda Chemical Industries | Dechloromaytansinoids, their pharmaceutical compositions and method of use |
| JPS55164687A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55102583A (en) | 1979-01-31 | 1980-08-05 | Takeda Chem Ind Ltd | 20-acyloxy-20-demethylmaytansinoid compound |
| JPS55162791A (en) | 1979-06-05 | 1980-12-18 | Takeda Chem Ind Ltd | Antibiotic c-15003pnd and its preparation |
| JPS55164685A (en) | 1979-06-08 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| JPS55164686A (en) | 1979-06-11 | 1980-12-22 | Takeda Chem Ind Ltd | Novel maytansinoid compound and its preparation |
| US4309428A (en) | 1979-07-30 | 1982-01-05 | Takeda Chemical Industries, Ltd. | Maytansinoids |
| JPS5645483A (en) | 1979-09-19 | 1981-04-25 | Takeda Chem Ind Ltd | C-15003phm and its preparation |
| EP0028683A1 (en) | 1979-09-21 | 1981-05-20 | Takeda Chemical Industries, Ltd. | Antibiotic C-15003 PHO and production thereof |
| JPS5645485A (en) | 1979-09-21 | 1981-04-25 | Takeda Chem Ind Ltd | Production of c-15003pnd |
| US4458066A (en) | 1980-02-29 | 1984-07-03 | University Patents, Inc. | Process for preparing polynucleotides |
| WO1982001188A1 (en) | 1980-10-08 | 1982-04-15 | Takeda Chemical Industries Ltd | 4,5-deoxymaytansinoide compounds and process for preparing same |
| US4450254A (en) | 1980-11-03 | 1984-05-22 | Standard Oil Company | Impact improvement of high nitrile resins |
| US4313946A (en) | 1981-01-27 | 1982-02-02 | The United States Of America As Represented By The Secretary Of Agriculture | Chemotherapeutically active maytansinoids from Trewia nudiflora |
| US4315929A (en) | 1981-01-27 | 1982-02-16 | The United States Of America As Represented By The Secretary Of Agriculture | Method of controlling the European corn borer with trewiasine |
| JPS57192389A (en) | 1981-05-20 | 1982-11-26 | Takeda Chem Ind Ltd | Novel maytansinoid |
| EP0092918B1 (en) | 1982-04-22 | 1988-10-19 | Imperial Chemical Industries Plc | Continuous release formulations |
| US4522811A (en) | 1982-07-08 | 1985-06-11 | Syntex (U.S.A.) Inc. | Serial injection of muramyldipeptides and liposomes enhances the anti-infective activity of muramyldipeptides |
| US5128326A (en) | 1984-12-06 | 1992-07-07 | Biomatrix, Inc. | Drug delivery systems based on hyaluronans derivatives thereof and their salts and methods of producing same |
| US5374548A (en) | 1986-05-02 | 1994-12-20 | Genentech, Inc. | Methods and compositions for the attachment of proteins to liposomes using a glycophospholipid anchor |
| MX9203291A (es) | 1985-06-26 | 1992-08-01 | Liposome Co Inc | Metodo para acoplamiento de liposomas. |
| AU600575B2 (en) | 1987-03-18 | 1990-08-16 | Sb2, Inc. | Altered antibodies |
| US4880078A (en) | 1987-06-29 | 1989-11-14 | Honda Giken Kogyo Kabushiki Kaisha | Exhaust muffler |
| US5677425A (en) | 1987-09-04 | 1997-10-14 | Celltech Therapeutics Limited | Recombinant antibody |
| US5108921A (en) | 1989-04-03 | 1992-04-28 | Purdue Research Foundation | Method for enhanced transmembrane transport of exogenous molecules |
| AU6430190A (en) | 1989-10-10 | 1991-05-16 | Pitman-Moore, Inc. | Sustained release composition for macromolecular proteins |
| US5208020A (en) | 1989-10-25 | 1993-05-04 | Immunogen Inc. | Cytotoxic agents comprising maytansinoids and their therapeutic use |
| EP0550436A1 (en) | 1989-11-06 | 1993-07-14 | Alkermes Controlled Therapeutics, Inc. | Protein microspheres and methods of using them |
| EP0583356B1 (en) | 1991-05-01 | 2002-07-31 | Henry M. Jackson Foundation For The Advancement Of Military Medicine | A method for treating infectious respiratory diseases |
| US5714350A (en) | 1992-03-09 | 1998-02-03 | Protein Design Labs, Inc. | Increasing antibody affinity by altering glycosylation in the immunoglobulin variable region |
| US5912015A (en) | 1992-03-12 | 1999-06-15 | Alkermes Controlled Therapeutics, Inc. | Modulated release from biocompatible polymers |
| CA2076465C (en) | 1992-03-25 | 2002-11-26 | Ravi V. J. Chari | Cell binding agent conjugates of analogues and derivatives of cc-1065 |
| EP0640094A1 (en) | 1992-04-24 | 1995-03-01 | The Board Of Regents, The University Of Texas System | Recombinant production of immunoglobulin-like domains in prokaryotic cells |
| US5798224A (en) * | 1992-12-29 | 1998-08-25 | Doheny Eye Institute | Nucleic acids encoding protocadherin |
| US5934272A (en) | 1993-01-29 | 1999-08-10 | Aradigm Corporation | Device and method of creating aerosolized mist of respiratory drug |
| EP0714409A1 (en) | 1993-06-16 | 1996-06-05 | Celltech Therapeutics Limited | Antibodies |
| US5834252A (en) | 1995-04-18 | 1998-11-10 | Glaxo Group Limited | End-complementary polymerase reaction |
| US5837458A (en) | 1994-02-17 | 1998-11-17 | Maxygen, Inc. | Methods and compositions for cellular and metabolic engineering |
| US5605793A (en) | 1994-02-17 | 1997-02-25 | Affymax Technologies N.V. | Methods for in vitro recombination |
| US6132764A (en) | 1994-08-05 | 2000-10-17 | Targesome, Inc. | Targeted polymerized liposome diagnostic and treatment agents |
| CA2207961A1 (en) | 1995-01-05 | 1996-07-11 | Robert J. Levy | Surface-modified nanoparticles and method of making and using same |
| US5641870A (en) | 1995-04-20 | 1997-06-24 | Genentech, Inc. | Low pH hydrophobic interaction chromatography for antibody purification |
| US6121022A (en) | 1995-04-14 | 2000-09-19 | Genentech, Inc. | Altered polypeptides with increased half-life |
| US6019968A (en) | 1995-04-14 | 2000-02-01 | Inhale Therapeutic Systems, Inc. | Dispersible antibody compositions and methods for their preparation and use |
| US5869046A (en) | 1995-04-14 | 1999-02-09 | Genentech, Inc. | Altered polypeptides with increased half-life |
| WO1997007788A2 (en) | 1995-08-31 | 1997-03-06 | Alkermes Controlled Therapeutics, Inc. | Composition for sustained release of an agent |
| WO1997032572A2 (en) | 1996-03-04 | 1997-09-12 | The Penn State Research Foundation | Materials and methods for enhancing cellular internalization |
| US5874064A (en) | 1996-05-24 | 1999-02-23 | Massachusetts Institute Of Technology | Aerodynamically light particles for pulmonary drug delivery |
| US5985309A (en) | 1996-05-24 | 1999-11-16 | Massachusetts Institute Of Technology | Preparation of particles for inhalation |
| US5855913A (en) | 1997-01-16 | 1999-01-05 | Massachusetts Instite Of Technology | Particles incorporating surfactants for pulmonary drug delivery |
| US6056973A (en) | 1996-10-11 | 2000-05-02 | Sequus Pharmaceuticals, Inc. | Therapeutic liposome composition and method of preparation |
| WO1998031346A1 (en) | 1997-01-16 | 1998-07-23 | Massachusetts Institute Of Technology | Preparation of particles for inhalation |
| US6277375B1 (en) | 1997-03-03 | 2001-08-21 | Board Of Regents, The University Of Texas System | Immunoglobulin-like domains with increased half-lives |
| AU736549B2 (en) | 1997-05-21 | 2001-08-02 | Merck Patent Gesellschaft Mit Beschrankter Haftung | Method for the production of non-immunogenic proteins |
| WO1998056915A2 (en) | 1997-06-12 | 1998-12-17 | Research Corporation Technologies, Inc. | Artificial antibody polypeptides |
| US5989463A (en) | 1997-09-24 | 1999-11-23 | Alkermes Controlled Therapeutics, Inc. | Methods for fabricating polymer-based controlled release devices |
| SE512663C2 (sv) | 1997-10-23 | 2000-04-17 | Biogram Ab | Inkapslingsförfarande för aktiv substans i en bionedbrytbar polymer |
| US6194551B1 (en) | 1998-04-02 | 2001-02-27 | Genentech, Inc. | Polypeptide variants |
| EP2180007B2 (en) | 1998-04-20 | 2017-08-30 | Roche Glycart AG | Glycosylation engineering of antibodies for improving antibody-dependent cellular cytotoxicity |
| ATE238768T1 (de) | 1998-06-24 | 2003-05-15 | Advanced Inhalation Res Inc | Grosse poröse partikel ausgestossen von einem inhalator |
| KR100609800B1 (ko) | 1998-12-16 | 2006-08-09 | 워너-램버트 캄파니 엘엘씨 | Mek 저해제를 사용한 관절염 치료 방법 |
| US6737056B1 (en) | 1999-01-15 | 2004-05-18 | Genentech, Inc. | Polypeptide variants with altered effector function |
| CN1763097B (zh) | 1999-01-15 | 2011-04-13 | 杰南技术公司 | 具有改变的效应功能的多肽变体 |
| CA2369292C (en) | 1999-04-09 | 2010-09-21 | Kyowa Hakko Kogyo Co. Ltd. | Method of modulating the activity of functional immune molecules |
| PT1242401E (pt) | 1999-11-24 | 2007-03-30 | Immunogen Inc | Agentes citotóxicos compreendendo taxanos e o seu uso terapêutico |
| EP1242438B1 (en) | 1999-12-29 | 2006-11-08 | Immunogen, Inc. | Cytotoxic agents comprising modified doxorubicins and daunorubicins and their therapeutic use |
| AU7349801A (en) | 2000-07-19 | 2002-01-30 | Warner Lambert Co | Oxygenated esters of 4-iodo phenylamino benzhydroxamic acids |
| US6411163B1 (en) | 2000-08-14 | 2002-06-25 | Intersil Americas Inc. | Transconductance amplifier circuit |
| US6333410B1 (en) | 2000-08-18 | 2001-12-25 | Immunogen, Inc. | Process for the preparation and purification of thiol-containing maytansinoids |
| US6441163B1 (en) | 2001-05-31 | 2002-08-27 | Immunogen, Inc. | Methods for preparation of cytotoxic conjugates of maytansinoids and cell binding agents |
| ES2326964T3 (es) | 2001-10-25 | 2009-10-22 | Genentech, Inc. | Composiciones de glicoproteina. |
| CA2472341C (en) | 2002-02-01 | 2011-06-21 | Ariad Gene Therapeutics, Inc. | Phosphorus-containing compounds & uses thereof |
| US7799827B2 (en) | 2002-03-08 | 2010-09-21 | Eisai Co., Ltd. | Macrocyclic compounds useful as pharmaceuticals |
| TWI275390B (en) | 2002-04-30 | 2007-03-11 | Wyeth Corp | Process for the preparation of 7-substituted-3- quinolinecarbonitriles |
| GB0215823D0 (en) | 2002-07-09 | 2002-08-14 | Astrazeneca Ab | Quinazoline derivatives |
| AU2003265276A1 (en) | 2002-07-15 | 2004-02-02 | The Trustees Of Princeton University | Iap binding compounds |
| CN1477122A (zh) * | 2002-08-23 | 2004-02-25 | 上海博德基因开发有限公司 | 一种多肽——人血管上皮钙粘着蛋白54和编码这种多肽的多核苷酸 |
| US8088387B2 (en) | 2003-10-10 | 2012-01-03 | Immunogen Inc. | Method of targeting specific cell populations using cell-binding agent maytansinoid conjugates linked via a non-cleavable linker, said conjugates, and methods of making said conjugates |
| WO2005001092A2 (en) * | 2003-05-20 | 2005-01-06 | Wyeth | Compositions and methods for diagnosing and treating cancers |
| US7399865B2 (en) | 2003-09-15 | 2008-07-15 | Wyeth | Protein tyrosine kinase enzyme inhibitors |
| US7932382B2 (en) | 2004-01-16 | 2011-04-26 | The Regents Of The University Of Michigan | Conformationally constrained Smac mimetics and the uses thereof |
| US20100093645A1 (en) | 2004-01-16 | 2010-04-15 | Shaomeng Wang | SMAC Peptidomimetics and the Uses Thereof |
| CA2558615C (en) | 2004-03-23 | 2013-10-29 | Genentech, Inc. | Azabicyclo-octane inhibitors of iap |
| SI2253614T1 (sl) | 2004-04-07 | 2013-01-31 | Novartis Ag | Inhibitorji za IAP |
| SI1778718T1 (sl) | 2004-07-02 | 2015-01-30 | Genentech, Inc. | Inhibitorji IAP |
| US7674787B2 (en) | 2004-07-09 | 2010-03-09 | The Regents Of The University Of Michigan | Conformationally constrained Smac mimetics and the uses thereof |
| EA200700225A1 (ru) | 2004-07-12 | 2008-02-28 | Айдан Фармасьютикалз, Инк. | Аналоги тетрапептида |
| CA2574040C (en) | 2004-07-15 | 2014-05-06 | Tetralogic Pharmaceuticals Corporation | Iap binding compounds |
| EA019420B1 (ru) | 2004-12-20 | 2014-03-31 | Дженентех, Инк. | Пирролидиновые ингибиторы иап (ингибиторов апоптоза) |
| CA2597407C (en) | 2005-02-11 | 2013-09-10 | Immunogen, Inc. | Process for preparing stable drug conjugates |
| US20110166319A1 (en) | 2005-02-11 | 2011-07-07 | Immunogen, Inc. | Process for preparing purified drug conjugates |
| GB0510390D0 (en) | 2005-05-20 | 2005-06-29 | Novartis Ag | Organic compounds |
| EP1912636B1 (en) | 2005-07-21 | 2014-06-25 | Ardea Biosciences, Inc. | N-(arylamino)-sulfonamide inhibitors of mek |
| JP5350792B2 (ja) | 2005-08-24 | 2013-11-27 | イムノゲン インコーポレーティッド | メイタンシノイド(maytansinoid)抗体複合体の調製方法 |
| WO2007030642A2 (en) | 2005-09-07 | 2007-03-15 | Medimmune, Inc. | Toxin conjugated eph receptor antibodies |
| JO2660B1 (en) | 2006-01-20 | 2012-06-17 | نوفارتيس ايه جي | Pi-3 inhibitors and methods of use |
| TWI432212B (zh) | 2007-04-30 | 2014-04-01 | Genentech Inc | Iap抑制劑 |
| RU2523890C2 (ru) | 2007-09-12 | 2014-07-27 | Дженентек, Инк. | Комбинации ингибиторов фосфоинозитид 3-киназы и химиотерапевтических агентов и способы применения |
| ES2439705T3 (es) | 2007-10-25 | 2014-01-24 | Genentech, Inc. | Proceso para la preparación de compuestos de tienopirimidina |
| NO2281006T3 (ko) | 2008-04-30 | 2017-12-30 | ||
| US8168784B2 (en) | 2008-06-20 | 2012-05-01 | Abbott Laboratories | Processes to make apoptosis promoters |
| SI2437790T1 (sl) | 2009-06-03 | 2019-07-31 | Immunogen, Inc. | Konjugacijske metode |
| KR20120080611A (ko) * | 2009-10-06 | 2012-07-17 | 이뮤노젠 아이엔씨 | 효능 있는 접합체 및 친수성 링커 |
| AR083847A1 (es) | 2010-11-15 | 2013-03-27 | Novartis Ag | Variantes de fc (fragmento constante) silenciosas de los anticuerpos anti-cd40 |
| BR112013025228A2 (pt) | 2011-03-29 | 2018-09-25 | Immunogen Inc | processo para fabricar conjugados de homogeneidade aperfeçoada |
| TR201902180T4 (tr) | 2011-03-29 | 2019-03-21 | Immunogen Inc | Tek adimli bi̇r i̇şlem i̇le maytansi̇noi̇d anti̇kor konjugatlarinin hazirlanmasi |
| US9999680B2 (en) * | 2013-02-28 | 2018-06-19 | Immunogen, Inc. | Conjugates comprising cell-binding agents and maytansinoids as cytotoxic agents |
| US9789203B2 (en) | 2013-03-15 | 2017-10-17 | Novartis Ag | cKIT antibody drug conjugates |
| AU2015302959B2 (en) * | 2014-08-12 | 2018-09-20 | Novartis Ag | Anti-CDH6 antibody drug conjugates |
| US11605302B2 (en) | 2020-11-10 | 2023-03-14 | Rockwell Collins, Inc. | Time-critical obstacle avoidance path planning in uncertain environments |
-
2015
- 2015-08-07 AU AU2015302959A patent/AU2015302959B2/en not_active Ceased
- 2015-08-07 CA CA2954599A patent/CA2954599A1/en not_active Abandoned
- 2015-08-07 PE PE2017000173A patent/PE20170903A1/es unknown
- 2015-08-07 SG SG11201700254QA patent/SG11201700254QA/en unknown
- 2015-08-07 EP EP15766632.2A patent/EP3180360A1/en not_active Withdrawn
- 2015-08-07 MX MX2017001946A patent/MX2017001946A/es unknown
- 2015-08-07 TN TN2016000577A patent/TN2016000577A1/en unknown
- 2015-08-07 KR KR1020177003507A patent/KR20170040249A/ko unknown
- 2015-08-07 US US15/502,478 patent/US10238748B2/en not_active Expired - Fee Related
- 2015-08-07 MA MA040513A patent/MA40513A/fr unknown
- 2015-08-07 AP AP2017009719A patent/AP2017009719A0/en unknown
- 2015-08-07 EA EA201790334A patent/EA201790334A1/ru unknown
- 2015-08-07 BR BR112017001588A patent/BR112017001588A2/pt not_active Application Discontinuation
- 2015-08-07 CN CN201580043072.7A patent/CN106659790A/zh active Pending
- 2015-08-07 US US14/820,897 patent/US9982045B2/en not_active Expired - Fee Related
- 2015-08-07 WO PCT/IB2015/056032 patent/WO2016024195A1/en active Application Filing
- 2015-08-07 JP JP2017507835A patent/JP2017524364A/ja active Pending
- 2015-08-11 TW TW104126127A patent/TW201613643A/zh unknown
- 2015-08-11 UY UY0001036266A patent/UY36266A/es not_active Application Discontinuation
- 2015-08-12 AR ARP150102601A patent/AR104664A1/es unknown
-
2017
- 2017-01-03 PH PH12017500002A patent/PH12017500002A1/en unknown
- 2017-01-08 IL IL250189A patent/IL250189A0/en unknown
- 2017-01-23 CL CL2017000170A patent/CL2017000170A1/es unknown
- 2017-02-08 CO CONC2017/0001191A patent/CO2017001191A2/es unknown
- 2017-02-09 DO DO2017000045A patent/DOP2017000045A/es unknown
- 2017-02-10 NI NI201700016A patent/NI201700016A/es unknown
- 2017-02-10 CU CUP2017000014A patent/CU20170014A7/es unknown
- 2017-02-25 EC ECIEPI201711657A patent/ECSP17011657A/es unknown
Cited By (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| KR20200006061A (ko) * | 2017-05-15 | 2020-01-17 | 다이이찌 산쿄 가부시키가이샤 | 항 cdh6 항체 및 항 cdh6 항체-약물 콘주게이트 |
| US12233155B2 (en) | 2017-05-15 | 2025-02-25 | Daiichi Sankyo Company, Limited | Polynucleotides encoding antibodies which bind the EC3 domain of cadherin-6 (CDH6) and possess internalization ability |
Also Published As
| Publication number | Publication date |
|---|---|
| US20160046711A1 (en) | 2016-02-18 |
| AU2015302959B2 (en) | 2018-09-20 |
| CN106659790A (zh) | 2017-05-10 |
| JP2017524364A (ja) | 2017-08-31 |
| SG11201700254QA (en) | 2017-02-27 |
| MX2017001946A (es) | 2017-06-19 |
| TN2016000577A1 (en) | 2018-04-04 |
| US9982045B2 (en) | 2018-05-29 |
| AP2017009719A0 (en) | 2017-01-31 |
| US10238748B2 (en) | 2019-03-26 |
| US20170224836A1 (en) | 2017-08-10 |
| DOP2017000045A (es) | 2017-05-15 |
| CU20170014A7 (es) | 2017-04-05 |
| PH12017500002A1 (en) | 2017-05-15 |
| CL2017000170A1 (es) | 2017-07-14 |
| TW201613643A (en) | 2016-04-16 |
| WO2016024195A1 (en) | 2016-02-18 |
| PE20170903A1 (es) | 2017-07-12 |
| AU2015302959A1 (en) | 2017-02-02 |
| MA40513A (fr) | 2017-06-21 |
| CA2954599A1 (en) | 2016-02-18 |
| AR104664A1 (es) | 2017-08-09 |
| EP3180360A1 (en) | 2017-06-21 |
| BR112017001588A2 (pt) | 2017-11-21 |
| CO2017001191A2 (es) | 2017-04-28 |
| ECSP17011657A (es) | 2018-03-31 |
| UY36266A (es) | 2016-04-01 |
| IL250189A0 (en) | 2017-03-30 |
| EA201790334A1 (ru) | 2017-06-30 |
| NI201700016A (es) | 2017-03-08 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2015302959B2 (en) | Anti-CDH6 antibody drug conjugates | |
| CN110869389B (zh) | 抗ror1抗体及其制备和使用方法 | |
| AU2017202150B2 (en) | Antibody drug conjugates and corresponding antibodies | |
| KR102096224B1 (ko) | 폴리펩티드 구축물 및 이의 용도 | |
| KR102569813B1 (ko) | Cd73에 대항한 항체 및 그의 용도 | |
| KR20210134300A (ko) | 항-sars-cov-2 스파이크 당단백질 항체 및 항원-결합 단편 | |
| KR20220033522A (ko) | 항-cd73 항체와의 조합 요법 | |
| TW202323292A (zh) | 抗體藥物結合物 | |
| KR20170084202A (ko) | 항체 약물 접합체 | |
| CN114163531A (zh) | 组织因子途径抑制剂抗体及其用途 | |
| US20240189439A1 (en) | Antibodies to pmel17 and conjugates thereof | |
| WO2018185618A1 (en) | Anti-cdh6 antibody drug conjugates and anti-gitr antibody combinations and methods of treatment | |
| KR20230017815A (ko) | 항-sars-cov-2 스파이크 당단백질 항체 및 항원-결합 단편 | |
| KR20150122730A (ko) | 암의 치료에서 항-gcc 항체-약물 콘주게이트 및 dna 손상 제제의 투여 | |
| IL294921A (en) | Anti-e-selectin antibodies, compositions and methods of use | |
| RU2831785C2 (ru) | Антитела к рмel17 и конъюгаты на их основе | |
| RU2777662C2 (ru) | Конъюгаты антитела к ccr7 и лекарственного средства | |
| KR20230002910A (ko) | 암 치료를 위한 ccr7 항체 약물 접합체 | |
| KR20230117379A (ko) | 이종이량체 psma 및 cd3-결합 이중특이적 항체 | |
| CN117242096A (zh) | 结合psma和cd3的异二聚体双特异性抗体 | |
| NZ790996A (en) | Anti-ccr7 antibody drug conjugates |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| PA0105 | International application |
Patent event date: 20170208 Patent event code: PA01051R01D Comment text: International Patent Application |
|
| PG1501 | Laying open of application | ||
| PC1203 | Withdrawal of no request for examination |