ES2811345T3 - Anticuerpos contra ICOS - Google Patents
Anticuerpos contra ICOS Download PDFInfo
- Publication number
- ES2811345T3 ES2811345T3 ES16769535T ES16769535T ES2811345T3 ES 2811345 T3 ES2811345 T3 ES 2811345T3 ES 16769535 T ES16769535 T ES 16769535T ES 16769535 T ES16769535 T ES 16769535T ES 2811345 T3 ES2811345 T3 ES 2811345T3
- Authority
- ES
- Spain
- Prior art keywords
- seq
- amino acid
- acid sequence
- antibody
- icos
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 125000003275 alpha amino acid group Chemical group 0.000 claims abstract description 837
- 241000282414 Homo sapiens Species 0.000 claims abstract description 209
- 210000004027 cell Anatomy 0.000 claims description 293
- 206010028980 Neoplasm Diseases 0.000 claims description 190
- 210000001744 T-lymphocyte Anatomy 0.000 claims description 125
- 238000000034 method Methods 0.000 claims description 105
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 97
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 94
- 108010074708 B7-H1 Antigen Proteins 0.000 claims description 90
- 229920001184 polypeptide Polymers 0.000 claims description 90
- 210000003289 regulatory T cell Anatomy 0.000 claims description 88
- 230000014509 gene expression Effects 0.000 claims description 87
- 208000002154 non-small cell lung carcinoma Diseases 0.000 claims description 59
- 208000029729 tumor suppressor gene on chromosome 11 Diseases 0.000 claims description 59
- 201000011510 cancer Diseases 0.000 claims description 53
- 238000010186 staining Methods 0.000 claims description 53
- 239000003814 drug Substances 0.000 claims description 50
- 241000124008 Mammalia Species 0.000 claims description 43
- 208000003721 Triple Negative Breast Neoplasms Diseases 0.000 claims description 40
- 229940124597 therapeutic agent Drugs 0.000 claims description 40
- 208000022679 triple-negative breast carcinoma Diseases 0.000 claims description 40
- 239000013598 vector Substances 0.000 claims description 33
- 238000003364 immunohistochemistry Methods 0.000 claims description 32
- 229940022399 cancer vaccine Drugs 0.000 claims description 25
- 238000009566 cancer vaccine Methods 0.000 claims description 25
- 150000007523 nucleic acids Chemical class 0.000 claims description 25
- 102000039446 nucleic acids Human genes 0.000 claims description 24
- 108020004707 nucleic acids Proteins 0.000 claims description 24
- 230000007423 decrease Effects 0.000 claims description 23
- 206010041823 squamous cell carcinoma Diseases 0.000 claims description 22
- 201000001441 melanoma Diseases 0.000 claims description 20
- 208000017572 squamous cell neoplasm Diseases 0.000 claims description 18
- 125000000539 amino acid group Chemical group 0.000 claims description 17
- 208000031671 Large B-Cell Diffuse Lymphoma Diseases 0.000 claims description 16
- 208000006265 Renal cell carcinoma Diseases 0.000 claims description 16
- 208000005718 Stomach Neoplasms Diseases 0.000 claims description 16
- 206010012818 diffuse large B-cell lymphoma Diseases 0.000 claims description 16
- 206010017758 gastric cancer Diseases 0.000 claims description 16
- 201000011549 stomach cancer Diseases 0.000 claims description 16
- 206010033128 Ovarian cancer Diseases 0.000 claims description 15
- 206010061535 Ovarian neoplasm Diseases 0.000 claims description 15
- 206010005003 Bladder cancer Diseases 0.000 claims description 11
- 208000007097 Urinary Bladder Neoplasms Diseases 0.000 claims description 11
- 201000005112 urinary bladder cancer Diseases 0.000 claims description 11
- 230000004913 activation Effects 0.000 claims description 10
- 210000003162 effector t lymphocyte Anatomy 0.000 claims description 9
- 208000017604 Hodgkin disease Diseases 0.000 claims description 8
- 208000021519 Hodgkin lymphoma Diseases 0.000 claims description 8
- 208000010747 Hodgkins lymphoma Diseases 0.000 claims description 8
- 229960005486 vaccine Drugs 0.000 claims description 7
- 102000008096 B7-H1 Antigen Human genes 0.000 claims description 6
- 210000000952 spleen Anatomy 0.000 claims description 6
- 208000009956 adenocarcinoma Diseases 0.000 claims description 5
- 241000700605 Viruses Species 0.000 claims description 4
- 108010041986 DNA Vaccines Proteins 0.000 claims description 3
- 229940021995 DNA vaccine Drugs 0.000 claims description 3
- 229940030325 tumor cell vaccine Drugs 0.000 claims description 3
- 238000012258 culturing Methods 0.000 claims description 2
- 210000004899 c-terminal region Anatomy 0.000 claims 1
- 101710205775 Inducible T-cell costimulator Proteins 0.000 description 264
- 102100021317 Inducible T-cell costimulator Human genes 0.000 description 264
- 108010047041 Complementarity Determining Regions Proteins 0.000 description 166
- 230000027455 binding Effects 0.000 description 89
- 102100024216 Programmed cell death 1 ligand 1 Human genes 0.000 description 85
- 102100036011 T-cell surface glycoprotein CD4 Human genes 0.000 description 74
- 238000011282 treatment Methods 0.000 description 72
- 238000003556 assay Methods 0.000 description 62
- 239000000427 antigen Substances 0.000 description 57
- 108091007433 antigens Proteins 0.000 description 55
- 102000036639 antigens Human genes 0.000 description 55
- 101000946843 Homo sapiens T-cell surface glycoprotein CD8 alpha chain Proteins 0.000 description 50
- 102100034922 T-cell surface glycoprotein CD8 alpha chain Human genes 0.000 description 50
- 108090000623 proteins and genes Proteins 0.000 description 47
- 241000699670 Mus sp. Species 0.000 description 45
- 230000000694 effects Effects 0.000 description 45
- 235000001014 amino acid Nutrition 0.000 description 44
- 230000037396 body weight Effects 0.000 description 42
- 239000000523 sample Substances 0.000 description 39
- 239000000203 mixture Substances 0.000 description 38
- 102000004169 proteins and genes Human genes 0.000 description 38
- 241000699666 Mus <mouse, genus> Species 0.000 description 37
- 235000018102 proteins Nutrition 0.000 description 37
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 35
- 108010012236 Chemokines Proteins 0.000 description 34
- 238000002560 therapeutic procedure Methods 0.000 description 33
- 102000019034 Chemokines Human genes 0.000 description 32
- 239000000556 agonist Substances 0.000 description 31
- 239000012636 effector Substances 0.000 description 31
- 108091033319 polynucleotide Proteins 0.000 description 31
- 102000040430 polynucleotide Human genes 0.000 description 31
- 239000002157 polynucleotide Substances 0.000 description 31
- 102100034980 ICOS ligand Human genes 0.000 description 30
- 229940024606 amino acid Drugs 0.000 description 30
- 150000001413 amino acids Chemical class 0.000 description 30
- 230000010056 antibody-dependent cellular cytotoxicity Effects 0.000 description 30
- 230000006870 function Effects 0.000 description 30
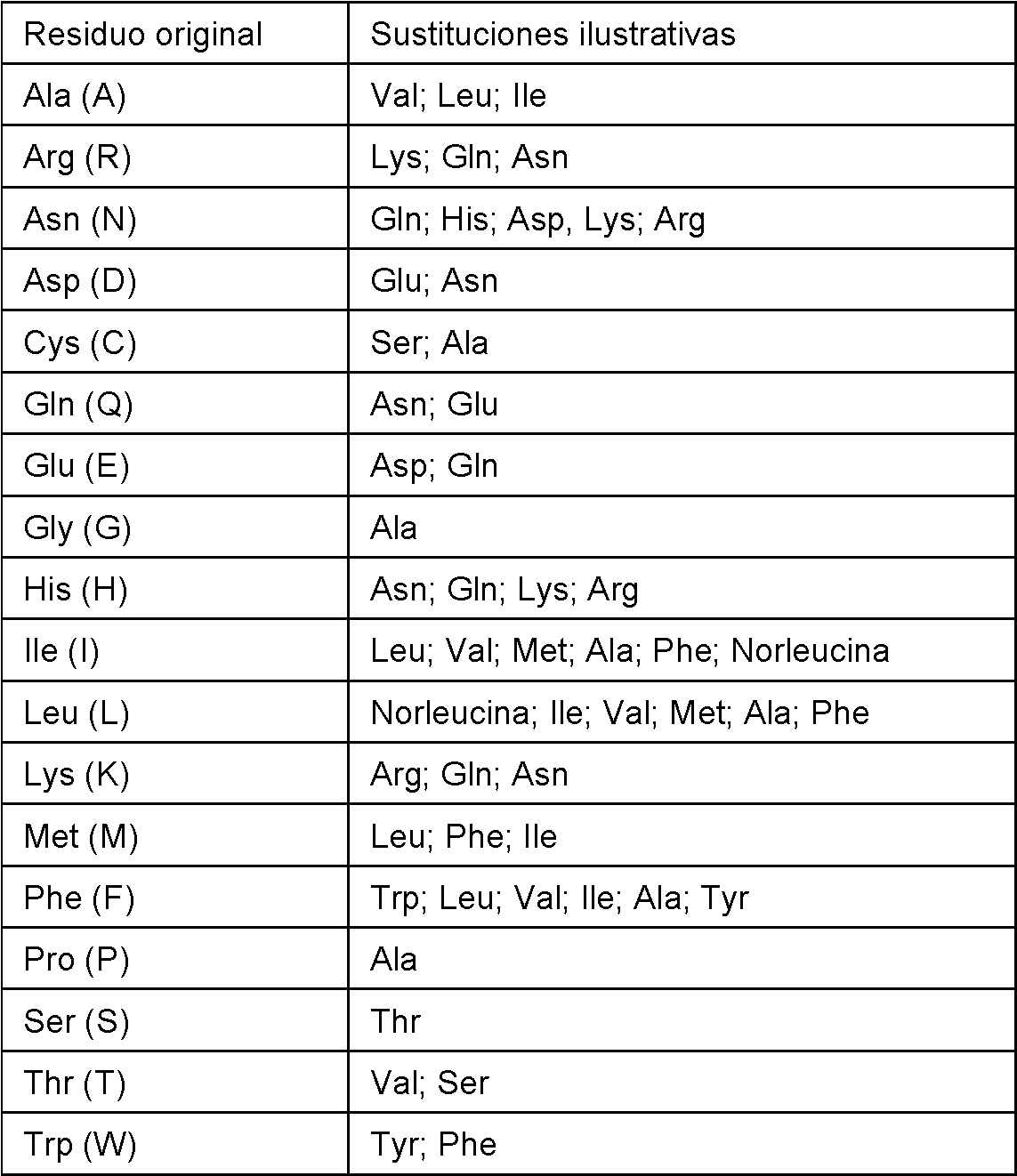
- 238000006467 substitution reaction Methods 0.000 description 30
- 201000010099 disease Diseases 0.000 description 28
- 241000699800 Cricetinae Species 0.000 description 27
- 230000000670 limiting effect Effects 0.000 description 27
- 101710093458 ICOS ligand Proteins 0.000 description 25
- 210000001519 tissue Anatomy 0.000 description 25
- 102000005962 receptors Human genes 0.000 description 23
- 108020003175 receptors Proteins 0.000 description 23
- 230000004481 post-translational protein modification Effects 0.000 description 22
- 239000008194 pharmaceutical composition Substances 0.000 description 21
- 241000282693 Cercopithecidae Species 0.000 description 20
- 239000012634 fragment Substances 0.000 description 20
- 229940127089 cytotoxic agent Drugs 0.000 description 18
- 102000006639 indoleamine 2,3-dioxygenase Human genes 0.000 description 17
- 108020004201 indoleamine 2,3-dioxygenase Proteins 0.000 description 17
- 108020004999 messenger RNA Proteins 0.000 description 17
- 230000001225 therapeutic effect Effects 0.000 description 17
- 102000004127 Cytokines Human genes 0.000 description 16
- 108090000695 Cytokines Proteins 0.000 description 16
- 238000001727 in vivo Methods 0.000 description 16
- 210000003819 peripheral blood mononuclear cell Anatomy 0.000 description 16
- 210000004881 tumor cell Anatomy 0.000 description 16
- 108060003951 Immunoglobulin Proteins 0.000 description 15
- 239000003795 chemical substances by application Substances 0.000 description 15
- 102000018358 immunoglobulin Human genes 0.000 description 15
- 230000004614 tumor growth Effects 0.000 description 15
- 241001465754 Metazoa Species 0.000 description 14
- 210000004671 cell-free system Anatomy 0.000 description 14
- 231100000599 cytotoxic agent Toxicity 0.000 description 14
- 208000020816 lung neoplasm Diseases 0.000 description 14
- 239000012528 membrane Substances 0.000 description 14
- 241000894007 species Species 0.000 description 14
- 102100025279 C-X-C motif chemokine 11 Human genes 0.000 description 13
- 101000858060 Homo sapiens C-X-C motif chemokine 11 Proteins 0.000 description 13
- 239000002254 cytotoxic agent Substances 0.000 description 13
- 238000003018 immunoassay Methods 0.000 description 13
- 238000004519 manufacturing process Methods 0.000 description 13
- 239000000126 substance Substances 0.000 description 13
- 108010087819 Fc receptors Proteins 0.000 description 12
- 102000009109 Fc receptors Human genes 0.000 description 12
- 230000001965 increasing effect Effects 0.000 description 12
- 239000002773 nucleotide Substances 0.000 description 12
- 125000003729 nucleotide group Chemical group 0.000 description 12
- 229960002621 pembrolizumab Drugs 0.000 description 12
- 206010006187 Breast cancer Diseases 0.000 description 11
- 208000026310 Breast neoplasm Diseases 0.000 description 11
- 229940045513 CTLA4 antagonist Drugs 0.000 description 11
- 101001042104 Homo sapiens Inducible T-cell costimulator Proteins 0.000 description 11
- 241000700159 Rattus Species 0.000 description 11
- 238000013459 approach Methods 0.000 description 11
- 239000000872 buffer Substances 0.000 description 11
- 238000002474 experimental method Methods 0.000 description 11
- 238000000684 flow cytometry Methods 0.000 description 11
- 238000000338 in vitro Methods 0.000 description 11
- 230000006698 induction Effects 0.000 description 11
- 208000037841 lung tumor Diseases 0.000 description 11
- 102100039620 Granulocyte-macrophage colony-stimulating factor Human genes 0.000 description 10
- 101100005713 Homo sapiens CD4 gene Proteins 0.000 description 10
- 108010021625 Immunoglobulin Fragments Proteins 0.000 description 10
- 102000008394 Immunoglobulin Fragments Human genes 0.000 description 10
- 108010002350 Interleukin-2 Proteins 0.000 description 10
- 102000000588 Interleukin-2 Human genes 0.000 description 10
- 238000004458 analytical method Methods 0.000 description 10
- 238000012575 bio-layer interferometry Methods 0.000 description 10
- 210000004369 blood Anatomy 0.000 description 10
- 239000008280 blood Substances 0.000 description 10
- 230000003247 decreasing effect Effects 0.000 description 10
- 238000012217 deletion Methods 0.000 description 10
- 230000037430 deletion Effects 0.000 description 10
- 238000005516 engineering process Methods 0.000 description 10
- 102000043396 human ICOS Human genes 0.000 description 10
- 239000003112 inhibitor Substances 0.000 description 10
- 239000003446 ligand Substances 0.000 description 10
- 229960003301 nivolumab Drugs 0.000 description 10
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 10
- 102100039498 Cytotoxic T-lymphocyte protein 4 Human genes 0.000 description 9
- 108020004414 DNA Proteins 0.000 description 9
- 101100179075 Mus musculus Icos gene Proteins 0.000 description 9
- 230000004540 complement-dependent cytotoxicity Effects 0.000 description 9
- 238000011161 development Methods 0.000 description 9
- 230000018109 developmental process Effects 0.000 description 9
- 238000010494 dissociation reaction Methods 0.000 description 9
- 230000005593 dissociations Effects 0.000 description 9
- 210000004408 hybridoma Anatomy 0.000 description 9
- 239000013641 positive control Substances 0.000 description 9
- 230000035755 proliferation Effects 0.000 description 9
- 230000009467 reduction Effects 0.000 description 9
- 102100025248 C-X-C motif chemokine 10 Human genes 0.000 description 8
- 108010021064 CTLA-4 Antigen Proteins 0.000 description 8
- 101000858088 Homo sapiens C-X-C motif chemokine 10 Proteins 0.000 description 8
- 108010004217 Natural Cytotoxicity Triggering Receptor 1 Proteins 0.000 description 8
- 102100032870 Natural cytotoxicity triggering receptor 1 Human genes 0.000 description 8
- 108010076504 Protein Sorting Signals Proteins 0.000 description 8
- 210000003719 b-lymphocyte Anatomy 0.000 description 8
- 238000011156 evaluation Methods 0.000 description 8
- 238000009472 formulation Methods 0.000 description 8
- 230000008595 infiltration Effects 0.000 description 8
- 238000001764 infiltration Methods 0.000 description 8
- 239000003550 marker Substances 0.000 description 8
- 230000004048 modification Effects 0.000 description 8
- 238000012986 modification Methods 0.000 description 8
- 210000000822 natural killer cell Anatomy 0.000 description 8
- 238000012360 testing method Methods 0.000 description 8
- 102000017420 CD3 protein, epsilon/gamma/delta subunit Human genes 0.000 description 7
- 101150063370 Gzmb gene Proteins 0.000 description 7
- 101000746373 Homo sapiens Granulocyte-macrophage colony-stimulating factor Proteins 0.000 description 7
- 101000914514 Homo sapiens T-cell-specific surface glycoprotein CD28 Proteins 0.000 description 7
- 101100179076 Rattus norvegicus Icos gene Proteins 0.000 description 7
- 102100027213 T-cell-specific surface glycoprotein CD28 Human genes 0.000 description 7
- 238000001514 detection method Methods 0.000 description 7
- FOCAHLGSDWHSAH-UHFFFAOYSA-N difluoromethanethione Chemical compound FC(F)=S FOCAHLGSDWHSAH-UHFFFAOYSA-N 0.000 description 7
- 208000035475 disorder Diseases 0.000 description 7
- 229940079593 drug Drugs 0.000 description 7
- 239000003937 drug carrier Substances 0.000 description 7
- 238000003780 insertion Methods 0.000 description 7
- 230000037431 insertion Effects 0.000 description 7
- 230000003993 interaction Effects 0.000 description 7
- 210000004962 mammalian cell Anatomy 0.000 description 7
- 150000002482 oligosaccharides Chemical class 0.000 description 7
- 230000002829 reductive effect Effects 0.000 description 7
- 230000004044 response Effects 0.000 description 7
- 230000009870 specific binding Effects 0.000 description 7
- ZADWXFSZEAPBJS-SNVBAGLBSA-N (2r)-2-amino-3-(1-methylindol-3-yl)propanoic acid Chemical compound C1=CC=C2N(C)C=C(C[C@@H](N)C(O)=O)C2=C1 ZADWXFSZEAPBJS-SNVBAGLBSA-N 0.000 description 6
- 102100036170 C-X-C motif chemokine 9 Human genes 0.000 description 6
- 206010009944 Colon cancer Diseases 0.000 description 6
- SHZGCJCMOBCMKK-UHFFFAOYSA-N D-mannomethylose Natural products CC1OC(O)C(O)C(O)C1O SHZGCJCMOBCMKK-UHFFFAOYSA-N 0.000 description 6
- AOJJSUZBOXZQNB-TZSSRYMLSA-N Doxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-TZSSRYMLSA-N 0.000 description 6
- 102000004190 Enzymes Human genes 0.000 description 6
- 108090000790 Enzymes Proteins 0.000 description 6
- PNNNRSAQSRJVSB-SLPGGIOYSA-N Fucose Natural products C[C@H](O)[C@@H](O)[C@H](O)[C@H](O)C=O PNNNRSAQSRJVSB-SLPGGIOYSA-N 0.000 description 6
- 241000282412 Homo Species 0.000 description 6
- 101000947172 Homo sapiens C-X-C motif chemokine 9 Proteins 0.000 description 6
- 101001019455 Homo sapiens ICOS ligand Proteins 0.000 description 6
- 101000917858 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-A Proteins 0.000 description 6
- 101000917839 Homo sapiens Low affinity immunoglobulin gamma Fc region receptor III-B Proteins 0.000 description 6
- FADYJNXDPBKVCA-UHFFFAOYSA-N L-Phenylalanyl-L-lysin Natural products NCCCCC(C(O)=O)NC(=O)C(N)CC1=CC=CC=C1 FADYJNXDPBKVCA-UHFFFAOYSA-N 0.000 description 6
- SHZGCJCMOBCMKK-DHVFOXMCSA-N L-fucopyranose Chemical compound C[C@@H]1OC(O)[C@@H](O)[C@H](O)[C@@H]1O SHZGCJCMOBCMKK-DHVFOXMCSA-N 0.000 description 6
- 102100029185 Low affinity immunoglobulin gamma Fc region receptor III-B Human genes 0.000 description 6
- PXIPVTKHYLBLMZ-UHFFFAOYSA-N Sodium azide Chemical compound [Na+].[N-]=[N+]=[N-] PXIPVTKHYLBLMZ-UHFFFAOYSA-N 0.000 description 6
- 230000003213 activating effect Effects 0.000 description 6
- 239000002246 antineoplastic agent Substances 0.000 description 6
- 230000004071 biological effect Effects 0.000 description 6
- -1 c Xc L9 Proteins 0.000 description 6
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 6
- 239000000306 component Substances 0.000 description 6
- 229940088598 enzyme Drugs 0.000 description 6
- 230000004927 fusion Effects 0.000 description 6
- 230000036541 health Effects 0.000 description 6
- 238000010166 immunofluorescence Methods 0.000 description 6
- 210000004072 lung Anatomy 0.000 description 6
- 229920001542 oligosaccharide Polymers 0.000 description 6
- 238000003752 polymerase chain reaction Methods 0.000 description 6
- 238000012216 screening Methods 0.000 description 6
- 235000000346 sugar Nutrition 0.000 description 6
- 230000002483 superagonistic effect Effects 0.000 description 6
- 230000003827 upregulation Effects 0.000 description 6
- FTCGGKNCJZOPNB-WHFBIAKZSA-N Asn-Gly-Ser Chemical compound NC(=O)C[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O FTCGGKNCJZOPNB-WHFBIAKZSA-N 0.000 description 5
- 102100025277 C-X-C motif chemokine 13 Human genes 0.000 description 5
- 102000014914 Carrier Proteins Human genes 0.000 description 5
- 238000002965 ELISA Methods 0.000 description 5
- 201000008808 Fibrosarcoma Diseases 0.000 description 5
- 101000858064 Homo sapiens C-X-C motif chemokine 13 Proteins 0.000 description 5
- 101000738771 Homo sapiens Receptor-type tyrosine-protein phosphatase C Proteins 0.000 description 5
- DAYQSYGBCUKVKT-VOAKCMCISA-N Leu-Thr-Lys Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(O)=O DAYQSYGBCUKVKT-VOAKCMCISA-N 0.000 description 5
- 206010058467 Lung neoplasm malignant Diseases 0.000 description 5
- MQVFHOPCKNTHGT-MELADBBJSA-N Phe-Asp-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)O)NC(=O)[C@H](CC2=CC=CC=C2)N)C(=O)O MQVFHOPCKNTHGT-MELADBBJSA-N 0.000 description 5
- NAIPAPCKKRCMBL-JYJNAYRXSA-N Pro-Pro-Phe Chemical compound C([C@@H](C(=O)O)NC(=O)[C@H]1N(CCC1)C(=O)[C@H]1NCCC1)C1=CC=CC=C1 NAIPAPCKKRCMBL-JYJNAYRXSA-N 0.000 description 5
- 102100037422 Receptor-type tyrosine-protein phosphatase C Human genes 0.000 description 5
- 241000283984 Rodentia Species 0.000 description 5
- 238000007792 addition Methods 0.000 description 5
- 239000012491 analyte Substances 0.000 description 5
- 239000005557 antagonist Substances 0.000 description 5
- 230000000875 corresponding effect Effects 0.000 description 5
- 210000004443 dendritic cell Anatomy 0.000 description 5
- 230000001419 dependent effect Effects 0.000 description 5
- 229950009791 durvalumab Drugs 0.000 description 5
- 108010082286 glycyl-seryl-alanine Proteins 0.000 description 5
- 230000012010 growth Effects 0.000 description 5
- 230000028993 immune response Effects 0.000 description 5
- 230000005764 inhibitory process Effects 0.000 description 5
- 108010034529 leucyl-lysine Proteins 0.000 description 5
- 201000005202 lung cancer Diseases 0.000 description 5
- 229960001592 paclitaxel Drugs 0.000 description 5
- 230000008823 permeabilization Effects 0.000 description 5
- 230000000069 prophylactic effect Effects 0.000 description 5
- 230000011664 signaling Effects 0.000 description 5
- 108010077037 tyrosyl-tyrosyl-phenylalanine Proteins 0.000 description 5
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 4
- NFGXHKASABOEEW-UHFFFAOYSA-N 1-methylethyl 11-methoxy-3,7,11-trimethyl-2,4-dodecadienoate Chemical compound COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C NFGXHKASABOEEW-UHFFFAOYSA-N 0.000 description 4
- XQJAFSDFQZPYCU-UWJYBYFXSA-N Ala-Asn-Tyr Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)O)N XQJAFSDFQZPYCU-UWJYBYFXSA-N 0.000 description 4
- CMSMOCZEIVJLDB-UHFFFAOYSA-N Cyclophosphamide Chemical compound ClCCN(CCCl)P1(=O)NCCCO1 CMSMOCZEIVJLDB-UHFFFAOYSA-N 0.000 description 4
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 4
- OUYCCCASQSFEME-QMMMGPOBSA-N L-tyrosine Chemical compound OC(=O)[C@@H](N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-QMMMGPOBSA-N 0.000 description 4
- KDXKERNSBIXSRK-UHFFFAOYSA-N Lysine Natural products NCCCCC(N)C(O)=O KDXKERNSBIXSRK-UHFFFAOYSA-N 0.000 description 4
- 101000686985 Mouse mammary tumor virus (strain C3H) Protein PR73 Proteins 0.000 description 4
- 241000699660 Mus musculus Species 0.000 description 4
- 210000004460 N cell Anatomy 0.000 description 4
- OVRNDRQMDRJTHS-UHFFFAOYSA-N N-acelyl-D-glucosamine Natural products CC(=O)NC1C(O)OC(CO)C(O)C1O OVRNDRQMDRJTHS-UHFFFAOYSA-N 0.000 description 4
- MBLBDJOUHNCFQT-LXGUWJNJSA-N N-acetylglucosamine Natural products CC(=O)N[C@@H](C=O)[C@@H](O)[C@H](O)[C@H](O)CO MBLBDJOUHNCFQT-LXGUWJNJSA-N 0.000 description 4
- 241000283973 Oryctolagus cuniculus Species 0.000 description 4
- RVRRHFPCEOVRKQ-KKUMJFAQSA-N Phe-His-Asn Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CN=CN2)C(=O)N[C@@H](CC(=O)N)C(=O)O)N RVRRHFPCEOVRKQ-KKUMJFAQSA-N 0.000 description 4
- 241000288906 Primates Species 0.000 description 4
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 4
- 108010003723 Single-Domain Antibodies Proteins 0.000 description 4
- 230000006052 T cell proliferation Effects 0.000 description 4
- 230000004075 alteration Effects 0.000 description 4
- 230000000890 antigenic effect Effects 0.000 description 4
- 230000009286 beneficial effect Effects 0.000 description 4
- 108091008324 binding proteins Proteins 0.000 description 4
- 230000015572 biosynthetic process Effects 0.000 description 4
- 150000001720 carbohydrates Chemical class 0.000 description 4
- 239000003153 chemical reaction reagent Substances 0.000 description 4
- 150000001875 compounds Chemical class 0.000 description 4
- 230000021615 conjugation Effects 0.000 description 4
- 230000000139 costimulatory effect Effects 0.000 description 4
- 229960004397 cyclophosphamide Drugs 0.000 description 4
- 230000002255 enzymatic effect Effects 0.000 description 4
- 210000003527 eukaryotic cell Anatomy 0.000 description 4
- 230000003053 immunization Effects 0.000 description 4
- 238000002649 immunization Methods 0.000 description 4
- 230000006872 improvement Effects 0.000 description 4
- 238000011534 incubation Methods 0.000 description 4
- 230000002401 inhibitory effect Effects 0.000 description 4
- 239000007924 injection Substances 0.000 description 4
- 238000002347 injection Methods 0.000 description 4
- 229940043355 kinase inhibitor Drugs 0.000 description 4
- 238000002794 lymphocyte assay Methods 0.000 description 4
- 230000003287 optical effect Effects 0.000 description 4
- 230000001575 pathological effect Effects 0.000 description 4
- 239000003757 phosphotransferase inhibitor Substances 0.000 description 4
- 229920000642 polymer Polymers 0.000 description 4
- 239000002243 precursor Substances 0.000 description 4
- 230000002265 prevention Effects 0.000 description 4
- 239000000047 product Substances 0.000 description 4
- 230000028327 secretion Effects 0.000 description 4
- 210000002966 serum Anatomy 0.000 description 4
- 150000008163 sugars Chemical class 0.000 description 4
- 231100000617 superantigen Toxicity 0.000 description 4
- 239000006228 supernatant Substances 0.000 description 4
- 238000002198 surface plasmon resonance spectroscopy Methods 0.000 description 4
- 208000024891 symptom Diseases 0.000 description 4
- RCINICONZNJXQF-MZXODVADSA-N taxol Chemical compound O([C@@H]1[C@@]2(C[C@@H](C(C)=C(C2(C)C)[C@H](C([C@]2(C)[C@@H](O)C[C@H]3OC[C@]3([C@H]21)OC(C)=O)=O)OC(=O)C)OC(=O)[C@H](O)[C@@H](NC(=O)C=1C=CC=CC=1)C=1C=CC=CC=1)O)C(=O)C1=CC=CC=C1 RCINICONZNJXQF-MZXODVADSA-N 0.000 description 4
- 108010061238 threonyl-glycine Proteins 0.000 description 4
- 238000011277 treatment modality Methods 0.000 description 4
- 210000003171 tumor-infiltrating lymphocyte Anatomy 0.000 description 4
- 239000003981 vehicle Substances 0.000 description 4
- STQGQHZAVUOBTE-UHFFFAOYSA-N 7-Cyan-hept-2t-en-4,6-diinsaeure Natural products C1=2C(O)=C3C(=O)C=4C(OC)=CC=CC=4C(=O)C3=C(O)C=2CC(O)(C(C)=O)CC1OC1CC(N)C(O)C(C)O1 STQGQHZAVUOBTE-UHFFFAOYSA-N 0.000 description 3
- VCSABYLVNWQYQE-UHFFFAOYSA-N Ala-Lys-Lys Natural products NCCCCC(NC(=O)C(N)C)C(=O)NC(CCCCN)C(O)=O VCSABYLVNWQYQE-UHFFFAOYSA-N 0.000 description 3
- OPEPUCYIGFEGSW-WDSKDSINSA-N Asn-Gly-Glu Chemical compound [H]N[C@@H](CC(N)=O)C(=O)NCC(=O)N[C@@H](CCC(O)=O)C(O)=O OPEPUCYIGFEGSW-WDSKDSINSA-N 0.000 description 3
- DJIMLSXHXKWADV-CIUDSAMLSA-N Asn-Leu-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](N)CC(N)=O DJIMLSXHXKWADV-CIUDSAMLSA-N 0.000 description 3
- NCXTYSVDWLAQGZ-ZKWXMUAHSA-N Asn-Ser-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC(N)=O NCXTYSVDWLAQGZ-ZKWXMUAHSA-N 0.000 description 3
- 102100029822 B- and T-lymphocyte attenuator Human genes 0.000 description 3
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 3
- 108010019670 Chimeric Antigen Receptors Proteins 0.000 description 3
- 208000001333 Colorectal Neoplasms Diseases 0.000 description 3
- 239000004971 Cross linker Substances 0.000 description 3
- YZKOXEJTLWZOQL-GUBZILKMSA-N Cys-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CS)N YZKOXEJTLWZOQL-GUBZILKMSA-N 0.000 description 3
- MSWBLPLBSLQVME-XIRDDKMYSA-N Cys-Trp-Leu Chemical compound C1=CC=C2C(C[C@@H](C(=O)N[C@@H](CC(C)C)C(O)=O)NC(=O)[C@@H](N)CS)=CNC2=C1 MSWBLPLBSLQVME-XIRDDKMYSA-N 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 102100027581 Forkhead box protein P3 Human genes 0.000 description 3
- HHQCBFGKQDMWSP-GUBZILKMSA-N Gln-Leu-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CCC(=O)N)N HHQCBFGKQDMWSP-GUBZILKMSA-N 0.000 description 3
- XEKAJTCACGEBOK-KKUMJFAQSA-N Glu-Met-Phe Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CCC(=O)O)N XEKAJTCACGEBOK-KKUMJFAQSA-N 0.000 description 3
- GVVKYKCOFMMTKZ-WHFBIAKZSA-N Gly-Cys-Ala Chemical compound OC(=O)[C@H](C)NC(=O)[C@H](CS)NC(=O)CN GVVKYKCOFMMTKZ-WHFBIAKZSA-N 0.000 description 3
- OLPPXYMMIARYAL-QMMMGPOBSA-N Gly-Gly-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)CNC(=O)CN OLPPXYMMIARYAL-QMMMGPOBSA-N 0.000 description 3
- RIYIFUFFFBIOEU-KBPBESRZSA-N Gly-Tyr-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)CN)CC1=CC=C(O)C=C1 RIYIFUFFFBIOEU-KBPBESRZSA-N 0.000 description 3
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 3
- 108010017213 Granulocyte-Macrophage Colony-Stimulating Factor Proteins 0.000 description 3
- LMMPTUVWHCFTOT-GARJFASQSA-N His-Asp-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)O)NC(=O)[C@H](CC2=CN=CN2)N)C(=O)O LMMPTUVWHCFTOT-GARJFASQSA-N 0.000 description 3
- 101000864344 Homo sapiens B- and T-lymphocyte attenuator Proteins 0.000 description 3
- 101000861452 Homo sapiens Forkhead box protein P3 Proteins 0.000 description 3
- 101001057504 Homo sapiens Interferon-stimulated gene 20 kDa protein Proteins 0.000 description 3
- 101001055144 Homo sapiens Interleukin-2 receptor subunit alpha Proteins 0.000 description 3
- 101150106931 IFNG gene Proteins 0.000 description 3
- 102100027268 Interferon-stimulated gene 20 kDa protein Human genes 0.000 description 3
- RRSLQOLASISYTB-CIUDSAMLSA-N Leu-Cys-Asp Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CS)C(=O)N[C@@H](CC(O)=O)C(O)=O RRSLQOLASISYTB-CIUDSAMLSA-N 0.000 description 3
- NHHKSOGJYNQENP-SRVKXCTJSA-N Leu-Cys-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CCCCN)C(=O)O)N NHHKSOGJYNQENP-SRVKXCTJSA-N 0.000 description 3
- KZZCOWMDDXDKSS-CIUDSAMLSA-N Leu-Ser-Asn Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(O)=O KZZCOWMDDXDKSS-CIUDSAMLSA-N 0.000 description 3
- VDIARPPNADFEAV-WEDXCCLWSA-N Leu-Thr-Gly Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)NCC(O)=O VDIARPPNADFEAV-WEDXCCLWSA-N 0.000 description 3
- JPYPRVHMKRFTAT-KKUMJFAQSA-N Lys-Phe-Cys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CCCCN)N JPYPRVHMKRFTAT-KKUMJFAQSA-N 0.000 description 3
- IOQWIOPSKJOEKI-SRVKXCTJSA-N Lys-Ser-Leu Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O IOQWIOPSKJOEKI-SRVKXCTJSA-N 0.000 description 3
- SQRLLZAQNOQCEG-KKUMJFAQSA-N Lys-Tyr-Ser Chemical compound NCCCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CO)C(O)=O)CC1=CC=C(O)C=C1 SQRLLZAQNOQCEG-KKUMJFAQSA-N 0.000 description 3
- RIPJMCFGQHGHNP-RHYQMDGZSA-N Lys-Val-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](C(C)C)NC(=O)[C@H](CCCCN)N)O RIPJMCFGQHGHNP-RHYQMDGZSA-N 0.000 description 3
- FBKMWOJEPMPVTQ-UHFFFAOYSA-N N'-(3-bromo-4-fluorophenyl)-N-hydroxy-4-[2-(sulfamoylamino)ethylamino]-1,2,5-oxadiazole-3-carboximidamide Chemical compound NS(=O)(=O)NCCNC1=NON=C1C(=NO)NC1=CC=C(F)C(Br)=C1 FBKMWOJEPMPVTQ-UHFFFAOYSA-N 0.000 description 3
- OVRNDRQMDRJTHS-RTRLPJTCSA-N N-acetyl-D-glucosamine Chemical compound CC(=O)N[C@H]1C(O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-RTRLPJTCSA-N 0.000 description 3
- YGACXVRLDHEXKY-WXRXAMBDSA-N O[C@H](C[C@H]1c2c(cccc2F)-c2cncn12)[C@H]1CC[C@H](O)CC1 Chemical compound O[C@H](C[C@H]1c2c(cccc2F)-c2cncn12)[C@H]1CC[C@H](O)CC1 YGACXVRLDHEXKY-WXRXAMBDSA-N 0.000 description 3
- 229930012538 Paclitaxel Natural products 0.000 description 3
- 102100033810 RAC-alpha serine/threonine-protein kinase Human genes 0.000 description 3
- BPMRXBZYPGYPJN-WHFBIAKZSA-N Ser-Gly-Asn Chemical compound [H]N[C@@H](CO)C(=O)NCC(=O)N[C@@H](CC(N)=O)C(O)=O BPMRXBZYPGYPJN-WHFBIAKZSA-N 0.000 description 3
- RRVFEDGUXSYWOW-BZSNNMDCSA-N Ser-Phe-Phe Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O RRVFEDGUXSYWOW-BZSNNMDCSA-N 0.000 description 3
- JLNMFGCJODTXDH-WEDXCCLWSA-N Thr-Lys-Gly Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O JLNMFGCJODTXDH-WEDXCCLWSA-N 0.000 description 3
- MGJLBZFUXUGMML-VOAKCMCISA-N Thr-Lys-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)O)N)O MGJLBZFUXUGMML-VOAKCMCISA-N 0.000 description 3
- 108091023040 Transcription factor Proteins 0.000 description 3
- 102000040945 Transcription factor Human genes 0.000 description 3
- AYHSJESDFKREAR-KKUMJFAQSA-N Tyr-Asn-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 AYHSJESDFKREAR-KKUMJFAQSA-N 0.000 description 3
- ZRPLVTZTKPPSBT-AVGNSLFASA-N Tyr-Glu-Ser Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CO)C(O)=O ZRPLVTZTKPPSBT-AVGNSLFASA-N 0.000 description 3
- FWOVTJKVUCGVND-UFYCRDLUSA-N Tyr-Met-Phe Chemical compound CSCC[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)O)NC(=O)[C@H](CC2=CC=C(C=C2)O)N FWOVTJKVUCGVND-UFYCRDLUSA-N 0.000 description 3
- UPODKYBYUBTWSV-BZSNNMDCSA-N Tyr-Phe-Cys Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CS)C(O)=O)C1=CC=C(O)C=C1 UPODKYBYUBTWSV-BZSNNMDCSA-N 0.000 description 3
- XJPXTYLVMUZGNW-IHRRRGAJSA-N Tyr-Pro-Asp Chemical compound [H]N[C@@H](CC1=CC=C(O)C=C1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CC(O)=O)C(O)=O XJPXTYLVMUZGNW-IHRRRGAJSA-N 0.000 description 3
- TYGHOWWWMTWVKM-HJOGWXRNSA-N Tyr-Tyr-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC(O)=CC=1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 TYGHOWWWMTWVKM-HJOGWXRNSA-N 0.000 description 3
- PVPAOIGJYHVWBT-KKHAAJSZSA-N Val-Asn-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(=O)N)NC(=O)[C@H](C(C)C)N)O PVPAOIGJYHVWBT-KKHAAJSZSA-N 0.000 description 3
- 229940028652 abraxane Drugs 0.000 description 3
- 210000004102 animal cell Anatomy 0.000 description 3
- 238000010171 animal model Methods 0.000 description 3
- 230000000259 anti-tumor effect Effects 0.000 description 3
- 238000002820 assay format Methods 0.000 description 3
- 229950002916 avelumab Drugs 0.000 description 3
- 230000008901 benefit Effects 0.000 description 3
- 230000000903 blocking effect Effects 0.000 description 3
- 235000014633 carbohydrates Nutrition 0.000 description 3
- 210000004970 cd4 cell Anatomy 0.000 description 3
- 230000001413 cellular effect Effects 0.000 description 3
- 238000012512 characterization method Methods 0.000 description 3
- 238000006243 chemical reaction Methods 0.000 description 3
- 238000002648 combination therapy Methods 0.000 description 3
- 230000002596 correlated effect Effects 0.000 description 3
- 108010004073 cysteinylcysteine Proteins 0.000 description 3
- 230000001086 cytosolic effect Effects 0.000 description 3
- 229960000975 daunorubicin Drugs 0.000 description 3
- STQGQHZAVUOBTE-VGBVRHCVSA-N daunorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(C)=O)[C@H]1C[C@H](N)[C@H](O)[C@H](C)O1 STQGQHZAVUOBTE-VGBVRHCVSA-N 0.000 description 3
- 230000002950 deficient Effects 0.000 description 3
- 238000009826 distribution Methods 0.000 description 3
- 231100000673 dose–response relationship Toxicity 0.000 description 3
- 229960004679 doxorubicin Drugs 0.000 description 3
- 239000013604 expression vector Substances 0.000 description 3
- 230000033581 fucosylation Effects 0.000 description 3
- 230000013595 glycosylation Effects 0.000 description 3
- 238000006206 glycosylation reaction Methods 0.000 description 3
- 108010078326 glycyl-glycyl-valine Proteins 0.000 description 3
- 206010073071 hepatocellular carcinoma Diseases 0.000 description 3
- 210000002865 immune cell Anatomy 0.000 description 3
- 230000002163 immunogen Effects 0.000 description 3
- 229950009034 indoximod Drugs 0.000 description 3
- 238000007912 intraperitoneal administration Methods 0.000 description 3
- 210000000265 leukocyte Anatomy 0.000 description 3
- 150000002632 lipids Chemical class 0.000 description 3
- 208000014018 liver neoplasm Diseases 0.000 description 3
- 238000005259 measurement Methods 0.000 description 3
- 230000001404 mediated effect Effects 0.000 description 3
- 210000001616 monocyte Anatomy 0.000 description 3
- 230000035772 mutation Effects 0.000 description 3
- 239000013642 negative control Substances 0.000 description 3
- 210000000440 neutrophil Anatomy 0.000 description 3
- 230000036961 partial effect Effects 0.000 description 3
- 230000037361 pathway Effects 0.000 description 3
- 238000002823 phage display Methods 0.000 description 3
- 125000002924 primary amino group Chemical group [H]N([H])* 0.000 description 3
- 230000008569 process Effects 0.000 description 3
- 230000002062 proliferating effect Effects 0.000 description 3
- 238000011321 prophylaxis Methods 0.000 description 3
- 238000001959 radiotherapy Methods 0.000 description 3
- 230000010076 replication Effects 0.000 description 3
- 230000002441 reversible effect Effects 0.000 description 3
- 230000035945 sensitivity Effects 0.000 description 3
- 239000007787 solid Substances 0.000 description 3
- 239000012128 staining reagent Substances 0.000 description 3
- 230000000638 stimulation Effects 0.000 description 3
- 238000001356 surgical procedure Methods 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- 238000012546 transfer Methods 0.000 description 3
- 230000035899 viability Effects 0.000 description 3
- 229960004528 vincristine Drugs 0.000 description 3
- OGWKCGZFUXNPDA-XQKSVPLYSA-N vincristine Chemical compound C([N@]1C[C@@H](C[C@]2(C(=O)OC)C=3C(=CC4=C([C@]56[C@H]([C@@]([C@H](OC(C)=O)[C@]7(CC)C=CCN([C@H]67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)C[C@@](C1)(O)CC)CC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-XQKSVPLYSA-N 0.000 description 3
- OGWKCGZFUXNPDA-UHFFFAOYSA-N vincristine Natural products C1C(CC)(O)CC(CC2(C(=O)OC)C=3C(=CC4=C(C56C(C(C(OC(C)=O)C7(CC)C=CCN(C67)CC5)(O)C(=O)OC)N4C=O)C=3)OC)CN1CCC1=C2NC2=CC=CC=C12 OGWKCGZFUXNPDA-UHFFFAOYSA-N 0.000 description 3
- YBJHBAHKTGYVGT-ZKWXMUAHSA-N (+)-Biotin Chemical compound N1C(=O)N[C@@H]2[C@H](CCCCC(=O)O)SC[C@@H]21 YBJHBAHKTGYVGT-ZKWXMUAHSA-N 0.000 description 2
- FDKXTQMXEQVLRF-ZHACJKMWSA-N (E)-dacarbazine Chemical compound CN(C)\N=N\c1[nH]cnc1C(N)=O FDKXTQMXEQVLRF-ZHACJKMWSA-N 0.000 description 2
- IAKHMKGGTNLKSZ-INIZCTEOSA-N (S)-colchicine Chemical compound C1([C@@H](NC(C)=O)CC2)=CC(=O)C(OC)=CC=C1C1=C2C=C(OC)C(OC)=C1OC IAKHMKGGTNLKSZ-INIZCTEOSA-N 0.000 description 2
- UEJJHQNACJXSKW-UHFFFAOYSA-N 2-(2,6-dioxopiperidin-3-yl)-1H-isoindole-1,3(2H)-dione Chemical compound O=C1C2=CC=CC=C2C(=O)N1C1CCC(=O)NC1=O UEJJHQNACJXSKW-UHFFFAOYSA-N 0.000 description 2
- HZAXFHJVJLSVMW-UHFFFAOYSA-N 2-Aminoethan-1-ol Chemical compound NCCO HZAXFHJVJLSVMW-UHFFFAOYSA-N 0.000 description 2
- FPQQSJJWHUJYPU-UHFFFAOYSA-N 3-(dimethylamino)propyliminomethylidene-ethylazanium;chloride Chemical compound Cl.CCN=C=NCCCN(C)C FPQQSJJWHUJYPU-UHFFFAOYSA-N 0.000 description 2
- AOJJSUZBOXZQNB-VTZDEGQISA-N 4'-epidoxorubicin Chemical compound O([C@H]1C[C@@](O)(CC=2C(O)=C3C(=O)C=4C=CC=C(C=4C(=O)C3=C(O)C=21)OC)C(=O)CO)[C@H]1C[C@H](N)[C@@H](O)[C@H](C)O1 AOJJSUZBOXZQNB-VTZDEGQISA-N 0.000 description 2
- LZRNYBIJOSKKRJ-XVYDVKMFSA-N Ala-Asp-His Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)O)C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)N LZRNYBIJOSKKRJ-XVYDVKMFSA-N 0.000 description 2
- VCSABYLVNWQYQE-SRVKXCTJSA-N Ala-Lys-Lys Chemical compound NCCCC[C@H](NC(=O)[C@@H](N)C)C(=O)N[C@@H](CCCCN)C(O)=O VCSABYLVNWQYQE-SRVKXCTJSA-N 0.000 description 2
- XSLGWYYNOSUMRM-ZKWXMUAHSA-N Ala-Val-Asn Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CC(N)=O)C(O)=O XSLGWYYNOSUMRM-ZKWXMUAHSA-N 0.000 description 2
- 102000002260 Alkaline Phosphatase Human genes 0.000 description 2
- 108020004774 Alkaline Phosphatase Proteins 0.000 description 2
- 102100021266 Alpha-(1,6)-fucosyltransferase Human genes 0.000 description 2
- 108010032595 Antibody Binding Sites Proteins 0.000 description 2
- DTBPLQNKYCYUOM-JYJNAYRXSA-N Arg-Met-Phe Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](CCSC)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 DTBPLQNKYCYUOM-JYJNAYRXSA-N 0.000 description 2
- 239000004475 Arginine Substances 0.000 description 2
- NLCDVZJDEXIDDL-BIIVOSGPSA-N Asn-Asn-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)N)NC(=O)[C@H](CC(=O)N)N)C(=O)O NLCDVZJDEXIDDL-BIIVOSGPSA-N 0.000 description 2
- 108091008875 B cell receptors Proteins 0.000 description 2
- 241000894006 Bacteria Species 0.000 description 2
- 208000003174 Brain Neoplasms Diseases 0.000 description 2
- 102000000844 Cell Surface Receptors Human genes 0.000 description 2
- 108010001857 Cell Surface Receptors Proteins 0.000 description 2
- 108091035707 Consensus sequence Proteins 0.000 description 2
- 241000699679 Cricetulus migratorius Species 0.000 description 2
- UPJGYXRAPJWIHD-CIUDSAMLSA-N Cys-Asn-Leu Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O UPJGYXRAPJWIHD-CIUDSAMLSA-N 0.000 description 2
- WQZGKKKJIJFFOK-QTVWNMPRSA-N D-mannopyranose Chemical compound OC[C@H]1OC(O)[C@@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-QTVWNMPRSA-N 0.000 description 2
- KCXVZYZYPLLWCC-UHFFFAOYSA-N EDTA Chemical compound OC(=O)CN(CC(O)=O)CCN(CC(O)=O)CC(O)=O KCXVZYZYPLLWCC-UHFFFAOYSA-N 0.000 description 2
- HTIJFSOGRVMCQR-UHFFFAOYSA-N Epirubicin Natural products COc1cccc2C(=O)c3c(O)c4CC(O)(CC(OC5CC(N)C(=O)C(C)O5)c4c(O)c3C(=O)c12)C(=O)CO HTIJFSOGRVMCQR-UHFFFAOYSA-N 0.000 description 2
- 241000283073 Equus caballus Species 0.000 description 2
- ZHNUHDYFZUAESO-UHFFFAOYSA-N Formamide Chemical compound NC=O ZHNUHDYFZUAESO-UHFFFAOYSA-N 0.000 description 2
- 229940032072 GVAX vaccine Drugs 0.000 description 2
- LVNILKSSFHCSJZ-IHRRRGAJSA-N Gln-Gln-Phe Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCC(=O)N)N LVNILKSSFHCSJZ-IHRRRGAJSA-N 0.000 description 2
- BLOXULLYFRGYKZ-GUBZILKMSA-N Gln-Glu-Arg Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O BLOXULLYFRGYKZ-GUBZILKMSA-N 0.000 description 2
- IULKWYSYZSURJK-AVGNSLFASA-N Gln-Leu-Lys Chemical compound NC(=O)CC[C@H](N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(O)=O IULKWYSYZSURJK-AVGNSLFASA-N 0.000 description 2
- QPTNELDXWKRIFX-YFKPBYRVSA-N Gly-Gly-Gln Chemical compound NCC(=O)NCC(=O)N[C@H](C(O)=O)CCC(N)=O QPTNELDXWKRIFX-YFKPBYRVSA-N 0.000 description 2
- INLIXXRWNUKVCF-JTQLQIEISA-N Gly-Gly-Tyr Chemical compound NCC(=O)NCC(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 INLIXXRWNUKVCF-JTQLQIEISA-N 0.000 description 2
- JPAACTMBBBGAAR-HOTGVXAUSA-N Gly-Leu-Trp Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](NC(=O)CN)CC(C)C)C(O)=O)=CNC2=C1 JPAACTMBBBGAAR-HOTGVXAUSA-N 0.000 description 2
- NGRPGJGKJMUGDM-XVKPBYJWSA-N Gly-Val-Gln Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O NGRPGJGKJMUGDM-XVKPBYJWSA-N 0.000 description 2
- AFMOTCMSEBITOE-YEPSODPASA-N Gly-Val-Thr Chemical compound NCC(=O)N[C@@H](C(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O AFMOTCMSEBITOE-YEPSODPASA-N 0.000 description 2
- 241000238631 Hexapoda Species 0.000 description 2
- MJNWEIMBXKKCSF-XVYDVKMFSA-N His-Ala-Asn Chemical compound C[C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)O)NC(=O)[C@H](CC1=CN=CN1)N MJNWEIMBXKKCSF-XVYDVKMFSA-N 0.000 description 2
- PLCAEMGSYOYIPP-GUBZILKMSA-N His-Ser-Gln Chemical compound NC(=O)CC[C@@H](C(O)=O)NC(=O)[C@H](CO)NC(=O)[C@@H](N)CC1=CN=CN1 PLCAEMGSYOYIPP-GUBZILKMSA-N 0.000 description 2
- 101000819490 Homo sapiens Alpha-(1,6)-fucosyltransferase Proteins 0.000 description 2
- 101001037261 Homo sapiens Indoleamine 2,3-dioxygenase 2 Proteins 0.000 description 2
- 108010001336 Horseradish Peroxidase Proteins 0.000 description 2
- 102100026120 IgG receptor FcRn large subunit p51 Human genes 0.000 description 2
- 101710177940 IgG receptor FcRn large subunit p51 Proteins 0.000 description 2
- 108010067060 Immunoglobulin Variable Region Proteins 0.000 description 2
- 102000017727 Immunoglobulin Variable Region Human genes 0.000 description 2
- 102100040062 Indoleamine 2,3-dioxygenase 2 Human genes 0.000 description 2
- 208000008839 Kidney Neoplasms Diseases 0.000 description 2
- ODKSFYDXXFIFQN-BYPYZUCNSA-P L-argininium(2+) Chemical compound NC(=[NH2+])NCCC[C@H]([NH3+])C(O)=O ODKSFYDXXFIFQN-BYPYZUCNSA-P 0.000 description 2
- DCXYFEDJOCDNAF-REOHCLBHSA-N L-asparagine Chemical compound OC(=O)[C@@H](N)CC(N)=O DCXYFEDJOCDNAF-REOHCLBHSA-N 0.000 description 2
- CKLJMWTZIZZHCS-REOHCLBHSA-N L-aspartic acid Chemical compound OC(=O)[C@@H](N)CC(O)=O CKLJMWTZIZZHCS-REOHCLBHSA-N 0.000 description 2
- 239000005411 L01XE02 - Gefitinib Substances 0.000 description 2
- 239000005551 L01XE03 - Erlotinib Substances 0.000 description 2
- 239000002146 L01XE16 - Crizotinib Substances 0.000 description 2
- QJUWBDPGGYVRHY-YUMQZZPRSA-N Leu-Gly-Cys Chemical compound CC(C)C[C@@H](C(=O)NCC(=O)N[C@@H](CS)C(=O)O)N QJUWBDPGGYVRHY-YUMQZZPRSA-N 0.000 description 2
- LXKNSJLSGPNHSK-KKUMJFAQSA-N Leu-Leu-Lys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)O)N LXKNSJLSGPNHSK-KKUMJFAQSA-N 0.000 description 2
- YESNGRDJQWDYLH-KKUMJFAQSA-N Leu-Phe-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CS)C(=O)O)N YESNGRDJQWDYLH-KKUMJFAQSA-N 0.000 description 2
- 108060001084 Luciferase Proteins 0.000 description 2
- 239000005089 Luciferase Substances 0.000 description 2
- NCTDKZKNBDZDOL-GARJFASQSA-N Lys-Asn-Pro Chemical compound C1C[C@@H](N(C1)C(=O)[C@H](CC(=O)N)NC(=O)[C@H](CCCCN)N)C(=O)O NCTDKZKNBDZDOL-GARJFASQSA-N 0.000 description 2
- FHIAJWBDZVHLAH-YUMQZZPRSA-N Lys-Gly-Ser Chemical compound NCCCC[C@H](N)C(=O)NCC(=O)N[C@@H](CO)C(O)=O FHIAJWBDZVHLAH-YUMQZZPRSA-N 0.000 description 2
- WKUXWMWQTOYTFI-SRVKXCTJSA-N Lys-Met-Gln Chemical compound CSCC[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)O)NC(=O)[C@H](CCCCN)N WKUXWMWQTOYTFI-SRVKXCTJSA-N 0.000 description 2
- NQSFIPWBPXNJII-PMVMPFDFSA-N Lys-Phe-Trp Chemical compound C([C@H](NC(=O)[C@@H](N)CCCCN)C(=O)N[C@@H](CC=1C2=CC=CC=C2NC=1)C(O)=O)C1=CC=CC=C1 NQSFIPWBPXNJII-PMVMPFDFSA-N 0.000 description 2
- 241000282553 Macaca Species 0.000 description 2
- 241000282567 Macaca fascicularis Species 0.000 description 2
- DLAFCQWUMFMZSN-GUBZILKMSA-N Met-Arg-Ala Chemical compound CSCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](C)C(O)=O)CCCN=C(N)N DLAFCQWUMFMZSN-GUBZILKMSA-N 0.000 description 2
- RBGLBUDVQVPTEG-DCAQKATOSA-N Met-Leu-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](CCSC)N RBGLBUDVQVPTEG-DCAQKATOSA-N 0.000 description 2
- CGUYGMFQZCYJSG-DCAQKATOSA-N Met-Lys-Ser Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CO)C(O)=O CGUYGMFQZCYJSG-DCAQKATOSA-N 0.000 description 2
- 206010027476 Metastases Diseases 0.000 description 2
- ZDZOTLJHXYCWBA-VCVYQWHSSA-N N-debenzoyl-N-(tert-butoxycarbonyl)-10-deacetyltaxol Chemical compound O([C@H]1[C@H]2[C@@](C([C@H](O)C3=C(C)[C@@H](OC(=O)[C@H](O)[C@@H](NC(=O)OC(C)(C)C)C=4C=CC=CC=4)C[C@]1(O)C3(C)C)=O)(C)[C@@H](O)C[C@H]1OC[C@]12OC(=O)C)C(=O)C1=CC=CC=C1 ZDZOTLJHXYCWBA-VCVYQWHSSA-N 0.000 description 2
- 108091007491 NSP3 Papain-like protease domains Proteins 0.000 description 2
- 229940124060 PD-1 antagonist Drugs 0.000 description 2
- 102000038030 PI3Ks Human genes 0.000 description 2
- 108091007960 PI3Ks Proteins 0.000 description 2
- 229940032310 PROSTVAC vaccine Drugs 0.000 description 2
- 229930040373 Paraformaldehyde Natural products 0.000 description 2
- 206010057249 Phagocytosis Diseases 0.000 description 2
- FUVBEZJCRMHWEM-FXQIFTODSA-N Pro-Asn-Ser Chemical compound [H]N1CCC[C@H]1C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CO)C(O)=O FUVBEZJCRMHWEM-FXQIFTODSA-N 0.000 description 2
- 238000003559 RNA-seq method Methods 0.000 description 2
- 238000011529 RT qPCR Methods 0.000 description 2
- 108020004511 Recombinant DNA Proteins 0.000 description 2
- 206010038389 Renal cancer Diseases 0.000 description 2
- 241000725643 Respiratory syncytial virus Species 0.000 description 2
- 239000006146 Roswell Park Memorial Institute medium Substances 0.000 description 2
- 206010039491 Sarcoma Diseases 0.000 description 2
- WDXYVIIVDIDOSX-DCAQKATOSA-N Ser-Arg-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@@H](N)CO)CCCN=C(N)N WDXYVIIVDIDOSX-DCAQKATOSA-N 0.000 description 2
- OOKCGAYXSNJBGQ-ZLUOBGJFSA-N Ser-Asn-Asn Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](CC(N)=O)C(O)=O OOKCGAYXSNJBGQ-ZLUOBGJFSA-N 0.000 description 2
- KMWFXJCGRXBQAC-CIUDSAMLSA-N Ser-Cys-Lys Chemical compound C(CCN)C[C@@H](C(=O)O)NC(=O)[C@H](CS)NC(=O)[C@H](CO)N KMWFXJCGRXBQAC-CIUDSAMLSA-N 0.000 description 2
- OJPHFSOMBZKQKQ-GUBZILKMSA-N Ser-Gln-Leu Chemical compound CC(C)C[C@@H](C(O)=O)NC(=O)[C@H](CCC(N)=O)NC(=O)[C@@H](N)CO OJPHFSOMBZKQKQ-GUBZILKMSA-N 0.000 description 2
- VGQVAVQWKJLIRM-FXQIFTODSA-N Ser-Ser-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(O)=O VGQVAVQWKJLIRM-FXQIFTODSA-N 0.000 description 2
- JGUWRQWULDWNCM-FXQIFTODSA-N Ser-Val-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O JGUWRQWULDWNCM-FXQIFTODSA-N 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- 229940126547 T-cell immunoglobulin mucin-3 Drugs 0.000 description 2
- 210000000662 T-lymphocyte subset Anatomy 0.000 description 2
- BPEGJWRSRHCHSN-UHFFFAOYSA-N Temozolomide Chemical compound O=C1N(C)N=NC2=C(C(N)=O)N=CN21 BPEGJWRSRHCHSN-UHFFFAOYSA-N 0.000 description 2
- 210000000447 Th1 cell Anatomy 0.000 description 2
- 210000004241 Th2 cell Anatomy 0.000 description 2
- JXGUUJMPCRXMSO-HJOGWXRNSA-N Tyr-Phe-Phe Chemical compound C([C@H](N)C(=O)N[C@@H](CC=1C=CC=CC=1)C(=O)N[C@@H](CC=1C=CC=CC=1)C(O)=O)C1=CC=C(O)C=C1 JXGUUJMPCRXMSO-HJOGWXRNSA-N 0.000 description 2
- QKXAEWMHAAVVGS-KKUMJFAQSA-N Tyr-Pro-Glu Chemical compound N[C@@H](Cc1ccc(O)cc1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(O)=O)C(O)=O QKXAEWMHAAVVGS-KKUMJFAQSA-N 0.000 description 2
- SYSWVVCYSXBVJG-RHYQMDGZSA-N Val-Leu-Thr Chemical compound C[C@H]([C@@H](C(=O)O)NC(=O)[C@H](CC(C)C)NC(=O)[C@H](C(C)C)N)O SYSWVVCYSXBVJG-RHYQMDGZSA-N 0.000 description 2
- ZHWZDZFWBXWPDW-GUBZILKMSA-N Val-Val-Cys Chemical compound CC(C)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CS)C(O)=O ZHWZDZFWBXWPDW-GUBZILKMSA-N 0.000 description 2
- 238000012452 Xenomouse strains Methods 0.000 description 2
- RJURFGZVJUQBHK-UHFFFAOYSA-N actinomycin D Natural products CC1OC(=O)C(C(C)C)N(C)C(=O)CN(C)C(=O)C2CCCN2C(=O)C(C(C)C)NC(=O)C1NC(=O)C1=C(N)C(=O)C(C)=C2OC(C(C)=CC=C3C(=O)NC4C(=O)NC(C(N5CCCC5C(=O)N(C)CC(=O)N(C)C(C(C)C)C(=O)OC4C)=O)C(C)C)=C3N=C21 RJURFGZVJUQBHK-UHFFFAOYSA-N 0.000 description 2
- 229960001686 afatinib Drugs 0.000 description 2
- ULXXDDBFHOBEHA-CWDCEQMOSA-N afatinib Chemical compound N1=CN=C2C=C(O[C@@H]3COCC3)C(NC(=O)/C=C/CN(C)C)=CC2=C1NC1=CC=C(F)C(Cl)=C1 ULXXDDBFHOBEHA-CWDCEQMOSA-N 0.000 description 2
- 238000005349 anion exchange Methods 0.000 description 2
- 230000006023 anti-tumor response Effects 0.000 description 2
- 210000000612 antigen-presenting cell Anatomy 0.000 description 2
- 229940127079 antineoplastic immunimodulatory agent Drugs 0.000 description 2
- ODKSFYDXXFIFQN-UHFFFAOYSA-N arginine Natural products OC(=O)C(N)CCCNC(N)=N ODKSFYDXXFIFQN-UHFFFAOYSA-N 0.000 description 2
- 229960003852 atezolizumab Drugs 0.000 description 2
- 230000003115 biocidal effect Effects 0.000 description 2
- 238000007622 bioinformatic analysis Methods 0.000 description 2
- 125000004057 biotinyl group Chemical group [H]N1C(=O)N([H])[C@]2([H])[C@@]([H])(SC([H])([H])[C@]12[H])C([H])([H])C([H])([H])C([H])([H])C([H])([H])C(*)=O 0.000 description 2
- 229960004562 carboplatin Drugs 0.000 description 2
- 190000008236 carboplatin Chemical compound 0.000 description 2
- 230000003197 catalytic effect Effects 0.000 description 2
- 238000005341 cation exchange Methods 0.000 description 2
- 230000020411 cell activation Effects 0.000 description 2
- 238000004113 cell culture Methods 0.000 description 2
- 239000002771 cell marker Substances 0.000 description 2
- 238000002659 cell therapy Methods 0.000 description 2
- 230000008859 change Effects 0.000 description 2
- 229960004316 cisplatin Drugs 0.000 description 2
- DQLATGHUWYMOKM-UHFFFAOYSA-L cisplatin Chemical compound N[Pt](N)(Cl)Cl DQLATGHUWYMOKM-UHFFFAOYSA-L 0.000 description 2
- 208000029742 colonic neoplasm Diseases 0.000 description 2
- 230000024203 complement activation Effects 0.000 description 2
- 230000000295 complement effect Effects 0.000 description 2
- 238000004590 computer program Methods 0.000 description 2
- 230000001268 conjugating effect Effects 0.000 description 2
- 230000004940 costimulation Effects 0.000 description 2
- 230000008878 coupling Effects 0.000 description 2
- 238000010168 coupling process Methods 0.000 description 2
- 238000005859 coupling reaction Methods 0.000 description 2
- 229960005061 crizotinib Drugs 0.000 description 2
- KTEIFNKAUNYNJU-GFCCVEGCSA-N crizotinib Chemical compound O([C@H](C)C=1C(=C(F)C=CC=1Cl)Cl)C(C(=NC=1)N)=CC=1C(=C1)C=NN1C1CCNCC1 KTEIFNKAUNYNJU-GFCCVEGCSA-N 0.000 description 2
- 230000009260 cross reactivity Effects 0.000 description 2
- 231100000433 cytotoxic Toxicity 0.000 description 2
- 230000001472 cytotoxic effect Effects 0.000 description 2
- 229960002465 dabrafenib Drugs 0.000 description 2
- BFSMGDJOXZAERB-UHFFFAOYSA-N dabrafenib Chemical compound S1C(C(C)(C)C)=NC(C=2C(=C(NS(=O)(=O)C=3C(=CC=CC=3F)F)C=CC=2)F)=C1C1=CC=NC(N)=N1 BFSMGDJOXZAERB-UHFFFAOYSA-N 0.000 description 2
- 229960003901 dacarbazine Drugs 0.000 description 2
- 230000034994 death Effects 0.000 description 2
- 238000013461 design Methods 0.000 description 2
- 238000003745 diagnosis Methods 0.000 description 2
- 239000003085 diluting agent Substances 0.000 description 2
- 229960003668 docetaxel Drugs 0.000 description 2
- 239000002552 dosage form Substances 0.000 description 2
- 230000003828 downregulation Effects 0.000 description 2
- 229960001904 epirubicin Drugs 0.000 description 2
- 229960001433 erlotinib Drugs 0.000 description 2
- AAKJLRGGTJKAMG-UHFFFAOYSA-N erlotinib Chemical compound C=12C=C(OCCOC)C(OCCOC)=CC2=NC=NC=1NC1=CC=CC(C#C)=C1 AAKJLRGGTJKAMG-UHFFFAOYSA-N 0.000 description 2
- 239000012091 fetal bovine serum Substances 0.000 description 2
- 239000012894 fetal calf serum Substances 0.000 description 2
- GNBHRKFJIUUOQI-UHFFFAOYSA-N fluorescein Chemical compound O1C(=O)C2=CC=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 GNBHRKFJIUUOQI-UHFFFAOYSA-N 0.000 description 2
- MHMNJMPURVTYEJ-UHFFFAOYSA-N fluorescein-5-isothiocyanate Chemical compound O1C(=O)C2=CC(N=C=S)=CC=C2C21C1=CC=C(O)C=C1OC1=CC(O)=CC=C21 MHMNJMPURVTYEJ-UHFFFAOYSA-N 0.000 description 2
- 238000001943 fluorescence-activated cell sorting Methods 0.000 description 2
- 230000002538 fungal effect Effects 0.000 description 2
- 229960002584 gefitinib Drugs 0.000 description 2
- XGALLCVXEZPNRQ-UHFFFAOYSA-N gefitinib Chemical compound C=12C=C(OCCCN3CCOCC3)C(OC)=CC2=NC=NC=1NC1=CC=C(F)C(Cl)=C1 XGALLCVXEZPNRQ-UHFFFAOYSA-N 0.000 description 2
- SDUQYLNIPVEERB-QPPQHZFASA-N gemcitabine Chemical compound O=C1N=C(N)C=CN1[C@H]1C(F)(F)[C@H](O)[C@@H](CO)O1 SDUQYLNIPVEERB-QPPQHZFASA-N 0.000 description 2
- 229960005277 gemcitabine Drugs 0.000 description 2
- 208000005017 glioblastoma Diseases 0.000 description 2
- RWSXRVCMGQZWBV-WDSKDSINSA-N glutathione Chemical compound OC(=O)[C@@H](N)CCC(=O)N[C@@H](CS)C(=O)NCC(O)=O RWSXRVCMGQZWBV-WDSKDSINSA-N 0.000 description 2
- 108010010096 glycyl-glycyl-tyrosine Proteins 0.000 description 2
- 239000001963 growth medium Substances 0.000 description 2
- 239000000710 homodimer Substances 0.000 description 2
- 238000009396 hybridization Methods 0.000 description 2
- 230000002209 hydrophobic effect Effects 0.000 description 2
- 238000010191 image analysis Methods 0.000 description 2
- 230000001900 immune effect Effects 0.000 description 2
- 230000036039 immunity Effects 0.000 description 2
- 230000005847 immunogenicity Effects 0.000 description 2
- 230000016784 immunoglobulin production Effects 0.000 description 2
- 229940072221 immunoglobulins Drugs 0.000 description 2
- 230000002055 immunohistochemical effect Effects 0.000 description 2
- 108091008042 inhibitory receptors Proteins 0.000 description 2
- 238000001990 intravenous administration Methods 0.000 description 2
- 229960005386 ipilimumab Drugs 0.000 description 2
- 201000010982 kidney cancer Diseases 0.000 description 2
- 229960004942 lenalidomide Drugs 0.000 description 2
- GOTYRUGSSMKFNF-UHFFFAOYSA-N lenalidomide Chemical compound C1C=2C(N)=CC=CC=2C(=O)N1C1CCC(=O)NC1=O GOTYRUGSSMKFNF-UHFFFAOYSA-N 0.000 description 2
- 108010057821 leucylproline Proteins 0.000 description 2
- 239000007788 liquid Substances 0.000 description 2
- 201000007270 liver cancer Diseases 0.000 description 2
- 108010054155 lysyllysine Proteins 0.000 description 2
- 210000002540 macrophage Anatomy 0.000 description 2
- 238000013507 mapping Methods 0.000 description 2
- 239000000463 material Substances 0.000 description 2
- 229910052751 metal Inorganic materials 0.000 description 2
- 239000002184 metal Substances 0.000 description 2
- 229960001156 mitoxantrone Drugs 0.000 description 2
- KKZJGLLVHKMTCM-UHFFFAOYSA-N mitoxantrone Chemical compound O=C1C2=C(O)C=CC(O)=C2C(=O)C2=C1C(NCCNCCO)=CC=C2NCCNCCO KKZJGLLVHKMTCM-UHFFFAOYSA-N 0.000 description 2
- 238000010369 molecular cloning Methods 0.000 description 2
- 238000010172 mouse model Methods 0.000 description 2
- 231100000252 nontoxic Toxicity 0.000 description 2
- 230000003000 nontoxic effect Effects 0.000 description 2
- 210000000056 organ Anatomy 0.000 description 2
- 238000004806 packaging method and process Methods 0.000 description 2
- 229920002866 paraformaldehyde Polymers 0.000 description 2
- 229960005079 pemetrexed Drugs 0.000 description 2
- QOFFJEBXNKRSPX-ZDUSSCGKSA-N pemetrexed Chemical compound C1=N[C]2NC(N)=NC(=O)C2=C1CCC1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 QOFFJEBXNKRSPX-ZDUSSCGKSA-N 0.000 description 2
- 230000008782 phagocytosis Effects 0.000 description 2
- 230000003285 pharmacodynamic effect Effects 0.000 description 2
- 229950010773 pidilizumab Drugs 0.000 description 2
- 229960000688 pomalidomide Drugs 0.000 description 2
- UVSMNLNDYGZFPF-UHFFFAOYSA-N pomalidomide Chemical compound O=C1C=2C(N)=CC=CC=2C(=O)N1C1CCC(=O)NC1=O UVSMNLNDYGZFPF-UHFFFAOYSA-N 0.000 description 2
- 238000002360 preparation method Methods 0.000 description 2
- 238000012545 processing Methods 0.000 description 2
- 210000001236 prokaryotic cell Anatomy 0.000 description 2
- 210000002307 prostate Anatomy 0.000 description 2
- 230000005180 public health Effects 0.000 description 2
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 239000002096 quantum dot Substances 0.000 description 2
- 238000011160 research Methods 0.000 description 2
- PYWVYCXTNDRMGF-UHFFFAOYSA-N rhodamine B Chemical compound [Cl-].C=12C=CC(=[N+](CC)CC)C=C2OC2=CC(N(CC)CC)=CC=C2C=1C1=CC=CC=C1C(O)=O PYWVYCXTNDRMGF-UHFFFAOYSA-N 0.000 description 2
- 229950011529 rilimogene galvacirepvec Drugs 0.000 description 2
- 229950000431 rilimogene glafolivec Drugs 0.000 description 2
- 238000007423 screening assay Methods 0.000 description 2
- 238000013207 serial dilution Methods 0.000 description 2
- 230000009450 sialylation Effects 0.000 description 2
- 229960000714 sipuleucel-t Drugs 0.000 description 2
- 239000011780 sodium chloride Substances 0.000 description 2
- 239000000243 solution Substances 0.000 description 2
- 239000002904 solvent Substances 0.000 description 2
- UCSJYZPVAKXKNQ-HZYVHMACSA-N streptomycin Chemical compound CN[C@H]1[C@H](O)[C@@H](O)[C@H](CO)O[C@H]1O[C@@H]1[C@](C=O)(O)[C@H](C)O[C@H]1O[C@@H]1[C@@H](NC(N)=N)[C@H](O)[C@@H](NC(N)=N)[C@H](O)[C@H]1O UCSJYZPVAKXKNQ-HZYVHMACSA-N 0.000 description 2
- 239000005720 sucrose Substances 0.000 description 2
- 230000002459 sustained effect Effects 0.000 description 2
- 230000008685 targeting Effects 0.000 description 2
- 229960004964 temozolomide Drugs 0.000 description 2
- 229960003433 thalidomide Drugs 0.000 description 2
- 231100000331 toxic Toxicity 0.000 description 2
- 230000002588 toxic effect Effects 0.000 description 2
- 239000003053 toxin Substances 0.000 description 2
- 231100000765 toxin Toxicity 0.000 description 2
- 108700012359 toxins Proteins 0.000 description 2
- 229960004066 trametinib Drugs 0.000 description 2
- LIRYPHYGHXZJBZ-UHFFFAOYSA-N trametinib Chemical compound CC(=O)NC1=CC=CC(N2C(N(C3CC3)C(=O)C3=C(NC=4C(=CC(I)=CC=4)F)N(C)C(=O)C(C)=C32)=O)=C1 LIRYPHYGHXZJBZ-UHFFFAOYSA-N 0.000 description 2
- 230000009261 transgenic effect Effects 0.000 description 2
- 238000011830 transgenic mouse model Methods 0.000 description 2
- 108010044292 tryptophyltyrosine Proteins 0.000 description 2
- OUYCCCASQSFEME-UHFFFAOYSA-N tyrosine Natural products OC(=O)C(N)CC1=CC=C(O)C=C1 OUYCCCASQSFEME-UHFFFAOYSA-N 0.000 description 2
- 229960003862 vemurafenib Drugs 0.000 description 2
- GPXBXXGIAQBQNI-UHFFFAOYSA-N vemurafenib Chemical compound CCCS(=O)(=O)NC1=CC=C(F)C(C(=O)C=2C3=CC(=CN=C3NC=2)C=2C=CC(Cl)=CC=2)=C1F GPXBXXGIAQBQNI-UHFFFAOYSA-N 0.000 description 2
- GBABOYUKABKIAF-GHYRFKGUSA-N vinorelbine Chemical compound C1N(CC=2C3=CC=CC=C3NC=22)CC(CC)=C[C@H]1C[C@]2(C(=O)OC)C1=CC([C@]23[C@H]([C@]([C@H](OC(C)=O)[C@]4(CC)C=CCN([C@H]34)CC2)(O)C(=O)OC)N2C)=C2C=C1OC GBABOYUKABKIAF-GHYRFKGUSA-N 0.000 description 2
- 229960002066 vinorelbine Drugs 0.000 description 2
- OZFAFGSSMRRTDW-UHFFFAOYSA-N (2,4-dichlorophenyl) benzenesulfonate Chemical compound ClC1=CC(Cl)=CC=C1OS(=O)(=O)C1=CC=CC=C1 OZFAFGSSMRRTDW-UHFFFAOYSA-N 0.000 description 1
- OOIBFPKQHULHSQ-UHFFFAOYSA-N (3-hydroxy-1-adamantyl) 2-methylprop-2-enoate Chemical compound C1C(C2)CC3CC2(O)CC1(OC(=O)C(=C)C)C3 OOIBFPKQHULHSQ-UHFFFAOYSA-N 0.000 description 1
- GZCWLCBFPRFLKL-UHFFFAOYSA-N 1-prop-2-ynoxypropan-2-ol Chemical compound CC(O)COCC#C GZCWLCBFPRFLKL-UHFFFAOYSA-N 0.000 description 1
- 108010058566 130-nm albumin-bound paclitaxel Proteins 0.000 description 1
- FWBHETKCLVMNFS-UHFFFAOYSA-N 4',6-Diamino-2-phenylindol Chemical compound C1=CC(C(=N)N)=CC=C1C1=CC2=CC=C(C(N)=N)C=C2N1 FWBHETKCLVMNFS-UHFFFAOYSA-N 0.000 description 1
- FWMNVWWHGCHHJJ-SKKKGAJSSA-N 4-amino-1-[(2r)-6-amino-2-[[(2r)-2-[[(2r)-2-[[(2r)-2-amino-3-phenylpropanoyl]amino]-3-phenylpropanoyl]amino]-4-methylpentanoyl]amino]hexanoyl]piperidine-4-carboxylic acid Chemical compound C([C@H](C(=O)N[C@H](CC(C)C)C(=O)N[C@H](CCCCN)C(=O)N1CCC(N)(CC1)C(O)=O)NC(=O)[C@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 FWMNVWWHGCHHJJ-SKKKGAJSSA-N 0.000 description 1
- 102000013563 Acid Phosphatase Human genes 0.000 description 1
- 108010051457 Acid Phosphatase Proteins 0.000 description 1
- 229920001817 Agar Polymers 0.000 description 1
- 230000007730 Akt signaling Effects 0.000 description 1
- IHMCQESUJVZTKW-UBHSHLNASA-N Ala-Phe-Val Chemical compound CC(C)[C@@H](C(O)=O)NC(=O)[C@@H](NC(=O)[C@H](C)N)CC1=CC=CC=C1 IHMCQESUJVZTKW-UBHSHLNASA-N 0.000 description 1
- REWSWYIDQIELBE-FXQIFTODSA-N Ala-Val-Ser Chemical compound [H]N[C@@H](C)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O REWSWYIDQIELBE-FXQIFTODSA-N 0.000 description 1
- 108010012934 Albumin-Bound Paclitaxel Proteins 0.000 description 1
- 101001007348 Arachis hypogaea Galactose-binding lectin Proteins 0.000 description 1
- GOWZVQXTHUCNSQ-NHCYSSNCSA-N Arg-Glu-Val Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(=O)N[C@@H](C(C)C)C(O)=O GOWZVQXTHUCNSQ-NHCYSSNCSA-N 0.000 description 1
- JEOCWTUOMKEEMF-RHYQMDGZSA-N Arg-Leu-Thr Chemical compound [H]N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O JEOCWTUOMKEEMF-RHYQMDGZSA-N 0.000 description 1
- FXGMURPOWCKNAZ-JYJNAYRXSA-N Arg-Val-Phe Chemical compound NC(N)=NCCC[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@H](C(O)=O)CC1=CC=CC=C1 FXGMURPOWCKNAZ-JYJNAYRXSA-N 0.000 description 1
- ZYPWIUFLYMQZBS-SRVKXCTJSA-N Asn-Lys-Lys Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CCCCN)C(=O)O)NC(=O)[C@H](CC(=O)N)N ZYPWIUFLYMQZBS-SRVKXCTJSA-N 0.000 description 1
- FAEIQWHBRBWUBN-FXQIFTODSA-N Asp-Arg-Ser Chemical compound C(C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC(=O)O)N)CN=C(N)N FAEIQWHBRBWUBN-FXQIFTODSA-N 0.000 description 1
- ICZWAZVKLACMKR-CIUDSAMLSA-N Asp-His-Ser Chemical compound OC(=O)C[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CO)C(O)=O)CC1=CN=CN1 ICZWAZVKLACMKR-CIUDSAMLSA-N 0.000 description 1
- MGSVBZIBCCKGCY-ZLUOBGJFSA-N Asp-Ser-Ser Chemical compound [H]N[C@@H](CC(O)=O)C(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O MGSVBZIBCCKGCY-ZLUOBGJFSA-N 0.000 description 1
- 206010003571 Astrocytoma Diseases 0.000 description 1
- 108090001008 Avidin Proteins 0.000 description 1
- 235000014469 Bacillus subtilis Nutrition 0.000 description 1
- 102000004506 Blood Proteins Human genes 0.000 description 1
- 108010017384 Blood Proteins Proteins 0.000 description 1
- 229920002799 BoPET Polymers 0.000 description 1
- 241000283690 Bos taurus Species 0.000 description 1
- LWFDUMPGOAKHKC-UHFFFAOYSA-N C1=CC=C2C=C(C(=O)C(C(O)=C3O)=O)C3=CC2=C1 Chemical compound C1=CC=C2C=C(C(=O)C(C(O)=C3O)=O)C3=CC2=C1 LWFDUMPGOAKHKC-UHFFFAOYSA-N 0.000 description 1
- 241000282465 Canis Species 0.000 description 1
- 241000283707 Capra Species 0.000 description 1
- 201000009030 Carcinoma Diseases 0.000 description 1
- 208000010667 Carcinoma of liver and intrahepatic biliary tract Diseases 0.000 description 1
- 108010078791 Carrier Proteins Proteins 0.000 description 1
- 101150015280 Cel gene Proteins 0.000 description 1
- 206010008342 Cervix carcinoma Diseases 0.000 description 1
- 241000699802 Cricetulus griseus Species 0.000 description 1
- HIPHJNWPLMUBQQ-ACZMJKKPSA-N Cys-Cys-Gln Chemical compound SC[C@H](N)C(=O)N[C@@H](CS)C(=O)N[C@H](C(O)=O)CCC(N)=O HIPHJNWPLMUBQQ-ACZMJKKPSA-N 0.000 description 1
- GGRDJANMZPGMNS-CIUDSAMLSA-N Cys-Ser-Leu Chemical compound [H]N[C@@H](CS)C(=O)N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(O)=O GGRDJANMZPGMNS-CIUDSAMLSA-N 0.000 description 1
- 238000000116 DAPI staining Methods 0.000 description 1
- 108010092160 Dactinomycin Proteins 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- BWGNESOTFCXPMA-UHFFFAOYSA-N Dihydrogen disulfide Chemical compound SS BWGNESOTFCXPMA-UHFFFAOYSA-N 0.000 description 1
- 206010061818 Disease progression Diseases 0.000 description 1
- 206010061819 Disease recurrence Diseases 0.000 description 1
- 239000012591 Dulbecco’s Phosphate Buffered Saline Substances 0.000 description 1
- 206010058314 Dysplasia Diseases 0.000 description 1
- 241000196324 Embryophyta Species 0.000 description 1
- MBYXEBXZARTUSS-QLWBXOBMSA-N Emetamine Natural products O(C)c1c(OC)cc2c(c(C[C@@H]3[C@H](CC)CN4[C@H](c5c(cc(OC)c(OC)c5)CC4)C3)ncc2)c1 MBYXEBXZARTUSS-QLWBXOBMSA-N 0.000 description 1
- 206010014733 Endometrial cancer Diseases 0.000 description 1
- 206010014759 Endometrial neoplasm Diseases 0.000 description 1
- 241000588724 Escherichia coli Species 0.000 description 1
- 101000867232 Escherichia coli Heat-stable enterotoxin II Proteins 0.000 description 1
- 208000000461 Esophageal Neoplasms Diseases 0.000 description 1
- 241000282324 Felis Species 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 229920001917 Ficoll Polymers 0.000 description 1
- GHASVSINZRGABV-UHFFFAOYSA-N Fluorouracil Chemical compound FC1=CNC(=O)NC1=O GHASVSINZRGABV-UHFFFAOYSA-N 0.000 description 1
- 229910052688 Gadolinium Inorganic materials 0.000 description 1
- 101001066288 Gallus gallus GATA-binding factor 3 Proteins 0.000 description 1
- 206010017993 Gastrointestinal neoplasms Diseases 0.000 description 1
- 201000010915 Glioblastoma multiforme Diseases 0.000 description 1
- KVXVVDFOZNYYKZ-DCAQKATOSA-N Gln-Gln-Leu Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(C)C)C(O)=O KVXVVDFOZNYYKZ-DCAQKATOSA-N 0.000 description 1
- SMLDOQHTOAAFJQ-WDSKDSINSA-N Gln-Gly-Ser Chemical compound [H]N[C@@H](CCC(N)=O)C(=O)NCC(=O)N[C@@H](CO)C(O)=O SMLDOQHTOAAFJQ-WDSKDSINSA-N 0.000 description 1
- CGYDXNKRIMJMLV-GUBZILKMSA-N Glu-Arg-Glu Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O CGYDXNKRIMJMLV-GUBZILKMSA-N 0.000 description 1
- DAHLWSFUXOHMIA-FXQIFTODSA-N Glu-Ser-Gln Chemical compound OC(=O)CC[C@H](N)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(O)=O DAHLWSFUXOHMIA-FXQIFTODSA-N 0.000 description 1
- QEJKKJNDDDPSMU-KKUMJFAQSA-N Glu-Tyr-Met Chemical compound [H]N[C@@H](CCC(O)=O)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(=O)N[C@@H](CCSC)C(O)=O QEJKKJNDDDPSMU-KKUMJFAQSA-N 0.000 description 1
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 1
- 108010024636 Glutathione Proteins 0.000 description 1
- UXJHNZODTMHWRD-WHFBIAKZSA-N Gly-Asn-Ala Chemical compound [H]NCC(=O)N[C@@H](CC(N)=O)C(=O)N[C@@H](C)C(O)=O UXJHNZODTMHWRD-WHFBIAKZSA-N 0.000 description 1
- FMNHBTKMRFVGRO-FOHZUACHSA-N Gly-Asn-Thr Chemical compound C[C@@H](O)[C@@H](C(O)=O)NC(=O)[C@H](CC(N)=O)NC(=O)CN FMNHBTKMRFVGRO-FOHZUACHSA-N 0.000 description 1
- WCORRBXVISTKQL-WHFBIAKZSA-N Gly-Ser-Ser Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@@H](CO)C(O)=O WCORRBXVISTKQL-WHFBIAKZSA-N 0.000 description 1
- FKYQEVBRZSFAMJ-QWRGUYRKSA-N Gly-Ser-Tyr Chemical compound NCC(=O)N[C@@H](CO)C(=O)N[C@H](C(O)=O)CC1=CC=C(O)C=C1 FKYQEVBRZSFAMJ-QWRGUYRKSA-N 0.000 description 1
- TVTZEOHWHUVYCG-KYNKHSRBSA-N Gly-Thr-Thr Chemical compound [H]NCC(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H]([C@@H](C)O)C(O)=O TVTZEOHWHUVYCG-KYNKHSRBSA-N 0.000 description 1
- 108010026389 Gramicidin Proteins 0.000 description 1
- 102100028972 HLA class I histocompatibility antigen, A alpha chain Human genes 0.000 description 1
- HTTJABKRGRZYRN-UHFFFAOYSA-N Heparin Chemical compound OC1C(NC(=O)C)C(O)OC(COS(O)(=O)=O)C1OC1C(OS(O)(=O)=O)C(O)C(OC2C(C(OS(O)(=O)=O)C(OC3C(C(O)C(O)C(O3)C(O)=O)OS(O)(=O)=O)C(CO)O2)NS(O)(=O)=O)C(C(O)=O)O1 HTTJABKRGRZYRN-UHFFFAOYSA-N 0.000 description 1
- 206010073069 Hepatic cancer Diseases 0.000 description 1
- DGLAHESNTJWGDO-SRVKXCTJSA-N His-Ser-His Chemical compound C1=C(NC=N1)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N DGLAHESNTJWGDO-SRVKXCTJSA-N 0.000 description 1
- 241001272567 Hominoidea Species 0.000 description 1
- 101100166600 Homo sapiens CD28 gene Proteins 0.000 description 1
- 101000889276 Homo sapiens Cytotoxic T-lymphocyte protein 4 Proteins 0.000 description 1
- 101001103039 Homo sapiens Inactive tyrosine-protein kinase transmembrane receptor ROR1 Proteins 0.000 description 1
- 101001037256 Homo sapiens Indoleamine 2,3-dioxygenase 1 Proteins 0.000 description 1
- 101001002657 Homo sapiens Interleukin-2 Proteins 0.000 description 1
- 101000878605 Homo sapiens Low affinity immunoglobulin epsilon Fc receptor Proteins 0.000 description 1
- 101001005719 Homo sapiens Melanoma-associated antigen 3 Proteins 0.000 description 1
- 101001133056 Homo sapiens Mucin-1 Proteins 0.000 description 1
- 101000581981 Homo sapiens Neural cell adhesion molecule 1 Proteins 0.000 description 1
- 101001103036 Homo sapiens Nuclear receptor ROR-alpha Proteins 0.000 description 1
- 101001117317 Homo sapiens Programmed cell death 1 ligand 1 Proteins 0.000 description 1
- 101000713602 Homo sapiens T-box transcription factor TBX21 Proteins 0.000 description 1
- 108090000144 Human Proteins Proteins 0.000 description 1
- 238000009015 Human TaqMan MicroRNA Assay kit Methods 0.000 description 1
- 102000037982 Immune checkpoint proteins Human genes 0.000 description 1
- 108091008036 Immune checkpoint proteins Proteins 0.000 description 1
- 102000009786 Immunoglobulin Constant Regions Human genes 0.000 description 1
- 108010009817 Immunoglobulin Constant Regions Proteins 0.000 description 1
- 108010054477 Immunoglobulin Fab Fragments Proteins 0.000 description 1
- 102000001706 Immunoglobulin Fab Fragments Human genes 0.000 description 1
- 108700005091 Immunoglobulin Genes Proteins 0.000 description 1
- 102000006496 Immunoglobulin Heavy Chains Human genes 0.000 description 1
- 108010019476 Immunoglobulin Heavy Chains Proteins 0.000 description 1
- 108010079585 Immunoglobulin Subunits Proteins 0.000 description 1
- 102000012745 Immunoglobulin Subunits Human genes 0.000 description 1
- 206010062016 Immunosuppression Diseases 0.000 description 1
- 102100039615 Inactive tyrosine-protein kinase transmembrane receptor ROR1 Human genes 0.000 description 1
- 102100040061 Indoleamine 2,3-dioxygenase 1 Human genes 0.000 description 1
- 241001138401 Kluyveromyces lactis Species 0.000 description 1
- HGCNKOLVKRAVHD-UHFFFAOYSA-N L-Met-L-Phe Natural products CSCCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 HGCNKOLVKRAVHD-UHFFFAOYSA-N 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- FFEARJCKVFRZRR-BYPYZUCNSA-N L-methionine Chemical compound CSCC[C@H](N)C(O)=O FFEARJCKVFRZRR-BYPYZUCNSA-N 0.000 description 1
- FBOZXECLQNJBKD-ZDUSSCGKSA-N L-methotrexate Chemical compound C=1N=C2N=C(N)N=C(N)C2=NC=1CN(C)C1=CC=C(C(=O)N[C@@H](CCC(O)=O)C(O)=O)C=C1 FBOZXECLQNJBKD-ZDUSSCGKSA-N 0.000 description 1
- LRQKBLKVPFOOQJ-YFKPBYRVSA-N L-norleucine Chemical compound CCCC[C@H]([NH3+])C([O-])=O LRQKBLKVPFOOQJ-YFKPBYRVSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- 241000880493 Leptailurus serval Species 0.000 description 1
- GZAUZBUKDXYPEH-CIUDSAMLSA-N Leu-Cys-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CS)C(=O)O)N GZAUZBUKDXYPEH-CIUDSAMLSA-N 0.000 description 1
- KWURTLAFFDOTEQ-GUBZILKMSA-N Leu-Cys-Glu Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)N[C@@H](CCC(=O)O)C(=O)O)N KWURTLAFFDOTEQ-GUBZILKMSA-N 0.000 description 1
- XWOBNBRUDDUEEY-UWVGGRQHSA-N Leu-His Chemical compound CC(C)C[C@H](N)C(=O)N[C@H](C(O)=O)CC1=CNC=N1 XWOBNBRUDDUEEY-UWVGGRQHSA-N 0.000 description 1
- MPSBSKHOWJQHBS-IHRRRGAJSA-N Leu-His-Met Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CCSC)C(=O)O)N MPSBSKHOWJQHBS-IHRRRGAJSA-N 0.000 description 1
- IEWBEPKLKUXQBU-VOAKCMCISA-N Leu-Leu-Thr Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H]([C@@H](C)O)C(O)=O IEWBEPKLKUXQBU-VOAKCMCISA-N 0.000 description 1
- LVTJJOJKDCVZGP-QWRGUYRKSA-N Leu-Lys-Gly Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)NCC(O)=O LVTJJOJKDCVZGP-QWRGUYRKSA-N 0.000 description 1
- QNTJIDXQHWUBKC-BZSNNMDCSA-N Leu-Lys-Phe Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=CC=C1)C(O)=O QNTJIDXQHWUBKC-BZSNNMDCSA-N 0.000 description 1
- GCXGCIYIHXSKAY-ULQDDVLXSA-N Leu-Phe-Arg Chemical compound [H]N[C@@H](CC(C)C)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCNC(N)=N)C(O)=O GCXGCIYIHXSKAY-ULQDDVLXSA-N 0.000 description 1
- ICYRCNICGBJLGM-HJGDQZAQSA-N Leu-Thr-Asp Chemical compound CC(C)C[C@H](N)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@H](C(O)=O)CC(O)=O ICYRCNICGBJLGM-HJGDQZAQSA-N 0.000 description 1
- FPFOYSCDUWTZBF-IHPCNDPISA-N Leu-Trp-Leu Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H]([NH3+])CC(C)C)C(=O)N[C@@H](CC(C)C)C([O-])=O)=CNC2=C1 FPFOYSCDUWTZBF-IHPCNDPISA-N 0.000 description 1
- OZTZJMUZVAVJGY-BZSNNMDCSA-N Leu-Tyr-His Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CC=C(C=C1)O)C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)N OZTZJMUZVAVJGY-BZSNNMDCSA-N 0.000 description 1
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 1
- 102100038007 Low affinity immunoglobulin epsilon Fc receptor Human genes 0.000 description 1
- 208000007433 Lymphatic Metastasis Diseases 0.000 description 1
- 206010025323 Lymphomas Diseases 0.000 description 1
- PFZWARWVRNTPBR-IHPCNDPISA-N Lys-Leu-Trp Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CC1=CNC2=CC=CC=C21)C(=O)O)NC(=O)[C@H](CCCCN)N PFZWARWVRNTPBR-IHPCNDPISA-N 0.000 description 1
- WBSCNDJQPKSPII-KKUMJFAQSA-N Lys-Lys-Lys Chemical compound NCCCC[C@H](N)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(O)=O WBSCNDJQPKSPII-KKUMJFAQSA-N 0.000 description 1
- BXPHMHQHYHILBB-BZSNNMDCSA-N Lys-Lys-Tyr Chemical compound [H]N[C@@H](CCCCN)C(=O)N[C@@H](CCCCN)C(=O)N[C@@H](CC1=CC=C(O)C=C1)C(O)=O BXPHMHQHYHILBB-BZSNNMDCSA-N 0.000 description 1
- URBJRJKWSUFCKS-AVGNSLFASA-N Lys-Met-Arg Chemical compound CSCC[C@@H](C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)NC(=O)[C@H](CCCCN)N URBJRJKWSUFCKS-AVGNSLFASA-N 0.000 description 1
- WQDKIVRHTQYJSN-DCAQKATOSA-N Lys-Ser-Arg Chemical compound C(CCN)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCN=C(N)N)C(=O)O)N WQDKIVRHTQYJSN-DCAQKATOSA-N 0.000 description 1
- GILLQRYAWOMHED-DCAQKATOSA-N Lys-Val-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](N)CCCCN GILLQRYAWOMHED-DCAQKATOSA-N 0.000 description 1
- 239000004472 Lysine Substances 0.000 description 1
- 108700018351 Major Histocompatibility Complex Proteins 0.000 description 1
- 206010025654 Malignant melanoma of sites other than skin Diseases 0.000 description 1
- 102100025082 Melanoma-associated antigen 3 Human genes 0.000 description 1
- 241000699673 Mesocricetus auratus Species 0.000 description 1
- YRAWWKUTNBILNT-FXQIFTODSA-N Met-Ala-Ala Chemical compound CSCC[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@@H](C)C(O)=O YRAWWKUTNBILNT-FXQIFTODSA-N 0.000 description 1
- WDTLNWHPIPCMMP-AVGNSLFASA-N Met-Arg-Leu Chemical compound [H]N[C@@H](CCSC)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CC(C)C)C(O)=O WDTLNWHPIPCMMP-AVGNSLFASA-N 0.000 description 1
- VOOINLQYUZOREH-SRVKXCTJSA-N Met-Gln-Leu Chemical compound CC(C)C[C@@H](C(=O)O)NC(=O)[C@H](CCC(=O)N)NC(=O)[C@H](CCSC)N VOOINLQYUZOREH-SRVKXCTJSA-N 0.000 description 1
- IRVONVRHHJXWTK-RWMBFGLXSA-N Met-Lys-Pro Chemical compound CSCC[C@@H](C(=O)N[C@@H](CCCCN)C(=O)N1CCC[C@@H]1C(=O)O)N IRVONVRHHJXWTK-RWMBFGLXSA-N 0.000 description 1
- ZACMJPCWVSLCNS-JYJNAYRXSA-N Met-Phe-Met Chemical compound CSCC[C@H](N)C(=O)N[C@H](C(=O)N[C@@H](CCSC)C(O)=O)CC1=CC=CC=C1 ZACMJPCWVSLCNS-JYJNAYRXSA-N 0.000 description 1
- 206010027458 Metastases to lung Diseases 0.000 description 1
- 206010027459 Metastases to lymph nodes Diseases 0.000 description 1
- 108020005196 Mitochondrial DNA Proteins 0.000 description 1
- 229930192392 Mitomycin Natural products 0.000 description 1
- 108090000143 Mouse Proteins Proteins 0.000 description 1
- 102100034256 Mucin-1 Human genes 0.000 description 1
- 241001529936 Murinae Species 0.000 description 1
- 101100179071 Mus musculus Icoslg gene Proteins 0.000 description 1
- 101100407308 Mus musculus Pdcd1lg2 gene Proteins 0.000 description 1
- 239000005041 Mylar™ Substances 0.000 description 1
- NWIBSHFKIJFRCO-WUDYKRTCSA-N Mytomycin Chemical compound C1N2C(C(C(C)=C(N)C3=O)=O)=C3[C@@H](COC(N)=O)[C@@]2(OC)[C@@H]2[C@H]1N2 NWIBSHFKIJFRCO-WUDYKRTCSA-N 0.000 description 1
- NQTADLQHYWFPDB-UHFFFAOYSA-N N-Hydroxysuccinimide Chemical compound ON1C(=O)CCC1=O NQTADLQHYWFPDB-UHFFFAOYSA-N 0.000 description 1
- YBAFDPFAUTYYRW-UHFFFAOYSA-N N-L-alpha-glutamyl-L-leucine Natural products CC(C)CC(C(O)=O)NC(=O)C(N)CCC(O)=O YBAFDPFAUTYYRW-UHFFFAOYSA-N 0.000 description 1
- SITLTJHOQZFJGG-UHFFFAOYSA-N N-L-alpha-glutamyl-L-valine Natural products CC(C)C(C(O)=O)NC(=O)C(N)CCC(O)=O SITLTJHOQZFJGG-UHFFFAOYSA-N 0.000 description 1
- XZFYRXDAULDNFX-UHFFFAOYSA-N N-L-cysteinyl-L-phenylalanine Natural products SCC(N)C(=O)NC(C(O)=O)CC1=CC=CC=C1 XZFYRXDAULDNFX-UHFFFAOYSA-N 0.000 description 1
- OVRNDRQMDRJTHS-FMDGEEDCSA-N N-acetyl-beta-D-glucosamine Chemical compound CC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O OVRNDRQMDRJTHS-FMDGEEDCSA-N 0.000 description 1
- 102100027347 Neural cell adhesion molecule 1 Human genes 0.000 description 1
- 108091028043 Nucleic acid sequence Proteins 0.000 description 1
- 206010030155 Oesophageal carcinoma Diseases 0.000 description 1
- 108010038807 Oligopeptides Proteins 0.000 description 1
- 102000015636 Oligopeptides Human genes 0.000 description 1
- 238000012408 PCR amplification Methods 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 206010061902 Pancreatic neoplasm Diseases 0.000 description 1
- 108090000526 Papain Proteins 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 229930182555 Penicillin Natural products 0.000 description 1
- JGSARLDLIJGVTE-MBNYWOFBSA-N Penicillin G Chemical compound N([C@H]1[C@H]2SC([C@@H](N2C1=O)C(O)=O)(C)C)C(=O)CC1=CC=CC=C1 JGSARLDLIJGVTE-MBNYWOFBSA-N 0.000 description 1
- 102000057297 Pepsin A Human genes 0.000 description 1
- 108090000284 Pepsin A Proteins 0.000 description 1
- 241000009328 Perro Species 0.000 description 1
- 108010081690 Pertussis Toxin Proteins 0.000 description 1
- MPGJIHFJCXTVEX-KKUMJFAQSA-N Phe-Arg-Glu Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCCNC(N)=N)C(=O)N[C@@H](CCC(O)=O)C(O)=O MPGJIHFJCXTVEX-KKUMJFAQSA-N 0.000 description 1
- MMYUOSCXBJFUNV-QWRGUYRKSA-N Phe-Gly-Cys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)NCC(=O)N[C@@H](CS)C(=O)O)N MMYUOSCXBJFUNV-QWRGUYRKSA-N 0.000 description 1
- SZYBZVANEAOIPE-UBHSHLNASA-N Phe-Met-Ala Chemical compound [H]N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](C)C(O)=O SZYBZVANEAOIPE-UBHSHLNASA-N 0.000 description 1
- AXIOGMQCDYVTNY-ACRUOGEOSA-N Phe-Phe-Leu Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)C(O)=O)NC(=O)[C@@H](N)CC=1C=CC=CC=1)C1=CC=CC=C1 AXIOGMQCDYVTNY-ACRUOGEOSA-N 0.000 description 1
- JDMKQHSHKJHAHR-UHFFFAOYSA-N Phe-Phe-Leu-Tyr Natural products C=1C=C(O)C=CC=1CC(C(O)=O)NC(=O)C(CC(C)C)NC(=O)C(NC(=O)C(N)CC=1C=CC=CC=1)CC1=CC=CC=C1 JDMKQHSHKJHAHR-UHFFFAOYSA-N 0.000 description 1
- IPFXYNKCXYGSSV-KKUMJFAQSA-N Phe-Ser-Lys Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)N[C@@H](CCCCN)C(=O)O)N IPFXYNKCXYGSSV-KKUMJFAQSA-N 0.000 description 1
- APZNYJFGVAGFCF-JYJNAYRXSA-N Phe-Val-Val Chemical compound CC(C)[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)Cc1ccccc1)C(C)C)C(O)=O APZNYJFGVAGFCF-JYJNAYRXSA-N 0.000 description 1
- 108010004729 Phycoerythrin Proteins 0.000 description 1
- 208000007913 Pituitary Neoplasms Diseases 0.000 description 1
- 206010035226 Plasma cell myeloma Diseases 0.000 description 1
- 241000276498 Pollachius virens Species 0.000 description 1
- FUOGXAQMNJMBFG-WPRPVWTQSA-N Pro-Val-Gly Chemical compound OC(=O)CNC(=O)[C@H](C(C)C)NC(=O)[C@@H]1CCCN1 FUOGXAQMNJMBFG-WPRPVWTQSA-N 0.000 description 1
- 108700030875 Programmed Cell Death 1 Ligand 2 Proteins 0.000 description 1
- 102100024213 Programmed cell death 1 ligand 2 Human genes 0.000 description 1
- 206010060862 Prostate cancer Diseases 0.000 description 1
- 208000000236 Prostatic Neoplasms Diseases 0.000 description 1
- 239000004365 Protease Substances 0.000 description 1
- 102100039641 Protein MFI Human genes 0.000 description 1
- 238000002123 RNA extraction Methods 0.000 description 1
- 238000011530 RNeasy Mini Kit Methods 0.000 description 1
- 108010025216 RVF peptide Proteins 0.000 description 1
- 108010008281 Recombinant Fusion Proteins Proteins 0.000 description 1
- 102000007056 Recombinant Fusion Proteins Human genes 0.000 description 1
- AUVVAXYIELKVAI-UHFFFAOYSA-N SJ000285215 Natural products N1CCC2=CC(OC)=C(OC)C=C2C1CC1CC2C3=CC(OC)=C(OC)C=C3CCN2CC1CC AUVVAXYIELKVAI-UHFFFAOYSA-N 0.000 description 1
- 206010061934 Salivary gland cancer Diseases 0.000 description 1
- YUJLIIRMIAGMCQ-CIUDSAMLSA-N Ser-Leu-Ser Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CC(C)C)C(=O)N[C@@H](CO)C(O)=O YUJLIIRMIAGMCQ-CIUDSAMLSA-N 0.000 description 1
- WOJYIMBIKTWKJO-KKUMJFAQSA-N Ser-Phe-His Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CC2=CN=CN2)C(=O)O)NC(=O)[C@H](CO)N WOJYIMBIKTWKJO-KKUMJFAQSA-N 0.000 description 1
- FZXOPYUEQGDGMS-ACZMJKKPSA-N Ser-Ser-Gln Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCC(N)=O)C(O)=O FZXOPYUEQGDGMS-ACZMJKKPSA-N 0.000 description 1
- BDMWLJLPPUCLNV-XGEHTFHBSA-N Ser-Thr-Val Chemical compound [H]N[C@@H](CO)C(=O)N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(O)=O BDMWLJLPPUCLNV-XGEHTFHBSA-N 0.000 description 1
- SYCFMSYTIFXWAJ-DCAQKATOSA-N Ser-Val-His Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)O)NC(=O)[C@H](CO)N SYCFMSYTIFXWAJ-DCAQKATOSA-N 0.000 description 1
- 206010041067 Small cell lung cancer Diseases 0.000 description 1
- VMHLLURERBWHNL-UHFFFAOYSA-M Sodium acetate Chemical compound [Na+].CC([O-])=O VMHLLURERBWHNL-UHFFFAOYSA-M 0.000 description 1
- 208000021712 Soft tissue sarcoma Diseases 0.000 description 1
- 108010090804 Streptavidin Proteins 0.000 description 1
- 241000282898 Sus scrofa Species 0.000 description 1
- 230000005867 T cell response Effects 0.000 description 1
- 102100036840 T-box transcription factor TBX21 Human genes 0.000 description 1
- 208000024313 Testicular Neoplasms Diseases 0.000 description 1
- 206010057644 Testis cancer Diseases 0.000 description 1
- ZUXQFMVPAYGPFJ-JXUBOQSCSA-N Thr-Ala-Lys Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C)C(=O)N[C@H](C(O)=O)CCCCN ZUXQFMVPAYGPFJ-JXUBOQSCSA-N 0.000 description 1
- JTEICXDKGWKRRV-HJGDQZAQSA-N Thr-Asn-Lys Chemical compound C[C@H]([C@@H](C(=O)N[C@@H](CC(=O)N)C(=O)N[C@@H](CCCCN)C(=O)O)N)O JTEICXDKGWKRRV-HJGDQZAQSA-N 0.000 description 1
- NWECYMJLJGCBOD-UNQGMJICSA-N Thr-Phe-Val Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](C(C)C)C(O)=O NWECYMJLJGCBOD-UNQGMJICSA-N 0.000 description 1
- QGVBFDIREUUSHX-IFFSRLJSSA-N Thr-Val-Gln Chemical compound [H]N[C@@H]([C@@H](C)O)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CCC(N)=O)C(O)=O QGVBFDIREUUSHX-IFFSRLJSSA-N 0.000 description 1
- MNYNCKZAEIAONY-XGEHTFHBSA-N Thr-Val-Ser Chemical compound C[C@@H](O)[C@H](N)C(=O)N[C@@H](C(C)C)C(=O)N[C@@H](CO)C(O)=O MNYNCKZAEIAONY-XGEHTFHBSA-N 0.000 description 1
- 208000024770 Thyroid neoplasm Diseases 0.000 description 1
- 101710120037 Toxin CcdB Proteins 0.000 description 1
- GWBWCGITOYODER-YTQUADARSA-N Trp-Leu-Pro Chemical compound CC(C)C[C@@H](C(=O)N1CCC[C@@H]1C(=O)O)NC(=O)[C@H](CC2=CNC3=CC=CC=C32)N GWBWCGITOYODER-YTQUADARSA-N 0.000 description 1
- GIAMKIPJSRZVJB-IHPCNDPISA-N Trp-Phe-Ser Chemical compound C1=CC=C(C=C1)C[C@@H](C(=O)N[C@@H](CO)C(=O)O)NC(=O)[C@H](CC2=CNC3=CC=CC=C32)N GIAMKIPJSRZVJB-IHPCNDPISA-N 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- PDSLRCZINIDLMU-QWRGUYRKSA-N Tyr-Glu Chemical compound OC(=O)CC[C@@H](C(O)=O)NC(=O)[C@@H](N)CC1=CC=C(O)C=C1 PDSLRCZINIDLMU-QWRGUYRKSA-N 0.000 description 1
- AZGZDDNKFFUDEH-QWRGUYRKSA-N Tyr-Gly-Ser Chemical compound OC[C@@H](C(O)=O)NC(=O)CNC(=O)[C@@H](N)CC1=CC=C(O)C=C1 AZGZDDNKFFUDEH-QWRGUYRKSA-N 0.000 description 1
- FBHBVXUBTYVCRU-BZSNNMDCSA-N Tyr-His-Leu Chemical compound C([C@@H](C(=O)N[C@@H](CC(C)C)C(O)=O)NC(=O)[C@@H](N)CC=1C=CC(O)=CC=1)C1=CN=CN1 FBHBVXUBTYVCRU-BZSNNMDCSA-N 0.000 description 1
- 229940127174 UCHT1 Drugs 0.000 description 1
- 101150013568 US16 gene Proteins 0.000 description 1
- 208000025865 Ulcer Diseases 0.000 description 1
- 208000006105 Uterine Cervical Neoplasms Diseases 0.000 description 1
- 101150117115 V gene Proteins 0.000 description 1
- HURRXSNHCCSJHA-AUTRQRHGSA-N Val-Gln-Gln Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CCC(=O)N)C(=O)N[C@@H](CCC(=O)N)C(=O)O)N HURRXSNHCCSJHA-AUTRQRHGSA-N 0.000 description 1
- WJVLTYSHNXRCLT-NHCYSSNCSA-N Val-His-Asp Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CN=CN1)C(=O)N[C@@H](CC(=O)O)C(=O)O)N WJVLTYSHNXRCLT-NHCYSSNCSA-N 0.000 description 1
- BMOFUVHDBROBSE-DCAQKATOSA-N Val-Leu-Cys Chemical compound CC(C)C[C@@H](C(=O)N[C@@H](CS)C(=O)O)NC(=O)[C@H](C(C)C)N BMOFUVHDBROBSE-DCAQKATOSA-N 0.000 description 1
- YQMILNREHKTFBS-IHRRRGAJSA-N Val-Phe-Cys Chemical compound CC(C)[C@@H](C(=O)N[C@@H](CC1=CC=CC=C1)C(=O)N[C@@H](CS)C(=O)O)N YQMILNREHKTFBS-IHRRRGAJSA-N 0.000 description 1
- LCHZBEUVGAVMKS-RHYQMDGZSA-N Val-Thr-Leu Chemical compound CC(C)C[C@H](NC(=O)[C@@H](NC(=O)[C@@H](N)C(C)C)[C@@H](C)O)C(O)=O LCHZBEUVGAVMKS-RHYQMDGZSA-N 0.000 description 1
- 241000251539 Vertebrata <Metazoa> Species 0.000 description 1
- JXLYSJRDGCGARV-WWYNWVTFSA-N Vinblastine Natural products O=C(O[C@H]1[C@](O)(C(=O)OC)[C@@H]2N(C)c3c(cc(c(OC)c3)[C@]3(C(=O)OC)c4[nH]c5c(c4CCN4C[C@](O)(CC)C[C@H](C3)C4)cccc5)[C@@]32[C@H]2[C@@]1(CC)C=CCN2CC3)C JXLYSJRDGCGARV-WWYNWVTFSA-N 0.000 description 1
- 206010047741 Vulval cancer Diseases 0.000 description 1
- 208000004354 Vulvar Neoplasms Diseases 0.000 description 1
- 230000002159 abnormal effect Effects 0.000 description 1
- 230000021736 acetylation Effects 0.000 description 1
- 238000006640 acetylation reaction Methods 0.000 description 1
- 239000002253 acid Substances 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- DZBUGLKDJFMEHC-UHFFFAOYSA-N acridine Chemical class C1=CC=CC2=CC3=CC=CC=C3N=C21 DZBUGLKDJFMEHC-UHFFFAOYSA-N 0.000 description 1
- RJURFGZVJUQBHK-IIXSONLDSA-N actinomycin D Chemical compound C[C@H]1OC(=O)[C@H](C(C)C)N(C)C(=O)CN(C)C(=O)[C@@H]2CCCN2C(=O)[C@@H](C(C)C)NC(=O)[C@H]1NC(=O)C1=C(N)C(=O)C(C)=C2OC(C(C)=CC=C3C(=O)N[C@@H]4C(=O)N[C@@H](C(N5CCC[C@H]5C(=O)N(C)CC(=O)N(C)[C@@H](C(C)C)C(=O)O[C@@H]4C)=O)C(C)C)=C3N=C21 RJURFGZVJUQBHK-IIXSONLDSA-N 0.000 description 1
- 239000012190 activator Substances 0.000 description 1
- 239000004480 active ingredient Substances 0.000 description 1
- 239000002671 adjuvant Substances 0.000 description 1
- 239000008272 agar Substances 0.000 description 1
- 230000001270 agonistic effect Effects 0.000 description 1
- 208000037883 airway inflammation Diseases 0.000 description 1
- 235000004279 alanine Nutrition 0.000 description 1
- 229940100198 alkylating agent Drugs 0.000 description 1
- 239000002168 alkylating agent Substances 0.000 description 1
- 230000000735 allogeneic effect Effects 0.000 description 1
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 description 1
- 210000004381 amniotic fluid Anatomy 0.000 description 1
- 210000004727 amygdala Anatomy 0.000 description 1
- 238000000540 analysis of variance Methods 0.000 description 1
- 239000004037 angiogenesis inhibitor Substances 0.000 description 1
- 238000005571 anion exchange chromatography Methods 0.000 description 1
- 229940045799 anthracyclines and related substance Drugs 0.000 description 1
- 239000003242 anti bacterial agent Substances 0.000 description 1
- 230000000340 anti-metabolite Effects 0.000 description 1
- 230000009830 antibody antigen interaction Effects 0.000 description 1
- 238000011091 antibody purification Methods 0.000 description 1
- 230000005875 antibody response Effects 0.000 description 1
- 238000009175 antibody therapy Methods 0.000 description 1
- 229940100197 antimetabolite Drugs 0.000 description 1
- 239000002256 antimetabolite Substances 0.000 description 1
- 239000003080 antimitotic agent Substances 0.000 description 1
- 230000001640 apoptogenic effect Effects 0.000 description 1
- 230000006907 apoptotic process Effects 0.000 description 1
- 238000011190 asparagine deamidation Methods 0.000 description 1
- 125000000613 asparagine group Chemical group N[C@@H](CC(N)=O)C(=O)* 0.000 description 1
- 229940009098 aspartate Drugs 0.000 description 1
- 108010093581 aspartyl-proline Proteins 0.000 description 1
- 230000001363 autoimmune Effects 0.000 description 1
- 230000001580 bacterial effect Effects 0.000 description 1
- 239000011324 bead Substances 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- SQVRNKJHWKZAKO-UHFFFAOYSA-N beta-N-Acetyl-D-neuraminic acid Natural products CC(=O)NC1C(O)CC(O)(C(O)=O)OC1C(O)C(O)CO SQVRNKJHWKZAKO-UHFFFAOYSA-N 0.000 description 1
- 239000003124 biologic agent Substances 0.000 description 1
- 239000012620 biological material Substances 0.000 description 1
- 230000033228 biological regulation Effects 0.000 description 1
- 239000012472 biological sample Substances 0.000 description 1
- 239000000090 biomarker Substances 0.000 description 1
- 229960002685 biotin Drugs 0.000 description 1
- 235000020958 biotin Nutrition 0.000 description 1
- 239000011616 biotin Substances 0.000 description 1
- 201000000053 blastoma Diseases 0.000 description 1
- 239000012503 blood component Substances 0.000 description 1
- 210000000988 bone and bone Anatomy 0.000 description 1
- 210000000481 breast Anatomy 0.000 description 1
- 239000007975 buffered saline Substances 0.000 description 1
- 125000000484 butyl group Chemical group [H]C([*])([H])C([H])([H])C([H])([H])C([H])([H])[H] 0.000 description 1
- 238000010805 cDNA synthesis kit Methods 0.000 description 1
- 239000001506 calcium phosphate Substances 0.000 description 1
- 229910000389 calcium phosphate Inorganic materials 0.000 description 1
- 235000011010 calcium phosphates Nutrition 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 238000005277 cation exchange chromatography Methods 0.000 description 1
- 239000012930 cell culture fluid Substances 0.000 description 1
- 239000006143 cell culture medium Substances 0.000 description 1
- 230000022131 cell cycle Effects 0.000 description 1
- 230000024245 cell differentiation Effects 0.000 description 1
- 230000003915 cell function Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000013592 cell lysate Substances 0.000 description 1
- 230000004663 cell proliferation Effects 0.000 description 1
- 238000001516 cell proliferation assay Methods 0.000 description 1
- 230000010001 cellular homeostasis Effects 0.000 description 1
- 238000005119 centrifugation Methods 0.000 description 1
- 201000010881 cervical cancer Diseases 0.000 description 1
- NDAYQJDHGXTBJL-MWWSRJDJSA-N chembl557217 Chemical compound C1=CC=C2C(C[C@H](NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](CC=3C4=CC=CC=C4NC=3)NC(=O)[C@@H](C(C)C)NC(=O)[C@H](C(C)C)NC(=O)[C@@H](C(C)C)NC(=O)[C@H](C)NC(=O)[C@H](NC(=O)CNC(=O)[C@@H](NC=O)C(C)C)CC(C)C)C(=O)NCCO)=CNC2=C1 NDAYQJDHGXTBJL-MWWSRJDJSA-N 0.000 description 1
- 238000002144 chemical decomposition reaction Methods 0.000 description 1
- 238000002512 chemotherapy Methods 0.000 description 1
- 208000006990 cholangiocarcinoma Diseases 0.000 description 1
- 238000004587 chromatography analysis Methods 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 238000010367 cloning Methods 0.000 description 1
- 239000011248 coating agent Substances 0.000 description 1
- 238000000576 coating method Methods 0.000 description 1
- 229960001338 colchicine Drugs 0.000 description 1
- 210000001072 colon Anatomy 0.000 description 1
- 238000004737 colorimetric analysis Methods 0.000 description 1
- 238000007398 colorimetric assay Methods 0.000 description 1
- 238000012875 competitive assay Methods 0.000 description 1
- 230000009137 competitive binding Effects 0.000 description 1
- 230000004154 complement system Effects 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 238000010276 construction Methods 0.000 description 1
- 239000000356 contaminant Substances 0.000 description 1
- 239000003431 cross linking reagent Substances 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 125000000151 cysteine group Chemical class N[C@@H](CS)C(=O)* 0.000 description 1
- JVHIPYJQMFNCEK-UHFFFAOYSA-N cytochalasin Natural products N1C(=O)C2(C(C=CC(C)CC(C)CC=C3)OC(C)=O)C3C(O)C(=C)C(C)C2C1CC1=CC=CC=C1 JVHIPYJQMFNCEK-UHFFFAOYSA-N 0.000 description 1
- ZMAODHOXRBLOQO-UHFFFAOYSA-N cytochalasin-A Natural products N1C(=O)C23OC(=O)C=CC(=O)CCCC(C)CC=CC3C(O)C(=C)C(C)C2C1CC1=CC=CC=C1 ZMAODHOXRBLOQO-UHFFFAOYSA-N 0.000 description 1
- 230000009089 cytolysis Effects 0.000 description 1
- 210000001151 cytotoxic T lymphocyte Anatomy 0.000 description 1
- 230000003013 cytotoxicity Effects 0.000 description 1
- 231100000135 cytotoxicity Toxicity 0.000 description 1
- 239000002619 cytotoxin Substances 0.000 description 1
- 229960000640 dactinomycin Drugs 0.000 description 1
- 230000007547 defect Effects 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000006735 deficit Effects 0.000 description 1
- 230000003111 delayed effect Effects 0.000 description 1
- 229940029030 dendritic cell vaccine Drugs 0.000 description 1
- CFCUWKMKBJTWLW-UHFFFAOYSA-N deoliosyl-3C-alpha-L-digitoxosyl-MTM Natural products CC=1C(O)=C2C(O)=C3C(=O)C(OC4OC(C)C(O)C(OC5OC(C)C(O)C(OC6OC(C)C(O)C(C)(O)C6)C5)C4)C(C(OC)C(=O)C(O)C(C)O)CC3=CC2=CC=1OC(OC(C)C1O)CC1OC1CC(O)C(O)C(C)O1 CFCUWKMKBJTWLW-UHFFFAOYSA-N 0.000 description 1
- 230000000779 depleting effect Effects 0.000 description 1
- 230000001627 detrimental effect Effects 0.000 description 1
- 239000008121 dextrose Substances 0.000 description 1
- UQLDLKMNUJERMK-UHFFFAOYSA-L di(octadecanoyloxy)lead Chemical compound [Pb+2].CCCCCCCCCCCCCCCCCC([O-])=O.CCCCCCCCCCCCCCCCCC([O-])=O UQLDLKMNUJERMK-UHFFFAOYSA-L 0.000 description 1
- 206010012601 diabetes mellitus Diseases 0.000 description 1
- 230000029087 digestion Effects 0.000 description 1
- 238000010790 dilution Methods 0.000 description 1
- 239000012895 dilution Substances 0.000 description 1
- 239000000539 dimer Substances 0.000 description 1
- 230000005750 disease progression Effects 0.000 description 1
- 230000009429 distress Effects 0.000 description 1
- 238000012377 drug delivery Methods 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000008030 elimination Effects 0.000 description 1
- 238000003379 elimination reaction Methods 0.000 description 1
- 201000008184 embryoma Diseases 0.000 description 1
- AUVVAXYIELKVAI-CKBKHPSWSA-N emetine Chemical compound N1CCC2=CC(OC)=C(OC)C=C2[C@H]1C[C@H]1C[C@H]2C3=CC(OC)=C(OC)C=C3CCN2C[C@@H]1CC AUVVAXYIELKVAI-CKBKHPSWSA-N 0.000 description 1
- 229960002694 emetine Drugs 0.000 description 1
- AUVVAXYIELKVAI-UWBTVBNJSA-N emetine Natural products N1CCC2=CC(OC)=C(OC)C=C2[C@H]1C[C@H]1C[C@H]2C3=CC(OC)=C(OC)C=C3CCN2C[C@H]1CC AUVVAXYIELKVAI-UWBTVBNJSA-N 0.000 description 1
- 230000002357 endometrial effect Effects 0.000 description 1
- 210000002889 endothelial cell Anatomy 0.000 description 1
- 239000003623 enhancer Substances 0.000 description 1
- 231100000655 enterotoxin Toxicity 0.000 description 1
- 229960003649 eribulin Drugs 0.000 description 1
- UFNVPOGXISZXJD-XJPMSQCNSA-N eribulin Chemical compound C([C@H]1CC[C@@H]2O[C@@H]3[C@H]4O[C@H]5C[C@](O[C@H]4[C@H]2O1)(O[C@@H]53)CC[C@@H]1O[C@H](C(C1)=C)CC1)C(=O)C[C@@H]2[C@@H](OC)[C@@H](C[C@H](O)CN)O[C@H]2C[C@@H]2C(=C)[C@H](C)C[C@H]1O2 UFNVPOGXISZXJD-XJPMSQCNSA-N 0.000 description 1
- 210000003743 erythrocyte Anatomy 0.000 description 1
- 201000004101 esophageal cancer Diseases 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- VJJPUSNTGOMMGY-MRVIYFEKSA-N etoposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@H](C)OC[C@H]4O3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 VJJPUSNTGOMMGY-MRVIYFEKSA-N 0.000 description 1
- 229960005420 etoposide Drugs 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 210000003754 fetus Anatomy 0.000 description 1
- 239000000945 filler Substances 0.000 description 1
- 238000009459 flexible packaging Methods 0.000 description 1
- 238000002875 fluorescence polarization Methods 0.000 description 1
- 229960002949 fluorouracil Drugs 0.000 description 1
- 230000003325 follicular Effects 0.000 description 1
- UIWYJDYFSGRHKR-UHFFFAOYSA-N gadolinium atom Chemical compound [Gd] UIWYJDYFSGRHKR-UHFFFAOYSA-N 0.000 description 1
- 229930182830 galactose Natural products 0.000 description 1
- 201000010175 gallbladder cancer Diseases 0.000 description 1
- 201000007487 gallbladder carcinoma Diseases 0.000 description 1
- 210000000609 ganglia Anatomy 0.000 description 1
- 201000006585 gastric adenocarcinoma Diseases 0.000 description 1
- 239000000499 gel Substances 0.000 description 1
- 238000003209 gene knockout Methods 0.000 description 1
- 210000004602 germ cell Anatomy 0.000 description 1
- 210000001280 germinal center Anatomy 0.000 description 1
- 108010078144 glutaminyl-glycine Proteins 0.000 description 1
- 229960003180 glutathione Drugs 0.000 description 1
- 230000036252 glycation Effects 0.000 description 1
- 108010010147 glycylglutamine Proteins 0.000 description 1
- 210000003714 granulocyte Anatomy 0.000 description 1
- 239000003102 growth factor Substances 0.000 description 1
- 201000010536 head and neck cancer Diseases 0.000 description 1
- 208000014829 head and neck neoplasm Diseases 0.000 description 1
- 210000002443 helper t lymphocyte Anatomy 0.000 description 1
- 230000002489 hematologic effect Effects 0.000 description 1
- 210000003958 hematopoietic stem cell Anatomy 0.000 description 1
- 229960002897 heparin Drugs 0.000 description 1
- 229920000669 heparin Polymers 0.000 description 1
- 108010040030 histidinoalanine Proteins 0.000 description 1
- 108010025306 histidylleucine Proteins 0.000 description 1
- 230000013632 homeostatic process Effects 0.000 description 1
- 102000046492 human ICOSLG Human genes 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 238000004191 hydrophobic interaction chromatography Methods 0.000 description 1
- 210000000987 immune system Anatomy 0.000 description 1
- 229940127121 immunoconjugate Drugs 0.000 description 1
- 108010023260 immunoglobulin Fv Proteins 0.000 description 1
- 238000002991 immunohistochemical analysis Methods 0.000 description 1
- 238000011532 immunohistochemical staining Methods 0.000 description 1
- 238000013388 immunohistochemistry analysis Methods 0.000 description 1
- 239000002955 immunomodulating agent Substances 0.000 description 1
- 238000001114 immunoprecipitation Methods 0.000 description 1
- 230000001506 immunosuppresive effect Effects 0.000 description 1
- 230000001771 impaired effect Effects 0.000 description 1
- 238000002513 implantation Methods 0.000 description 1
- 238000000099 in vitro assay Methods 0.000 description 1
- 238000011503 in vivo imaging Methods 0.000 description 1
- 238000010348 incorporation Methods 0.000 description 1
- 230000006882 induction of apoptosis Effects 0.000 description 1
- 208000015181 infectious disease Diseases 0.000 description 1
- 238000001802 infusion Methods 0.000 description 1
- 239000004615 ingredient Substances 0.000 description 1
- 230000002452 interceptive effect Effects 0.000 description 1
- 238000005305 interferometry Methods 0.000 description 1
- 230000014828 interferon-gamma production Effects 0.000 description 1
- 238000001361 intraarterial administration Methods 0.000 description 1
- 230000003834 intracellular effect Effects 0.000 description 1
- 238000010212 intracellular staining Methods 0.000 description 1
- 239000007928 intraperitoneal injection Substances 0.000 description 1
- 230000002601 intratumoral effect Effects 0.000 description 1
- 238000011835 investigation Methods 0.000 description 1
- 238000004255 ion exchange chromatography Methods 0.000 description 1
- 229960004768 irinotecan Drugs 0.000 description 1
- UWKQSNNFCGGAFS-XIFFEERXSA-N irinotecan Chemical compound C1=C2C(CC)=C3CN(C(C4=C([C@@](C(=O)OC4)(O)CC)C=4)=O)C=4C3=NC2=CC=C1OC(=O)N(CC1)CCC1N1CCCCC1 UWKQSNNFCGGAFS-XIFFEERXSA-N 0.000 description 1
- 230000002427 irreversible effect Effects 0.000 description 1
- 239000013038 irreversible inhibitor Substances 0.000 description 1
- 238000006317 isomerization reaction Methods 0.000 description 1
- 229960002014 ixabepilone Drugs 0.000 description 1
- FABUFPQFXZVHFB-CFWQTKTJSA-N ixabepilone Chemical compound C/C([C@@H]1C[C@@H]2O[C@]2(C)CCC[C@@H]([C@@H]([C@H](C)C(=O)C(C)(C)[C@H](O)CC(=O)N1)O)C)=C\C1=CSC(C)=N1 FABUFPQFXZVHFB-CFWQTKTJSA-N 0.000 description 1
- 238000012004 kinetic exclusion assay Methods 0.000 description 1
- 238000011813 knockout mouse model Methods 0.000 description 1
- 229910052747 lanthanoid Inorganic materials 0.000 description 1
- 150000002602 lanthanoids Chemical class 0.000 description 1
- 230000002045 lasting effect Effects 0.000 description 1
- 108010009932 leucyl-alanyl-glycyl-valine Proteins 0.000 description 1
- 208000032839 leukemia Diseases 0.000 description 1
- 201000002250 liver carcinoma Diseases 0.000 description 1
- 230000005923 long-lasting effect Effects 0.000 description 1
- 201000005249 lung adenocarcinoma Diseases 0.000 description 1
- 210000002751 lymph Anatomy 0.000 description 1
- 210000001165 lymph node Anatomy 0.000 description 1
- 230000001926 lymphatic effect Effects 0.000 description 1
- 210000005210 lymphoid organ Anatomy 0.000 description 1
- 239000008176 lyophilized powder Substances 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 229920002521 macromolecule Polymers 0.000 description 1
- 230000014759 maintenance of location Effects 0.000 description 1
- 230000003211 malignant effect Effects 0.000 description 1
- 208000015486 malignant pancreatic neoplasm Diseases 0.000 description 1
- 238000004949 mass spectrometry Methods 0.000 description 1
- 230000008774 maternal effect Effects 0.000 description 1
- 238000001840 matrix-assisted laser desorption--ionisation time-of-flight mass spectrometry Methods 0.000 description 1
- 238000002844 melting Methods 0.000 description 1
- 230000008018 melting Effects 0.000 description 1
- 230000009401 metastasis Effects 0.000 description 1
- 229930182817 methionine Natural products 0.000 description 1
- 108010022588 methionyl-lysyl-proline Proteins 0.000 description 1
- 108010068488 methionylphenylalanine Proteins 0.000 description 1
- 229960000485 methotrexate Drugs 0.000 description 1
- 238000002493 microarray Methods 0.000 description 1
- 244000005700 microbiome Species 0.000 description 1
- 230000003278 mimic effect Effects 0.000 description 1
- CFCUWKMKBJTWLW-BKHRDMLASA-N mithramycin Chemical compound O([C@@H]1C[C@@H](O[C@H](C)[C@H]1O)OC=1C=C2C=C3C[C@H]([C@@H](C(=O)C3=C(O)C2=C(O)C=1C)O[C@@H]1O[C@H](C)[C@@H](O)[C@H](O[C@@H]2O[C@H](C)[C@H](O)[C@H](O[C@@H]3O[C@H](C)[C@@H](O)[C@@](C)(O)C3)C2)C1)[C@H](OC)C(=O)[C@@H](O)[C@@H](C)O)[C@H]1C[C@@H](O)[C@H](O)[C@@H](C)O1 CFCUWKMKBJTWLW-BKHRDMLASA-N 0.000 description 1
- 229960004857 mitomycin Drugs 0.000 description 1
- 238000012434 mixed-mode chromatography Methods 0.000 description 1
- 239000003607 modifier Substances 0.000 description 1
- 238000012544 monitoring process Methods 0.000 description 1
- 230000000869 mutational effect Effects 0.000 description 1
- 201000000050 myeloid neoplasm Diseases 0.000 description 1
- 229950006780 n-acetylglucosamine Drugs 0.000 description 1
- 239000002105 nanoparticle Substances 0.000 description 1
- 230000017095 negative regulation of cell growth Effects 0.000 description 1
- 108010068617 neonatal Fc receptor Proteins 0.000 description 1
- 230000002314 neuroinflammatory effect Effects 0.000 description 1
- 230000007935 neutral effect Effects 0.000 description 1
- 210000004940 nucleus Anatomy 0.000 description 1
- 238000002515 oligonucleotide synthesis Methods 0.000 description 1
- 244000309459 oncolytic virus Species 0.000 description 1
- DWAFYCQODLXJNR-BNTLRKBRSA-L oxaliplatin Chemical compound O1C(=O)C(=O)O[Pt]11N[C@@H]2CCCC[C@H]2N1 DWAFYCQODLXJNR-BNTLRKBRSA-L 0.000 description 1
- 229960001756 oxaliplatin Drugs 0.000 description 1
- 239000003002 pH adjusting agent Substances 0.000 description 1
- 210000002741 palatine tonsil Anatomy 0.000 description 1
- 201000002528 pancreatic cancer Diseases 0.000 description 1
- 208000008443 pancreatic carcinoma Diseases 0.000 description 1
- 229940055729 papain Drugs 0.000 description 1
- 235000019834 papain Nutrition 0.000 description 1
- 230000007170 pathology Effects 0.000 description 1
- 239000013610 patient sample Substances 0.000 description 1
- 229940049954 penicillin Drugs 0.000 description 1
- 229940111202 pepsin Drugs 0.000 description 1
- 229940023041 peptide vaccine Drugs 0.000 description 1
- 210000004976 peripheral blood cell Anatomy 0.000 description 1
- 201000002628 peritoneum cancer Diseases 0.000 description 1
- 239000000546 pharmaceutical excipient Substances 0.000 description 1
- 229940124531 pharmaceutical excipient Drugs 0.000 description 1
- 125000001997 phenyl group Chemical group [H]C1=C([H])C([H])=C(*)C([H])=C1[H] 0.000 description 1
- 108010083476 phenylalanyltryptophan Proteins 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 230000026731 phosphorylation Effects 0.000 description 1
- 238000006366 phosphorylation reaction Methods 0.000 description 1
- 201000002511 pituitary cancer Diseases 0.000 description 1
- 239000000902 placebo Substances 0.000 description 1
- 229940068196 placebo Drugs 0.000 description 1
- 210000002381 plasma Anatomy 0.000 description 1
- 239000004033 plastic Substances 0.000 description 1
- 229960003171 plicamycin Drugs 0.000 description 1
- 229940071643 prefilled syringe Drugs 0.000 description 1
- 230000000644 propagated effect Effects 0.000 description 1
- 230000004845 protein aggregation Effects 0.000 description 1
- 238000001742 protein purification Methods 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 229950010131 puromycin Drugs 0.000 description 1
- 238000010791 quenching Methods 0.000 description 1
- 230000000171 quenching effect Effects 0.000 description 1
- 238000003127 radioimmunoassay Methods 0.000 description 1
- 230000008707 rearrangement Effects 0.000 description 1
- 230000001105 regulatory effect Effects 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 230000001177 retroviral effect Effects 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 239000013037 reversible inhibitor Substances 0.000 description 1
- 238000012552 review Methods 0.000 description 1
- 229960005560 rindopepimut Drugs 0.000 description 1
- 238000009666 routine test Methods 0.000 description 1
- 201000003804 salivary gland carcinoma Diseases 0.000 description 1
- 238000000926 separation method Methods 0.000 description 1
- 108010048818 seryl-histidine Proteins 0.000 description 1
- 238000004904 shortening Methods 0.000 description 1
- SQVRNKJHWKZAKO-OQPLDHBCSA-N sialic acid Chemical compound CC(=O)N[C@@H]1[C@@H](O)C[C@@](O)(C(O)=O)OC1[C@H](O)[C@H](O)CO SQVRNKJHWKZAKO-OQPLDHBCSA-N 0.000 description 1
- 230000007781 signaling event Effects 0.000 description 1
- 238000002741 site-directed mutagenesis Methods 0.000 description 1
- 208000000587 small cell lung carcinoma Diseases 0.000 description 1
- 239000001632 sodium acetate Substances 0.000 description 1
- 235000017281 sodium acetate Nutrition 0.000 description 1
- 210000004872 soft tissue Anatomy 0.000 description 1
- 210000004989 spleen cell Anatomy 0.000 description 1
- 239000003381 stabilizer Substances 0.000 description 1
- 238000010561 standard procedure Methods 0.000 description 1
- 238000007619 statistical method Methods 0.000 description 1
- 210000000130 stem cell Anatomy 0.000 description 1
- 239000008223 sterile water Substances 0.000 description 1
- 238000003860 storage Methods 0.000 description 1
- 229960005322 streptomycin Drugs 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 239000000758 substrate Substances 0.000 description 1
- 239000004094 surface-active agent Substances 0.000 description 1
- 230000004083 survival effect Effects 0.000 description 1
- 210000001179 synovial fluid Anatomy 0.000 description 1
- NRUKOCRGYNPUPR-QBPJDGROSA-N teniposide Chemical compound COC1=C(O)C(OC)=CC([C@@H]2C3=CC=4OCOC=4C=C3[C@@H](O[C@H]3[C@@H]([C@@H](O)[C@@H]4O[C@@H](OC[C@H]4O3)C=3SC=CC=3)O)[C@@H]3[C@@H]2C(OC3)=O)=C1 NRUKOCRGYNPUPR-QBPJDGROSA-N 0.000 description 1
- 229960001278 teniposide Drugs 0.000 description 1
- 201000003120 testicular cancer Diseases 0.000 description 1
- 201000002510 thyroid cancer Diseases 0.000 description 1
- 239000003104 tissue culture media Substances 0.000 description 1
- 230000002463 transducing effect Effects 0.000 description 1
- 238000010361 transduction Methods 0.000 description 1
- 230000026683 transduction Effects 0.000 description 1
- 238000003146 transient transfection Methods 0.000 description 1
- 238000013519 translation Methods 0.000 description 1
- 229960000575 trastuzumab Drugs 0.000 description 1
- QORWJWZARLRLPR-UHFFFAOYSA-H tricalcium bis(phosphate) Chemical compound [Ca+2].[Ca+2].[Ca+2].[O-]P([O-])([O-])=O.[O-]P([O-])([O-])=O QORWJWZARLRLPR-UHFFFAOYSA-H 0.000 description 1
- 239000013638 trimer Substances 0.000 description 1
- 108010080629 tryptophan-leucine Proteins 0.000 description 1
- 229960001005 tuberculin Drugs 0.000 description 1
- 230000036269 ulceration Effects 0.000 description 1
- 238000011144 upstream manufacturing Methods 0.000 description 1
- 210000002700 urine Anatomy 0.000 description 1
- 206010046766 uterine cancer Diseases 0.000 description 1
- 208000012991 uterine carcinoma Diseases 0.000 description 1
- 238000002255 vaccination Methods 0.000 description 1
- 238000010200 validation analysis Methods 0.000 description 1
- 235000013311 vegetables Nutrition 0.000 description 1
- 229960003048 vinblastine Drugs 0.000 description 1
- JXLYSJRDGCGARV-XQKSVPLYSA-N vincaleukoblastine Chemical compound C([C@@H](C[C@]1(C(=O)OC)C=2C(=CC3=C([C@]45[C@H]([C@@]([C@H](OC(C)=O)[C@]6(CC)C=CCN([C@H]56)CC4)(O)C(=O)OC)N3C)C=2)OC)C[C@@](C2)(O)CC)N2CCC2=C1NC1=CC=CC=C21 JXLYSJRDGCGARV-XQKSVPLYSA-N 0.000 description 1
- 239000013603 viral vector Substances 0.000 description 1
- 230000000007 visual effect Effects 0.000 description 1
- 201000005102 vulva cancer Diseases 0.000 description 1
- 238000005406 washing Methods 0.000 description 1
- 230000003442 weekly effect Effects 0.000 description 1
- 239000000080 wetting agent Substances 0.000 description 1
- 229940055760 yervoy Drugs 0.000 description 1
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K16/00—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies
- C07K16/18—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans
- C07K16/28—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants
- C07K16/2803—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily
- C07K16/2818—Immunoglobulins [IGs], e.g. monoclonal or polyclonal antibodies against material from animals or humans against receptors, cell surface antigens or cell surface determinants against the immunoglobulin superfamily against CD28 or CD152
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P35/00—Antineoplastic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61K—PREPARATIONS FOR MEDICAL, DENTAL OR TOILETRY PURPOSES
- A61K39/00—Medicinal preparations containing antigens or antibodies
- A61K2039/505—Medicinal preparations containing antigens or antibodies comprising antibodies
- A61K2039/507—Comprising a combination of two or more separate antibodies
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/20—Immunoglobulins specific features characterized by taxonomic origin
- C07K2317/24—Immunoglobulins specific features characterized by taxonomic origin containing regions, domains or residues from different species, e.g. chimeric, humanized or veneered
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/30—Immunoglobulins specific features characterized by aspects of specificity or valency
- C07K2317/33—Crossreactivity, e.g. for species or epitope, or lack of said crossreactivity
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/50—Immunoglobulins specific features characterized by immunoglobulin fragments
- C07K2317/56—Immunoglobulins specific features characterized by immunoglobulin fragments variable (Fv) region, i.e. VH and/or VL
- C07K2317/565—Complementarity determining region [CDR]
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/70—Immunoglobulins specific features characterized by effect upon binding to a cell or to an antigen
- C07K2317/75—Agonist effect on antigen
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2317/00—Immunoglobulins specific features
- C07K2317/90—Immunoglobulins specific features characterized by (pharmaco)kinetic aspects or by stability of the immunoglobulin
- C07K2317/92—Affinity (KD), association rate (Ka), dissociation rate (Kd) or EC50 value
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K2319/00—Fusion polypeptide
- C07K2319/30—Non-immunoglobulin-derived peptide or protein having an immunoglobulin constant or Fc region, or a fragment thereof, attached thereto
Landscapes
- Health & Medical Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Immunology (AREA)
- Organic Chemistry (AREA)
- Life Sciences & Earth Sciences (AREA)
- General Health & Medical Sciences (AREA)
- Medicinal Chemistry (AREA)
- Biochemistry (AREA)
- Biophysics (AREA)
- Genetics & Genomics (AREA)
- Molecular Biology (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Chemical Kinetics & Catalysis (AREA)
- General Chemical & Material Sciences (AREA)
- Nuclear Medicine, Radiotherapy & Molecular Imaging (AREA)
- Pharmacology & Pharmacy (AREA)
- Animal Behavior & Ethology (AREA)
- Public Health (AREA)
- Veterinary Medicine (AREA)
- Medicines Containing Antibodies Or Antigens For Use As Internal Diagnostic Agents (AREA)
- Peptides Or Proteins (AREA)
- Medicines That Contain Protein Lipid Enzymes And Other Medicines (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Preparation Of Compounds By Using Micro-Organisms (AREA)
Abstract
Un anticuerpo monoclonal aislado que se une a ICOS, en donde el anticuerpo comprende (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; ∈ LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67, y en donde el anticuerpo comprende una región constante de cadena pesada de IgG1 humana o una región constante de cadena pesada de IgG3 humana.
Description
DESCRIPCIÓN
Anticuerpos contra ICOS
Solicitudes relacionadas
Esta solicitud reivindica el beneficio de prioridad de las solicitudes provisionales de Estados Unidos núms. 62/137,034 presentada el 23 de marzo de 2015; 62/147,484 presentada el 14 de abril de 2015; 62/156,588 presentada el 4 de mayo de 2015; 62/242,489 presentada el 16 de octubre de 2015; y 62/255,635 presentada el 16 de noviembre de 2015.
Campo de la invención
Se proporcionan anticuerpos que se unen a un coestimulador inducible de células T (ICOS) así como también el uso de estos en métodos de tratamiento que comprenden administrar los anticuerpos anti-ICOS.
Antecedentes
ICOS es un miembro de la superfamilia de inmunoglobulinas B7/CD28/CTLA-4 y se expresa específicamente en las células T. A diferencia de CD28, que se expresa constitutivamente en las células T y proporciona señales coestimuladoras necesarias para la activación completa de las células T en reposo, el ICOS se expresa solo después de la activación inicial de las células T.
ICOS se ha implicado en aspectos diversos de las respuestas de células T (revisado en Simpson y otros, 2010, Curr. Opin. Immunol., 22: 326-332). Tiene un papel en la formación de centros germinales, la colaboración de células T/B y el cambio de clase de inmunoglobulinas. Los ratones deficientes de ICOS muestran una afectación en la formación de centros germinales y tienen disminución en la producción de interleucina IL-10. Estos defectos se han vinculado específicamente a deficiencias en células T cooperadoras foliculares.
ICOS también desempeña un papel en el desarrollo y la función de otros subconjuntos de células T, que incluyen Th1, Th2 y Th17. Cabe destacar que ICOS coestimula la proliferación de células T y la secreción de citocinas asociadas con las células Th1 y Th2. En consecuencia, los ratones con desactivación de ICOS demuestran una afectación del desarrollo de fenotipos autoinmunitarios en una variedad de modelos de enfermedad, que incluyen diabetes (Th1), inflamación de las vías respiratorias (Th2) y modelos neuroinflamatorios de EAE (Th17).
Además de su papel en la modulación de la función de células T efectoras (Tef), ICOS modula además las células T reguladoras (Tregs). El ICOS se expresa a altos niveles en las células Tregs, y se ha implicado en la homeostasis y función de las células Treg.
Tras la activación, ICOS, un homodímero enlazado por disulfuro, induce una señal a través de las vías PI3K y AKT. Los eventos de señalización posteriores resultan en la expresión de factores de transcripción específicos del linaje (por ejemplo, T-bet, GATA-3) y, a su vez, efectos sobre la proliferación y supervivencia de las células T.
El ligando de ICOS (ICOSL; B7-H2; B7RP1; CD275; GL50), además un miembro de la superfamilia B7, es el único ligando para ICOS y se expresa en la superficie celular de células B, macrófagos y células dendríticas. ICOSL funciona como un homodímero enlazado no covalentemente en la superficie celular en su interacción con ICOS. Se ha informado que el ICOSL humano, aunque no el ICOSL de ratón, se une a CD28 humano y CTLA-4 (Yao y otros, 2011, Immunity, 34: 729-740).
Breve descripción
La invención proporciona un anticuerpo monoclonal aislado que se une a ICOS, en donde el anticuerpo comprende (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67; y en donde el anticuerpo comprende una región constante de cadena pesada de IgG1 humana o una región constante de cadena pesada de IgG3 humana.
Se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo es un agonista de células T CD4 (tales como células T CD4 efectoras (Tef)). En algunos casos, se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo es un agonista de células T CD4 (tales como células Tef CD4) y agota las células T reguladoras (Treg). En algunos casos, se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo agota las células Treg, pero no agota las células Tef. En algunos casos, se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo induce la señalización de pAKT en las células T CD4. En algunos casos, se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo induce la señalización de pAKT en las células T CD4 y
agota las células Treg. En algunos casos, se describe un anticuerpo aislado que se une a ICOS, en donde el anticuerpo comprende:
i) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17; o
ii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47; o
iii) (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 22, 62, 72, 82, 92, 102 y 112; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 23, 63, 73, 83, 93, 103 y 113; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 24, 64, 74, 84, 94, 104 y 114; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 25, 65, 75, 85, 95, 105 y 115; (e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 26, 66, 76, 86, 96, 106 y 116; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 27, 67, 77, 87, 97, 107 y 117; o
iv) (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 32, 162, 172 y 182; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 33, 163, 173 y 183; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 34, 164, 174 y 184; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 35, 165, 175 y 185; (e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 36, 166, 176 y 186; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 37, 167, 177 y 187; o v) (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 52, 122, 132, 142 y 152; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 53, 123, 133, 143 y 153; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 54, 124, 134, 144 y 154; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 55, 125, 135, 145 y 155; (e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 56, 126, 136, 146 y 156; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 57, 127, 137, 147 y 157; o
vi) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27; o
vii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37; o
viii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57; o
ix) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77; o
x) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87; o
xi) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97; o
xii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (e) LCDR2 que
comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107; o
xiii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117; o
xiv) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127; o
xv) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137; o (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147; o
xvi) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 154; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157; o
xvii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167; o
xviii) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177; o
xix) (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunas modalidades, el anticuerpo comprende una región variable de cadena pesada (Vh) y una región variable de cadena ligera (Vl), en donde la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 60 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 %, o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 61. En otros casos descritos:
i) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 10 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 11; o
ii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 20 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 21; o
iii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 30 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 31; o
iv) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 40 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 41; o
v) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 50 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 51; o
vi) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 70 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 71; o
vii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 80 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 81; o
viii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 90 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 91; o
ix) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 100 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 101; o
x) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 110 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 111; o
xi) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 120 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 121; o
xii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 130 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 131; o
xiii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 140 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 141; o
xiv) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 150 y Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 151; o
xv) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 160 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 161; o
xvi) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 170 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 171; o
xvii) la Vh es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 180 y la Vl es al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 181.
En algunas modalidades, el anticuerpo comprende una región variable de cadena pesada (Vh) y una región variable de cadena ligera (Vl), en donde la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 60 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 61. También se describen anticuerpos en donde:
i) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 10 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 11; o
ii) la VH comprende la secuencia de aminoácidos de la SEQ ID NO: 20 y la VL comprende la secuencia de aminoácidos de la SEQ ID NO: 21; o
iii) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 30 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 31; o
iv) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 40 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 41; o
v) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 50 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 51; o
vi) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 70 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 71; o
vii) la VH comprende la secuencia de aminoácidos de la SEQ ID NO: 80 y la VL comprende la secuencia de aminoácidos de la SEQ ID NO: 81; o
viii) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 90 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 91; o
ix) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 100 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 101; o
x) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 110 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 111; o
xi) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 120 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 121; o
xii) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 130 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 131; o
xiii) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 140 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 141; o
xiv) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 150 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 151; o
xv) la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 160 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 161; o
xvi) la VH comprende la secuencia de aminoácidos de la SEQ ID NO: 170 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 171; o
xvii) la VH comprende la secuencia de aminoácidos de la SEQ ID NO: 180 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 181.
El anticuerpo es un anticuerpo monoclonal. En algunas modalidades, el anticuerpo es un anticuerpo quimérico o un anticuerpo humanizado. En algunas modalidades, el anticuerpo es un fragmento de anticuerpo seleccionado de un Fab, Fab', Fv, scFv o un fragmento (Fab')2. En algunas modalidades, el anticuerpo es un anticuerpo de longitud completa.
En algunas modalidades, se proporciona un anticuerpo que se une a ICOS, en donde el anticuerpo comprende una cadena pesada que comprende la secuencia de aminoácidos de la SEQ ID NO: 188 y una cadena ligera que comprende la secuencia de aminoácidos de la SEQ ID NO: 189.
En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado un aumento en las células T efectoras (Tef) en el mamífero. En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado la activación de células T efectoras (Tef) en el mamífero. En algunas modalidades, la administración del anticuerpo a un mamífero aumenta la relación de células Tef con respecto a las células Treg. En algunas modalidades, las células Tef son células T CD4+ FoxP3-. En algunas modalidades, las células Tef son células T CD4+ FoxP3- y células T CD8+. En algunas modalidades, las células Tef son células T CD8+. En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado una disminución en las células T reguladoras (Treg) en el mamífero. En algunas modalidades, las células Treg son células T CD4+ FoxP3+.
En algunos casos, se proporciona un anticuerpo aislado que se une al ICOS humano, en donde el anticuerpo se une, además, al ICOS de ratón y/o ICOS de rata. En algunos casos, el anticuerpo aislado se une al ICOS humano con una afinidad (Kd) de menos de 5 nM. En algunos casos, el anticuerpo aislado se une al ICOS de rata con una afinidad (Kd) de menos de 10 nM. En algunos casos, la afinidad se determina mediante interferometría de biocapa (ver, por ejemplo, Abdiche y otros, 2008, Anal Biochem, 377: 209-217; y el sistema ForteBio Octet®). En algunos casos, el anticuerpo se une al ICOS humano, ICOS de ratón, e ICOS de rata. En algunos casos, el anticuerpo se une al ICOS de macaco cangrejero. En algunas modalidades, el anticuerpo es un anticuerpo quimérico o un anticuerpo humanizado. En algunas modalidades, el anticuerpo es un fragmento de anticuerpo seleccionado de un Fab, Fab', Fv, scFv o un fragmento (Fab')2. En algunas modalidades, el anticuerpo es un anticuerpo de longitud completa. En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado un aumento en las células T efectoras (Tef) en el mamífero. En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado la activación de células T efectoras (Tef) en el mamífero. En algunas modalidades, las células Tef son células T CD4+ FoxP3-. En algunas modalidades, las células Tef son células T CD4+ FoxP3- y células T CD8+. En algunas modalidades, las células Tef son células T CD8+. En algunas modalidades, la administración del anticuerpo a un mamífero da como resultado una disminución en las células T reguladoras (Treg) en el mamífero. En algunas modalidades, las células Treg son células T CD4+ FoxP3+. En algunas modalidades, el mamífero se selecciona de un ratón, rata, mono cangrejero y ser humano.
En algunos casos, después del tratamiento del tejido tumoral pulmonar con un anticuerpo proporcionado en la presente, el nivel de una quimiocina o citocina seleccionada de GZMa, GZMb, CSF2, IL2, CXCL9, CXCL10, CXCL11 y CXCL13 es de al menos 2 veces o al menos 3 veces más alto que el nivel de la quimiocina después del tratamiento del tejido tumoral pulmonar con un anticuerpo control. En algunos casos, el nivel es el nivel de un ARNm. En algunas modalidades, el nivel es el nivel de una proteína. En algunos casos, un anticuerpo de control es un anticuerpo de isotipo similar que se une a un antígeno no relacionado, y que no se espera que tenga un efecto sobre los niveles de quimiocinas. En algunos casos, el nivel de la quimiocina se mide 1 hora, 2 horas, 3 horas, 4 horas, 5 horas o 6 horas después del tratamiento. En algunos casos, la quimiocina es CXCL11. En algunos casos, el nivel de la quimiocina se mide 6 horas, 8 horas, 10 horas, 12 horas, 14 horas, 16 horas, 18 horas, 20 horas, 22 horas, 24 horas, 36 horas o 48 horas después del tratamiento. En algunos casos, el nivel de la quimiocina se mide 24 horas después del tratamiento. En algunos casos, el tejido tumoral pulmonar es tejido tumoral pulmonar humano.
En algunos casos, un anticuerpo proporcionado en la presente aumenta el nivel de al menos una quimiocina y/o citocina seleccionada de GZMa, GZMb, CSF2, IL2, Cx Cl 9, CXCL10, CXCL11 y CXCL13 en un mamífero al que se ha administrado el anticuerpo al menos 2 veces. En algunos casos, el nivel de la quimiocina se mide 1 hora, 2 horas, 3 horas, 4 horas, 5 horas o 6 horas después de la administración del anticuerpo. En algunos casos, el nivel es el nivel de un ARNm. En algunos casos, el nivel es el nivel de una proteína. En algunos casos, la quimiocina es CXCL11. En algunos casos, el nivel de la quimiocina se mide 6 horas, 8 horas, 10 horas, 12 horas, 14 horas, 16 horas, 18 horas, 20 horas, 22 horas, 24 horas, 36 horas o 48 horas después de la administración del anticuerpo. En algunos casos, el nivel de la quimiocina se mide 24 horas después de la administración del anticuerpo.
En algunos casos, se proporciona un anticuerpo agonista anti-ICOS, donde el anticuerpo aumenta el nivel de al menos
una quimiocina y/o citocina seleccionada de GZMa, GZMb, CSF2, IL2, CXCL9, CXCL10, CXCL11 y CXCL13 en un mamífero al que se ha administrado el anticuerpo al menos 2 veces. En algunos casos, se proporciona un anticuerpo agonista anti-ICOS, en donde el anticuerpo aumenta el nivel de al menos una quimiocina y/o citocina seleccionada de GZMa, GZMb, CSF2, IL2, CXCL9, CXCL10 y CXCL11 en un mamífero al que se ha administrado el anticuerpo al menos 2 veces. En algunos casos, el nivel de la quimiocina se mide 1 hora, 2 horas, 3 horas, 4 horas, 5 horas o 6 horas después de la administración del anticuerpo. En algunos casos, el nivel es el nivel de un ARNm. En algunas modalidades, el nivel es el nivel de una proteína. En algunos casos, la quimiocina es CXCL11. En algunas modalidades, el nivel de la quimiocina se mide 6 horas, 8 horas, 10 horas, 12 horas, 14 horas, 16 horas, 18 horas, 20 horas, 22 horas, 24 horas, 36 horas o 48 horas después de la administración del anticuerpo. En algunos casos, el nivel de la quimiocina se mide 24 horas después de la administración del anticuerpo. En algunas modalidades, el mamífero es un ser humano. En algunas modalidades, el ser humano tiene cáncer. En algunas modalidades, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunas modalidades, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
En algunos casos, se describe un anticuerpo que se une a ICOS, en donde el anticuerpo aumenta el nivel del ligando de NKp46 (NKp46-L) en las células T. En algunos casos, el aumento del nivel de NKp46-L en las células T se determina mediante un dominio extracelular de NKp46 soluble en un ensayo de citometría de flujo. En algunos casos, el anticuerpo aumenta el nivel de NKp46-L en las células Treg más que el aumento del nivel de NKp46-L en las células Tef. En algunos casos, el anticuerpo aumenta la pérdida de CD16 en las células NK.
En algunas modalidades, se proporciona un ácido nucleico aislado que codifica un anticuerpo descrito en el presente documento. En algunas modalidades, se proporciona un vector que comprende el ácido nucleico. En algunas modalidades, se proporciona una célula huésped que comprende el vector. En algunas modalidades, se proporciona una célula huésped que produce un anticuerpo descrito en el presente documento. En algunas modalidades, se proporciona un método para producir un anticuerpo anti-ICOS, que comprende cultivar la célula huésped en condiciones adecuadas para la expresión del anticuerpo y recuperar el anticuerpo producido por la célula huésped.
En algunos casos, se describe una composición farmacéutica, que comprende un anticuerpo anti-ICOS descrito en la presente y un vehículo farmacéuticamente aceptable.
En algunas modalidades, el anticuerpo anti-ICOS descrito en el presente documento es para usar en métodos para tratar un cáncer, que comprende administrar una cantidad eficaz de un anticuerpo anti-ICOS descrito en el presente documento o una composición farmacéutica descrita en este documento. En algunas modalidades, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunas modalidades, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
En algunos casos, se describe un método para aumentar la cantidad de células T efectoras (Tef) en un mamífero, que comprende administrar una cantidad eficaz de un anticuerpo anti-ICOS descrito en el presente documento o una composición farmacéutica descrita en este documento. En algunos casos, el método comprende además activar las células Tef. En algunos casos, se describe un método para activar células T efectoras (Tef) en un mamífero, que comprende administrar una cantidad eficaz de un anticuerpo anti-ICOS descrito en el presente documento o una composición farmacéutica descrita en este documento. En algunos casos, se describe un método para aumentar la relación de células Tef con respecto a las células Treg en un mamífero, que comprende administrar una cantidad eficaz de un anticuerpo anti-ICOS descrito en el presente documento o una composición farmacéutica descrita en este documento. En algunos casos, las células Tef son células T CD4+ FoxP3-. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+. En algunos casos, las células Tef son células T CD8+. En algunos casos, el método comprende además disminuir la cantidad de células T reguladoras (Treg).
En algunos casos, se describe un método para disminuir la cantidad de células T reguladoras (Treg) en un mamífero, que comprende administrar una cantidad eficaz de un anticuerpo anti-ICOS descrito en el presente documento o una composición farmacéutica descrita en este documento. En algunos casos, las células Treg son células T CD4+ FoxP3+.
En algunos casos, se describe un método para aumentar el nivel de al menos una quimiocina y/o citocina seleccionadas de GZMa, GZMb, CSF2, IL2, c Xc L9, CXCL10, CXCL11 y CXCL13 en un mamífero, que comprende administrar al mamífero un anticuerpo proporcionado en la presente. En algunos casos, el nivel de al menos una quimiocina aumenta en al menos 2 veces o al menos 3 veces. En algunos casos, el nivel de la quimiocina se mide 1 hora, 2 horas, 3 horas, 4 horas, 5 horas o 6 horas después de la administración del anticuerpo. En algunos casos, el nivel es el nivel de un ARNm. En algunos casos, el nivel es el nivel de una proteína. En algunos casos, la quimiocina es CXCL11. En algunos casos, el nivel de la quimiocina se mide 6 horas, 8 horas, 10 horas, 12 horas, 14 horas, 16
horas, 18 horas, 20 horas, 22 horas, 24 horas, 36 horas o 48 horas después de la administración del anticuerpo. En algunos casos, el nivel de la quimiocina se mide 24 horas después de la administración del anticuerpo. En algunas modalidades, el mamífero es un ser humano. En algunas modalidades, el ser humano tiene cáncer. En algunas modalidades, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunas modalidades, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
En algunas modalidades, el mamífero es un ser humano.
En algunas modalidades, el mamífero se administra con al menos un agente terapéutico adicional. En algunos casos, el agente terapéutico adicional se administra simultáneamente o secuencialmente con el anticuerpo anti-ICOS. En algunos casos, el agente terapéutico adicional es una terapia de PD-1. En algunas modalidades, el agente terapéutico adicional se selecciona de un anticuerpo anti-PD-1 y un anticuerpo anti-PD-L1. En algunos casos, un anticuerpo anti ICOS proporcionado en la presente descripción se administra con nivolumab. En algunos casos, un anticuerpo anti ICOS proporcionado en la presente descripción se administra con pembrolizumab. En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con atezolizumab. En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con avelumab. En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con durvalumab.
En algunas modalidades, el agente terapéutico adicional es una vacuna contra el cáncer. En algunas de estas modalidades, la vacuna contra el cáncer se desarrolla mediante el uso de un neoantígeno. En algunas modalidades, la vacuna contra el cáncer es una vacuna de ADN. En algunas modalidades, la vacuna contra el cáncer es un virus modificado genéticamente que comprende un antígeno del cáncer, como PROSTVAC (rilimogene galvacirepvec/rilimogene glafolivec). En algunas modalidades, la vacuna contra el cáncer comprende células tumorales modificadas genéticamente, tales como GVAX.
En algunos casos, el anticuerpo anti-ICOS proporcionado en la presente se administra con un anticuerpo agonista anti-OX40. En algunos casos, el anticuerpo anti-ICOS proporcionado en la presente descripción se administra con un anticuerpo anti-CTLA4. En algunos casos, el anticuerpo anti-ICOS proporcionado en la presente se administra con ipilimumab.
En algunos casos, el agente terapéutico adicional es un agente quimioterapéutico. Los agentes quimioterapéuticos ilustrativos no limitantes incluyen capectiabina, ciclofosfamida, dacarbazina, temozolomida, ciclofosfamida, docetaxel, doxorrubicina, daunorrubicina, cisplatino, carboplatino, epirubicina, eribulina, 5-FU, gemcitabina, irinotecán, ixabepilona, metotrexato, mitoxantrona, oxaliplatino, paclitaxel, nab-paclitaxel, ABRAXANE® (paclitaxel unido a proteínas), pemetrexed, vinorelbina y vincristina. En algunos casos, el anticuerpo anti-ICOS proporcionado en la presente descripción se administra con ABRAXANE® (Celgene). En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con al menos un inhibidor de quinasa. Los inhibidores de quinasas ilustrativos no limitantes incluyen erlotinib, afatinib, gefitinib, crizotinib, dabrafenib, trametinib, vemurafenib y cobimetanib.
En algunos casos, el agente terapéutico adicional es un inhibidor de IDO. Los ejemplos no limitantes de inhibidores de IDO incluyen Indoximod (New Link Genetics), INCB024360 (Incyte Corp), 1-metil-D-triptófano (New Link Genetics) y GDC- 0919 (Genentech). En algunos casos, el agente terapéutico adicional es un fármaco modificador inmunitario (IMiD). Los IMiD ilustrativos no limitantes incluyen talidomida, lenalidomida y pomalidomida.
En algunos casos, el mamífero recibe terapia CAR-T además de la administración de un anticuerpo anti-ICOS descrito en el presente documento.
En algunos casos, el mamífero experimenta cirugía y/o radioterapia además de la administración de un anticuerpo anti-ICOS descrito en el presente documento, con o sin un agente terapéutico adicional. En algunos casos, el mamífero experimenta radioterapia además de la administración de un anticuerpo anti-ICOS descrito en el presente documento, con o sin un agente terapéutico adicional.
En algunos casos, se proporciona el uso de un anticuerpo descrito en el presente documento para la fabricación de un medicamento para tratar el cáncer. En algunos casos, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunos casos, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC). En algunos casos, el medicamento es para la administración con al menos un agente terapéutico adicional. En algunos casos, el agente terapéutico adicional se selecciona de un anticuerpo anti-PD-1 y un anticuerpo anti-PD-L1.
En algunos casos, se proporciona el uso de un anticuerpo descrito en el presente documento o una composición farmacéutica descrita en este documento para tratar el cáncer. En algunos casos, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunos casos, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
En algunos casos, se proporciona el uso de un anticuerpo descrito en el presente documento o una composición farmacéutica descrita en este documento y al menos un agente terapéutico adicional para tratar el cáncer. En algunos casos, el agente terapéutico adicional se selecciona de un anticuerpo anti-PD-1 y un anticuerpo anti-PD-L1. En algunos casos, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunos casos, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
En algunas modalidades, se proporciona un anticuerpo descrito en el presente documento o una composición farmacéutica descrita en este documento para el uso en el tratamiento del cáncer. En algunas modalidades, el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC) y cáncer de mama triple negativo (TNBC). En algunas modalidades, el cáncer se selecciona de melanoma, cáncer gástrico, cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC).
Breve descripción de las figuras
Figura 1A-B. Niveles de ARNm de ICOS en múltiples tumores humanos. A) Se representan la intensidad media y los intervalos de confianza del 75 % de los niveles normalizados de ARNm de ICOS en diversas indicaciones médicas. Se indican las muestras con intensidades fuera del intervalo de confianza del 75 % (puntos). B) El porcentaje de cada tipo de tumor indicado que muestra una tinción para ICOS de 0, 1+, 2+ o 3+ mediante inmunohistoquímica (IHC).
Figura 2. Correlación de la expresión de ICOS con infiltración de células T. Los niveles de ARNm de ICOS de ~ 450 tumores HNSCC se compararon con una puntuación de la firma de quimiocinas asociada con 12 genes de células T o los niveles de ARNm de FoxP3. Los niveles de la firma de quimiocinas normalizada o el ARNm de FoxP3 para cada tumor se grafican en el eje Y, los niveles de ARNm de ICOS se representan en el eje X. La correlación de Spearman (R) de la asociación se muestra en el gráfico [Corr(S)]. Una correlación de >0,75 (R de Spearman) se observa como corte para una correlación fuerte.
Figura 3. La expresión de ARNm de ICOS se comparó con ~450 tumores HNSCC mediante el uso de datos de secuenciación de ARN TCGA (NCI). Los niveles de ARNm de CTLA-4 o PD-1 o PD-L1 normalizados para cada tumor se grafican en el eje Y, los niveles de ARNm de ICOS se representan en el eje X. La correlación de Spearman (R) de la asociación se muestra en el gráfico [Corr(S)]. Los niveles de expresión de ICOS se correlacionan significativamente con la expresión de las moléculas de puntos de control CTLA-4, PD-1. Se observó una débil correlación entre ICOS y PD-L1. Una correlación de >0,75 (R de Spearman) se observa como corte para una correlación fuerte.
Figura 4. Imágenes representativas de las diferentes intensidades de una tinción para ICOS en un tumor de NSCLC humano.
Figura 5. Distribución de la densidad de células con ICOS de tumores humanos. La densidad de células con ICOS se determinó en cada uno de los tumores humanos, y la densidad media de ICOS de cada tipo de tumor se grafica en el eje Y [NSCLC (N=100); HNSCC (N=102); cáncer de mama, todos los subtipos principales (N=94); subtipo triple negativo de cáncer de mama, TNBC (N=95); cáncer de ovario (N=94)]. El análisis estadístico se realizó mediante el uso de ANOVA.
Figura 6. Diversidad de la densidad de células con ICOS en muestras de NSCLC. A) La densidad de la expresión de ICOS se evaluó en un panel de muestras de tumores pulmonares (N=98) y los tumores se clasificaron sobre la base de la densidad de células positivas ICOS+ / mm2. B) Diversidad de la expresión de ICOS en una segunda cohorte independiente de muestras clínicas de NSCLC (N=204).
Figura 7. Distribución de la densidad de células con ICOS en diferentes subconjuntos de células T de cáncer humano. A) Imagen representativa que representa la tinción para ICOS en los distintos compartimentos de células T. Las flechas señalan células Treg ICOS+ FOXP3+ o células efectoras CD4 ICOS+ CD8-. B) La densidad de células con ICOS de tumores individuales se analizó en células Tregs CD4 positivas para FoxP3 o células T positivas para CD8 o células CD4 negativas para CD8 y negativas para FoxP3. Se representan la densidad media de ICOS y la desviación estándar de cada uno de estos tipos de tumor. [Cáncer de pulmón (N=100); HNSCC (N=102); Subtipo triple negativo de cáncer de mama, TNBC (N=95); Cáncer de ovario (N=94)].
Figura 8A-B. Se observa una expresión elevada de ICOS en tumores NSCLC con alta expresión de PD-L1.
Mediante la utilización de la IHC múltiple para PD-L1/ICOS/PD-1 se evaluaron los niveles de ICOS y PD-L1 en un conjunto de adenocarcinoma de NSCLC (n=150). A) Imágenes representativas de un tumor con alto contenido de PD-L1 (panel izquierdo) o con bajo contenido de PD-L1 (panel derecho) teñidos para ICOS, PD-1 y PD-L1. B) Cuantificación de la densidad de células con ICOS en carcinoma de células escamosas (panel izquierdo) y adenocarcinoma (panel derecho) con alto contenido de PD-L1 (>5 % de las células son positivas para PD-L1) o bajo contenido de PD-L1 (<5 % de las células son positivas para PD-L1).
Figura 9A-C. El análisis de TIL de tumores humanos muestra la expresión de ICOS en células Treg y efectores CD4. A) Gráficas de contornos representativos de la expresión de PD-1 e ICOS en diferentes subconjuntos de células T de pacientes con HNSCC (N=4). B) Se muestra la frecuencia de las células positivas para ICOS solamente o las células dobles positivas para ICOS y PD-1 en el compartimento de células T (HNSCC N=4; NSCLC N=3; Ovario N=4). C) Comparación de los niveles de ICOS en células Treg CD4 y Tef CD4. Se grafica la intensidad de la tinción de ICOS según se mide por la Intensidad fluorescente media (MFI; o media geométrica de ICOS) en los subconjuntos de células T CD4 de muestras de tumores de pacientes.
Figura 10A-C. A) Se muestra el efecto de los anticuerpos anti-ICOS sobre la proliferación de células T CD4+ humanas primarias en un formato unido a la placa en presencia de anti-CD3 subóptimo. Se grafica el porcentaje de células divididas. B) Se muestra el efecto de los anticuerpos anti-ICOS sobre la proliferación de células T CD4+ humanas en formato soluble en presencia de PMA subóptimo. Se grafica el porcentaje de células divididas. C) Efecto del anticuerpo anti-ICOS 37A10S713-hIgG1 en la proliferación de células T CD4+ humanas primarias en un formato unido a la placa en presencia de anti-CD3 subóptimo.
Figura 11A-B. Se muestra la evaluación de anticuerpos anti-ICOS en un ensayo indicador de NF-Kb. Los gráficos muestran el porcentaje de células GFP+.
Figura 12. Se muestra la evaluación de anticuerpos anti-ICOS solubles en un ensayo con PBMC con estimulación con superantígeno (SEB). La lectura es la producción de IFNg.
Figura 13. Los anticuerpos anti-ICOS se evaluaron para determinar el posible superagonismo en un ensayo de proliferación de células T humanas en ausencia de anti-CD3. La lectura en este ensayo es el porcentaje de proliferación.
Figura 14A-B. Evaluación de un anticuerpo anti-ICOS en un ensayo de fosfo-AKT (pAKT) en presencia o ausencia de un enlazador cruzado secundario. La lectura es el por ciento de células T CD4 que son positivas para pAKT. A) Resultados en ausencia de un enlazador cruzado secundario. B) Resultados en presencia de un enlazador cruzado secundario.
Figura 15A-B. Se evaluaron los anticuerpos anti-ICOS en el modelo de tumor singénico de fibrosarcoma Sa1/N. El crecimiento tumoral se grafica en el eje y. A) Las líneas discontinuas indican el crecimiento tumoral de ratones individuales; la línea continua indica la curva de crecimiento promedio para el grupo. Se indica la cantidad de ratones libres de tumor por grupo. B) Volumen tumoral promedio en cada grupo de tratamiento.
Figura 16. Los ratones sin tumor tratados previamente con anti-ICOS 7F12 se volvieron a retar con tumores Sa1/N El crecimiento tumoral se graficó en el eje y.
Figura 17. Efecto del anticuerpo anti-ICOS de hámster 37A10 con Fc mG1 y mG2a sobre el crecimiento de tumores Sa1/N. Las líneas discontinuas indican el crecimiento tumoral individual de ratones individuales; la línea continua indica la curva promedio de crecimiento tumoral para el grupo. Se indica la cantidad de ratones libres de tumor por grupo.
Figura 18. Evaluación de anticuerpos anti-ICOS como agentes únicos o en combinación con anti-PDl en el modelo de tumor singénico CT26. Las líneas discontinuas indican ratones individuales; la línea continua indica la curva de crecimiento promedio para el grupo. Se indica la cantidad de ratones libres de tumor por grupo.
Figura 19. Agotamiento de células Treg FoxP3+ en tumores Sa1/N tras el tratamiento con anticuerpo anti-ICOS. Se muestra la frecuencia de células CD8, CD4 Tef, y Treg, en bazo y tumor, y la cantidad de células Treg por mg de tumor. Cada forma indica un ratón individual.
Figura 20. Agotamiento de células Treg y activación de células Tef en tumores Sa1/N tras el tratamiento con anticuerpo anti-ICOS. Fila superior: Frecuencia de células CD8, Tef CD4 y Treg, relación de CD8 a Treg y frecuencia de células T CD8 PD-1+ en los tumores. Fila inferior: Frecuencia de las células Ki-67+ CD8 y CD4 Tef en división, y células Tef CD4 Tbet+ entre las células CD3+ en los tumores. Cada forma indica un ratón individual. Figura 21. Evaluación de un anticuerpo anti-ICOS en el modelo tumoral Sa1/N después del agotamiento de las células T. Se grafica el crecimiento tumoral en el tiempo. Se indica la cantidad de ratones libres de tumor.
Figura 22A-B. A) Reducción de las células Tregs en un ensayo con PBMC tras el tratamiento con un anticuerpo anti-ICOS humanizado. B) Las células Treg y Tef expresan niveles similares de ICOS después de cinco días de tratamiento con IL-2.
Figura 23. Nuevo reto tumoral después del tratamiento con anticuerpo anti-ICOS. El panel izquierdo muestra el volumen tumoral en los ratones después de la administración de una o dos dosis de anticuerpo anti-ICOS. El panel derecho muestra el volumen tumoral en ratones control o ratones que estaban libres de tumor después de la administración del anticuerpo anti-ICOS, después del nuevo reto tumoral.
Figura 24A-B. Aumento en la expresión de ICOSL en ratones que portan tumor Sa1/N (A) y monos cangrejeros (B) administrados con anticuerpo anti-ICOS.
Figura 25. Cambio en la expresión de quimiocinas y citocinas de Th-1 después del tratamiento del tejido tumoral pulmonar con anticuerpo anti- ICOS (paneles derechos) o anticuerpo anti-PD-1 (paneles izquierdos), a las 6 horas (paneles superiores) o 24 horas (paneles inferiores).
Figura 26. Niveles del ligando de NKp46 en células Tef (A, C, E) y células Treg (B, D, F) de tres donantes diferentes, después del tratamiento con anticuerpos agonistas y antagonistas anti-ICOS.
Figura 27. Pérdida de CD16 (disminución de CD16) en células NK tratadas con anticuerpo agonista anti-ICOS.
Descripción detallada de algunas modalidades
Se proporcionan anticuerpos que se unen a ICOS. Se proporcionan, además, cadenas pesadas y cadenas ligeras de un anticuerpo, capaces de formar anticuerpos que se unen a ICOS. Además, se proporcionan anticuerpos, cadenas pesadas y cadenas ligeras que comprenden una o más regiones determinantes de la complementariedad (CDR) particulares. Se proporcionan polinucleótidos que codifican anticuerpos para ICOS. Se proporcionan, además, polinucleótidos que codifican cadenas pesadas o cadenas ligeras de un anticuerpo. Se proporcionan métodos para producir y/o purificar anticuerpos contra ICOS. Se proporcionan métodos de tratamiento con anticuerpos contra ICOS. Tales métodos incluyen, pero sin limitarse a, métodos para tratar el cáncer. Se proporcionan métodos para detectar ICOS. Dichos métodos incluyen métodos para identificar a un individuo que puede beneficiarse del tratamiento con un anticuerpo anti-ICOS, para monitorear el tratamiento de un individuo con un anticuerpo anti-ICOS y para mejorar la eficacia terapéutica de un anticuerpo anti-ICOS en un individuo.
Los títulos de las secciones que se usan en el presente documento son solamente para propósitos organizativos y no deben interpretarse como limitantes de la materia descrita.
Las técnicas y procedimientos descritos o referenciados en la presente descripción generalmente se conocen bien y comúnmente se emplean con el uso de metodología convencional por los expertos en la técnica, tales como, por ejemplo, las metodologías ampliamente utilizadas descritas en Sambrook y otros, Molecular Cloning: A Laboratory Manual 3ra. edición (2001) Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. CURRENT PROTOCOl S IN MOLECULAR BIOLOGY (F. M. Ausubel, y otros, eds., (2003)); la serie METHODS IN ENZYMOLOGY (Academic Press, Inc.): PCR 2: A PRACTICAL APPROACH (M. J. MacPherson, B. D. Hames y G. R. Taylor eds. (1995)), Harlow y Lane, eds. (1988) ANTIBODIES, A LABORATORY MANUAL, y ANIMAL CELL CULTURE (R. I. Freshney, ed. (1987)); Oligonucleotide Synthesis (M. J. Gait, ed., 1984); Methods in Molecular Biology, Humana Press; Cell Biology: A Laboratory Notebook (J. E. Cellis, ed., 1998) Academic Press; Animal Cell Culture (R. I. Freshney), ed., 1987); Introduction to Cell and Tissue Culture (J. P. Mather y P. E. Roberts, 1998) Plenum Press; Cell and Tissue Culture Laboratory Procedures (A. Doyle, J. B. Griffiths, y D. G. Newell, eds., 1993-8) J. Wiley and Sons; Handbook of Experimental Immunology (D. M. Weir y C. C. Blackwell, eds.); Gene Transfer Vectors for Mammalian Cells (J. M. Miller y M. P. Calos, eds., 1987); PCR: The Polymerase Chain Reaction, (Mullis y otros, eds., 1994); Current Protocols in Immunology (J. E. Coligan y otros, eds., 1991); Short Protocols in Molecular Biology (Wiley and Sons, 1999); Immunobiology (C. A. Janeway y P. Travers, 1997); Antibodies (P. Finch, 1997); Antibodies: A Practical Approach (D. Catty., ed., IRL Press, 1988-1989); Monoclonal Antibodies: A Practical Approach (P. Shepherd y C. Dean, eds., Oxford University Press, 2000); Using Antibodies: A Laboratory Manual (E. Harlow y D. Lane (Cold Spring Harbor Laboratory Press, 1999); The Antibodies (M. Zanetti y J. D. Capra, eds., Harwood Academic Publishers, 1995); y Cancer: Principles and Practice of Oncology (V. T. Devita y otros, eds., J.B. Lippincott Company, 1993); y versiones actualizadas.
I. Definiciones
A menos que se defina lo contrario, los términos científicos y técnicos usados en relación con la presente descripción tendrán los significados que comúnmente entienden los expertos en la técnica. Además, a menos que se requiera de cualquier otra manera por contexto o se indique expresamente, los términos en singular incluirán pluralidades y los términos en plural incluirán el singular. Para cualquier conflicto en las definiciones entre varias fuentes o referencias, prevalecerá la definición proporcionada en la presente descripción.
Se entiende que las modalidades de la invención descritas en este documento incluyen modalidades "que consisten en" y/o "que consisten esencialmente en". Como se usa en la presente descripción, las formas en singular "un", "una" y "el/la" incluyen las referencias en plural a menos que se indique de cualquier otra manera. El uso del término "o" en la presente no pretende implicar que las alternativas sean mutuamente excluyentes.
En esta solicitud, el uso de "o" significa "y/o" a menos que se indique expresamente o se comprenda por un experto en la técnica. En el contexto de una reivindicación dependiente múltiple, el uso de "o" se refiere a más de una reivindicación independiente o dependiente anterior.
Como entiende un experto en la técnica, la referencia a "aproximadamente" un valor o parámetro en la presente descripción incluye (y describe) modalidades que se dirigen a ese valor o parámetro per se. Por ejemplo, la descripción con referencia a "aproximadamente X" incluye la descripción de "X".
Los términos "molécula de ácido nucleico", "ácido nucleico" y "polinucleótido" pueden usarse indistintamente, y referirse a un polímero de nucleótidos. Tales polímeros de nucleótidos pueden contener nucleótidos naturales y/o no naturales e incluyen, entre otros, ADN, ARN y PNA. "Secuencia de ácido nucleico" se refiere a la secuencia lineal de nucleótidos que comprende la molécula de ácido nucleico o polinucleótido.
Los términos "polipéptido" y "proteína" se usan indistintamente para referirse a un polímero de residuos de
aminoácidos, y no se limitan a una longitud mínima. Tales polímeros de residuos de aminoácidos pueden contener residuos de aminoácidos naturales o no naturales, e incluyen, pero no se limitan a, péptidos, oligopéptidos, dímeros, trímeros y multímeros de residuos de aminoácidos. Tanto las proteínas de longitud completa como los fragmentos de estas se abarcan en la definición. Los términos incluyen además modificaciones después de la expresión del polipéptido, por ejemplo, glicosilación, sialilación, acetilación, fosforilación, y similares. Además, para los propósitos de la presente descripción, un "polipéptido" se refiere a una proteína que incluye modificaciones, tales como deleciones, adiciones y sustituciones (generalmente de naturaleza conservadora), a la secuencia nativa, siempre y cuando la proteína mantenga la actividad deseada. Estas modificaciones pueden ser deliberadas, como a través de mutagénesis dirigida a un sitio, o pueden ser accidentales, como a través de mutaciones de hospederos que producen las proteínas o errores debido a la amplificación por PCR.
"ICOS" y "coestimulador inducible de células T" como se usa en la presente descripción se refieren a cualquier ICOS nativo que resulta de la expresión y el procesamiento de ICOS en una célula. El término incluye ICOS de cualquier fuente de vertebrado, que incluye mamíferos tales como primates (por ejemplo, seres humanos y monos cangrejeros) y roedores (por ejemplo, ratones y ratas), a menos que se indique de cualquier otra manera. El término incluye, además, variantes de origen natural de ICOS, por ejemplo, variantes de corte y empalme o variantes alélicas. La secuencia de aminoácidos de una proteína precursora de ICOS humano ilustrativa, con secuencia señal (con secuencia señal, aminoácidos 1-20) se muestra en la SEQ ID NO: 1. La secuencia de aminoácidos de un ICOS humano maduro ilustrativo se muestra en la SEQ ID NO: 2. La secuencia de aminoácidos de una proteína precursora de ICOS de ratón ilustrativa, con la secuencia señal (con secuencia señal, aminoácidos 1-20) se muestra en la SEQ ID NO: 3. La secuencia de aminoácidos de un ICOS de ratón maduro ilustrativo se muestra en la SEQ ID NO: 4. La secuencia de aminoácidos de una proteína precursora de ICOS de rata ilustrativa, con la secuencia señal (con secuencia señal, aminoácidos 1-20) se muestra en la SEQ ID NO: 190. La secuencia de aminoácidos de un ICOS de rata maduro ilustrativo se muestra en la SEQ ID NO: 191. La secuencia de aminoácidos de un ejemplo de proteína precursora de ICOS de mono cangrejero, con la secuencia señal (con secuencia señal, aminoácidos 1-20) se muestra en la SEQ ID NO: 5. La secuencia de aminoácidos de un ejemplo de ICOS maduro de mono cangrejero se muestra en la SEQ ID NO: 6.
El término "se une específicamente" a un antígeno o epítopo es un término que se entiende bien en la técnica, y los métodos para determinar dicha unión específica también se conocen bien en la técnica. Se dice que una molécula presenta "unión específica" o "unión preferencial" si reacciona o se asocia con mayor frecuencia, con mayor rapidez, con mayor duración y/o con mayor afinidad con una célula o sustancia particular que su unión a otras células o sustancias. Un anticuerpo "se une específicamente" o "se une preferentemente" a un objetivo si se une con mayor afinidad, avidez, más fácilmente, y/o con mayor duración que su unión a otras sustancias. Por ejemplo, un anticuerpo que se une específicamente o preferentemente a un epítopo de ICOS es un anticuerpo que se une a este epítopo con mayor afinidad, avidez, más fácilmente, y/o con mayor duración que su unión a otros epítopos de ICOS o epítopos que no son de ICOS. Al leer esta definición se entiende, además, que, por ejemplo, un anticuerpo (o porción o epítopo) que se une específica o preferentemente a un primer objetivo puede o no unirse específica o preferentemente a un segundo objetivo. Como tal, una “unión específica” o “unión preferencial” no requiere necesariamente (aunque puede incluir) una unión exclusiva. Generalmente, pero no necesariamente, la referencia a la unión se refiere a una unión preferencial. "Especificidad" se refiere a la capacidad de una proteína de unión para unirse selectivamente a un antígeno.
Como se usa en la presente descripción, "esencialmente puro" se refiere a un material que es al menos 50 % puro (es decir, libre de contaminantes), con mayor preferencia, al menos 90 % puro, con mayor preferencia, al menos 95 % puro, aún con mayor preferencia, al menos 98 % puro, y con la máxima preferencia, al menos 99 % puro.
Como se usa en la presente, el término "epítopo" se refiere a un sitio en una molécula objetivo (por ejemplo, un antígeno, tal como una proteína, ácido nucleico, carbohidrato o lípido) al cual se une una molécula de unión a antígeno (por ejemplo, un anticuerpo, fragmento de anticuerpo o proteína de soporte que contiene regiones de unión a anticuerpos). Los epítopos a menudo incluyen un grupo superficial químicamente activo de moléculas tales como aminoácidos, polipéptidos o cadenas laterales de azúcar y tienen características estructurales tridimensionales específicas así como características de carga específicas. Los epítopos pueden formarse a partir de residuos contiguos y/o yuxtapuestos no continuos (por ejemplo, aminoácidos, nucleótidos, azúcares, porción de lípido) de la molécula objetivo. Los epítopos formados a partir de residuos contiguos (por ejemplo, aminoácidos, nucleótidos, azúcares, porción de lípido) típicamente se retienen al exponerse a solventes desnaturalizantes mientras que los epítopos formados por el plegamiento terciario típicamente se pierden en el tratamiento con disolventes desnaturalizantes. Un epítopo puede incluir, entre otros, al menos 3, al menos 5 u 8-10 residuos (por ejemplo, aminoácidos o nucleótidos). En algunos ejemplos, un epítopo tiene menos de 20 residuos (por ejemplo, aminoácidos o nucleótidos) de longitud, menos de 15 residuos o menos de 12 residuos. Dos anticuerpos pueden unirse al mismo epítopo dentro de un antígeno si muestran una unión competitiva por el antígeno. En algunas modalidades, un epítopo puede identificarse por una distancia mínima determinada a un residuo de CDR en la molécula de unión al antígeno. En algunas modalidades, un epítopo puede identificarse por la distancia anterior, y limitarse además a los residuos implicados en un enlace (por ejemplo, un enlace de hidrógeno) entre un residuo del anticuerpo y un residuo del antígeno. Un epítopo puede identificarse mediante diversos barridos, por ejemplo, un barrido de alanina o arginina puede indicar uno o más residuos con los que la molécula de unión al antígeno puede interactuar. A menos que se
indique explícitamente, un conjunto de residuos como un epítopo no excluye que otros residuos sean parte del epítopo para un anticuerpo particular. Más bien, la presencia de tal conjunto designa una serie mínima (o conjunto de especies) de epítopos. Por lo tanto, en algunas modalidades, un conjunto de residuos identificados como un epítopo designa un epítopo mínimo de relevancia para el antígeno, en lugar de una lista exclusiva de residuos para un epítopo en un antígeno.
Un "epítopo no lineal" o "epítopo conformacional" comprende polipéptidos, aminoácidos y/o azúcares no contiguos dentro de la proteína antigénica a la que se une un anticuerpo específico para el epítopo. En algunas modalidades, al menos uno de los residuos será no contiguo con los otros residuos señalados del epítopo; sin embargo, uno o más residuos pueden ser también contiguos con los otros residuos.
Un "epítopo lineal" comprende polipéptidos, aminoácidos y/o azúcares contiguos dentro de la proteína antigénica a la que se une un anticuerpo específico para el epítopo. Se observa que, en algunas modalidades, no todos los residuos dentro de los epítopos lineales necesitan unirse directamente (o estar implicados en un enlace) con el anticuerpo. En algunas modalidades, los epítopos lineales pueden ser de inmunizaciones con un péptido que consistió de manera eficaz en la secuencia del epítopo lineal, o de secciones estructurales de una proteína que están relativamente aisladas del resto de la proteína (de manera que el anticuerpo puede interactuar, al menos principalmente), con esa sección de secuencia.
El término "anticuerpo" en la presente descripción se usa en el sentido más amplio y abarca varias estructuras de anticuerpos, que incluyen pero no se limitan a anticuerpos monoclonales, anticuerpos policlonales, anticuerpos multiespecíficos (por ejemplo, anticuerpos biespecíficos (como anticuerpos acopladores de células T biespecíficos) y triespecíficos) y fragmentos de anticuerpo siempre que exhiban la actividad deseada de unión al antígeno.
El término anticuerpo incluye, pero sin limitarse a, fragmentos que son capaces de unirse a un antígeno, tal como Fv, Fv de cadena simple (scFv), Fab, Fab', di-scFv, sdAb (anticuerpo de dominio único) y (Fab')2 (que incluye un F(ab')2 unido químicamente). La digestión de anticuerpos con papaína produce dos fragmentos idénticos de unión al antígeno, llamados fragmentos "Fab", cada uno con un solo sitio de unión al antígeno, y un fragmento "Fc" residual, cuyo nombre refleja su capacidad de cristalizar fácilmente. El tratamiento con pepsina produce un fragmento F(ab')2 que tiene dos sitios de combinación al antígeno y aún es capaz de unirse de manera cruzada al antígeno. El término anticuerpo incluye, además, pero sin limitarse a, anticuerpos quiméricos, anticuerpos humanizados y anticuerpos de diversas especies tales como ratón, ser humano, monos cangrejeros, etc. Además, para todas las construcciones de anticuerpo proporcionadas en la presente descripción, se contemplan además variantes que tienen las secuencias de otros organismos. Por lo tanto, si se describe una versión humana de un anticuerpo, un experto en la técnica apreciará cómo transformar el anticuerpo basado en la secuencia humana en una secuencia de ratón, rata, gato, perro, caballo, etc. Los fragmentos de anticuerpo incluyen, además, cualquier orientación de scFv de cadena simple, di-scFv en tándem, diacuerpos, tri-sdcFv en tándem, minicuerpos, etc. Los fragmentos de anticuerpo incluyen, además, nanocuerpos (sdAb, un anticuerpo que tiene un dominio monomérico único, tal como un par de dominios variables de cadenas pesadas, sin una cadena ligera). Un fragmento de anticuerpo puede denominarse como específico de una especie en algunas modalidades (por ejemplo, scFv humano o un scFv de ratón). Esto denota las secuencias de al menos parte de las regiones que no son CDR, en lugar de la fuente de la construcción.
El término "anticuerpo monoclonal" se refiere a un anticuerpo de una población sustancialmente homogénea de anticuerpos, es decir, los anticuerpos individuales que comprenden la población son idénticos excepto por posibles mutaciones de origen natural que pueden estar presentes en cantidades menores. Los anticuerpos monoclonales son altamente específicos, dirigidos contra un único sitio antigénico. Además, a diferencia de preparaciones de anticuerpos policlonales, que incluyen típicamente diferentes anticuerpos dirigidos contra diferentes determinantes (epítopos), cada anticuerpo monoclonal se dirige contra un solo determinante en el antígeno. Por lo tanto, una muestra de anticuerpos monoclonales puede unirse al mismo epítopo en el antígeno. El modificador "monoclonal" indica el carácter del anticuerpo que se obtiene a partir de una población sustancialmente homogénea de anticuerpos y no debe interpretarse como que requiere la producción del anticuerpo por ningún método particular. Por ejemplo, los anticuerpos monoclonales pueden producirse por el método de hibridomas descrito primero por Kohler y Milstein, 1975, Nature 256:495, o pueden producirse mediante métodos de ADN recombinante tales como los descritos en la patente de Estados Unidos Núm. 4,816,567. Los anticuerpos monoclonales pueden aislarse, además, a partir de genotecas de fagos generadas mediante el uso de las técnicas descritas en McCafferty y otros, 1990, Nature 348:552-554, por ejemplo.
El término "CDR" denota una región determinante de la complementariedad como se define por al menos una manera de identificación para un experto en la técnica. En algunas modalidades, las CDR pueden definirse de acuerdo con cualquiera de los esquemas de numeración de Chothia, el esquema de numeración de Kabat, una combinación de Kabat y Chothia, la definición de AbM, la definición de contacto, y/o una combinación de las definiciones de Kabat, Chothia, AbM, y/o de contacto. Las CDR ilustrativas (CDR-L1, CDRL2, CDR-L3, CDR-H1, CDR-H2 y CDR-H3) se producen en los residuos de aminoácidos 24-34 de L1, 50-56 de L2, 89-97 de L3, 31-35B de H1, 50-65 de H2 y 95 102 de H3. (Kabat y otros, Sequences of Proteins of Immunological Interest, 5a Ed. Public Health Service, Instituto Nacional de Salud, Bethesda, Md (1991)). La definición de AbM puede incluir, por ejemplo, las CDR (CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 y Cd R-H3) en los residuos de aminoácidos 24-34 de L1, 50-56 de L2, 89-97 de L3, H26-H35B de H1, 50-58 de H2 y 95-102 de H3. La definición por contacto puede incluir, por ejemplo, las CDR (CDRL1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 y CDR-H3) en los residuos de aminoácidos 30-36 de L1 ,46-55 de L2, 89-96 de L3, 30-35 de H 1,47-58 de H2 y 93-101 de H3. La definición de Chothia puede incluir, por ejemplo, las CDR (CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 y CDR-H3) en los residuos de aminoácidos 24-34 de L1, 50-56 de L2, 89-97 de L3, 26-32...34 de H1, 52-56 de H2 y 95-102 de H3. Las CDR pueden proporcionarse, además, como se muestra en cualquiera o más de las figuras acompañantes. Con la excepción de CDR1 en Vh, las CDR generalmente comprenden los residuos de aminoácidos que forman los lazos hipervariables. Las diversas CDR dentro de un anticuerpo pueden designarse por su número adecuado y tipo de cadena, que incluyen, sin limitarse a: a) CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 y CDR-H3; b) CDRL1, CDRL2, CDRL3, CDRH1, CDRH2 y CDRH3; c) LCDR-1, LCDR-2, LCDR-3, HCDR-1, HCDR-2 y HCDR-3; o d) LCDR1, LCDR2, LCDR3, HCDR1, HCDR2 y HCDR3; etc. El término "CDR" se usa en la presente descripción para abarcar además HVR o una "región hipervariable", que incluye lazos hipervariables. Los lazos hipervariables ilustrativos se producen en los residuos de aminoácidos 26-32 (L1), 50 52 (L2), 91-96 (L3), 26-32 (HI), 53-55 (H2) y 96-101 (H3). (Chothia y Lesk, J. Mol. Biol. 196:901-917 (1987)).
El término "región variable de cadena pesada" como se usa en la presente descripción se refiere a una región que comprende al menos tres CDR de cadena pesada. En algunas modalidades, la región variable de cadena pesada incluye las tres CDR y al menos FR2 y FR3. En algunas modalidades, la región variable de cadena pesada incluye al menos HCDR1 de cadena pesada, un marco (FR) 2, HCDR2, FR3 y HCDR3. En algunas modalidades, una región variable de cadena pesada comprende además al menos una porción de un FR1 y/o al menos una porción de un FR4.
El término "región constante de cadena pesada" como se usa en la presente descripción se refiere a una región que comprende al menos tres dominios constantes de cadena pesada, Ch1, Ch2 y Ch3. Por supuesto, las deleciones y modificaciones dentro de los dominios que no alteran la función se incluyen dentro del alcance del término "región constante de cadena pesada", a menos que se designe de cualquier otra manera. Las regiones constantes de cadena pesada ilustrativas no limitantes incluyen y, 6 y a. Las regiones constantes de cadena pesada ilustrativas no limitantes incluyen, además, £ y m. Cada región constante pesada corresponde a un isotipo de anticuerpo. Por ejemplo, un anticuerpo que comprende una región constante de y es un anticuerpo IgG, un anticuerpo que comprende una región constante 6 es un anticuerpo IgD, y un anticuerpo que comprende una región constante a es un anticuerpo IgA. Además, un anticuerpo que comprende una región constante m es un anticuerpo IgM, y un anticuerpo que comprende una región constante £ es un anticuerpo IgE. Determinados isotipos pueden subdividirse adicionalmente en subclases. Por ejemplo, los anticuerpos IgG incluyen, pero sin limitarse a, anticuerpos IgG1 (que comprenden una región constante de y1), IgG2 (que comprenden una región constante de y2), IgG3 (que comprenden una región constante Y3), e IgG4 (que comprenden una región constante y4); los anticuerpos IgA incluyen, pero sin limitarse a, anticuerpos IgA1 (que comprenden una región constante y1) e IgA2 (que comprenden una región constante a2); y los anticuerpos IgM incluyen, pero sin limitarse a, IgM1 e IgM2.
El término "cadena pesada" como se usa en la presente descripción se refiere a un polipéptido que comprende al menos una región variable de cadena pesada, con una secuencia líder o sin ella. En algunas modalidades, una cadena pesada comprende al menos una porción de una región constante de cadena pesada. El término "cadena pesada de longitud completa" como se usa en la presente descripción se refiere a un polipéptido que comprende una región variable de cadena pesada y una región constante de cadena pesada, con una secuencia líder o sin ella.
El término "región variable de cadena ligera" como se usa en la presente descripción se refiere a una región que comprende al menos tres CDR de cadena ligera. En algunas modalidades, la región variable de cadena ligera incluye las tres CDR y al menos FR2 y FR3. En algunas modalidades, la región variable de cadena ligera incluye al menos LCDR1 de cadena ligera, un marco (FR) 2, LCDR2, FR3 y LCDR3. Por ejemplo, una región variable de cadena ligera puede comprender CDR1 de cadena ligera, marco (FR) 2, CDR2, FR3 y CDR3. En algunas modalidades, una región variable de cadena ligera comprende, además, al menos una porción de un FR1 y/o al menos una porción de un FR4.
El término "región constante de cadena ligera" como se usa en la presente descripción se refiere a una región que comprende un dominio constante de cadena ligera, Cl. Las regiones constantes de cadena ligera ilustrativas no limitantes incluyen A y k. Por supuesto, las eliminaciones y modificaciones dentro de los dominios que no alteran la función se incluyen dentro del alcance del término "región constante de cadena ligera", a menos que se designe de cualquier otra manera.
El término "cadena ligera" como se usa en la presente descripción se refiere a un polipéptido que comprende al menos una región variable de cadena ligera, con una secuencia líder o sin ella. En algunas modalidades, una cadena ligera comprende al menos una porción de una región constante de cadena ligera. El término "cadena ligera de longitud completa" como se usa en la presente descripción se refiere a un polipéptido que comprende una región variable de cadena ligera y una región constante de cadena ligera, con una secuencia líder o sin ella.
Un "marco humano aceptor" para los propósitos en la presente descripción es un marco que comprende la secuencia de aminoácidos de un marco de un dominio variable de cadena ligera (Vl) o un marco de un dominio variable de cadena pesada (Vh) derivado de un marco de inmunoglobulina humana o un marco consenso humano, como se define a continuación. Un marco humano aceptor derivado de un marco de inmunoglobulina humana o un marco consenso humano puede comprender su misma secuencia de aminoácidos, o puede contener cambios en la secuencia de aminoácidos. En algunas modalidades, la cantidad de cambios de aminoácidos es 10 o menos, 9 o menos, 8 o menos,
7 o menos, 6 o menos, 5 o menos, 4 o menos, 3 o menos, o 2 o menos. En algunas modalidades, el marco humano aceptor de Vl tiene una secuencia idéntica a la secuencia del marco de Vl de inmunoglobulina humana o una secuencia de marco consenso humano.
"Afinidad" se refiere a la fuerza de la suma total de las interacciones no covalentes entre un solo sitio de unión de una molécula (por ejemplo, un anticuerpo) y su pareja de unión (por ejemplo, un antígeno). La afinidad de una molécula X por su pareja Y puede representarse generalmente por la constante de disociación (Kd ). La afinidad puede medirse mediante métodos comunes conocidos en la técnica (tales como, por ejemplo, Kd por ELISA, KinExA, interferometría de biocapa (BLI) y/o dispositivos de resonancia de plasmones superficiales (como un dispositivo BIAcore®), que incluye los descritos en la presente descripción).
El término "Kd", como se usa en la presente descripción, se refiere a la constante de disociación de equilibrio de una interacción anticuerpo-antígeno.
En algunas modalidades, la Kd", "Kd", "Kd" o "valor de Kd" del anticuerpo se mide mediante el uso de ensayos de resonancia de plasmones superficiales usando un BIACORE®-2000 o BIACORE®-3000 (BIAcore, Inc., Piscataway, N.J.) a 25 °C con chips CM5 de antígenos inmovilizados en ~10 unidades de respuesta (RU). Brevemente, los chips de biosensores de dextrano carboximetilado (CM5, BIACORE, Inc.) se activan con N-etil-N'-(3-dimetilaminopropil)-carbodiimida clorhidrato (EDC) y N-hidroxisuccinimida (NHS) de conformidad con las instrucciones del proveedor. El antígeno se diluye con acetato de sodio 10 mM, pH 4,8, a 5 mg/ml (~0,2 mM) antes de la inyección a una velocidad de flujo de 5 ml/minuto para lograr aproximadamente 10 unidades de respuesta (RU) de la proteína acoplada. Después de la inyección del antígeno, se inyecta etanolamina 1 M para bloquear los grupos sin reaccionar. Para las mediciones de cinética, se inyectan diluciones en serie del polipéptido, por ejemplo, anticuerpo de longitud completa, en PBS con tensioactivo Tw Ee N-20™ al 0,05 % (PBST) a 25 °C a una velocidad de flujo de aproximadamente 25 ml/min. Las velocidades de asociación (kon) y las velocidades de disociación (koff) se calculan mediante el uso de un modelo simple de unión de Langmuir de uno a uno (Software de evaluación BIACORE® versión 3.2) mediante el ajuste simultáneo de los sensorgramas de asociación y disociación. La constante de disociación de equilibrio (Kd) se calcula como la relación kf /kon. Véase, por ejemplo, Chen y otros, J. Mol. Biol. 293:865-881 (1999). Si la velocidad de asociación excede 106 M'V mediante el ensayo de resonancia de plasmones superficiales anterior, entonces la velocidad de asociación puede determinarse mediante el uso de una técnica de inactivación fluorescente que mide el aumento o disminución de la intensidad de emisión de fluorescencia (excitación=295 nm; emisión=340 nm, paso de banda de 16 nm) a 25 °C de un anticuerpo anti-antígeno 20 nM en PBS, pH 7,2, en presencia de concentraciones crecientes de antígeno según se mide en un espectrómetro, tal como un espectrofotómetro con parada de flujo (Aviv Instruments) o un espectrofotómetro SLM-AMINCo ™ serie 8000 (ThermoSpectronic) con una cubeta agitada.
En algunas modalidades, la diferencia entre dichos dos valores (por ejemplo, los valores de Kd) es sustancialmente la misma, por ejemplo, menos de aproximadamente 50 %, menos de aproximadamente 40 %, menos de aproximadamente 30 %, menos de aproximadamente 20 % y/o menos de aproximadamente 10 % como una función del valor de referencia/comparación.
En algunas modalidades, la diferencia entre dichos dos valores (por ejemplo, los valores de Kd) es sustancialmente diferente, por ejemplo, mayor que aproximadamente 10 %, mayor que aproximadamente 20 %, mayor que aproximadamente 30 %, mayor que aproximadamente 40 % y/o mayor que aproximadamente 50 % en función del valor para la molécula de referencia/comparación.
"Resonancia de plasmones superficiales" denota un fenómeno óptico que permite el análisis de interacciones bioespecíficas en tiempo real mediante la detección de alteraciones en concentraciones de proteínas dentro de una matriz de biosensores, por ejemplo mediante el uso del sistema BIAcore™ (BIAcore International AB, una empresa de GE Healthcare, Uppsala, Suecia y Piscataway, N.J.). Para obtener más descripciones, véase Jonsson y otros (1993) Ann. Biol. Clin. 51:19-26.
"Interferometría de biocapa" se refiere a una técnica analítica óptica que analiza el patrón de interferencia de la luz reflejada desde una capa de proteína inmovilizada en una punta del biosensor y una capa de referencia interna. Los cambios en el número de moléculas unidas a la punta del biosensor provocan cambios en el patrón de interferencia que pueden medirse en tiempo real. Un dispositivo ilustrativo no limitante para la interferometría de biocapa es el sistema ForteBio Octet® RED96 (Pall Corporation). Véase, por ejemplo, Abdiche y otros, 2008, Anal. Biochem. 377: 209-277.
El término "kon", como se usa en la presente descripción, se refiere a la constante de velocidad para la asociación de un anticuerpo a un antígeno. Específicamente, las constantes de velocidad (kon y koff) y las constantes de disociación de equilibrio se miden mediante el uso de IgG (bivalentes) con antígeno ICOS monovalente. "Kon", "kon", “constante de velocidad de asociación" o "ka", se usan indistintamente en la presente descripción. El valor indica la velocidad de unión de una proteína de unión a su antígeno objetivo o la velocidad de formación de complejos entre un anticuerpo y un antígeno, que se muestra por la ecuación:
Anticuerpo("Ac") Antígeno("Ag") ^ Ac-Ag.
El término "k"off", como se usa en la presente descripción, se refiere a la constante de velocidad para la disociación de un anticuerpo del complejo anticuerpo/antígeno. kf también se denota como "Koff" o la "constante de velocidad de disociación".
Este valor indica la velocidad de disociación de un anticuerpo de su antígeno objetivo o la separación del complejo Ac-Ag en el tiempo para dar el anticuerpo y el antígeno libres como se muestra por la ecuación:
Ac Ag ^ Ac-Ag.
El término "actividad biológica" se refiere a cualquiera de una o más propiedades biológicas de una molécula (ya sea presentes de manera natural como se encuentra in vivo, o proporcionadas o habilitadas por medios recombinantes). Las propiedades biológicas incluyen, pero no se limitan a, unión a un receptor, inducción de la proliferación celular, inhibición del crecimiento celular, inducción de otras citocinas, inducción de apoptosis y actividad enzimática. En algunas modalidades, la actividad biológica de una proteína ICOS incluye, por ejemplo, la coestimulación de la proliferación de células T y la secreción de citocinas asociadas con células Th1 y Th2; la modulación de células Treg; los efectos sobre la diferenciación de las células T que incluyen la modulación de la expresión génica de factores de transcripción; la inducción de la señalización a través de las vías PI3K y AKT; y la mediación de ADCC.
Un anticuerpo "con maduración de la afinidad" se refiere a un anticuerpo con una o más alteraciones en una o más CDR en comparación con un anticuerpo original que no posee tales alteraciones, dichas alteraciones resultan en una mejora en la afinidad del anticuerpo por el antígeno.
Un "anticuerpo quimérico" como se usa en la presente descripción se refiere a un anticuerpo en el que una porción de la cadena pesada y/o ligera se deriva de una fuente o especie particulares, mientras que al menos una parte del resto de la cadena pesada y/o ligera se deriva de una fuente o especie diferentes. En algunas modalidades, un anticuerpo quimérico se refiere a un anticuerpo que comprende al menos una región variable de una primera especie (tal como ratón, rata, macaco cangrejero, etc.) y al menos una región constante de una segunda especie (tal como un ser humano, mono cangrejero, etc.). En algunas modalidades, un anticuerpo quimérico comprende al menos una región variable de ratón y al menos una región constante humana. En algunas modalidades, un anticuerpo quimérico comprende al menos una región variable de mono cangrejero y al menos una región constante humana. En algunas modalidades, todas las regiones variables de un anticuerpo quimérico son de una primera especie y todas las regiones constantes del anticuerpo quimérico son de una segunda especie. La construcción quimérica también puede ser un fragmento funcional, como se señaló anteriormente.
Un "anticuerpo humanizado" como se usa en la presente descripción se refiere a un anticuerpo en el que al menos un aminoácido en una región marco de una región variable no humana se ha sustituido con el aminoácido correspondiente de una región variable humana. En algunas modalidades, un anticuerpo humanizado comprende al menos una región constante humana o fragmento de esta. En algunas modalidades, un anticuerpo humanizado es un fragmento de anticuerpo, tal como Fab, un scFv, un (Fab')2, etc. El término humanizado denota, además, formas de anticuerpos no humanos (por ejemplo, murinos) que son inmunoglobulinas quiméricas, cadenas de inmunoglobulinas o fragmentos de estos (tales como Fv, Fab, Fab', F(ab')2 u otras subsecuencias de unión al antígeno de los anticuerpos) que contienen una secuencia mínima de inmunoglobulina no humana. Los anticuerpos humanizados pueden incluir inmunoglobulinas humanas (anticuerpo receptor) en las que los residuos de una región determinante de la complementariedad (CDR) del receptor se sustituyen por residuos de una CDR de una especie no humana (anticuerpo donante) tal como ratón, rata o conejo que tiene la especificidad, afinidad y capacidad deseadas. En algunos casos, los residuos de la región del marco Fv (FR) de la inmunoglobulina humana se sustituyen por los correspondientes residuos no humanos. Además, el anticuerpo humanizado puede comprender residuos que no se encuentran ni en el anticuerpo receptor ni en las secuencias de CDR o marco importadas, pero se incluyen para refinar y optimizar adicionalmente el rendimiento del anticuerpo. En general, el anticuerpo humanizado puede comprender sustancialmente al menos uno, y típicamente dos, dominios variables, en los que todas o sustancialmente todas las regiones CDR corresponden a las de una inmunoglobulina no humana y todas o sustancialmente todas las regiones FR son las de una secuencia consenso de inmunoglobulina humana. En algunas modalidades, el anticuerpo humanizado puede comprender, además, al menos una porción de una región o dominio constante de inmunoglobulina (Fc), típicamente el de una inmunoglobulina humana. Otras formas de anticuerpos humanizados tienen una o más CDR (CDR L1, CDR L2, CDR L3, CDR H1, CDR H2 y/o CDR H3) que se alteran con respecto al anticuerpo original, que se denomina además una o más CDR "derivadas de" una o más CDR del anticuerpo original. Como se apreciará, una secuencia humanizada puede identificarse por su secuencia primaria y no necesariamente denota el proceso mediante el cual se creó el anticuerpo.
Un "anticuerpo injertado con CDR" como se usa en la presente descripción se refiere a un anticuerpo humanizado en el que una o más regiones determinantes de la complementariedad (CDR) de una primera especie (no humana) se han injertado en las regiones del marco (FR) de una segunda especie (humana).
Un "anticuerpo humano" como se usa en la presente descripción, abarca anticuerpos producidos en seres humanos, anticuerpos producidos en animales no humanos que comprenden genes de inmunoglobulinas humanas, tales como ratones XenOmouse® y anticuerpos seleccionados mediante el uso de métodos in vitro, tales como la presentación
en fagos (Vaughan y otros, 1996, Nature Biotechnology, 14:309-314; Sheets y otros, 1998, Proc. Natl. Acad. Sci. (USA) 95:6157-6162; Hoogenboom y Winter, 1991, J. Mol. Biol., 227:381; Marks y otros, 1991, J. Mol. Biol., 222:581), en donde el repertorio de anticuerpos se basa en una secuencia de inmunoglobulina humana. El término "anticuerpo humano" denota el género de secuencias que son secuencias humanas. Por lo tanto, el término no designa el proceso mediante el cual se creó el anticuerpo, sino el género de las secuencias que son de interés.
Una "región Fc funcional" posee una "función efectora" de una región Fc de secuencia nativa. Las "funciones efectoras" ilustrativas incluyen la unión al receptor de Fc; unión de C1q; CDC; ADCC; fagocitosis; regulación negativa de receptores de la superficie celular (por ejemplo, receptor de células B; BCR), etc. Tales funciones efectoras requieren generalmente que la región Fc se combine con un dominio de unión (por ejemplo, un dominio variable de anticuerpo) y puede evaluarse mediante el uso de varios ensayos.
Una "región Fc de secuencia nativa" comprende una secuencia de aminoácidos idéntica a la secuencia de aminoácidos de una región Fc que se encuentra en la naturaleza. Las regiones Fc humanas de secuencia nativa incluyen una región Fc de IgG1 humana de secuencia nativa (alotipos A y diferentes de A); una región Fc de IgG2 humana de secuencia nativa; una región Fc de IgG3 humana de secuencia nativa; y una región Fc de IgG4 humana de secuencia nativa así como sus variantes de origen natural.
Una "variante de la región Fc" comprende una secuencia de aminoácidos que difiere de la de una región Fc de secuencia nativa debido a al menos una modificación de aminoácidos. En algunas modalidades, una "variante de la región Fc" comprende una secuencia de aminoácidos que difiere de la de una región Fc de secuencia nativa debido a al menos una modificación de aminoácidos, aunque mantiene al menos una función efectora de la región Fc de secuencia nativa. En algunas modalidades, la variante de la región Fc tiene al menos una sustitución de aminoácidos en comparación con una región Fc de secuencia nativa o con la región Fc de un polipéptido original, por ejemplo, de aproximadamente una a aproximadamente diez sustituciones de aminoácidos, y preferentemente, de aproximadamente una a aproximadamente cinco sustituciones de aminoácidos en una región Fc de secuencia nativa o en la región Fc del polipéptido original. En algunas modalidades, la variante de la región Fc en la presente descripción poseerá al menos aproximadamente 80 % de identidad de secuencia con una región Fc de secuencia nativa y/o con una región Fc de un polipéptido original, al menos aproximadamente 90 % de identidad de secuencia con ella, al menos aproximadamente 95 %, al menos aproximadamente 96 %, al menos aproximadamente 97 %, al menos aproximadamente 98 % o al menos aproximadamente 99 % de identidad de secuencia con ella.
"Receptor de Fc" o "FcR" describe un receptor que se une a la región Fc de un anticuerpo. En algunas modalidades, un FcyR es un FcR humano nativo. En algunas modalidades, un FcR es uno que se une a un anticuerpo IgG (un receptor gamma) e incluye receptores de las subclases FcyRI, FcyRII y FcyRIII, que incluyen variantes alélicas y formas de empalmes alternativos de esos receptores. Los receptores FcyRII incluyen FcyRIIA (un "receptor activador") y FcyRIIB (un "receptor inhibidor"), que tienen secuencias de aminoácidos similares que difieren principalmente en sus dominios citoplasmáticos. El receptor activador FcyRIIA contiene un motivo de activación basado en tirosina del inmunorreceptor (ITAM) en su dominio citoplasmático. El receptor inhibidor FcyRIIB contiene un motivo de inhibición basado en tirosina del inmunorreceptor (ITIM) en su dominio citoplasmático (ver, por ejemplo, Daeron, Annu. Rev. Immunol. 15:203-234 (1997)). Los FcR se revisan, por ejemplo, en Ravetch y Kinet, Annu. Rev. Immunol 9:457-92 (1991); Capel y otros, Immunomethods 4:25-34 (1994); y de Haas y otros, J. Lab. Clin. Med. 126:330-41 (1995). Otros FcR, incluidos los que se identificarán en el futuro, se incluyen en el término "FcR" en la presente.
El término "receptor de Fc" o "FcR" incluye además el receptor neonatal, FcRn, que es responsable de la transferencia de IgG maternas al feto (Guyer y otros, J. Immunol. 117:587 (1976) y Kim y otros, J. Immunol. 24:249 (1994)) y la regulación de la homeostasis de las inmunoglobulinas. Los métodos para medir la unión a FcRn se conocen (ver, por ejemplo, Ghetie y Ward., Immunol. Today 18(12):592-598 (1997); Ghetie y otros, Nature Biotechnology, 15(7):637-640 (1997); Hinton y otros, J. Biol. Chem. 279(8):6213-6216 (2004); documento WO 2004/92219 (Hinton y otros).
Las "funciones efectoras" se refieren a actividades biológicas atribuibles a la región Fc de un anticuerpo, que varían con el isotipo de anticuerpo. Los ejemplos de funciones efectoras de anticuerpos incluyen: Unión a Clq y citotoxicidad dependiente del complemento (CDC); unión al receptor de Fc; citotoxicidad mediada por células dependiente de anticuerpos (ADCC); fagocitosis; regulación negativa de receptores de la superficie celular (por ejemplo, receptor de células B); y activación de células B.
Las "células efectoras humanas" son leucocitos que expresan uno o más FcR y realizan funciones efectoras. En algunas modalidades, las células expresan al menos FcyRIII y realizan la(s) función(es) efectora(s) de ADCC. Los ejemplos de leucocitos humanos que median la ADCC incluyen células mononucleares de sangre periférica (PBMC), células asesinas naturales (NK), monocitos, células T citotóxicas y neutrófilos. Las células efectoras pueden aislarse de una fuente natural, por ejemplo, de sangre.
"Citotoxicidad mediada por células dependiente de anticuerpos" o "ADCC" se refiere a una forma de citotoxicidad en la que una Ig secretada se une a receptores de Fc (FcR) presentes en determinadas células citotóxicas (por ejemplo, células NK, neutrófilos y macrófagos) lo que permite que estas células efectoras citotóxicas se unan específicamente a una célula objetivo portadora de antígenos y posteriormente matan a la célula objetivo con citotoxinas. Las células
primarias para mediar la ADCC, las células NK, expresan únicamente FcyRIII, mientras que los monocitos expresan FcyRI, FcyRII y FcyRIII. La expresión de FcR en células hematopoyéticas se resume en la Tabla 3 en la página 464 de Ravetch y Kinet, Annu. Rev. Immunol 9:457-92 (1991). Para evaluar la actividad de ADCC de una molécula de interés, puede llevarse a cabo un ensayo de ADCC in vitro, tal como el descrito en las patentes de los Estados Unidos núms. 5,500,362 o 5,821,337 o la patente de Estados Unidos núm. 6,737,056 (Presta). Las células efectoras útiles para tales ensayos incluyen células PBMC y NK. Alternativa o adicionalmente, la actividad ADCC de la molécula de interés puede evaluarse in vivo, por ejemplo, en un modelo animal tal como el descrito en Clynes y otros Proc. Natl. Acad. Sci. (USA) 95:652-656 (1998). Las variantes polipeptídicas adicionales con secuencias de aminoácidos alteradas de la región Fc (polipéptidos con una variante de la región Fc) y la actividad de ADCC aumentada o disminuida se describen, por ejemplo, en la patente de Estados Unidos núm. 7,923,538 y la patente de Estados Unidos núm. 7,994,290.
"Citotoxicidad dependiente del complemento" o "CDC" se refiere a la lisis de una célula objetivo en presencia del complemento. La activación de la vía clásica del complemento se inicia por la unión del primer componente del sistema del complemento (C1q) a anticuerpos (de la subclase apropiada), que se unen a su antígeno afín. Para evaluar la activación del complemento, se puede realizar un ensayo de CDC, por ejemplo, como se describe en Gazzano-Santoro y otros, J. Immunol. Methods 202:163 (1996). Las variantes polipeptídicas con secuencias de aminoácidos alteradas de la región Fc (polipéptidos con una variante de la región Fc) y la capacidad de unión a C1q aumentada o disminuida se describen, por ejemplo, en la patente de Estados Unidos núm. 6,194,551 B1, la patente de Estados Unidos núm.
7,923,538, la patente de Estados Unidos núm. 7,994,290 y el documento WO 1999/51642. Véase además, por ejemplo, Idusogie y otros, J. Immunol. 164: 4178-4184 (2000).
Una variante del polipéptido con alteración de la afinidad de unión a FcR o de la actividad ADCC es aquella que tiene aumento o disminución de la actividad de unión a FcR y/o actividad de ADCC en comparación con un polipéptido original o con un polipéptido que comprende una región Fc de secuencia nativa. La variante del polipéptido que "muestra una mayor unión" a un FcR se une a al menos un FcR con mejor afinidad que el polipéptido original. La variante del polipéptido que "muestra una reducción de la unión" a un FcR, se une al menos a un FcR con menor afinidad que un polipéptido original. Tales variantes que muestran disminución de la unión a un FcR pueden poseer poca o ninguna unión apreciable a un FcR, por ejemplo, 0-20 % de unión al FcR en comparación con una región Fc de IgG de secuencia nativa.
La variante del polipéptido que "media la citotoxicidad mediada por células dependiente de anticuerpos (ADCC) en presencia de células efectoras humanas de manera más eficaz" que un anticuerpo original es aquella que in vitro o in vivo es más eficaz en la mediación de ADCC, cuando las cantidades de la variante del polipéptido y el anticuerpo original usados en el ensayo son esencialmente iguales. Generalmente, tales variantes se identificarán mediante el uso del ensayo de ADCC in vitro como se describe en el presente documento, pero se contemplan otros ensayos o métodos para determinar la actividad de ADCC, por ejemplo en un modelo animal etc.
El término "sustancialmente similar" o "sustancialmente el mismo", como se usa en la presente descripción, denota un grado de similitud suficientemente alto entre dos o más valores numéricos de manera que un experto en la técnica consideraría que la diferencia entre los dos o más valores es de poca o ninguna significación biológica y/o estadística dentro del contexto de la característica biológica medida por dicho valor. En algunas modalidades los dos o más valores sustancialmente similares difieren en no más de cualquiera de aproximadamente 5 %, 10 %, 15 %, 20 %, 25 % o 50 %.
La frase "sustancialmente diferente" como se usa en la presente descripción, denota un grado de diferencia suficientemente alto entre dos valores numéricos de manera que un experto en la técnica consideraría que la diferencia entre los dos valores es de significación estadística dentro del contexto de la característica biológica medida por dichos valores. En algunas modalidades, los dos valores numéricos sustancialmente diferentes difieren en más de aproximadamente cualquiera de 10 %, 20 %, 25 %, 30 %, 35 %, 40 %, 45 %, 50 %, 60 %, 70 %, 80 %, 90 % o 100 %.
La frase "sustancialmente reducida", como se usa en la presente descripción, denota un grado suficientemente alto de reducción entre un valor numérico y un valor numérico de referencia de manera que un experto en la técnica consideraría que la diferencia entre los dos valores es de significación estadística dentro del contexto de la característica biológica medida por dichos valores. En algunas modalidades, los valores numéricos sustancialmente reducidos están reducidos por más de cualquiera de aproximadamente 10 %, 20 %, 25 %, 30 %, 35 %, 40 %, 45 %, 50 %, 60 %, 70 %, 80 %, 90 % o 100 % comparado con el valor de referencia.
El término "secuencia líder" se refiere a una secuencia de residuos de aminoácidos localizados en el extremo N-terminal de un polipéptido que facilita la secreción de un polipéptido de una célula de mamífero. Una secuencia líder puede escindirse tras la exportación del polipéptido de la célula de mamífero, para formar una proteína madura. Las secuencias líderes pueden ser naturales o sintéticas, y pueden ser heterólogas u homólogas a la proteína a la que están unidas.
Un polipéptido de "secuencia nativa" comprende un polipéptido que tiene la misma secuencia de aminoácidos que un polipéptido que se encuentra en la naturaleza. Por lo tanto, un polipéptido de secuencia nativa puede tener la
secuencia de aminoácidos de un polipéptido de origen natural de cualquier mamífero. Dicho polipéptido de secuencia nativa puede aislarse de la naturaleza o puede producirse mediante medios recombinantes o sintéticos. El término polipéptido de "secuencia nativa" abarca específicamente las formas truncadas o secretadas de origen natural del polipéptido (por ejemplo, una secuencia de dominio extracelular), formas de variantes de origen natural (por ejemplo, formas empalmadas alternativamente) y variantes alélicas de origen natural del polipéptido.
Una "variante" de polipéptido se refiere a un polipéptido biológicamente activo que tiene al menos aproximadamente 80 % de identidad de secuencia de aminoácidos con el polipéptido de secuencia nativa después de alinear las secuencias e introducir espacios, si es necesario, para lograr el porcentaje máximo de identidad de secuencia, y sin considerar ninguna sustitución conservadora como parte de la identidad de secuencia. Tales variantes incluyen, por ejemplo, polipéptidos en donde se añaden o se eliminan uno o más residuos de aminoácidos en el extremo N o C terminal del polipéptido. En algunas modalidades, una variante tendrá al menos aproximadamente 80 % de identidad de secuencia de aminoácidos. En algunas modalidades, una variante tendrá al menos aproximadamente 90 % de identidad de secuencia de aminoácidos. En algunas modalidades, una variante tendrá al menos aproximadamente 95 % de identidad de secuencia de aminoácidos con el polipéptido de secuencia nativa.
Como se usa en la presente descripción, "Por ciento (%) de identidad de secuencia de aminoácidos" y "homología" con respecto a una secuencia de péptido, polipéptido o anticuerpo se definen como el porcentaje de residuos de aminoácidos en una secuencia candidata que son idénticos a los residuos de aminoácidos en la secuencia peptídica o polipeptídica específica, después de alinear las secuencias e introducir espacios, si es necesario, para lograr el porcentaje máximo de identidad de secuencia, y sin considerar ninguna sustitución conservadora como parte de la identidad de secuencia. La alineación con fines de determinar el por ciento de identidad de secuencia de aminoácidos puede lograrse de diversas maneras que se encuentran dentro de la técnica, por ejemplo, mediante el uso de software informático disponible públicamente tal como BLAST, BLAST-2, ALIGN o MEGALIGN™ (DNASTAR). Los expertos en la técnica pueden determinar los parámetros apropiados para medir la alineación, lo que incluye cualquier algoritmo necesario para lograr una alineación máxima sobre toda la longitud de las secuencias que se están comparando.
Una sustitución de aminoácidos puede incluir, entre otros, el reemplazo de un aminoácido en un polipéptido con otro aminoácido. En la Tabla 1 se muestran sustituciones ilustrativas. Las sustituciones de aminoácidos pueden introducirse en un anticuerpo de interés y los productos se analizan para determinar una actividad deseada, por ejemplo, retención/mejora de la unión al antígeno, menor inmunogenicidad o mejor ADCC o CDC.
TABLA 1
(continuación)
Los aminoácidos pueden agruparse de acuerdo con las propiedades comunes de la cadena lateral:
(1) hidrofóbicos: Norleucina, Met, Ala, Val, Leu, Ile;
(2) hidrófilos neutros: Cys, Ser, Thr, Asn, Gln;
(3) ácidos: Asp, Glu;
(4) básicos: His, Lys, Arg;
(5) residuos que influyen en la orientación de la cadena: Gly, Pro;
(6) aromáticos: Trp, Tyr, Phe.
Las sustituciones no conservadoras implicarán intercambiar un miembro de una de estas clases para otra clase.
El término "vector" se usa para describir un polinucleótido que puede modificarse genéticamente para contener un polinucleótido o polinucleótidos clonados que pueden propagarse en una célula huésped. Un vector puede incluir uno o más de los siguientes elementos: un origen de replicación, una o más secuencias reguladoras (como, por ejemplo, promotores y/o potenciadores) que regulan la expresión del polipéptido de interés, y/o uno o más genes marcadores de selección (como, por ejemplo, genes de resistencia a antibióticos y genes que pueden usarse en ensayos colorimétricos, por ejemplo, p-galactosidasa). El término "vector de expresión" se refiere a un vector que se usa para expresar un polipéptido de interés en una célula huésped.
Una "célula huésped" se refiere a una célula que puede ser o ha sido un receptor de un vector o polinucleótido aislado. Las células huésped pueden ser células procariotas o células eucariotas. Las células eucariotas ilustrativas incluyen células de mamífero, como células animales de primate o no; células fúngicas, como de levadura; células vegetales; y células de insectos. Las células de mamíferos ilustrativas no limitantes incluyen, pero sin limitarse a, células NSO, células PER.C6® (Crucell) y células 293 y CHO y sus derivados, tales como células 293-6E y DG44, respectivamente. Las células huésped incluyen la progenie de una célula huésped única, y la progenie puede no necesariamente ser completamente idéntica (en morfología o en complemento de ADN genómico) a la célula original debido a una mutación natural, accidental o intencional. Una célula huésped incluye células transfectadas in vivo con polinucleótido(s) proporcionado(s) en la presente descripción.
El término "aislado" como se usa en la presente descripción se refiere a una molécula que se ha separado de al menos algunos de los componentes con los que típicamente se encuentra en la naturaleza o se produce. Por ejemplo, se hace referencia a un polipéptido como "aislado" cuando se separa de al menos algunos de los componentes de la célula en la que se produjo. Cuando un polipéptido es secretado por una célula después de su expresión, separar físicamente el sobrenadante que contiene el polipéptido de la célula que lo produjo se considera que es "aislar" el polipéptido. De manera similar, se refiere a un polinucleótido como "aislado" cuando no es parte del polinucleótido más grande (tal como, por ejemplo, ADN genómico o ADN mitocondrial, en el caso de un polinucleótido de ADN) en el que típicamente se encuentra en la naturaleza, o se separa de al menos algunos de los componentes de la célula en la que se produjo, por ejemplo, en el caso de un polinucleótido de ARN. Por lo tanto, un polinucleótido de ADN que está contenido en un vector dentro de una célula huésped puede denominarse "aislado".
Los términos "individuo" o "sujeto" se usan indistintamente en la presente para referirse a un animal; por ejemplo un mamífero. En algunas modalidades, se proporcionan métodos para tratar mamíferos, que incluyen, pero sin limitarse a, seres humanos, roedores, simios, felinos, caninos, equinos, bovinos, porcinos, ovinos, caprinos, animales mamíferos de laboratorio, animales mamíferos de granja, animales mamíferos deportivos y mascotas mamíferos. En algunos ejemplos, un "individuo" o "sujeto" se refiere a un individuo o sujeto que necesita tratamiento para una enfermedad o trastorno. En algunas modalidades, el sujeto que recibe el tratamiento puede ser un paciente, que designa el hecho de que el sujeto se ha identificado que tiene un trastorno de relevancia para el tratamiento, o que tiene un riesgo adecuado de contraer el trastorno.
Una "enfermedad" o "trastorno" como se usa en la presente descripción se refiere a una afección donde se necesita y/o se desea un tratamiento.
"Cáncer" y "tumor," como se usan en la presente descripción, son términos intercambiables que se refieren a cualquier
crecimiento o proliferación anormal de células o tejidos en un animal. Como se usa en la presente descripción, los términos "cáncer" y "tumor" abarcan cánceres sólidos y hematológicos/linfáticos y también abarcan un crecimiento maligno, premaligno y benigno, como la displasia. Los ejemplos de cáncer incluyen, entre otros, carcinoma, linfoma, blastoma, sarcoma y leucemia. Ejemplos no limitantes más particulares de tales cánceres incluyen cáncer de células escamosas, cáncer de pulmón de células pequeñas, cáncer de la hipófisis, cáncer esofágico, astrocitoma, sarcoma de tejido blando, cáncer de pulmón de células no pequeñas, adenocarcinoma del pulmón, carcinoma escamoso de pulmón, cáncer de peritoneo, cáncer hepatocelular, cáncer gastrointestinal, cáncer de páncreas, glioblastoma, cáncer de cuello uterino, cáncer de ovario, cáncer hepático, cáncer de vejiga, hepatoma, cáncer de mama, cáncer de colon, cáncer colorrectal, carcinoma uterino o endometrial, carcinoma de glándulas salivares, cáncer de riñón, cáncer renal, cáncer de hígado, cáncer de próstata, cáncer vulvar, cáncer de tiroides, carcinoma hepático, cáncer de encéfalo, cáncer de endometrio, cáncer de testículo, colangiocarcinoma, carcinoma de vesícula biliar, cáncer gástrico, melanoma, y varios tipos de cáncer de cabeza y cuello.
Como se usa en la presente descripción, "tratamiento" es un enfoque para obtener resultados clínicos beneficiosos o deseados. "Tratamiento" como se usa en la presente descripción, cubre cualquier administración o aplicación de un producto terapéutico para la enfermedad en un mamífero, que incluye un ser humano. Para los fines de esta descripción, los resultados clínicos beneficiosos o deseados incluyen, pero no se limitan a, uno o más de: alivio de uno o más síntomas, disminución de la extensión de la enfermedad, prevención o retraso de la propagación (por ejemplo, metástasis, por ejemplo, metástasis en el pulmón o en el ganglio linfático) de la enfermedad, prevención o retraso de la recurrencia de la enfermedad, retraso o ralentización del avance de la enfermedad, mejora del estado de la enfermedad, inhibición de la enfermedad o la progresión de la enfermedad, inhibición o ralentización de la enfermedad o su progresión, detención de su desarrollo, y remisión (ya sea parcial o total). El término “tratamiento” también abarca una reducción de la consecuencia patológica de una enfermedad proliferativa. Los métodos proporcionados en la presente descripción contemplan uno o más de estos aspectos del tratamiento. En línea con lo anterior, el término tratamiento no requiere cien por ciento de eliminación de todos los aspectos del trastorno.
“Mejorar” significa una reducción o mejora de uno o más síntomas en comparación con no administrar un anticuerpo anti-ICOS. La "mejora" incluye además acortamiento o reducción de la duración de un síntoma.
En el contexto de cáncer, el término "tratar" incluye cualquiera o todos los siguientes: inhibir el crecimiento de células cancerosas, inhibir la replicación de células cancerosas, disminuir la carga tumoral total y mejorar uno o más síntomas asociados con la enfermedad.
El término "muestra biológica" significa una cantidad de una sustancia de un organismo vivo o que estuvo vivo. Tales sustancias incluyen, pero no se limitan a, sangre, (por ejemplo, sangre completa), plasma, suero, orina, líquido amniótico, líquido sinovial, células endoteliales, leucocitos, monocitos, otras células, órganos, tejidos, médula ósea, ganglios linfáticos y bazo.
Una muestra que tiene un "nivel elevado de ICOS" o "que expresa ICOS a un nivel elevado" o es " ICOSALTO" significa que el nivel de ICOS es tal que un experto en la técnica concluiría que el cáncer puede tratarse con una terapia anti ICOS, como un anticuerpo proporcionado en la presente descripción. En algunas modalidades, un "nivel elevado de ICOS" es uno en el que 1 % de las células dentro de una muestra de tumor muestra tinción para ICOS. En algunas modalidades, un "alto nivel" con respecto a ICOS es una tinción de 1 % o más, por ejemplo, 1, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90 o 100 % de las células dentro de la muestra tumoral que muestran tinción. En algunas modalidades, los niveles de ICOS pueden medirse mediante IHC cromogénica o IHC de inmunofluorescencia (puntuación Aqua).
Una muestra que "expresa ICOS" o tiene "tinción positiva para ICOS" o es "ICOS positiva" significa que 1 % o más de las células en una muestra muestran tinción para ICOS. En algunas modalidades, una muestra que es positiva para ICOS presenta al menos una tinción celular débil, moderada y/o fuerte (sobre la base de la expresión de ICOS en la membrana). Una muestra con tinción de células moderada o fuerte para ICOS también se considera que es " ICOSALTO".
Una muestra que tiene un "nivel bajo de PD-L1" o expresa "PD-L1 a un nivel bajo" o es "p d -L1bajo" significa que el nivel de PD-L1 está por debajo del nivel umbral de expresión para un cáncer al que normalmente se indica un tratamiento con una terapia de P D -1. En algunas modalidades, un "bajo nivel de PD-L1" es uno en el que menos del 5 % de las células en el tumor muestran tinción de membrana para PD-L1. En algunas modalidades, un "nivel bajo" con respecto a PD-L1 es menos de 5 % de tinción, por ejemplo, 4 %, 3 %, 2 %, 1 % o 0 % de las células del tumor que muestran tinción. En algunas modalidades, los niveles de PD-L1 pueden medirse mediante IHC cromogénica o IHC de inmunofluorescencia (puntuación Aqua). Una muestra que no expresa PD-L1 detectable también puede decirse "que expresa un nivel bajo de PD-L1". Por lo tanto, el término "bajo" abarca cuando no hay PD-L1 detectable.
Una muestra que tiene un "nivel elevado de PD-L1" o "expresa PD-L1 a un nivel elevado" o es "PD-L1alto" significa que el nivel de PD-L1 es tal que un experto en la técnica concluiría que el cáncer puede tratarse con una terapia de PD-1. En algunas modalidades, un "nivel elevado de PD-L1" es uno en el que 5 % de las células en el tumor o más tienen tinción de membrana de PD-L1. En algunas modalidades, un "alto nivel" con respecto a PD-L1 es 5 % o más de tinción, por ejemplo, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90 o 100 % de las células del tumor que muestran tinción. En
algunas modalidades, los niveles de PD-L1 pueden medirse mediante IHC cromogénica o IHC de inmunofluorescencia (puntuación Aqua).
Una muestra que "expresa PD-L1" o tiene "tinción positiva para PD-L1" o es "positiva para PD-L1" significa que el 1 % o más de las células tienen tinción de membrana para PD-L1. En algunas modalidades, una muestra que es positiva para PD-L1 presenta al menos una tinción celular débil, moderada y/o fuerte (sobre la base de la expresión en membrana de PD-L1). Una muestra con tinción de células moderada o fuerte para PD-L1 también se considera que es "PD-L1alto".
Una muestra que "carece de expresión de PD-L1" o tiene "tinción negativa para PD-L1" o es "negativa para PD-L1" significa que la expresión de PD-L1 en la superficie de las células de la muestra es indetectable por IHC, tal como IHC cromogénica o iHc de inmunofluorescencia (puntuación Aqua). Una muestra negativa para PD-L1 también se considera que es "PD-L1BAJO".
En algunas modalidades, puede emplearse cualquier método para medir el nivel de PD-L1. En algunas modalidades, esto puede incluir usar la prueba de IHC para PD-L1 22C3 pharmDx (Dako Inc., Carpinteria, CA), que es una prueba clínicamente validada y aprobada por la FDA para la evaluación de la expresión de PD-L1 en el NSCLC. IHC para PD-L1 22C3 pharmDx es un ensayo inmunohistoquímico cualitativo que usa anticuerpo monoclonal anti-PD-L1 de ratón, (clon 22C3), que puede usarse en la detección de la proteína PD-L1 en tejidos con cáncer de pulmón de células no pequeñas (NSCLC), fijados en formalina y embebidos en parafina (FFPE). El ensayo puede llevarse a cabo en el sistema Autostainer Link 48 y visualizarse mediante el uso del sistema EnVision Fl Ex . La expresión de la proteína PD-L1 se califica mediante el uso de la Puntuación de Proporción del tumor (TPS), que es el porcentaje de células tumorales viables que muestran tinción de membrana parcial o completa. En algunas modalidades, la muestra se considera positiva para PD-L1 si TPS > 50 % de las células tumorales viables exhiben tinción de membrana en cualquier intensidad. IHC para PD-L1 22C3 pharmDx se indica como un auxiliar para identificar pacientes con NSCLC para el tratamiento con KEYTRUDA® (pembrolizumab). Los detalles adicionales del sistema de puntuación y la respuesta a pembrolizumab se describen en el artículo de Garon y otros (N Engl J Med 2015;372:2018-28). En algunas modalidades, las muestras de pacientes con NSCLC pueden considerarse positivas para la expresión de PD-L1 si la puntuación de proporción del tumor muestra que > 50 % de las células tumorales viables exhiben tinción de membrana (parcial o completa) con cualquier intensidad (es decir > 1+). En algunas modalidades, esto puede ser específico con respecto al clon de anticuerpo 22C3. En algunas modalidades, si TPS = 5 % a 50 % de las células tumorales viables exhiben tinción de membrana en cualquier intensidad, la muestra y/o el paciente se consideran positivos para PD-L1. En algunas modalidades, si TPS muestra que > 50 % de las células tumorales viables exhiben tinción de membrana en cualquier intensidad, la muestra y/o el paciente se consideran con PD-L1ALTO
El término "control" se refiere a una composición conocida por no contener un analito ("control negativo") o por contener un analito ("control positivo"). Un control positivo puede comprender una concentración conocida de un analito. "Control," "control positivo," y "calibrador" pueden usarse indistintamente en la presente descripción para referirse a una composición que comprende una concentración conocida de un analito. Un "control positivo" puede usarse para establecer características de desempeño del ensayo y es un indicador útil de la integridad de los reactivos (por ejemplo, los analitos).
"Valor de corte predeterminado" y "nivel predeterminado" se refieren generalmente a un valor de corte de un ensayo que se usa para evaluar los resultados de la eficacia diagnóstica/pronóstica/terapéutica mediante la comparación de los resultados del ensayo contra el valor de corte/nivel predeterminado, donde el valor de corte/nivel predeterminado ya se ha vinculado o asociado con diversos parámetros clínicos (por ejemplo, gravedad de la enfermedad, progresión/no progresión/mejora, etcétera). Si bien la presente descripción puede proporcionar niveles predeterminados ilustrativos, es bien conocido que los valores de corte pueden variar según la naturaleza del inmunoensayo (por ejemplo, anticuerpos empleados, etc.). Dentro de la habilidad de un experto en la técnica está, además, adaptar la descripción de este documento para otros inmunoensayos para obtener valores de corte específicos del inmunoensayo para esos otros inmunoensayos sobre la base de esta descripción. Si bien el valor preciso del valor de corte/nivel predeterminado puede variar entre ensayos, generalmente pueden aplicarse correlaciones como se describe en el presente documento (si existen).
Los términos "inhibición" o "inhibir" se refieren a una disminución o cese de cualquier característica fenotípica o a la disminución o cese en la incidencia, grado o probabilidad de esa característica. “Reducir” o “inhibir” es disminuir, reducir o detener una actividad, función, y/o cantidad en comparación con una referencia. En algunas modalidades, por “reducir” o “inhibir” se entiende la capacidad de provocar una disminución total de 20 % o más. En algunas modalidades, por “reducir” o “inhibir” se entiende la capacidad de provocar una disminución total de 50 % o más. En algunas modalidades, por “reducir” o “inhibir” se entiende la capacidad de provocar una disminución total de 75 %, 85 %, 90 %, 95 % o más. En algunas modalidades, se inhibe o disminuye la cantidad indicada anteriormente durante un período de tiempo, con respecto a una dosis de control (tal como un placebo) durante el mismo período de tiempo. Una "referencia" como se usa en la presente descripción, se refiere a cualquier muestra, patrón o nivel que se usa con fines de comparación. Una referencia puede obtenerse de una muestra saludable y/o no patológica. En algunos ejemplos, una referencia puede obtenerse de una muestra no tratada. En algunos ejemplos, una referencia se obtiene de una muestra no patológica o no tratada de un sujeto individual. En algunos ejemplos, una referencia se obtiene de
uno o más individuos sanos que no son el sujeto o el paciente.
Como se usa en la presente, "retrasar el desarrollo de una enfermedad" significa diferir, dificultar, ralentizar, retardar, estabilizar, suprimir y/o aplazar el desarrollo de la enfermedad (tal como cáncer). Este retraso puede ser de largos períodos de tiempo, según la historia de la enfermedad y/o individuo que se trata. Tal como es evidente para un experto en la técnica, un retraso suficiente o significativo puede, en efecto, abarcar la prevención, donde el individuo no desarrolla la enfermedad. Por ejemplo, puede retrasarse un cáncer en etapa tardía, tal como el desarrollo de metástasis.
"Prevenir" como se usa en la presente descripción, incluye proporcionar profilaxis con respecto a la aparición o recurrencia de una enfermedad en un sujeto que puede estar predispuesto a la enfermedad pero aún no ha sido diagnosticado con la enfermedad. A menos que se especifique lo contrario, los términos “reducir”, “ inhibir” o “prevenir” no indican ni requieren prevención completa en todo el tiempo.
Como se usa en la presente descripción, "suprimir" una función o actividad es reducir la función o actividad en comparación con cualquier otra condición similar excepto para una condición o parámetro de interés, o alternativamente, en comparación con otra condición. Por ejemplo, un anticuerpo que inhibe el crecimiento tumoral reduce la velocidad de crecimiento del tumor en comparación con la velocidad de crecimiento del tumor en ausencia del anticuerpo.
Una "cantidad con eficacia terapéutica" de una sustancia/molécula, agonista o antagonista puede variar de acuerdo con factores tales como el estado de la enfermedad, la edad, el sexo y el peso del individuo, y la capacidad de la sustancia/molécula, agonista o antagonista de inducir una respuesta deseada en el individuo. Una cantidad con eficacia terapéutica también es una en la que cualquier efecto tóxico o perjudicial de la sustancia/molécula, agonista 0 antagonista se sopesa con los efectos terapéuticamente beneficiosos. Una cantidad con eficacia terapéutica puede suministrarse en una o más administraciones. Una cantidad con eficacia terapéutica se refiere a una cantidad eficaz, en dosis y durante períodos de tiempo necesarios, para lograr el resultado terapéutico y/o profiláctico deseado.
Una "cantidad con eficacia profiláctica" se refiere a una cantidad eficaz, en dosis y durante períodos de tiempo necesarios, para lograr el resultado profiláctico deseado. Típicamente pero no necesariamente, dado que se usa una dosis profiláctica en sujetos antes de una etapa temprana de la enfermedad o en una etapa más temprana de esta, la cantidad con eficacia profiláctica será menor que la cantidad con eficacia terapéutica.
Los términos "formulación farmacéutica" y "composición farmacéutica" se refieren a una preparación que está en una forma tal que permite que la actividad biológica del (de los) ingrediente(s) activo(s) sea eficaz, y que no contenga componentes adicionales que sean inaceptablemente tóxicos para un sujeto a los que la formulación se administrará. Dichas formulaciones pueden ser estériles.
Un "portador farmacéuticamente aceptable" se refiere a un sólido, semisólido, o relleno líquido, diluyente, material de encapsulación, auxiliar de la formulación, o portador no tóxicos, convencionales en la técnica, para su uso con un agente terapéutico, que juntos comprenden una "composición farmacéutica" para su administración a un sujeto. Un portador farmacéuticamente aceptable no es tóxico para los receptores a las dosis y concentraciones empleadas y es compatible con otros ingredientes de la formulación. El portador farmacéuticamente aceptable es adecuado para la formulación empleada.
Una formulación "estéril" es aséptica o está esencialmente libre de microorganismos vivos y sus esporas.
Una "terapia de PD-1" abarca cualquier terapia que module la unión de PD-1 a PD-L1 y/o PD-L2. Las terapias de PD-1 pueden, por ejemplo, interactuar directamente con PD-1 y/o PD-L1. En algunas modalidades, una terapia de PD-1 incluye una molécula que se une directamente a y/o influye en la actividad de PD-1. En algunas modalidades, una terapia de PD-1 incluye una molécula que se une directamente a y/o influye en la actividad de PD-L1. Por lo tanto, un anticuerpo que se une a PD-1 o PD-L1 y bloquea la interacción de PD-1 a PD-L1 es un agente terapéutico de PD-1. Cuando se pretende un subtipo deseado de terapia de PD-1, se designará por la frase "específico para PD-1" para una terapia que implica una molécula que interactúa directamente con PD-1 o "específico para PD-L1" para una molécula que interactúa directamente con PD-L1, según sea apropiado. A menos que se designe de cualquier otra manera, toda la descripción contenida en este documento con respecto a la terapia de PD-1 se aplica en general a la terapia de PD-1, así como a terapias específicas de PD-1 y/o específicas de PD-L1. Los ejemplos no limitantes de terapias de PD-1 incluyen nivolumab (anticuerpo anti-PD-1; BMS-936558, M DX-1106, ONO-4538; OPDIVO®; Bristol-Myers Squibb); pidilizumab (anticuerpo anti-PD-1, CureTech), pembrolizumab (anticuerpo anti-PD-1; KEYTRUDA®, MK-3475, lambrolizumab); durvalumab (anticuerpo anti-PD-L1, MEDI-4736; AstraZeneca/Medlmmune); RG-7446; MSB-0010718C; AMP-224; BMS-936559 (un anticuerpo anti-PD-L1; Bristol-Myers Squibb); AMP-514; MDX-1105; ANB-011; anti-LAG-3/PD-1; Ac anti-PD-1 (CoStim); Ac anti-PD-1 (Kadmon Pharm.); Ac anti-PD-1 (Immunovo); Ac anti-TIM-3/PD-1 (AnaptysBio); Ac anti-PD-L1 (CoStim/Novartis); atezolizumab (un anticuerpo anti-PD-L1, Genentech/Roche); avelumab (un anticuerpo anti-PD-L1, MSB0010718C, Pfizer); Kd -033, antagonista de PD-1 (Agenus); STI-A1010; STI-A1110; TSR-042; y otros anticuerpos dirigidos contra la proteína de muerte programada 1 (PD-1) o el ligando 1 de muerte programada (PD-L1).
El término "inhibidor de IDO" se refiere a un agente capaz de inhibir la actividad de indolamina 2,3-dioxigenasa (IDO) e invertir de esta manera la inmunosupresión mediada por IDO. El inhibidor de IDO puede inhibir IDO1 y/o IDO2 (INDOL1). Un inhibidor de IDO puede ser un inhibidor de IDO reversible o irreversible. Un "inhibidor reversible de IDO" es un compuesto que inhibe de forma reversible la actividad enzimática IDO ya sea en el sitio catalítico o en un sitio no catalítico y un "inhibidor irreversible de IDO" es un compuesto que inhibe de forma irreversible la actividad enzimática IDO formando una unión covalente con la enzima. Los ejemplos no limitantes de inhibidores de IDO incluyen Indoximod (New Link Genetics), INCB024360 (Incyte Corp.), 1-metil-D triptofano (New Link Genetics) y GDC-0919 (Genentech).
Una "terapia de células T con receptor de antígeno quimérico" o "terapia CAR-T" se refiere a un agente terapéutico que comprende una célula T modificada genéticamente para expresar un receptor que reconoce un antígeno expresado por una célula tumoral. El antígeno puede ser un antígeno expresado específicamente por el tumor o un antígeno expresado tanto por células cancerosas como en tejido saludable. En algunas modalidades la terapia CAR-T es terapia CAR-T adoptiva, en la que se extraen células T del paciente y se modifican para expresar el receptor de antígeno quimérico, y después se regresan al paciente. Véase, por ejemplo, Dai y otros, 2016, J Natl Cancer Inst, 108 (7): djv439, doi: 10.1093/jnci/djv439; Gill y otros, 2015, Blood Rev, pii: S0268-960X(15)00080-6, doi: 10.1016/j.blre.2015.10.003; Gill y otros, 2015, Immunol Rev, 263(1):68-89. doi: 10.1111/imr.12243.
La administración "en combinación con" uno o más agentes terapéuticos adicionales incluye la administración simultánea (concurrente) y consecutiva o secuencial en cualquier orden.
El término "concurrentemente" se usa en la presente descripción para referirse a la administración de dos o más agentes terapéuticos, donde al menos parte de la administración coincide en el tiempo o donde la administración de un agente terapéutico cae dentro de un período corto de tiempo con relación a la administración del otro agente terapéutico. Por ejemplo, los dos o más agentes terapéuticos se administran con una separación de tiempo de no más que aproximadamente un número especificado de minutos.
El término "secuencialmente" se usa en la presente descripción para referirse a la administración de dos o más agentes terapéuticos donde la administración de uno o más agentes continúa después de interrumpir la administración de uno o más agentes, o en donde la administración de uno o más agentes comienza antes de la administración de uno o más de otros agentes. Por ejemplo, la administración de los dos o más agentes terapéuticos se administran con una separación de tiempo de más de aproximadamente un número especificado de minutos.
Como se usa en la presente descripción, "junto con" se refiere a la administración de una modalidad de tratamiento además de otra modalidad de tratamiento. Como tal, "junto con" se refiere a la administración de una modalidad de tratamiento antes, durante o después de la administración de la otra modalidad de tratamiento al individuo.
El término "prospecto" se usa para referirse a instrucciones incluidas habitualmente en paquetes comerciales de productos terapéuticos, que contienen información sobre indicaciones, uso, dosificación, administración, terapia de combinación, contraindicaciones y/o advertencias con respecto al uso de tales productos terapéuticos.
Un "artículo de fabricación" es cualquier fabricación (por ejemplo, un paquete o contenedor) o kit que comprende al menos un reactivo, por ejemplo, un medicamento para el tratamiento de una enfermedad o trastorno (por ejemplo, cáncer), o una sonda para detectar específicamente un biomarcador descrito en el presente documento. En algunas modalidades, la fabricación o kit se promociona, distribuye o comercializa como una unidad para llevar a cabo los métodos descritos en la presente descripción.
Los términos "marcador" y "marcador detectable" significan una porción unida a un anticuerpo o a su analito para hacer que una reacción (por ejemplo, la unión) entre los miembros del par de unión específico, sea detectable. El miembro marcado del par de unión específico se denomina "marcado de manera detectable". Por lo tanto, el término "proteína de unión marcada" se refiere a una proteína con un marcador incorporado que proporciona la identificación de la proteína de unión. En algunas modalidades, el marcador es un marcador detectable que puede producir una señal que es detectable por medios visuales o instrumentales, por ejemplo, incorporación de un aminoácido radiomarcado o unión a un polipéptido de restos de biotinilo que pueden detectarse por avidina marcada (por ejemplo, estreptavidina que contiene un marcador fluorescente o actividad enzimática que puede detectarse por métodos ópticos o colorimétricos). Los ejemplos de marcadores para polipéptidos incluyen, pero no se limitan a, los siguientes: radioisótopos o radionúclidos (por ejemplo, 3H, 14C,35S, 90Y, 99Tc, 111In, 125I, 131I, 177Lu, 166Ho o 153Sm); cromógenos, marcadores fluorescentes (por ejemplo, FITC, rodamina, fósforos de lantánidos), marcadores enzimáticos (por ejemplo, peroxidasa de rábano picante, luciferasa, fosfatasa alcalina); marcadores quimioluminiscentes; grupos biotinilo; epítopos de polipéptidos predeterminados reconocidos por un reportero secundario (por ejemplo, secuencias de pares de cremalleras de leucina, sitios de unión para anticuerpos secundarios, dominios de unión a metales, etiquetas de epítopos); y agentes magnéticos, tales como quelatos de gadolinio. Los ejemplos representativos de marcadores comúnmente empleados para inmunoensayos incluyen restos que producen luz, por ejemplo, compuestos de acridinio, y restos que producen fluorescencia, por ejemplo, fluoresceína. En este sentido, el resto en sí puede no estar marcado de manera detectable pero puede volverse detectable tras la reacción con otro resto.
El término "conjugado" se refiere a un anticuerpo que se une químicamente a un segundo resto químico, tal como un agente terapéutico o citotóxico. El término "agente" incluye un compuesto químico, una mezcla de compuestos químicos, una macromolécula biológica o un extracto obtenido a partir de materiales biológicos. En algunas modalidades, los agentes terapéuticos o citotóxicos incluyen, pero sin limitarse a, toxina pertussis, taxol, citocalasina
B, gramicidina D, bromuro de etidio, emetina, mitomicina, etopósido, tenopósido, vincristina, vinblastina, colchicina, doxorrubicina, daunorrubicina, dihidroxi antracenodiona, mitoxantrona, mitramicina, actinomicina D, 1-deshidrotestosterona, glucocorticoides, procaína, tetracaína, lidocaína, propranolol y puromicina y sus análogos u homólogos. Cuando se usa en el contexto de un inmunoensayo, el anticuerpo conjugado puede ser un anticuerpo marcado de manera detectable usado como el anticuerpo de detección.
II. Anticuerpos anti-ICOS
Se proporcionan anticuerpos novedosos dirigidos contra ICOS. Los anticuerpos anti-ICOS incluyen, pero no se limitan a, anticuerpos humanizados, anticuerpos quiméricos, anticuerpos de ratón, anticuerpos humanos y anticuerpos que comprenden las CDR de cadena pesada y/o cadena ligera analizadas en la presente descripción. En algunos casos, se proporciona un anticuerpo aislado que se une a ICOS. En algunos casos, se proporciona un anticuerpo monoclonal que se une a ICOS. En algunos casos, un anticuerpo anti-ICOS es un anticuerpo agonista anti-ICOS. En algunos casos, la administración de los anticuerpos anti-ICOS descritos en la presente aumenta la cantidad de células Tef; activa las células Tef; agota las células Treg en un sujeto; y/o aumenta la relación de las células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunos casos, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, un anticuerpo anti-ICOS comprende al menos una, dos, tres, cuatro, cinco o seis CDR que se seleccionan de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunos casos, un anticuerpo anti-ICOS comprende al menos una, dos, tres, cuatro, cinco o seis CDR que se seleccionan de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En la invención, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63;
(c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66;
y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67.
En algunos casos, un anticuerpo anti-ICOS comprende al menos una, dos, tres, cuatro, cinco o seis CDR que se seleccionan de (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 22, 62,
72, 82, 92, 102 y 112; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO:
23, 63, 73, 83, 93, 103 y 113; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID
NO: 24, 64, 74, 84, 94, 104 y 114; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las
SEQ ID NO: 25, 65, 75, 85, 95, 105 y 115; (e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 26, 66, 76, 86, 96, 106 y 116; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 27, 67, 77, 87, 97, 107 y 117.
En algunos casos, un anticuerpo anti-ICOS comprende al menos una, dos, tres, cuatro, cinco o seis CDR que se seleccionan de (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 32, 162,
172 y 182; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 33, 163, 173 y 183; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 34, 164, 174 y
184; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 35, 165, 175 y 185;
(e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 36, 166, 176 y 186; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 37, 167, 177 y 187.
En algunos casos, un anticuerpo anti-ICOS comprende al menos una, dos, tres, cuatro, cinco o seis CDR que se seleccionan de (a) HCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 52, 122, 132, 142 y 152; (b) HCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 53, 123, 133, 143 y 153; (c) HCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 54, 124, 134, 144 y 154; (d) LCDR1 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 55, 125, 135, 145 y 155; (e) LCDR2 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 56, 126,
136, 146 y 156; y (f) LCDR3 que comprende una secuencia de aminoácidos seleccionada de las SEQ ID NO: 57, 127, 137, 147 y 157.
En algunos casos, un anticuerpo anti-ICOS comprende una región variable de cadena pesada y una región variable de cadena ligera. En algunos casos, un anticuerpo anti-ICOS comprende al menos una cadena pesada que comprende una región variable de cadena pesada y al menos una porción de una región constante de cadena pesada, y al menos una cadena ligera que comprende una región variable de cadena ligera y al menos una porción de una región constante de cadena ligera. En algunos casos, un anticuerpo anti-ICOS comprende dos cadenas pesadas, en donde cada cadena pesada comprende una región variable de cadena pesada y al menos una porción de una región constante de cadena pesada, y dos cadenas ligeras, en donde cada cadena ligera comprende una región variable de cadena ligera y al menos una porción de una región constante de cadena ligera. Como se usa en la presente descripción, un Fv de cadena simple (scFv), o cualquier otro anticuerpo que comprenda, por ejemplo, una sola cadena polipeptídica que comprende las seis CDR (tres CDR de cadena pesada y tres CDR de cadena ligera) se considera que tiene una cadena pesada y una cadena ligera. En algunos casos, la cadena pesada es la región del anticuerpo anti-ICOS que comprende las tres CDR de cadena pesada. En algunos casos, la cadena ligera es la región del anticuerpo anti-ICOS que comprende las tres CDR de cadena ligera.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia
de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 154; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177.
En algunos casos, el anticuerpo anti-ICOS comprende seis CDR que incluyen (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunas modalidades, el anticuerpo anti-ICOS comprende las seis CDR como se describió anteriormente y se une a ICOS. En algunas modalidades, el anticuerpo anti-ICOS comprende las seis CDR como se describió anteriormente, se une a ICOS y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef con respecto a las células Treg en un mamífero, tal como un ser humano. En algunas modalidades, las células Treg son células T CD4+ FoxP3+. En algunas modalidades, las células Tef son células T CD8+. En algunas modalidades, las células Tef son células T CD4+ FoxP3- y/o células T CD8+.
En algunos casos, se proporciona un anticuerpo anti-ICOS que compite con un anticuerpo anti-ICOS descrito en el presente documento para unirse a ICOS. En algunos casos, puede producirse y/o usarse un anticuerpo que compite con cualquiera de los anticuerpos proporcionados en la presente descripción para su unión.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que
comprende la secuencia de aminoácidos de la SEQ ID NO: 13; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54.
En algunas modalidades, el anticuerpo anti-ICOS comprende las tres secuencias de CDR de VH (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 154.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174.
En algunos casos, el anticuerpo anti-ICOS comprende al menos una, al menos dos o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57.
En algunas modalidades, el anticuerpo comprende las tres secuencias de CDR de VL (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL
seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177.
En algunos casos, el anticuerpo comprende al menos una, al menos dos o las tres secuencias de CDR de VL seleccionadas de (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunos casos, cualquiera de las seis CDR proporcionadas en la presente pueden combinarse como subpartes con cualquiera de las otras CDR proporcionadas en la presente descripción, para un total de seis CDR en una construcción. Así, en algunos casos, dos CDR de un primer anticuerpo (por ejemplo, HCDR1 y HCDR2) pueden combinarse con cuatro CDR de un segundo anticuerpo (HCDR3, LCDR1, LCDR2 y LCDR3). En algunos casos, dos o menos residuos en una o más de las CDR pueden reemplazarse para obtener una variante de esta. En algunos casos, dos o menos residuos pueden reemplazarse en 1, 2, 3, 4, 5 o 6 de las CDR.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57.
En la invención, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende las tres secuencias de CDR de VH (a) HCDR1 que comprende la secuencia de aminoácidos de la s Eq ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; y (II) un dominio VL que comprende las tres secuencias de CDR de VL (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la
SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137.
En algunas modalidades, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 154; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende al menos una, al menos dos, o las tres secuencias de CDR de VH seleccionadas de (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184; y (II) un dominio VL que comprende al menos una, al menos dos, o las tres secuencias de CDR de VL seleccionadas de (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunas modalidades, un anticuerpo anti-ICOS comprende una secuencia del dominio variable de cadena pesada (VH) que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % de identidad de secuencia con la secuencia de aminoácidos de la SEQ ID NO: 60. En algunos casos, una secuencia de VH que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 % o 99 % de identidad contiene sustituciones (por
ejemplo, sustituciones conservadoras), inserciones o deleciones con relación a la secuencia de referencia, pero un anticuerpo anti-ICOS que comprende esa secuencia retiene la capacidad de unirse a ICOS. En algunos casos, un total de 1 a 10 aminoácidos se han sustituido, insertado y/o eliminado en las SEQ ID NO: 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 o 180. En algunos casos, las sustituciones, inserciones o deleciones se producen en regiones fuera de las CDR (es decir, en las FR). Opcionalmente, el anticuerpo anti-ICOS comprende la secuencia de VH en la SEQ ID NO: 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 o 180, que incluye modificaciones postraduccionales de esa secuencia.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54.
En la invención, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; y (c) HCDR3 que comprende la
secuencia de aminoácidos de la SEQ ID NO: 154.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164.
En algunos casos, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174.
En algunas modalidades, la VH comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184.
En algunas modalidades, se proporciona un anticuerpo anti-ICOS, en donde el anticuerpo comprende un dominio variable de cadena ligera (Vl ) que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % de identidad de secuencia con la secuencia de aminoácidos de la SEQ ID NO: 61. En algunos casos, una secuencia VL que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 % o 99 % de identidad contiene sustituciones (por ejemplo, sustituciones conservadoras), inserciones o deleciones con relación a la secuencia de referencia, pero un anticuerpo anti-ICOS que comprende esa secuencia retiene la capacidad de unirse a ICOS. En algunos casos, un total de 1 a 10 aminoácidos se han sustituido, insertado y/o eliminado en las SEQ ID NO: 11,21, 31, 41, 51, 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 o 181. En algunos casos, las sustituciones, inserciones o deleciones se producen en regiones fuera de las CDR (es decir, en las FR). Opcionalmente, el anticuerpo anti-ICOS comprende la secuencia VL en las SEQ ID NO: 11, 21, 31, 41, 51, 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 o 181, que incluye modificaciones postraduccionales de esa secuencia.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57.
En la invención, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177.
En algunos casos, la VL comprende: (a) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (b) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (c) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunas modalidades, un anticuerpo anti-ICOS comprende una secuencia del dominio variable de cadena pesada (VH) que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % de identidad de secuencia con la secuencia de aminoácidos de la SEQ ID NO: 60 y un dominio variable de cadena ligera (VL) que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 %, 99 % o 100 % de identidad de secuencia con la secuencia de aminoácidos de la SEQ ID NO: 61. En algunos casos, una secuencia de VH que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 % o 99 % de identidad contiene sustituciones (por ejemplo, sustituciones conservadoras), inserciones o deleciones con relación a la secuencia de referencia, y una secuencia de VL que tiene al menos 90 %, 91 %, 92 %, 93 %, 94 %, 95 %, 96 %, 97 %, 98 % o 99 % de identidad contiene sustituciones (por ejemplo, sustituciones conservadoras), inserciones o deleciones con relación a la secuencia de referencia, pero un anticuerpo anti-ICOS que comprende esa secuencia retiene la capacidad de unirse a ICOS. En algunos casos, un total de 1 a 10 aminoácidos se han sustituido, insertado y/o eliminado en las SEQ ID NO: 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 o 180. En algunos casos, un total de 1 a 10 aminoácidos se han sustituido, insertado y/o eliminado en la Se Q ID NO: 11,21,31,41,51,61,71,81,91, 101, 111, 121, 131, 141, 151, 161, 171 o 181. En algunos casos, las sustituciones, inserciones o deleciones se producen en regiones fuera de las CDR (es decir, en las FR). Opcionalmente, el anticuerpo anti-ICOS comprende la secuencia VH en las SEQ ID NO: 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 o 180 y la secuencia VL de las SEQ ID NO: 11,21,31,41,51,61,71,81,91,101,111,121,131,141,151, 161, 171 o 181, que incluyen modificaciones postraduccionales de una o ambas secuencias.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 12; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 13; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 14; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 15; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 16; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 17.
En algunas modalidades, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 22; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 23; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 24; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 25; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 26; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 27.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende
la secuencia de aminoácidos de la SEQ ID NO: 32; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 33; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 34; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 35; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 36; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 37.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 42; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 43; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 44; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 45; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 46; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 47.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 52; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 53; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 54; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 55; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 56; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 57.
En la invención, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 72; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 73; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 74; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 75; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 76; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 77.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 82; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 83; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 84; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 85; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 86; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 87.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 92; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 93; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 94; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 95; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 96; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 97.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 102; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 103; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 104; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 105; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 106; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 107.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 112; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 113; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 114; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 115; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 116; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 117.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 122; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 123; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 124; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 125; (e) LCDR2 que
comprende la secuencia de aminoácidos de la SEQ ID NO: 126; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 127.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 132; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 133; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 134; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 135; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 136; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 137.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 142; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 143; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 144; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 145; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 146; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 147.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 152; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 153; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 154; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 155; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 156; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 157.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 162; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 163; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 164; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 165; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 166; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 167.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 172; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 173; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 174; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 175; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 176; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 177.
En algunos casos, el anticuerpo anti-ICOS comprende (I) un dominio VH que comprende: (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 182; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 183; y (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 184; y (II) un dominio VL que comprende: (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 185; (e) LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 186; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 187.
En algunos casos, un anticuerpo anti-ICOS comprende una VH como en cualquiera de las modalidades proporcionadas en la presente descripción, y una VL como en cualquiera de las modalidades proporcionadas en la presente descripción. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 10 y la SEQ ID NO: 11, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la s Eq ID NO: 20 y la SEQ ID NO: 21, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 30 y la SEQ ID NO: 31, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 40 y la SEQ ID NO: 41, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 50 y la SEQ ID NO: 51, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En la invención, el anticuerpo comprende las secuencias VH y VL en la s Eq ID NO: 60 y la SEQ ID NO: 61, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 70 y la SEQ ID NO: 71, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 80 y la SEQ ID NO: 81, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 90 y la SEQ ID NO: 91, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 100 y la SEQ ID NO: 101, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 110 y la SEQ ID NO: 111, respectivamente, que incluyen modificaciones postraduccionales de esas
secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 120 y la SEQ ID NO: 121, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la s Eq ID NO: 130 y la SEQ ID NO: 131, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 140 y la SEQ ID NO: 141, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 150 y la SEQ ID NO: 151, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 160 y la SEQ ID NO: 161, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la s Eq ID NO: 170 y la SEQ ID NO: 171, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias. En algunos casos, el anticuerpo comprende las secuencias VH y VL en la SEQ ID NO: 180 y la SEQ ID NO: 181, respectivamente, que incluyen modificaciones postraduccionales de esas secuencias.
En algunos casos, se proporcionan anticuerpos que compiten con los anticuerpos anti-ICOS proporcionados en la presente descripción para unirse a ICOS. En algunos casos, los anticuerpos compiten con los anticuerpos anti-ICOS proporcionados en la presente descripción para unirse a un epítopo en ICOS.
En algunos casos, los ensayos de competencia pueden usarse para identificar un anticuerpo monoclonal que compite con un anticuerpo anti-ICOs descrito en el presente documento (tal como 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 o 35A9S89) para unirse a ICOS. Los ensayos de competencia pueden usarse para determinar si dos anticuerpos se unen al mismo epítopo mediante el reconocimiento de epítopos idénticos o solapados estéricamente o un anticuerpo inhibe de manera competitiva la unión de otro anticuerpo al antígeno. En algunos casos, un anticuerpo competidor de este tipo se une al mismo epítopo que se une un anticuerpo descrito en el presente documento. Los ensayos de competencia ilustrativos incluyen, pero sin limitarse a, ensayos de rutina tales como los proporcionados en Harlow y Lane (1988) Antibodies: A Laboratory Manual cap.14 (Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y.). Métodos ilustrativos detallados para mapear un epítopo al cual se une un anticuerpo se pueden encontrar en Morris (1996) "Epitope Mapping Protocols," en Methods in Molecular Biology vol. 66 (Humana Press, Totowa, N.J.). En algunos casos, se dice que dos anticuerpos se unen al mismo epítopo si cada uno bloquea la unión del otro en un 50 % o más. En algunos casos, el anticuerpo que compite con un anticuerpo anti-ICOS descrito en el presente documento es un anticuerpo quimérico, humanizado o humano. En algunos casos, se proporciona un anticuerpo que compite con un anticuerpo anti-ICOS quimérico, humanizado o humano como se describe en el presente documento.
En algunos casos, se proporcionan anticuerpos que se unen a cualquiera de uno o más de los epítopos que los anticuerpos proporcionados en la presente descripción. En algunos casos, se proporcionan anticuerpos que se unen y se superponen a un epítopo al que se unen los anticuerpos de la presente. En algunos casos, se proporciona un anticuerpo que compite con al menos uno de los anticuerpos proporcionados en la presente descripción. En algunos casos, se proporciona un anticuerpo que compite con al menos dos de los anticuerpos proporcionados en la presente descripción. En algunos casos, se proporciona un anticuerpo que compite con al menos tres de los anticuerpos proporcionados en la presente descripción. En algunos casos, el anticuerpo se une a un epítopo superpuesto como un anticuerpo descrito en los ejemplos en la presente descripción. En algunos casos, el anticuerpo competidor se une y/u obstruye todo el epítopo. En algunos casos, el anticuerpo competidor se une y/u obstruye una parte del epítopo. En algunos casos, el paratopo del anticuerpo competidor se une al menos a una parte del epítopo de un anticuerpo proporcionado en la presente descripción. En algunos casos, el paratopo del anticuerpo competidor se une al objetivo, y una sección diferente de la estructura del anticuerpo competidor obstruye al menos una parte del epítopo de un anticuerpo proporcionado en la presente descripción.
Anticuerpos quiméricos ilustrativos
En algunas modalidades, un anticuerpo proporcionado en la presente descripción es un anticuerpo quimérico. Determinados anticuerpos quiméricos se describen, por ejemplo, en la patente de Estados Unidos núm. 4,816,567 y Morrison y otros, (1984) Proc. Natl. Acad. Sci. USA, 81:6851-6855 (1984)). En un ejemplo, un anticuerpo quimérico comprende una región variable no humana (por ejemplo, una región variable derivada de un ratón, rata, hámster, conejo o primate no humano, como un mono) y una región constante humana. En un ejemplo adicional, un anticuerpo quimérico es un anticuerpo "con cambio de clase" en el que la clase o subclase se ha cambiado con respecto a la del anticuerpo original. Los anticuerpos quiméricos incluyen sus fragmentos de unión a antígeno.
Los anticuerpos quiméricos ilustrativos no limitantes incluyen anticuerpos quiméricos que comprenden las regiones variables de cadena pesada y/o ligera de un anticuerpo que se selecciona de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 o 35A9S89. Los anticuerpos quiméricos ilustrativos adicionales incluyen anticuerpos quiméricos que comprenden CDR1, CDR2 y CDR3 de cadena pesada, y/o CDR1, CDR2 y CDR3 de cadena ligera de un anticuerpo seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 o
35A9S89. Otros anticuerpos quiméricos ilustrativos no limitantes incluyen anticuerpos quiméricos que comprenden CDR1, CDR2 y CDR3 de cadena pesada, y/o CDR1, CDR2 y CDR3 de cadena ligera de un anticuerpo seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 o 35A9S89. En algunos casos, el anticuerpo anti-ICOS quimérico comprende las regiones variables descritas anteriormente y se une a ICOS. En algunos casos, el anticuerpo anti-ICOS quimérico comprende las regiones variables descritas anteriormente, se une a ICOS, y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunos casos, las células Tef son células T CD4+ FoxP3-. En algunos casos, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, un anticuerpo anti-ICOS quimérico comprende una cadena pesada que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia que se selecciona de las SEQ ID NO: 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 o 180, en donde el anticuerpo se une a ICOS. En algunos casos, un anticuerpo anti-ICOS quimérico comprende una cadena ligera que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia seleccionada de las SEQ ID NO: 11, 21, 31, 41, 51, 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 o 181, en donde el anticuerpo se une a ICOS. En algunas modalidades, un anticuerpo anti-ICOS quimérico comprende una cadena pesada que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia que se selecciona de las SEQ ID NO: 60; y una cadena ligera que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia seleccionada de las SEQ ID NO: 61; en donde el anticuerpo se une a ICOS.
Los anticuerpos anti-ICOS quiméricos ilustrativos incluyen, además, anticuerpos quiméricos que compiten por la unión a ICOS con un anticuerpo o fragmento de este descrito en el presente documento. Por lo tanto, en algunos casos, se proporciona un anticuerpo anti-ICOS quimérico que compite por la unión a ICOS con un anticuerpo seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89 o un fragmento de estos. En algunos casos, el anticuerpo compite por unirse a ICOS y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunos casos, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, un anticuerpo quimérico descrito en el presente documento comprende una o más regiones constantes humanas. En algunos casos, la región constante de cadena pesada humana es de un isotipo seleccionado de IgA, IgG e IgD. En algunos casos, la región constante de cadena ligera humana es de un isotipo seleccionado de k y A. En algunas modalidades, un anticuerpo quimérico descrito en el presente documento comprende una región constante de IgG1 humana. En algunos casos, un anticuerpo quimérico descrito en el presente documento comprende una región constante de cadena pesada de IgG4 humana. En algunos casos, un anticuerpo quimérico descrito en el presente documento comprende una región constante de IgG4 humana y una cadena ligera k humana.
Como se señaló anteriormente, si la función efectora es conveniente o no puede depender del método particular de tratamiento previsto para un anticuerpo. Por lo tanto, en algunos casos, cuando la función efectora es conveniente, se selecciona un anticuerpo anti-ICOS quimérico que comprende una región constante de cadena pesada IgG1 humana o una región constante de cadena pesada IgG3 humana. En algunas modalidades, cuando la función efectora no es conveniente, se selecciona un anticuerpo anti-ICOS quimérico que comprende una región constante de cadena pesada de IgG4 o IgG2 humana.
Anticuerpos humanizados ilustrativos
En algunas modalidades, se proporcionan anticuerpos humanizados que se unen a ICOS. Los anticuerpos humanizados son útiles como moléculas terapéuticas porque los anticuerpos humanizados reducen o eliminan la respuesta inmunitaria humana en comparación con los anticuerpos no humanos, lo que puede resultar en una respuesta inmunitaria contra un agente terapéutico (como la respuesta de anticuerpos anti-ratón humanos (HAMA)), y una disminución de la eficacia del agente terapéutico.
En algunas modalidades, un anticuerpo quimérico es un anticuerpo humanizado. Típicamente, un anticuerpo no humano se humaniza para reducir la inmunogenicidad para los seres humanos, a la vez que retiene la especificidad y la afinidad del anticuerpo no humano original. Generalmente, un anticuerpo humanizado comprende uno o más dominios variables en los que las CDR, (o porciones de estas) se derivan de un anticuerpo no humano, y las FR (o porciones de estas) se derivan de secuencias de anticuerpos humanos. Opcionalmente, un anticuerpo humanizado comprenderá, además, al menos una porción de una región constante humana. En algunas modalidades, algunos
residuos de FR en un anticuerpo humanizado se sustituyen con residuos correspondientes de un anticuerpo no humano (por ejemplo, el anticuerpo a partir del cual se derivan los residuos de CDR), por ejemplo, para restaurar o mejorar la especificidad o afinidad de los anticuerpos.
Los anticuerpos humanizados y los métodos para obtenerlos se revisan, por ejemplo, en Almagro y Fransson, (2008) Front. Biosci. 13: 1619-1633, y se describen además, por ejemplo, en Riechmann y otros, (1988) Nature 332:323-329; Queen y otros, (1989) Proc. Natl Acad. Sci. USA 86: 10029-10033; patentes de Estados Unidos núm. 5, 821,337, 7,527,791, 6,982,321 y 7,087,409; Kashmiri y otros, (2005) Methods 36:25-34; Padlan, (1991) Mol. Immunol. 28:489-498 (que describe la "remodelación de la superficie"); Dall'Acqua y otros, (2005) Methods 36:43-60 (que describe el "barajado de FR"); y Osbourn y otros, (2005) Methods 36:61-68 y Klimka y otros, (2000) Br. J. Cancer, 83:252-260 (que describe el enfoque de "selección guiada" para el barajado de FR).
Las regiones del marco humanas que pueden usarse para la humanización incluyen pero no se limitan a: regiones del marco seleccionadas con el uso del método de "mejor ajuste" (ver, por ejemplo, Sims y otros (1993) J. Immunol.
151:2296); regiones del marco derivadas de la secuencia consenso de anticuerpos humanos de un subgrupo particular de regiones variables de cadena pesada y ligera (ver, por ejemplo, Carter y otros (1992) Proc. Natl. Acad. Sci. USA, 89:4285; y Presta y otros (1993) J. Immunol, 151:2623); regiones del marco maduras humanas (mutadas somáticamente) o regiones del marco de la línea germinal humana (ver, por ejemplo, Almagro y Fransson, (2008) Front. Biosci. 13:1619-1633); y regiones del marco derivadas del tamizaje de genotecas de FR (ver, por ejemplo, Baca y otros, (1997) J. Biol. Chem. 272: 10678-10684 y Rosok y otros, (1996) J. Biol. Chem. 271:22611-22618).
Los anticuerpos humanizados ilustrativos no limitantes incluyen 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89, descritos en la presente. Los anticuerpos humanizados ilustrativos no limitantes incluyen, además, anticuerpos que comprenden una región variable de cadena pesada de un anticuerpo seleccionado de 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89 y/o una región variable de cadena ligera de un anticuerpo seleccionado de 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89. Los anticuerpos humanizados ilustrativos no limitantes incluyen anticuerpos que comprenden una región variable de cadena pesada seleccionada de las SEQ ID NO: 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 y 180 y/o una región variable de cadena ligera seleccionada de las SEQ ID NO: 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 y 181. Los anticuerpos humanizados ilustrativos incluyen, además, pero sin limitarse a, anticuerpos humanizados que comprenden CDR1, CDR2 y CDR3 de cadena pesada, y/o CDR1, CDR2 y CDR3 de cadena ligera de un anticuerpo seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89. En algunos casos, el anticuerpo anti-ICOS humanizado comprende las CDR descritas anteriormente y se une a ICOS. En algunos casos, el anticuerpo anti-ICOS humanizado comprende las CDR descritas anteriormente, se une a ICOS y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunas modalidades, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, un anticuerpo anti-ICOS humanizado comprende una CDR1, CDR2 y CDR3 de cadena pesada y/o una CDR1, CDR2 y CDR3 de cadena ligera de un anticuerpo seleccionado de 7F12, 37A10, 35A9, 36E10, 16g 10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89.
En algunos casos, un anticuerpo anti-ICOS humanizado comprende una cadena pesada que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia que se selecciona de las SEQ ID NO: 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170 y 180 y en donde el anticuerpo se une a ICOS. En algunos casos, un anticuerpo anti-ICOS humanizado comprende una cadena ligera que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia que se selecciona de las SEQ ID NO: 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 y 181, en donde el anticuerpo se une a ICOS. En algunas modalidades, un anticuerpo anti-ICOS humanizado comprende una cadena pesada que comprende una secuencia de región variable que es al menos 90%, al menos 91%, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia seleccionada de la SEQ ID NO: 60; y una cadena ligera que comprende una secuencia de región variable que es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia seleccionada de la SEQ ID NO: 61; en donde el anticuerpo se une a ICOS.
En algunos casos, se mantienen una o más de las secuencias de CDR proporcionadas en la presente descripción, mientras que la región restante de cadena pesada, ligera o pesada y ligera (es decir, FR1, f R2, FR3 y FR4) es al menos 90 %, al menos 91 %, al menos 92 %, al menos 93 %, al menos 94 %, al menos 95 %, al menos 96 %, al
menos 97 %, al menos 98 % o al menos 99 % idéntica a una secuencia seleccionada de las SEQ ID NO: 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 11, 21, 31,41, 51, 61, 71, 81, 91, 101, 111, 121, 131, 141, 151, 161, 171 y 181.
En algunos casos, un anticuerpo anti-ICOS humanizado comprende al menos una de las CDR descritas en la presente descripción. Es decir, en algunos casos, un anticuerpo anti-ICOS humanizado comprende al menos una CDR seleccionada de una CDR1 de cadena pesada analizada en este documento, una CDR2 de cadena pesada analizada en este documento, una CDR3 de cadena pesada analizada en este documento, una CDR1 de cadena ligera analizada en este documento, una CDR2 de cadena ligera analizada en este documento, y una CDR3 de cadena ligera analizada en este documento. Además, en algunos casos, un anticuerpo anti-ICOS humanizado comprende al menos una CDR mutada basada en una CDR analizada en la presente descripción, en donde la CDR mutada comprende 1, 2, 3 o 4 sustituciones de aminoácidos con relación a la CDR analizada en la presente descripción. En algunos casos, una o más de las sustituciones de aminoácidos son sustituciones de aminoácidos conservadoras. Un experto en la técnica puede seleccionar una o más sustituciones de aminoácidos conservadoras adecuadas para una secuencia de CDR particular, en donde las sustituciones conservadoras adecuadas de aminoácidos no están destinadas a alterar significativamente las propiedades de unión del anticuerpo que comprende la CDR mutada.
Los anticuerpos anti-ICOS humanizados ilustrativos incluyen, además, anticuerpos que compiten por la unión a ICOS con un anticuerpo o fragmento de este descrito en el presente documento. Por lo tanto, en algunos casos, se proporciona un anticuerpo anti-ICOS humanizado que compite por la unión a ICOS con un anticuerpo o fragmento de este seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89. En algunos casos, se proporciona un anticuerpo anti-ICOS humanizado que compite por la unión a ICOS con un anticuerpo o fragmento de este seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89 y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef a células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunas modalidades, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunas modalidades, un anticuerpo anti-ICOS humanizado comprende una cadena pesada que comprende la secuencia de aminoácidos de la SEQ ID NO: 188 y una cadena ligera que comprende la secuencia de aminoácidos de la SEQ ID NO: 189.
Anticuerpos humanos ilustrativos
En algunas modalidades, un anticuerpo anti-ICOS proporcionado en la presente es un anticuerpo humano. Los anticuerpos humanos pueden producirse mediante el uso de diversas técnicas conocidas en la materia. Los anticuerpos humanos se describen generalmente en van Dijk y van de Winkel, (2001) Curr. Opin. Pharmacol. 5:368-374 y Lonberg, (2008) Curr. Opin. Inmunol. 20:450-459. En algunas modalidades, el anticuerpo humano no es un anticuerpo de origen natural. En algunas modalidades, el anticuerpo humano es un anticuerpo monoclonal; por lo tanto, en algunas modalidades, cada uno de los anticuerpos humanos en un conjunto puede unirse al mismo epítopo en el antígeno.
Los anticuerpos humanos pueden prepararse mediante la administración de un inmunógeno a un animal transgénico que se ha modificado para producir anticuerpos humanos intactos o anticuerpos intactos con regiones variables humanas en respuesta a la estimulación antigénica. Tales animales contienen típicamente toda o una porción de los loci de inmunoglobulinas humanas, que remplazan los loci endógenos de inmunoglobulinas, o que están presentes de manera extracromosómica o integrada aleatoriamente en los cromosomas del animal. En tales ratones transgénicos, los loci endógenos de inmunoglobulinas se han inactivado generalmente. Para la revisión de métodos para obtener anticuerpos humanos a partir de animales transgénicos ver Lonberg, (2005) Nat. Biotech. 23: 1117-1125. Véase además, por ejemplo, las patentes de Estados Unidos núms. 6,075,181 y 6,150,584 que describen la tecnología XENOMOUSE™; la patente de Estados Unidos núm. 5,770,429 que describe la tecnología HUMAB® la patente de Estados Unidos núm. 7,041,870 que describe la tecnología K-M MOUSE®, y la publicación de solicitud de patente de Estados Unidos núm. US 2007/0061900, que describe la tecnología VELo C iMOUSE®). Las regiones variables humanas de anticuerpos intactos generados por tales animales pueden modificarse adicionalmente, por ejemplo, combinándose con una región constante humana diferente.
Los anticuerpos humanos pueden producirse, además, mediante métodos basados en hibridomas. Se han descrito líneas celulares de mieloma humano y heteromieloma humano-ratón para la producción de anticuerpos monoclonales humanos. (Ver, por ejemplo, Kozbor (1984) J. Immunol, 133: 3001; Brodeur y otros, Monoclonal Antibody Production Techniques and Applications, pp. 51-63 (Marcel Dekker, Inc., Nueva York, 1987); y Boerner y otros, (1991) J. Immunol., 147:86). Los anticuerpos humanos generados por medio de la tecnología de hibridoma de células B humanas se describen, además, en Li y otros, (2006) Proc. Natl. Acad. Sci. USA, 103:3557-3562. Otros métodos incluyen los descritos, por ejemplo, en la patente de Estados Unidos núm. 7,189,826 (que describe la producción de anticuerpos monoclonales IgM humanos a partir de líneas celulares de hibridoma) y Ni, (2006) Xiandai Mianyixue, 26(4):265-268
(que describe hibridomas humano-humano). La tecnología de hibridomas humanos (tecnología de Triomas) se describe además en Vollmers y Brandlein, (2005) Histology and Histopatology, 20(3):927-937 (2005) y Vollmers and Brandlein, (2005) Methods and Findings in Experimental and Clinical Pharmacology, 27(3): 185-191.
Los anticuerpos humanos pueden generarse, además, mediante el aislamiento de secuencias del dominio variable de clones de Fv, seleccionadas de genotecas de presentación en fagos derivadas de humanos. Tales secuencias del dominio variable pueden combinarse después con un dominio constante humano deseado. A continuación se describen técnicas para seleccionar anticuerpos humanos a partir de genotecas de anticuerpos.
Los anticuerpos pueden aislarse mediante análisis de genotecas combinatorias para anticuerpos con la actividad o actividades deseadas. Por ejemplo, en la técnica se conoce una variedad de métodos para generar genotecas de presentación en fagos y tamizar tales genotecas para anticuerpos que poseen las características de unión deseadas. Tales métodos se revisan, por ejemplo, en Hoogenboom y otros, en Methods in Molecular Biology 178: 1-37 (O'Brien y otros, ed., Human Press, Totowa, NJ, 2001) y se describen además, por ejemplo, en McCafferty y otros, (1990) Nature 348:552-554; Clackson y otros, (1991) Nature 352: 624-628; Marks y otros, (1992) J. Mol. Biol 222: 581-597; Marks y Bradbury, en Methods in Molecular Biology 248: 161-175 (Lo, ed., Human Press, Totowa, NJ, 2003); Sidhu y otros, (2004) J. Mol. Biol. 338(2): 299-310; Lee y otros, (2004) J. Mol. Biol. 340(5): 1073-1093; Fellouse, (2004) Proc. Natl. Acad. Sci. USA 101(34): 12467-12472; y Lee y otros, (2004) J. Immunol. Methods 284(1-2): 119-132 y la publicación PCT WO 99/10494.
En determinados métodos de presentación en fagos, los repertorios de los genes VH y VL se clonan por separado por reacción en cadena de la polimerasa (PCR) y se recombinan aleatoriamente en genotecas de fagos, que después pueden analizarse en busca de un fago con unión al antígeno como se describe en Winter y otros, (1994) Ann. Rev. Immunol, 12:433-455. Los fagos típicamente muestran fragmentos de anticuerpo, ya sea como fragmentos Fv de cadena simple (scFv) o como fragmentos Fab. Las genotecas de fuentes inmunizadas proporcionan anticuerpos de alta afinidad contra el inmunógeno sin el requisito de construir hibridomas. De manera alternativa, el repertorio virgen puede clonarse (por ejemplo, de ser humano) para proporcionar una única fuente de anticuerpos a una amplia variedad de antígenos no propios y además autoantígenos sin ninguna inmunización según lo descrito por Griffiths y otros, (1993) EMBO J 12:725-734. Finalmente, las genotecas vírgenes pueden hacerse, además, sintéticamente mediante la clonación de segmentos génicos V no reorganizados de células madre, y mediante el uso de cebadores de PCR que contienen secuencia aleatoria para codificar las regiones CDR3 altamente variables y para lograr la reorganización in vitro, como se describe por Hoogenboom y Winter (1992), J. Mol. Biol, 227:381-388. Las publicaciones de patentes que describen genotecas de fagos de anticuerpos humanos incluyen, por ejemplo: la patente de Estados Unidos núm.
5,750,373 y las publicaciones de patente de Estados Unidos núm. 2005/0079574, 2005/0119455, 2005/0266000, 2007/0117126, 2007/0160598, 2007/0237764, 2007/0292936 y 2009/0002360.
En algunos casos, un anticuerpo anti-ICOS humano se une a un polipéptido que tiene la secuencia de la SEQ ID NO: 1 o 2. En algunos casos, el anticuerpo anti-ICOS humano se une a ICOS y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunos casos, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
Los anticuerpos anti-ICOS humanos ilustrativos incluyen, además, anticuerpos que compiten por la unión a ICOS con un anticuerpo humano o fragmento de este descrito en el presente documento. Por lo tanto, en algunos casos, se proporciona un anticuerpo anti-ICOS humano que compite por la unión a ICOS con un anticuerpo o fragmento de este seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89. En algunos casos, se proporciona un anticuerpo anti-ICOS humano que compite por la unión a ICOS con un anticuerpo o fragmento de este seleccionado de 7F12, 37A10, 35A9, 36E10, 16G10, 37A10S713, 37A10S714, 37A10S715, 37A10S716, 37A10S717, 37A10S718, 16G10S71, 16G10S72, 16G10S73, 16G10S83, 35A9S79, 35A9S710 y 35A9S89 y aumenta la cantidad de células Tef y/o activa las células Tef y/o disminuye la cantidad de células Treg y/o aumenta la relación de células Tef a células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunos casos, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, se proporciona un anticuerpo anti-ICOS humano quimérico, donde el anticuerpo comprende la región variable de un anticuerpo humano que se une a ICOS y la región constante de un anticuerpo humano diferente. En algunos casos, se proporciona un anticuerpo anti-ICOS humano quimérico, donde el anticuerpo comprende las CDR de un anticuerpo humano que se une a ICOS y un marco de un anticuerpo humano diferente. En algunos casos, el anticuerpo no es un anticuerpo humano de origen natural.
En algunos casos, un anticuerpo anti-ICOS humano comprende una o más regiones constantes humanas. En algunos casos, la región constante de cadena pesada humana es de un isotipo seleccionado de IgA, IgG e IgD. En algunos casos, la región constante de cadena ligera humana es de un isotipo seleccionado de k y A. En algunas modalidades, un anticuerpo humano descrito en el presente documento comprende una región constante de IgG1 humana. En algunos casos, un anticuerpo humano descrito en el presente documento comprende una región constante de cadena pesada de IgG4 humana. En algunos casos, un anticuerpo humano descrito en el presente documento comprende
una región constante de IgG4 humana y una cadena ligera k humana.
En algunos casos, cuando la función efectora es conveniente, se selecciona un anticuerpo anti-ICOS humano que comprende una región constante de cadena pesada IgG1 humana o una región constante de cadena pesada IgG3 humana. En algunos casos, cuando la función efectora no es conveniente, se selecciona un anticuerpo anti-ICOS humano que comprende una región constante de cadena pesada IgG4 o IgG2 humana.
Como se señala en la presente descripción, el término "anticuerpo humano" denota el género de las secuencias posibles para la construcción del anticuerpo, en lugar de una fuente del anticuerpo.
Regiones constantes de anticuerpo ilustrativas
En algunos casos, un anticuerpo descrito en el presente documento comprende una o más regiones constantes humanas. En algunos casos, la región constante de cadena pesada humana es de un isotipo seleccionado de IgA, IgG e IgD. En algunos casos, la región constante de cadena ligera humana es de un isotipo seleccionado de k y A. En algunas modalidades, un anticuerpo descrito en el presente documento comprende una región constante de IgG1 humana. En algunos casos, un anticuerpo descrito en el presente documento comprende una región constante de cadena pesada de IgG4 humana. En algunos casos, un anticuerpo descrito en el presente documento comprende una región constante de IgG4 humana y una cadena ligera k humana.
A lo largo de la presente descripción y las reivindicaciones a menos que se indique explícitamente o sea conocido por un experto en la técnica, la numeración de los residuos en una cadena pesada de inmunoglobulina es la del índice de EU como en Kabat y otros, Sequences of Proteins of Immunological Interest, 5ta Ed. Public Health Service, Instituto Nacional de Salud, Bethesda, Md. (1991), incorporado expresamente en la presente descripción como referencia. El "índice de EU como en Kabat" se refiere a la numeración de residuos del anticuerpo IgG1 EU humano.
Como se señaló anteriormente, si la función efectora es conveniente o no puede depender del método particular de tratamiento previsto para un anticuerpo. Por lo tanto, en algunas modalidades, cuando la función efectora es conveniente, se selecciona un anticuerpo anti-ICOS que comprende una región constante de cadena pesada IgG1 humana o una región constante de cadena pesada IgG3 humana. En algunos casos, cuando la función efectora no es conveniente, se selecciona un anticuerpo anti-ICOS que comprende una región constante de cadena pesada IgG4 o IgG2 humana.
En algunos casos, un anticuerpo comprende una variante de región Fc que tiene al menos una sustitución de aminoácidos en comparación con la región Fc de una IgG natural o un anticuerpo natural. En algunos casos, la variante de la región Fc tiene dos o más sustituciones de aminoácidos en la región Fc del anticuerpo natural. En algunos casos, la variante de la región Fc tiene tres o más sustituciones de aminoácidos en la región Fc del anticuerpo natural. En algunos casos, la variante de la región Fc tiene al menos una, dos o tres o más sustituciones de aminoácidos de la región Fc, descritas en la presente descripción. En algunos casos, la variante de la región Fc en la presente descripción poseerá al menos aproximadamente 80 % de homología con una región Fc de secuencia nativa y/o con una región Fc de un polipéptido original. En algunos casos, la variante de la región Fc en la presente descripción poseerá al menos aproximadamente 90 % de homología con una región Fc de secuencia nativa y/o con una región Fc de un polipéptido original. En algunos casos, la variante de la región Fc en la presente descripción poseerá al menos aproximadamente 95 % de homología con una región Fc de secuencia nativa y/o con una región Fc de un polipéptido original.
En algunos casos, un anticuerpo proporcionado en la presente se modifica para aumentar o disminuir el grado en que el anticuerpo se glicosila. La adición o eliminación de sitios de glicosilación a un anticuerpo puede lograrse convenientemente modificando la secuencia de aminoácidos de manera que se crean o se eliminan uno o más sitios de glicosilación.
Cuando el anticuerpo comprende una región Fc, el carbohidrato unido a este puede modificarse. Los anticuerpos nativos producidos por células de mamífero comprenden típicamente un oligosacárido biantenario ramificado que se une generalmente por un enlace N a Asn297 del dominio CH2 de la región Fc. Véase, por ejemplo, Wright y otros TIBTECH 15:26-32 (1997). El oligosacárido puede incluir varios carbohidratos, por ejemplo, manosa, N-acetil glucosamina (GlcNAc), galactosa y ácido siálico, así como también una fucosa unida a una GlcNAc en el "tallo" de la estructura del oligosacárido biantenario. En algunos casos, las modificaciones del oligosacárido en un anticuerpo pueden realizarse para crear variantes de anticuerpos con determinadas propiedades mejoradas.
En algunos casos, se proporcionan variantes de anticuerpos que tienen una estructura de carbohidrato que carece de fucosa unida (directa o indirectamente) a una región Fc. Por ejemplo, la cantidad de fucosa en tal anticuerpo puede ser de 1 % a 80 %, de 1 % a 65 %, de 5 % a 65 % o de 20 % a 40 %. La cantidad de fucosa se determina mediante el cálculo de la cantidad promedio de fucosa dentro de la cadena de azúcar en Asn297, en relación con la suma de todas las glicoestructuras unidas a Asn 297 (por ejemplo, estructuras complejas, híbridas y con alto contenido de manosa) según se mide mediante espectrometría de masa MALDI-TOF, como se describe en el documento WO 2008/077546, por ejemplo. Asn297 se refiere al residuo de asparagina ubicado en aproximadamente la posición 297 en la región Fc (numeración de EU de los residuos de la región Fc); sin embargo, Asn297 también puede ubicarse aproximadamente
± 3 aminoácidos corriente arriba o corriente abajo de la posición 297, es decir, entre las posiciones 294 y 300, debido a variaciones menores de secuencia en los anticuerpos. Tales variantes de fucosilación pueden tener una mejor función de ADCC. Véase, por ejemplo, las publicaciones de patentes de Estados Unidos núm. US 2003/0157108 (Presta, L.); US 2004/0093621 (Kyowa Hakko Kogyo Co., Ltd). Los ejemplos de publicaciones relacionadas con variantes de anticuerpos "desfucosiladas" o "deficientes de fucosa" incluyen: US 2003/0157108; WO 2000/61739; WO 2001/29246; US 2003/0115614; US 2002/0164328; US 2004/0093621; US 2004/0132140; US 2004/0110704; US 2004/0110282; US 2004/0109865; WO 2003/085119; WO 2003/084570; WO 2005/035586; WO 2005/035778; WO2005/053742; WO2002/031140; Okazaki y otros, J. Mol. Biol. 336:1239-1249 (2004); Yamane-Ohnuki y otros Biotech. Bioeng. 87: 614 (2004). Los ejemplos de líneas celulares capaces de producir anticuerpos desfucosilados incluyen células CHO Lec13 deficientes en la fucosilación de proteínas (Ripka y otros, Arch. Biochem. Biophys.
249:533-545 (1986); solicitud de patente de Estados Unidos núm. US 2003/0157108 A1, Presta, L; y el documento WO 2004/056312 A1, Adams y otros,especialmente en el Ejemplo 11) y líneas celulares con genes desactivados, como células CHO con genes desactivados, gen de alfa- 1,6-fucosiltransferasa, FUT8 (véase, por ejemplo, Yamane-Ohnuki y otros, Biotech. Bioeng. 87: 614 (2004); Kanda, Y. y otros, Biotechnol. Bioeng., 94(4):680-688 (2006); y documento WO2003/085107).
Las variantes de anticuerpos se proporcionan además con oligosacáridos bisecados, por ejemplo, en los cuales un oligosacárido biantenario unido a la región Fc del anticuerpo está bisecado por GlcNAc. Tales variantes de anticuerpos pueden tener una fucosilación reducida y/o una mejor función de ADCC. Los ejemplos de tales variantes de anticuerpos se describen, por ejemplo, en el documento WO 2003/011878 (Jean-Mairet y otros); patente de Estados Unidos núm. 6,602,684 (Umana y otros); y US 2005/0123546 (Umana y otros). Se proporcionan, además, variantes de anticuerpo con al menos un residuo de galactosa en el oligosacárido unido a la región Fc. Tales variantes de anticuerpos pueden tener una mejor función CDC. Tales variantes de anticuerpos se describen, por ejemplo, en el documento WO 1997/30087 (Patel y otros); WO 1998/58964 (Raju, S.); y WO 1999/22764 (Raju, S.).
Las variantes de anticuerpos se proporcionan, además, con extensiones de líder amino-terminal. Por ejemplo, uno o más residuos de aminoácidos de la secuencia líder amino terminal están presentes en el extremo amino terminal de cualquiera de una o más cadenas pesadas o ligeras de un anticuerpo. Una extensión de líder amino terminal ilustrativa comprende o consiste en tres residuos de aminoácidos, VHS, presentes en una o ambas cadenas ligeras de una variante de anticuerpo.
El tiempo de vida media in vivo o en suero de polipéptidos de unión a FcRn humano de alta afinidad pueden analizarse, por ejemplo, en ratones transgénicos, en seres humanos o en primates no humanos a los que se administran los polipéptidos con una variante de región Fc. Véase además, por ejemplo, Petkova y otros International Immunology 18(12):1759-1769 (2006).
En algunos casos, la variante de anticuerpo media la ADCC en presencia de células efectoras humanas más eficazmente que un anticuerpo original. En algunos casos, la variante de anticuerpo es sustancialmente más eficaz en la mediación de ADCC in vitro, cuando las cantidades de la variante del polipéptido y el anticuerpo original utilizados en el ensayo son esencialmente iguales. En algunos casos, la variante de anticuerpo es sustancialmente más eficaz en la mediación de ADCC in vivo, cuando las cantidades de la variante del polipéptido y el anticuerpo original utilizadas en el ensayo son esencialmente iguales. Generalmente, tales variantes se identificarán mediante el uso del ensayo de ADCC in vitro como se describe en el presente documento, pero se contemplan otros ensayos o métodos para determinar la actividad de ADCC, por ejemplo en un modelo animal etc.
Conjugados de anticuerpo ilustrativos
En algunos casos, un anticuerpo anti-ICOS se conjuga a otra molécula. En algunos casos, la molécula adicional puede ser un marcador detectable, tal como una etiqueta. En algunos casos, la molécula adicional puede ser una molécula terapéutica, tal como un agente citotóxico. En algunos casos, una etiqueta y/o un agente citotóxico pueden conjugarse al anticuerpo. Como se usa en la presente descripción, una etiqueta es un resto que facilita la detección del anticuerpo y/o facilita la detección de una molécula a la que se une el anticuerpo. Los ejemplos de etiquetas no limitantes incluyen, pero no se limitan a, radioisótopos, grupos fluorescentes, grupos enzimáticos, grupos quimioluminiscentes, biotina, etiquetas de epítopos, etiquetas de unión a metales, etc. Un experto en la técnica puede seleccionar una etiqueta adecuada de conformidad con la aplicación específica.
Como se usa en la presente descripción, un agente citotóxico es un resto que reduce la capacidad proliferativa de una o más células. Una célula tiene una capacidad de proliferación reducida cuando la célula se vuelve menos capaz de proliferar, por ejemplo, debido a que la célula experimenta apoptosis o muere de cualquier otra manera, la célula no avanza a través del ciclo celular y/o no se divide, la célula se desdiferencia, etc. Los agentes citotóxicos ilustrativos no limitantes incluyen, pero no se limitan a, radioisótopos, toxinas y agentes quimioterapéuticos. Un experto en la técnica puede seleccionar un agente citotóxico adecuado de acuerdo con la aplicación prevista. En algunos casos, el agente citotóxico es al menos uno de un antimetabolito, un agente alquilante, un antibiótico, un factor de crecimiento, una citocina, un agente antiangiogénico, un agente antimitótico, una antraciclina, toxina o un agente apoptótico.
En algunos casos, una etiqueta y/o un agente citotóxico se conjuga a un anticuerpo mediante el uso de métodos
químicos in vitro. Los métodos químicos ilustrativos de conjugación no limitantes se conocen en la técnica, e incluyen servicios, métodos y/o reactivos disponibles comercialmente de, por ejemplo, Thermo Scientific Life Science Research Produces (anteriormente Pierce; Rockford, 111.), Prozyme (Hayward, Calif.), SACRI Antibody Services (Calgary, Canadá), AbD Serotec (Raleigh, N.C.), etc. En algunos casos, cuando una etiqueta y/o un agente citotóxico es un polipéptido, la etiqueta y/o agente citotóxico puede expresarse a partir del mismo vector de expresión con al menos una cadena de anticuerpo para producir un polipéptido que comprende la etiqueta y/o el agente citotóxico fusionado a una cadena de anticuerpo. Un experto en la técnica puede seleccionar un método adecuado para conjugar una etiqueta y/o agente citotóxico a un anticuerpo de acuerdo con la aplicación prevista.
En algunos casos, la conjugación puede ser covalente. En algunos casos, la conjugación puede ser no covalente. En algunos casos, la conjugación puede ser por medio de una interacción de unión específica, por ejemplo, a través de la unión de un anticuerpo secundario.
Secuencias líder ilustrativas
Para que algunas proteínas secretadas se expresen y secreten en grandes cantidades, puede ser conveniente una secuencia líder de una proteína heteróloga. En algunos casos, emplear secuencias líder heterólogas puede ser ventajoso ya que un polipéptido maduro resultante puede permanecer inalterado mientras que la secuencia líder se elimina en el Er durante el proceso de secreción. La adición de una secuencia líder heteróloga puede ser útil para expresar y secretar algunas proteínas.
Determinadas secuencias líder ilustrativas se describen, por ejemplo, en la base de datos de secuencias líder en línea mantenida por el Departamento de Bioquímica, Universidad Nacional de Singapur. Véase Choo y otros, BMC Bioinformatics, 6: 249 (2005); y publicación PCT núm. WO 2006/081430.
IV. Expresión y producción de anticuerpos
Moléculas de ácido nucleico que codifican anticuerpos anti-ICOS
En la presente se proporcionan moléculas de ácido nucleico que comprenden polinucleótidos que codifican una o más cadenas de un anticuerpo anti-ICOS. En algunos casos, una molécula de ácido nucleico comprende un polinucleótido que codifica una cadena pesada o una cadena ligera de un anticuerpo anti-ICOS. En algunas modalidades, una molécula de ácido nucleico comprende un polinucleótido que codifica una cadena pesada y un polinucleótido que codifica una cadena ligera, de un anticuerpo anti-ICOS. En algunas modalidades, una primera molécula de ácido nucleico comprende un primer polinucleótido que codifica una cadena pesada y una segunda molécula de ácido nucleico comprende un segundo polinucleótido que codifica una cadena ligera.
En algunas modalidades, la cadena pesada y la cadena ligera se expresan a partir de una molécula de ácido nucleico o de dos moléculas de ácido nucleico separadas, como dos polipéptidos separados. En algunas modalidades, como cuando un anticuerpo es un scFv, un único polinucleótido codifica un único polipéptido que comprende tanto una cadena pesada como una cadena ligera unidas entre sí.
En algunos casos, un polinucleótido que codifica una cadena pesada o cadena ligera de un anticuerpo anti-ICOS comprende una secuencia nucleotídica que codifica al menos una de las CDR proporcionadas en la presente descripción. En algunos casos, un polinucleótido que codifica una cadena pesada o cadena ligera de un anticuerpo anti-ICOS comprende una secuencia nucleotídica que codifica al menos 3 de las CDR proporcionadas en la presente descripción. En algunas modalidades, un polinucleótido que codifica una cadena pesada o cadena ligera de un anticuerpo anti-ICOS comprende una secuencia de nucleótidos que codifica al menos 6 de las CDR proporcionadas en la presente descripción. En algunos casos, un polinucleótido que codifica una cadena pesada o cadena ligera de un anticuerpo anti-ICOS comprende una secuencia de nucleótidos que codifica una secuencia líder, que, cuando se traduce, se ubica en el extremo N terminal de la cadena pesada o la cadena ligera. Como se analizó anteriormente, la secuencia líder puede ser la secuencia líder de cadena pesada o ligera natural, o puede ser otra secuencia líder heteróloga.
En algunos casos, el ácido nucleico es uno que codifica cualquiera de las secuencias de aminoácidos para los anticuerpos en la Tabla de secuencias en la presente descripción. En algunos casos, el ácido nucleico es uno que es al menos 80 % idéntico a un ácido nucleico que codifica cualquiera de las secuencias de aminoácidos para los anticuerpos en la Tabla de secuencias en la presente, por ejemplo, al menos 80, 85, 90, 91, 92, 93, 94, 95, 96, 97, 98 o 99 % idéntica. En algunos casos, el ácido nucleico es uno que se hibrida a cualquiera o más de las secuencias de ácidos nucleicos proporcionadas en la presente descripción. En algunos casos, la hibridación es en condiciones moderadas. En algunos casos, la hibridación es en condiciones altamente rigurosas, tales como: al menos aproximadamente 6X SSC y SDS al 1 % a 65 °C, con un primer lavado durante 10 minutos a aproximadamente 42 °C con aproximadamente 20 % (v/v) de formamida en SSC 0,1X, y con un lavado posterior con SSC 0,2 X y 0,1 % de SDS a 65 °C.
Las moléculas de ácido nucleico pueden construirse mediante el uso de técnicas de ADN recombinante
convencionales en la materia. En algunas modalidades, una molécula de ácido nucleico es un vector de expresión que es adecuado para la expresión en una célula huésped seleccionada.
Se proporcionan vectores que comprenden polinucleótidos que codifican cadenas pesadas anti-ICOS y/o cadenas ligeras anti-ICOS. Se proporcionan, además, vectores que comprenden polinucleótidos que codifican cadenas pesadas anti-ICOS y/o cadenas ligeras anti-ICOS. Tales vectores incluyen, pero sin limitarse a, vectores de ADN, vectores de fagos, vectores virales, vectores retrovirales, etc. En algunas modalidades, un vector comprende una primera secuencia de polinucleótidos que codifica una cadena pesada y una segunda secuencia de polinucleótidos que codifica una cadena ligera. En algunos casos, la cadena pesada y la cadena ligera se expresan a partir del vector como dos polipéptidos separados. En algunos casos, la cadena pesada y la cadena ligera se expresan como parte de un polipéptido único, tal como, por ejemplo, cuando el anticuerpo es un scFv.
En algunos casos, un primer vector comprende un polinucleótido que codifica una cadena pesada y un segundo vector comprende un polinucleótido que codifica una cadena ligera. En algunos casos, el primer vector y el segundo vector se transfectan en células huésped en cantidades similares (tales como cantidades molares similares o cantidades de masa similares). En algunos casos, una relación molar o de masa de entre 5:1 y 1:5 del primer vector y el segundo vector se transfecta en células huésped. En algunos casos, se usa una relación de masa de entre 1:1 y 1:5 para el vector que codifica la cadena pesada y el vector que codifica la cadena ligera. En algunos casos, se usa una relación de masa de 1:2 para el vector que codifica la cadena pesada y el vector que codifica la cadena ligera.
En algunos casos, se selecciona un vector que se optimiza para la expresión de polipéptidos en células CHO o derivadas de CHO o en células NSO. Tales vectores ilustrativos se describen, por ejemplo, en Running Deer y otros, Biotechnol. Prog. 20:880-889 (2004).
Células huésped
En algunos casos, las cadenas pesadas de anticuerpo anti-ICOS y/o las cadenas ligeras de anticuerpos anti-ICOS pueden expresarse en células procariotas, como células bacterianas; o en células eucariotas, como células fúngicas (como levadura), células vegetales, células de insectos y células de mamíferos. Dicha expresión puede llevarse a cabo, por ejemplo, de acuerdo con los procedimientos conocidos en la técnica. Las células eucariotas ilustrativas que pueden usarse para expresar polipéptidos incluyen, pero no se limitan a, células COS, que incluyen células COS 7; células 293, que incluyen células 293-6E; células Ch O, que incluyen CHO-S, células CHO DG44. Lecl3 y células CHO FUT8; células PER.C6® (Crucell); y células NSO. En algunos casos, las cadenas pesadas de anticuerpo anti-ICOS y/o las cadenas ligeras de anticuerpos anti-ICOS pueden expresarse en levadura. Véase, por ejemplo, la publicación de Estados Unidos núm. US 2006/0270045 A1. En algunos casos, una célula huésped eucariota particular se selecciona sobre la base de su capacidad para realizar modificaciones postraduccionales deseadas a las cadenas pesadas de anticuerpo anti-ICOS y/o las cadenas ligeras de anticuerpos anti-ICOS. Por ejemplo, en algunos casos, las células CHO producen polipéptidos que tienen un mayor nivel de sialilación que el mismo polipéptido producido en células 293.
La introducción de uno o más ácidos nucleicos en una célula huésped deseada puede lograrse mediante cualquier método, que incluye pero sin limitarse a, transfección con fosfato de calcio, transfección mediada por DEAE-dextrano, transfección mediada por lípidos catiónicos, electroporación, transducción, infección, etc. Los métodos ilustrativos no limitantes se describen, por ejemplo, en Sambrook y otros, Molecular Cloning, A Laboratory Manual, 3ra ed. Cold Spring Harbor Laboratory Press (2001). Los ácidos nucleicos pueden transfectarse transitoriamente o establemente en las células huésped deseadas, de conformidad con cualquier método adecuado.
Se proporcionan, además, células huésped que comprenden cualquiera de los polinucleótidos o vectores descritos en la presente descripción. En algunas modalidades, se proporciona una célula huésped que comprende un anticuerpo anti-ICOS. Cualquier célula huésped capaz de sobreexpresar ADN heterólogo puede usarse con el propósito de aislar los genes que codifican el anticuerpo, polipéptido o proteína de interés. Los ejemplos no limitantes de células huésped de mamífero incluyen, pero no se limitan a, células COS, HeLa y CHO. Véase también la publicación PCT núm. WO 87/04462. Las células huésped adecuadas que no son de mamífero incluyen procariotas (tales como E. coli o B. subtilis) y levadura (tal como S. cerevisae, S. pombe; o K. lactis).
Purificación de anticuerpos
Los anticuerpos anti-ICOS pueden purificarse mediante cualquier método adecuado. Estos métodos incluyen, pero no se limitan a, el uso de matrices de afinidad o cromatografía de interacción hidrofóbica. Los ligandos de afinidad adecuados incluyen el ECD ROR1 y los ligandos que se unen a las regiones constantes del anticuerpo. Por ejemplo, una columna de afinidad de Proteína A, Proteína G, Proteína A/G o de anticuerpos puede usarse para unirse a la región constante y purificar un anticuerpo anti-ICOS. La cromatografía interactiva hidrófoba, por ejemplo, una columna de butilo o fenilo, también puede ser adecuada para purificar algunos polipéptidos tales como anticuerpos. La cromatografía de intercambio iónico (por ejemplo cromatografía de intercambio aniónico y/o cromatografía de intercambio catiónico) también puede ser adecuada para purificar algunos polipéptidos tales como anticuerpos. La cromatografía en modo mixto (por ejemplo fase inversa/ intercambio de aniones, fase inversa/ intercambio de cationes,
interacción hidrofílica/intercambio de aniones, interacción hidrofílica/intercambio de cationes, etc.) también puede ser adecuada para purificar algunos polipéptidos tales como anticuerpos. En la técnica se conocen muchos métodos de purificación de polipéptidos.
Producción de anticuerpos libre de células
En algunos casos, un anticuerpo anti-ICOS se produce en un sistema libre de células. Los sistemas sin células ilustrativos no limitantes se describen, por ejemplo, en Sitaraman y otros, Methods Mol. Biol. 498: 229-44 (2009); Spirin, Trends Biotechnol. 22: 538-45 (2004); Endo y otros, Biotechnol. Adv. 21: 695-713 (2003).
Composiciones
En algunos casos, se describen anticuerpos preparados mediante los métodos descritos anteriormente. En algunos casos, el anticuerpo se prepara en una célula huésped. En algunos casos, el anticuerpo se prepara en un sistema libre de células. En algunos casos, el anticuerpo se purifica. En algunos casos, el anticuerpo preparado en una célula huésped o un sistema libre de células es un anticuerpo quimérico. En algunos casos, el anticuerpo preparado en una célula huésped o un sistema libre de células es un anticuerpo humanizado. En algunos casos, el anticuerpo preparado en una célula huésped o un sistema libre de células es un anticuerpo humano. En algunos casos, se proporciona un medio de cultivo celular que comprende un anticuerpo anti-ICOS. En algunos casos, se proporciona un fluido de cultivo celular huésped que comprende un anticuerpo anti-ICOS.
En algunos casos, se describen composiciones que comprenden anticuerpos preparados mediante los métodos descritos anteriormente. En algunos casos, la composición comprende un anticuerpo preparado en una célula huésped. En algunos casos, la composición comprende un anticuerpo preparado en un sistema libre de células. En algunos casos, la composición comprende un anticuerpo purificado. En algunos casos, la composición comprende un anticuerpo quimérico preparado en una célula huésped o un sistema libre de células. En algunos casos, la composición comprende un anticuerpo humanizado preparado en una célula huésped o un sistema libre de células. En algunos casos, la composición comprende un anticuerpo humano preparado en una célula huésped o un sistema libre de células.
En algunos casos, se proporciona una composición que comprende anticuerpo anti-ICOS a una concentración de más de aproximadamente cualquiera de 10 mg/ml, 20 mg/ml, 30 mg/ml, 40 mg/ml, 50 mg/ml, 60 mg/ml, 70 mg/ml, 80 mg/ml, 90 mg/ml, 100 mg/ml, 125 mg/ml, 150 mg/ml, 175 mg/ml, 200 mg/ml, 225 mg/ml o 250 mg/ml. En algunos casos, la composición comprende un anticuerpo quimérico preparado en una célula huésped o un sistema libre de células. En algunos casos, la composición comprende un anticuerpo humanizado preparado en una célula huésped o un sistema libre de células. En algunos casos, la composición comprende un anticuerpo humano preparado en una célula huésped o un sistema libre de células.
V. Composiciones y métodos terapéuticos
Métodos para tratar enfermedades mediante el uso de anticuerpos anti-ICOS
Los anticuerpos y composiciones que comprenden anticuerpos se proporcionan para usar en métodos de tratamiento para seres humanos o animales. Se describen, además, métodos para tratar una enfermedad, que comprenden administrar anticuerpos anti-ICOS. Las enfermedades ilustrativas no limitantes que pueden tratarse con anticuerpos anti-ICOS incluyen, pero no se limitan al cáncer.
En algunos casos, se proporciona un método para tratar un tumor, en donde las células dentro de una muestra del tumor expresan ICOS. En algunos casos, el tumor puede considerarse como positivo para ICOS o que expresa ICOS. La expresión de ICOS puede determinarse por IHC, por ejemplo, como se analiza en el presente documento. En algunas modalidades, se considera que un tumor expresa ICOS cuando una muestra del tumor muestra una tinción de ICOS de 1+, 2+ o 3+ mediante IHC. En algunas modalidades, la muestra del tumor muestra una tinción de ICOS de 2+ o 3+ mediante IHC. En algunos casos, una muestra de tumor de un sujeto se analiza para determinar la expresión de ICOS y el sujeto se selecciona para el tratamiento con un anticuerpo descrito en el presente documento si la muestra de tumor muestra expresión de ICOS. En algunos casos, el sujeto se selecciona si la muestra tumoral muestra una expresión elevada de ICOS.
En algunos casos, se selecciona un sujeto para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente si el tumor del sujeto es p d -L1bajo. En algunos casos, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente si el tumor del sujeto es ICOSalto/PD-L1bajo. En algunos casos, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente si el tumor del sujeto es ICOSalto/PD-L1alto.
El anticuerpo anti-ICOS puede administrarse a los sujetos según sea necesario. La determinación de la frecuencia de administración puede realizarse por los expertos en la técnica, como un médico especialista sobre la base de consideraciones de la afección que se trata, la edad del sujeto que se trata, la gravedad de la afección que se trata, el
estado general de salud del sujeto que se trata y similares. En algunas modalidades, una dosis eficaz de un anticuerpo anti-ICOS se administra a un sujeto una o más veces. En algunos casos, una dosis eficaz de un anticuerpo anti-ICOS se administra al sujeto una vez al mes, menos de una vez al mes, tal como, por ejemplo, cada dos meses o cada tres meses. En algunos casos, una dosis eficaz de un anticuerpo anti-ICOS se administra menos de una vez al mes, tal como, por ejemplo, una vez cada tres semanas, una vez cada dos semanas o una vez cada semana. Una dosis eficaz de un anticuerpo anti-ICOS se administra al sujeto al menos una vez. En algunos casos, la dosis eficaz de un anticuerpo anti-ICOS puede administrarse múltiples veces, que incluye durante períodos de al menos un mes, al menos seis meses o al menos un año.
En algunos casos, las composiciones farmacéuticas se administran en una cantidad eficaz para el tratamiento (que incluye profilaxis) de cáncer. La cantidad con eficacia terapéutica depende típicamente del peso del sujeto que se trata, su afección física o de salud, la capacidad de propagación de la afección a tratar o la edad del sujeto que se trata. En general, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 10 mg/kg de peso corporal a aproximadamente 100 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 50 mg/kg de peso corporal a aproximadamente 5 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 10 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,5 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis.
Las composiciones farmacéuticas se administran en una cantidad eficaz para aumentar la cantidad de células Tef; activar las células Tef; agotar las células Treg; y/o aumentar la relación de las células Tef con respecto a las células Treg. En algunos casos, las células Treg son células T CD4+ FoxP3+. En algunas modalidades, las células Tef son células T CD8+. En algunos casos, las células Tef son células T CD4+ FoxP3- y células T CD8+.
En algunos casos, el tratamiento con anticuerpo anti-ICOS da como resultado una lectura de la farmacodinámica, tal como la regulación positiva del ligando de ICOS (ICOSL). En algunos casos, se observa regulación positiva de ICOSL en la superficie de las células B. En algunos casos, se observa regulación positiva de ICOSL en la superficie de granulocitos. En algunos casos, se observa regulación positiva de ICOSL en la superficie de neutrófilos. La regulación positiva de ICOSL puede observarse en células en el tumor; en células del bazo; en células de sangre periférica. La regulación positiva de ICOSL en la superficie celular puede detectarse, por ejemplo, mediante citometría de flujo. En algunos casos, el ICOSL soluble aumenta en el suero después del tratamiento con anticuerpo anti-ICOS. El ICOSL soluble puede detectarse mediante métodos que incluyen, pero no se limitan a, ELISA, m Sd y espectrometría de masa. En algunos casos, el acoplamiento al objetivo ICOS, según se mide por disponibilidad del receptor libre, por anticuerpos anti-ICOS puede usarse, además, como una lectura de la farmacodinámica. En algunos casos, después del tratamiento con un anticuerpo anti-ICOS, puede cuantificarse la cantidad de receptores ICOS en la superficie de linfocitos T libres para unirse a otros anticuerpos. La disminución en los receptores disponibles observados puede servir como una indicación de que los anticuerpos anti-ICOS se unen a su molécula objetivo.
La cantidad con eficacia terapéutica depende típicamente del peso del sujeto que se trata, su afección física o de salud, la capacidad de propagación de la afección a tratar o la edad del sujeto que se trata. En general, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 10 mg/kg de peso corporal a aproximadamente 100 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 50 mg/kg de peso corporal a aproximadamente 5 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 10 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,5 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis.
Composiciones farmacéuticas
En algunos casos, las composiciones que comprenden anticuerpos anti-ICOS se proporcionan en formulaciones con una amplia variedad de portadores farmacéuticamente aceptables (véase, por ejemplo, Gennaro, Remington: The Science and Practice of Pharmacy with Facts and Comparisons: Drugfacts Plus, 20a ed. (2003); Ansel y otros, Pharmaceutical Dosing Forms and Drug Delivery Systems, 7a ed., Lippencott Williams and Wilkins (2004); Kibbe y otros, Handbook of Pharmaceutical Excipients, 3a ed., Pharmaceutical Press (2000)). Varios portadores farmacéuticamente aceptables, que incluyen vehículos, adyuvantes y diluyentes, están disponibles. Además, también están disponibles diversas sustancias auxiliares farmacéuticamente aceptables, tales como agentes de ajuste del pH y tampones, agentes de ajuste de la tonicidad, estabilizadores, agentes humectantes y similares. Los ejemplos no limitantes de portadores incluyen solución salina, solución salina tamponada, dextrosa, agua, glicerol, etanol y combinaciones de estos.
En algunos casos, se proporciona una composición farmacéutica que comprende un anticuerpo anti-ICOS. En algunos casos, la composición farmacéutica comprende un anticuerpo quimérico. En algunos casos, la composición farmacéutica comprende un anticuerpo humanizado. En algunos casos, la composición farmacéutica comprende un anticuerpo preparado en una célula huésped o sistema libre de células como se describe en el presente documento. En algunos casos, la composición farmacéutica comprende un portador farmacéuticamente aceptable.
En algunos casos, las composiciones farmacéuticas se administran en una cantidad eficaz para el tratamiento (que incluye profilaxis) de cáncer. La cantidad con eficacia terapéutica depende típicamente del peso del sujeto que se trata, su afección física o de salud, la capacidad de propagación de la afección a tratar o la edad del sujeto que se trata. En general, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,05 mg/kg de peso corporal a aproximadamente 100 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 10 mg/kg de peso corporal a aproximadamente 100 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 50 mg/kg de peso corporal a aproximadamente 5 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 10 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 100 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,5 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,5 mg/kg de peso corporal a aproximadamente 10 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,05 mg/kg de peso corporal a aproximadamente 20 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 0,05 mg/kg de peso corporal a aproximadamente 10 mg/kg de peso corporal por dosis. En algunos casos, los anticuerpos anti-ICOS pueden administrarse en una cantidad en el intervalo de aproximadamente 5 mg/kg de peso corporal o menor, por ejemplo menos de 4, menos de 3, menos de 2 o menos de 1 mg/kg del anticuerpo.
En algunos casos, los anticuerpos anti-ICOS pueden estar presentes en una cantidad en el intervalo de aproximadamente 50 mg/kg de peso corporal a aproximadamente 5 mg/kg de peso corporal por dosis. Por ejemplo, en algunos casos, una dosis para una persona de 20 kg puede estar dentro de un intervalo de aproximadamente 1 mg a aproximadamente 100 mg. En algunos casos, la dosis puede estar dentro de un intervalo de 2 mg a 200 mg del anticuerpo anti-ICOS. En algunos casos, la dosis puede estar dentro de un intervalo de 10 mg a 400 mg del anticuerpo anti-ICOS.
Vías de administración
En algunos casos, pueden administrarse anticuerpos anti-ICOS in vivo por diversas vías, que incluyen, pero no se limitan a, intravenosa, intraarterial, parenteral, intratumoral, intraperitoneal o subcutánea. La formulación y vía de administración apropiadas pueden seleccionarse de acuerdo con la aplicación prevista.
Terapia de combinación
Los anticuerpos anti-ICOS pueden administrarse solos o con otros modos de tratamiento. Pueden proporcionarse antes, sustancialmente de manera simultánea, y/o después de otros modos de tratamiento, por ejemplo, cirugía, quimioterapia, radioterapia o la administración de un agente biológico, como otro anticuerpo terapéutico. En algunas modalidades, un anticuerpo anti-ICOS se administra junto con otro agente anticanceroso.
En algunos casos, el anticuerpo anti-ICOS se administra simultáneamente con un segundo agente terapéutico. Por ejemplo, los dos o más agentes terapéuticos se administran con una separación temporal de no más de aproximadamente 60 minutos, tal como no más de aproximadamente 30, 15, 10, 5 o 1 minuto. En algunos casos, el anticuerpo anti-ICOS se administra secuencialmente con un segundo agente terapéutico. Por ejemplo, la administración de los dos o más agentes terapéuticos se realiza con una separación de tiempo de más de aproximadamente 15 minutos, tal como aproximadamente 20, 30, 40, 50 o 60 minutos, 1 día, 2 días, 3 días, 1 semana, 2 semanas o 1 mes o más.
En algunas modalidades, el anticuerpo anti-ICOS se administra con un segundo método terapéutico para el tratamiento. Por lo tanto, la administración de un anticuerpo proporcionado en la presente descripción puede estar en combinación con otro sistema de tratamiento.
En algunas modalidades, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con una terapia de PD-1. Los ejemplos de terapias de PD-1 incluyen, pero sin limitarse a, nivolumab (OPDIVO®, BMS-936558, MDX-1106, ONO-4538); pidilizumab, lambrolizumab/pembrolizumab (KEYTRUDA, MK-3475); durvalumab (anticuerpo anti-PD-L1, MEDI-4736; Astrazeneca/ Medimmune); RG-7446; avelumab (anticuerpo anti-PD-L1; MSB-0010718C; Pfizer); AMP-224; BMS-936559 (anticuerpo anti-PD-L1); AMP-514; MDX-1105; ANB-011; anti-LAG-3/PD-1; anticuerpo
anti-PD-1 (CoStim); anticuerpo anti-PD-1 (Kadmon Pharm.); anticuerpo anti-PD-1 (Immunovo); anticuerpo anti-TIM-3/PD-1 (AnaptysBio); anticuerpo anti-PD-LI (CoStim/Novartis); Rg 7446/MPd L3280A (anticuerpo anti-PD-LI, Genentech/Roche); KD-033, antagonista de PD-1 (Agenus); STI-A1010; STI-A1110; TSR-042; y otros anticuerpos que se dirigen contra la proteína de muerte programada -1 (PD-1) o el ligando de muerte programada 1 (PD-L1).
En algunas modalidades, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente descripción y una terapia de PD-1 si el tumor del sujeto expresa PD-L1. En algunas modalidades, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente descripción y una terapia de PD-1 si el tumor del sujeto es PD-L1ALTO. En algunas modalidades, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente descripción y una terapia de PD-1 si el tumor del sujeto expresa ICOS y PD-L1. En algunas modalidades, un sujeto se selecciona para el tratamiento con un anticuerpo anti-ICOS proporcionado en la presente descripción y una terapia de PD-1 si el tumor del sujeto es ICOSalto/PD-L1alto Determinar el nivel de PD-L1 y/o ICOS puede realizarse, por ejemplo, mediante el uso de IHC. Un tumor de un paciente se considera que expresa PD-L1, en algunos casos, cuando 1 % o más o 5 % o más, de las células tumorales en una muestra muestran tinción de la membrana para PD-L1 mediante IHC. En algunos casos, más del 50 % de las células tumorales en una muestra muestran tinción de membrana para PD-L1 mediante IHC. En algunos casos, el tumor del sujeto se considera PD-L1ALTO Se considera que el tumor de un paciente expresa ICOS, en algunos casos, cuando 1 % o más de las células en una muestra de tumor muestran tinción para ICOS por IHC. En algunos casos, un sujeto se trata primero con una terapia de PD-1, y después se trata con un anticuerpo anti-ICOS proporcionado en la presente descripción, con o sin continuar con la terapia de PD-1. Por lo tanto, los métodos proporcionados en la presente descripción incluyen el tratamiento de un sujeto con un anticuerpo anti-ICOS, en donde el sujeto ha sido tratado previamente con una terapia de PD-1.
En algunos casos, el anticuerpo anti-ICOS proporcionado en la presente se administra con un anticuerpo agonista anti-OX40 (tal como Medi6469, Medlmmune; MOXR0916/RG7888, Roche). En algunas modalidades, el anticuerpo anti-ICOS proporcionado en la presente se administra con un anticuerpo anti-CTLA4 (tal como ipilimumab, YERVOY®, BMS).
En algunas modalidades, un agente terapéutico adicional es un agente quimioterapéutico. Los agentes quimioterapéuticos ilustrativos que pueden combinarse con los anticuerpos anti-ICOS proporcionados en la presente incluyen, pero no se limitan a, capectiabina, ciclofosfamida, dacarbazina, temozolomida, ciclofosfamida, docetaxel, doxorrubicina, daunorrubicina, cisplatino, carboplatino, epirubicina, eribulina, 5-FU, gemcitabina, irinotecán, ixabepilona, metotrexato, mitoxantrona, oxaliplatino, paclitaxel, nab-paclitaxel, ABRAXANE® (paclitaxel unido a proteína), pemetrexed, vinorelbina y vincristina. En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra con al menos un inhibidor de quinasa. Los inhibidores de quinasas ilustrativos no limitantes incluyen erlotinib, afatinib, gefitinib, crizotinib, dabrafenib, trametinib, vemurafenib y cobimetanib.
En algunos casos, el agente terapéutico adicional es un inhibidor de IDO. Los inhibidores de IDO ilustrativos no limitantes se describen, por ejemplo, en los documentos US 2016/0060237 y US 2015/0352206. Los ejemplos no limitantes de inhibidores de IDO incluyen Indoximod (New Link Genetics), INCB024360 (Incyte Corp), 1-metil-D-triptófano (New Link Genetics) y GDC-0919 (Genentech).
En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente se administra en combinación con un fármaco modificador de la inmunidad (IMiD). Los IMiD ilustrativos no limitantes incluyen talidomida, lenalidomida y pomalidomida.
En algunas modalidades, un agente terapéutico adicional es una vacuna contra el cáncer. Las vacunas contra el cáncer se han investigado como un posible enfoque para la transferencia de antígenos y la activación de células dendríticas. En particular, la vacunación en combinación con puntos de control inmunológico o agonistas para las vías coestimuladoras ha demostrado evidencia de superar la tolerancia y generar una respuesta antitumoral aumentada. Se ha evaluado una variedad de vacunas contra el cáncer que emplean diferentes enfoques para promover una respuesta inmunitaria contra el tumor (ver, por ejemplo, Emens LA, Expert Opin Emerg Drugs 13(2): 295-308 (2008)). Se han diseñado enfoques para mejorar la respuesta de células B, células T o células presentadoras de antígeno profesionales contra los tumores. Los tipos ilustrativos de vacunas contra el cáncer incluyen, pero sin limitarse a, vacunas basadas en péptidos que emplean el direccionamiento a antígenos tumorales distintos, que pueden administrarse como péptidos/proteínas o como vectores de ADN genéticamente modificados, virus, bacterias o similares; y enfoques de biología celular, por ejemplo, para el desarrollo de vacunas contra el cáncer contra objetivos menos definidos, que incluyen, pero sin limitarse a, vacunas desarrolladas a partir de células dendríticas derivadas de pacientes, células tumorales autólogas o lisados celulares tumorales, células tumorales alogénicas, y similares.
Por lo tanto, en determinadas modalidades, los anticuerpos anti-ICOS proporcionados en la presente descripción pueden usarse en combinación con una vacuna contra el cáncer. Las vacunas contra el cáncer ilustrativas incluyen, pero sin limitarse a, vacunas de células dendríticas, virus oncolíticos, vacunas de células tumorales, etc. En algunos casos, tales vacunas aumentan la respuesta antitumoral. Los ejemplos de vacunas contra el cáncer que pueden usarse en combinación con los anticuerpos anti-ICOS proporcionados en la presente incluyen, pero sin limitarse a, vacuna de MAGE3 (por ejemplo, para el cáncer de melanoma y vejiga), vacuna de MUC1 (por ejemplo, para el cáncer de mama),
EGFRv3 (como Rindopepimut, por ejemplo, para el cáncer de cerebro, que incluye glioblastoma multiforme) o ALVAC-CEA (por ejemplo, para cánceres c Ea ).
Las vacunas contra el cáncer ilustrativas no limitantes incluyen, además, Sipuleucel-T, que se deriva de células mononucleares de sangre periférica autólogas (PBMC) que incluyen células presentadoras de antígeno (ver, por ejemplo, Kantoff PW y otros, N Engl J Med 363:411-22 (2010)). En la generación de Sipuleucel-T, las p Bm C del paciente se activan ex vivo con PA2024, una proteína de fusión recombinante de la fosfatasa ácida prostática (un antígeno prostático) y el factor estimulante de colonias de granulocitos y macrófagos (un activador de células inmunitarias). Otro enfoque de una vacuna candidata contra el cáncer es generar una respuesta inmunitaria contra péptidos específicos mutados en tejido tumoral, tal como melanoma (ver, por ejemplo, Carreno BM y otros, Science 348:6236 (2015)). Tales péptidos mutados, en algunos casos, pueden denominarse neoantígenos. Como ejemplo no limitante del uso de neoantígenos en vacunas tumorales, los neoantígenos en el tumor que se predice que se unen a la proteína del complejo mayor de histocompatibilidad HLA-A*02:01 se identifican para pacientes individuales con un cáncer, tal como melanoma. Las células dendríticas del paciente se maduran ex vivo, después se incuban con neoantígenos. Las células dendríticas activadas se administran después al paciente. En algunos casos, después de la administración de la vacuna contra el cáncer, puede detectarse una fuerte inmunidad de células T contra el neoantígeno.
En algunas de estas modalidades, la vacuna contra el cáncer se desarrolla mediante el uso de un neoantígeno. En algunas modalidades, la vacuna contra el cáncer es una vacuna de ADN. En algunas modalidades, la vacuna contra el cáncer es un virus modificado genéticamente que comprende un antígeno del cáncer, como PROSTVAC (rilimogene galvacirepvec/rilimogene glafolivec). En algunas modalidades, la vacuna contra el cáncer comprende células tumorales modificadas genéticamente, como GVAX, que es una vacuna de células tumorales transfectadas con el gen del factor estimulante de colonias de granulocitos y macrófagos (GMCSF) (ver, por ejemplo, Nemunaitis, 2005, Expert Rev Vaccines, 4: 259-74).
En algunos casos, un anticuerpo anti-ICOS descrito en el presente documento se administra antes, simultáneamente, y/o después de una vacuna contra el cáncer. En algunas modalidades, las vacunas contra el cáncer desarrolladas mediante el uso de neoantígenos se usan en combinación con los anticuerpos anti-ICOS descritos en la presente descripción. En algunos casos, la combinación se usa para tratar un cáncer con una carga mutacional alta, tal como melanoma, cáncer de pulmón, vejiga o colorrectal.
En algunos casos, un anticuerpo anti-ICOS proporcionado en la presente descripción se administra en combinación con una terapia con células T con receptor de antígeno quimérico (terapia CAR-T).
Usos de diagnóstico
En la presente se describen métodos para usar los anticuerpos anti-ICOS, polipéptidos y polinucleótidos para la detección, el diagnóstico y el control de una enfermedad, trastorno o afección asociados con la expresión de epítopos de anticuerpos anti-ICOS (ya sea aumentada o disminuida con relación a una muestra normal, y/o expresión inapropiada, tal como la presencia de expresión en tejido(s) y/o célula(s) que normalmente carecen de la expresión de los epítopos). En la presente se describen métodos para determinar si un paciente responderá a una terapia con anticuerpos anti-ICOS.
En algunos casos, el método comprende detectar si el paciente tiene células que expresan ICOS usando un anticuerpo anti-ICOS. En algunos casos, el método de detección comprende poner en contacto la muestra con un anticuerpo, polipéptido o polinucleótido y determinar si el nivel de unión difiere del de una muestra de referencia o de comparación (tal como un control). En algunos casos, el método puede ser útil para determinar si los anticuerpos o polipéptidos descritos en la presente son un tratamiento adecuado para el sujeto.
En algunos casos, las células o lisado celular/tisular se ponen en contacto con un anticuerpo anti-ICOS y se determina la unión entre el anticuerpo y la célula. Cuando las células de prueba muestran actividad de unión en comparación con una célula de referencia del mismo tipo de tejido, puede indicar que el sujeto podría beneficiarse del tratamiento con un anticuerpo anti-ICOS. En algunos casos, las células de prueba son de tejidos humanos.
Pueden usarse varios métodos conocidos en la técnica para detectar la unión específica entre un anticuerpo y un antígeno. Los inmunoensayos ilustrativos que pueden llevarse a cabo incluyen inmunoensayo de polarización de fluorescencia (FPIA), inmunoensayo de fluorescencia (FIA), inmunoensayo enzimático (EIA), inmunoensayo de inhibición nefelométrica (NIA), ensayo inmunoabsorbente ligado a enzimas (ELISA), y radioinmunoensayo (RIA). Un resto indicador o grupo marcador, puede unirse a los anticuerpos objetivo y se selecciona para satisfacer las necesidades de varios usos del método que se establecen frecuentemente por la disponibilidad del equipo de ensayo y los procedimientos de inmunoensayo compatibles. Las etiquetas adecuadas incluyen, sin limitación, radionúclidos (por ejemplo, 125I, 131I, 35S, 3H o 32P), enzimas (por ejemplo, fosfatasa alcalina, peroxidasa de rábano picante, luciferasa o p-galactosidasa), restos o proteínas fluorescentes (por ejemplo, fluoresceína, rodamina, ficoeritrina, GFP o BFP) o restos luminiscentes (por ejemplo, nanopartículas Qdot™ suministradas por Quantum Dot Corporation, Palo Alto, Calif.). Las técnicas generales para usar en la realización de los diversos inmunoensayos señalados anteriormente se
conocen por los expertos en la técnica.
Para propósitos de diagnóstico, el polipéptido que incluye anticuerpos puede marcarse con un resto detectable que incluye, entre otros, radioisótopos, etiquetas fluorescentes y diversas etiquetas enzima-sustrato, conocidos en la técnica. Los métodos de conjugación de etiquetas a un anticuerpo se conocen en la técnica.
En algunos casos, no es necesario que los anticuerpos anti-ICOS se etiqueten, y la presencia de estos puede detectarse mediante el uso de un segundo anticuerpo marcado que se une al primer anticuerpo anti-ICOS.
En algunos casos, el anticuerpo anti-ICOS puede emplearse en cualquier método de ensayo conocido, tal como ensayos de unión competitiva, ensayos sándwich directos e indirectos y ensayos de inmunoprecipitación. Zola, Monoclonal Antibodies: A Manual of Techniques, pp. 147-158 (CRC Press, Inc. 1987).
Los anticuerpos y polipéptidos anti-ICOS pueden usarse, además, para ensayos de diagnóstico in vivo, tales como la obtención de imágenes in vivo. Generalmente, el anticuerpo o el polipéptido se marca con un radionúclido (como 111In, 99Tc, 14C, 131I, 125I, 3H o cualquier otra etiqueta de radionúclido, que incluye las descritas en la presente descripción) de manera que las células o el tejido de interés pueden localizarse mediante el uso de inmunoscintiografía.
El anticuerpo puede usarse además como reactivo de tinción en patología mediante el uso de técnicas conocidas en la materia.
En algunos casos, se usa un primer anticuerpo para un diagnóstico y se usa un segundo anticuerpo como un agente terapéutico. En algunos casos, el primer y segundo anticuerpos son diferentes. En algunos casos, el primer anticuerpo no es de un ser humano, mientras que el agente terapéutico es de un ser humano. En algunos casos, el primer y segundo anticuerpos pueden unirse al antígeno al mismo tiempo, al unirse a epítopos separados.
Kits/artículos de fabricación
En la presente también se proporcionan kits, medicamentos, composiciones y formas de dosificación unitaria para usar en cualquiera de los métodos descritos en la presente.
Los kits pueden incluir uno o más contenedores que comprenden un anticuerpo anti-ICOS (o formas de dosificación unitaria y/o artículos de fabricación). En algunos casos, se proporciona una dosis unitaria en donde la dosis unitaria contiene una cantidad predeterminada de una composición que comprende un anticuerpo anti-ICOS, con o sin uno o más agentes adicionales. En algunos casos, tal dosificación unitaria se suministra en una jeringa precargada de un solo uso para inyección. En algunos casos, la composición contenida en la dosificación unitaria puede comprender solución salina, sacarosa o similares; un tampón, como fosfato o similares; y/o se formula dentro de un intervalo de pH estable y eficaz. En algunos casos, la composición puede proporcionarse como un polvo liofilizado que puede reconstituirse tras la adición de un líquido apropiado, por ejemplo, agua estéril. En algunos casos, la composición comprende una o más sustancias que inhiben la agregación de proteínas, que incluyen, entre otros, sacarosa y arginina. En algunos casos, una composición comprende heparina y/o un proteoglicano.
En algunos casos, la cantidad del anticuerpo anti-ICOS usada en la dosis unitaria puede ser cualquiera de las cantidades proporcionadas en la presente para los diversos métodos y/o composiciones descritos.
En algunos casos, los kits comprenden además instrucciones para usar en el tratamiento del cáncer de acuerdo con cualquiera de los métodos descritos en la presente. El kit puede comprender además una descripción de la selección de un individuo adecuado o tratamiento. Las instrucciones que se suministran en los kits son instrucciones por escrito típicamente en una etiqueta o prospecto (por ejemplo, una lámina de papel incluida en el kit), pero también son aceptables instrucciones legibles por una máquina (por ejemplo, instrucciones transportadas en un disco de almacenamiento magnético u óptico). En algunos casos, el kit comprende además otro agente terapéutico.
Los kits están en un empaque adecuado. El empaque adecuado incluye, pero no se limita a, frascos, botellas, botes, empaques flexibles (por ejemplo, bolsas Mylar o plásticas selladas) y similares. Los kits pueden proporcionar opcionalmente componentes adicionales tales como tampones e información interpretativa. La presente solicitud también proporciona artículos de fabricación, que incluyen frascos (como frascos sellados), botellas, botes, empaques flexibles y similares.
Ejemplos
Los ejemplos analizados a continuación son puramente ilustrativos de la invención y no deben considerarse que limitan la invención de ninguna manera. Los ejemplos no pretenden representar que los experimentos a continuación sean todos o solo los experimentos realizados. Se han realizado esfuerzos para asegurar la precisión con respecto a los números utilizados (por ejemplo, cantidades, temperatura, etc()) pero deben tenerse en cuenta algunos errores y desviaciones experimentales. A menos que se indique de cualquier otra manera, las partes son partes en peso, el peso molecular es peso molecular promedio en peso, la temperatura es en grados Centígrados y la presión es la
atmosférica o cercana a esta.
Ejemplo 1: Análisis bioinformático de la expresión de ARNm de ICOS en tumores humanos.
Mediante la utilización de los datos de secuenciación de ARN recopilados como parte de TCGA, la expresión de ICOS se comparó en ~ 7500 tumores en 24 indicaciones diferentes. Se encontraron altos niveles de ARNm de ICOS en subconjuntos de cáncer de células escamosas de cabeza y cuello (HNSCC), cáncer de pulmón de células no pequeñas (NSCLC) y cáncer de mama triple negativo (TNBC). Ver la Figura 1A.
Se investigó la asociación entre la infiltración de células T y los niveles de expresión de ICOS. La expresión de un conjunto de 12 genes de quimiocinas se ha asociado con altos niveles de infiltración de células T y la formación de estructuras tipo ganglio linfático (Messina y otros, 2012, Sci Reports. 2:765-771). La puntuación de la firma de quimiocinas se calculó para cada muestra sobre la base de la expresión promedio de estos 12 genes de quimiocinas. Esta puntuación de la firma se calculó en todas las muestras de cáncer de células escamosas de cabeza y cuello (HNSCC). Se correlacionaron los niveles de la firma de quimiocinas o un marcador de células Treg (FoxP3) con los niveles de ICOS en los tumores de HNSCC. Ver la Figura 2. Hubo una fuerte correlación entre la firma de quimiocinas y los niveles de ICOS (R=0,83; correlación de Spearman) o ICOS y FoxP3, un marcador de células Treg (R=0,88; correlación de Spearman). Estos datos demuestran que la expresión de ICOS se asocia estrechamente con la infiltración de células T y las células Treg. Se observaron datos similares en NSCLC y TNBC.
En los tumores HNSCC, la expresión de ICOS se correlacionó significativamente (R=0,93 y 0,78 respectivamente) con los niveles de expresión de otras moléculas de puntos de control como CTLA-4 y PD-1. Ver la Figura 3. La correlación de ICOS con PD-L1 fue débil (R=0,62). Se observaron datos similares en otras indicaciones tales como NSCLC. Estos datos sugieren que ICOS puede expresarse en las mismas células T que expresan otras moléculas de puntos de control como CTLA-4 y PD-1. La correlación más débil con PD-L1 e ICOS sugiere que podría haber un subconjunto de pacientes con ICOS alto que podrían ser de PD-L1 bajo o negativo. Estos datos apoyan a ICOS como un agente único en pacientes negativos para PD-L1 y también una estrategia de combinación con terapias anti-PD-1 o anti-CTLA-4.
Ejemplo 2: Análisis IHC de tumores humanos
Los niveles de expresión de la proteína ICOS se determinaron mediante el uso de un ensayo inmunohistoquímico (IHC). Este ensayo, que usa un anticuerpo monoclonal anti-ICOS de conejo (SP98, Spring Biosciences, Pleasanton, CA), se validó para determinar la especificidad de ensayo, la precisión del ensayo (intra-ejecuciones y entre ejecuciones, y reproducibilidad entre lotes) y la sensibilidad. Los estudios de validación se realizaron mediante el uso de secciones de tejido fijados en formalina, embebidos en parafina (FFPE) y líneas celulares control (CHO modificadas genéticamente para expresar ICOS humano (control positivo) o una línea celular control de vector CHO que no expresa ICOS (control negativo)). El ensayo se realizó en una plataforma de tinción automatizada Leica-Bond Rx y se detectó tinción específica de ICOS en las células CHO de control positivo y en subconjuntos de células T en la amígdala humana normal.
Los portaobjetos se calificaron por un patólogo capacitado mediante el uso de los siguientes criterios para la tinción cromogénica:
Frecuencia de células positivas para ICOS___________ Puntuación < 1 % de las células son positivas para ICOS 0
> 1 % pero < 5 % de las células son positivas para ICOS 1
> 5 % pero < 15 % de las células son positivas para ICOS 2+
> 15 % de las células son positivas para ICOS__________ 3+
Las micromatrices de tejidos de 11 tipos de tumor diferentes se tiñeron y clasificaron mediante el uso de este sistema de puntuación. Ver la Figura 1B. Los datos de IHC confirmaron los datos basados en ARNm en el hecho que HNSCC, NSCLC y TNBC contenían el mayor porcentaje de infiltración de células inmunitarias ICOS+ alto (es decir, 3+). Ver la Figura 1B. Además de estos tumores, los subconjuntos de pacientes con melanoma, cáncer colorrectal y adenocarcinoma gástrico tuvieron niveles moderados de infiltración celular positiva a ICOS. Ver la Figura 1B.
Para evaluar la prevalencia y naturaleza de las células T que expresan ICOS, se desarrolló un ensayo IHC de inmunofluorescencia múltiple para la detección de ICOS, FOXP3 y CD8. Se utilizó un marcador de ADN (DAPI) para contar la cantidad total de núcleos en las secciones tumorales humanas.
La expresión de ICOS se determinó mediante el uso de análisis inmunohistoquímico con el clon de anticuerpo monoclonal de conejo que reconoce ICOS humano (Spring Biosciences Inc. Pleasanton, CA). La especificidad y sensibilidad del ensayo IHC de ICOS se confirmó con el uso de amígdalas humanas y líneas celulares que sobreexpresan ICOS de manera constitutiva. La intensidad de la tinción fue evaluada por un patólogo capacitado con el uso de los siguientes criterios. A toda tinción positiva se le asignó una puntuación sobre la base de la expresión de membrana en al menos dos tercios de las células.
Una imagen representativa del esquema de puntuación se muestra en la Figura 4. La puntuación se realizó sobre la base de los siguientes criterios para la inmunofluorescencia:
0 (negativo) = No hay o hay menos de 0,1 % de células con tinción de la membrana
1+ (leve) = 0,1 a 5 % de las células son positivas
2+ (moderado) = 5 a 10 % de las células son positivas
3+ (Fuerte) = > 10 a 50 % en las células son positivas
La prevalencia de la expresión de ICOS en varios subconjuntos de NSCLC o muestras de pulmón normales adyacentes se resumen en la Tabla 2. En el tejido pulmonar normal adyacente en los pacientes con cáncer no se observó expresión fuerte de ICOS. Se observó una expresión fuerte de ICOS en todos los subtipos principales de cáncer de pulmón. Aproximadamente 29-31 % de los subtipos de NSCLC más comunes (adenocarcinoma o carcinoma de células escamosas) tuvieron una fuerte tinción para ICOS.
Tabla 2. Distribución de la tinción para ICOS en varios subconjuntos de muestras de NSCLC en función de la puntuación patológica.
Además, se empleó una metodología automatizada para medir las células positivas a ICOS, mediante el uso de un programa informático de análisis de imágenes (Strataquest de Tissuegnostics Inc., Tarzana, CA). La densidad de células positivas a ICOS en un área fija de tejido tumoral se calculó mediante la determinación de la cantidad de células positivas a ICOS en la región viable de tejido tumoral humano como se define por la región de tinción con DAPI. La densidad de células con ICOS se determinó a partir de un conjunto separado de aproximadamente 500 pacientes individuales en 4 tipos de tumor principales [NSCLC (N=100); HNSCC (N=102); cáncer de mama, todos los subtipos principales (N=94); subtipo triple negativo de cáncer de mama, TNBC (N=95); cáncer de ovario (N=94)]. Un resumen de los resultados del análisis se muestra en la Figura 5. De acuerdo con el análisis de ARNm de ICOS, los tumores de HNSCC y NSCLC tuvieron una densidad significativamente mayor de tumores positivos a ICOS en comparación con el cáncer de ovario o de mama. Aunque la expresión de ICOS es baja en el cáncer de mama, se observaron altos niveles de expresión de ICOS en el subtipo TNBC, que constituye aproximadamente 10 % del cáncer de mama. Ver la Figura 5. Este subtipo de cáncer de mama TNBC es el subtipo más agresivo con opciones de tratamiento limitadas y la mayor necesidad médica no satisfecha.
La variabilidad de paciente a paciente en la expresión de ICOS en dos cohortes diferentes de tipos tumorales que expresan ICOS altos (NSCLC) se muestra en las Figuras 6A y 6B. Un intervalo de densidad de células con ICOS se observó en NSCLC a partir de 98 muestras de cáncer de pulmón (Figura 6A) disponibles de un proveedor comercial. Se observó una diversidad similar en la expresión de ICOS mediante el uso de una cohorte clínica independiente (Figura 6B) de NSCLC (N=204).
Una IHC múltiple con ICOS, FoxP3 (un marcador de células Treg) y CD8 de las muestras de tumor humano descritas anteriormente se analizaron y cuantificaron mediante el uso de un programa informático de análisis de imágenes (Strataquest de Tissuegnostics Inc., Tarzana, CA). Una imagen representativa de la tinción múltiple para ICOS con estos marcadores de células T se muestra en la Figura 7A. Las células positivas a ICOS, que también son positivas para FOXP3, se denominan células Treg CD4 ICOS+. Las células positivas a ICOS y positivas a CD8 se denominan células CD8 ICOS+. Las positivas para ICOS pero negativas para CD8 y FoxP3 se denominan Tef CD4 ICOS+. La densidad de los diferentes subconjuntos de células T positivas a ICOS se cuantificó mediante el uso del software de análisis de imágenes como se describió anteriormente. En las muestras de pulmón (N=100) y TNBC (n=95) hubo una gran cantidad de efectores CD4 y células Treg CD4 que eran positivas a ICOS (Figura 7B). Por el contrario, no se observaron muchas células positivas a ICOS en el cáncer de ovario (n=94). Los tumores HNSCC (N=102) tuvieron una gran población de células Treg CD4 positivas a ICOS. Solo hubo una pequeña población de células T CD8 positivas a ICOS observadas en todos los tipos de tumores examinados. Estos datos sugieren que ICOS se expresa predominantemente en el compartimento CD4 en comparación con las células T CD8, y que la variación en las proporciones relativas de Treg vs Tef puede observarse en todas las indicaciones.
Para entender si la expresión de ICOS se asocia directamente con el estado de PD-L1, se evaluó la correlación entre los niveles de PDL1 y la expresión de ICOS. El análisis bioinformático sugirió que la expresión de PD-L1 y los niveles de ICOS se correlacionaron débilmente (R=0,62). Los niveles de PD-L1, ICOS y PD-1 se evaluaron por IHC múltiple. Se muestra una imagen representativa de IHC múltiple de tumor de pulmón con PD-L1 alto y bajo. Ver la Figura 8. Se evaluaron PD-L1 e ICOS en 154 tumores de NSCLC de subtipo de adenocarcinoma. Los tumores se subdividieron en tumores con PD-L1 alto o bajo sobre la base del 5 % de células positivas para PD-L1. Los resultados indican que los tumores positivos para PD-L1 tenían una mayor densidad de expresión de ICOS.
Ejemplo 3: Análisis de citometría de flu jo
Para confirmar los datos de la expresión de ICOS obtenidos por IHC y para comparar las intensidades relativas de ICOS expresado en diferentes poblaciones de células T, se evaluó la expresión de ICOS en linfocitos infiltrantes de tumores mediante el uso de citometría de flujo de múltiples colores. Se analizaron muestras de cuatro pacientes con HNSCC, tres tumores de pulmón, y cuatro pacientes con cáncer de ovario. De manera congruente con los datos de IHC, la expresión de ICOS se observó predominantemente en las células T CD4. Ver la Figura 9 (HNSCC). La frecuencia de células positivas a ICOS en la población de CD8 es muy baja en la mayoría de estos tumores. También observamos que la mayoría de las células efectoras CD4 coexpresan ICOS y PD-1. Estos datos respaldan el desarrollo de un agente terapéutico para ICOS en la clínica solo o en combinación con terapias anti-PD-1. La intensidad media de fluorescencia (MFI) de la tinción para ICOS proporciona una medida de la expresión de ICOS en las diferentes poblaciones de células T. La MFI de las células positivas a ICOS en las células Treg fue 2-3 veces mayor que los efectores CD4. Ver la Figura 9C. Debe señalarse que existen poblaciones pequeñas de efectores CD4 que tienen MFI de ICOS alto. La confirmación de la diferencia en las densidades del receptor ICOS en las células Tef contra las células Treg, junto con los datos de los ensayos de ADCC en curso para evaluar la sensibilidad diferencial a la disminución de Tef y Treg, apoyaría el desarrollo de un anticuerpo agonista con un Fc activo que podría agotar potencialmente las células Tregs.
Los estudios de traducción muestran altos niveles de células T positivas para ICOS, infiltrantes de tumores, en un subconjunto de tumores humanos (tales como HNSCC, NSCLC, etc.). La expresión de ICOS se correlaciona con la expresión de otros reguladores de puntos de control tales como CTLA-4 y PD-1. El análisis de los subconjuntos de células T mostró que la expresión de ICOS se restringe predominantemente al compartimento de células T CD4. El ICOS se expresa en las células Treg positivas para FoxP3 así como las células Tef CD4. Los estudios muestran, de acuerdo con la literatura, que el nivel de expresión de ICOS es mayor en las células Treg en comparación con las células Tef CD4.
Ejemplo 4: Generación de anticuerpos
Reactivos
Las proteínas ICOS que representan especies humanas, de ratón, rata y mono cangrejero se produjeron como fusiones Fc, homodiméricas (cadena principal de IgG1), y los ICOS-hFc humano y de ratón se usaron como antígenos para las inmunizaciones de roedores. ICOS humano-hFc incluyó los aminoácidos de ICOS humano 1 a 141 (21 a 141 en la construcción madura); ICOS de ratón-hFc incluyó los aminoácidos de ICOS de ratón 1 a 142 (21 a 142 en la construcción madura).
ICOS-Fc se generó como moléculas de fusión de Fc tanto bivalentes como monovalentes para evaluar la avidez y la unión monovalente de anticuerpos contra ICOS, respectivamente.
Para propósitos de tamizaje, se generaron líneas celulares CHO estables que sobreexpresan ICOS de ratón o humano de longitud completa (células CHO ICOS+ o "células CHO-ICOS") como las construcciones que codifican ICOS de rata o mono cangrejero de longitud completa para permitir el tamizaje después de la transfección transitoria.
Campaña de anticuerpo de roedor
Las campañas de roedor se realizaron con un anticuerpo de precisión. Los ratones (10), ratas (6), hámsteres sirios (6) y hámsteres armenios (6) se inmunizaron con hICOS-hFc o mICOS-hFc. Los hibridomas se generaron y los sobrenadantes se analizaron mediante ELISA para determinar la unión a hICOS y a mICOS, así como tamizaje múltiple mediante FACS para determinar la unión a células CHO-hICOS y CHO-mICOS. Los hibridomas se evaluaron adicionalmente para determinar la capacidad de bloquear la unión de ICOSL a las células CHO-ICOS. Los clones que dieron positivo para la unión a ICOS de ratón y humano se seleccionaron para la purificación de proteínas. Posteriormente, los anticuerpos purificados se volvieron a seleccionar en los ensayos de unión y bloqueo, y los anticuerpos que resultaron positivos se evaluaron in vitro como se describe a continuación. Todos los anticuerpos seleccionados para su investigación adicional a partir del enfoque de inmunización derivaron de fusiones de hámster armenio.
Caracterización bioquímica de los anticuerpos
Las mediciones de afinidad se llevaron a cabo mediante el uso de la tecnología de Interferometría de biocapa (BLI) (ForteBio Octet® RED96). Las afinidades monovalentes se generaron con versiones de IgG de los anticuerpos con formas monovalentes, heterodiméricas, del receptor ICOS. Las afinidades por avidez se generaron mediante el uso de IgG de longitud completa contra formas homodiméricas del receptor ICOS. Las afinidades de hICOS monomérico y bivalente de los anticuerpos de hámster seleccionados se muestran en la Tabla 3.
Tabla 3. Afinidad de anticuerpos derivados de hámster.
Además se determinó la afinidad de unión de los anticuerpos para ICOS de mono cangrejero, ratón y rata, y se encontró que los anticuerpos se unían a todas las especies con afinidad comparable. Las mediciones de reactividad cruzada se llevaron a cabo mediante el uso de tecnología BLI con paneles de anticuerpos que se analizan para determinar la unión a fusiones de ICOS-Fc humano, de ratón y mono cangrejero (formas homodiméricas, bivalentes). La Tabla 4 muestra datos de unión representativos para varios anticuerpos de hámster para ICOS humano y de mono cangrejero.
Tabla 4. Afinidades de unión bivalente de varios anticuerpos de hámster eontra ICOS-Fc humano y de mono cangrejero (cino).
Para evaluar la especificidad, se analizó individualmente la unión a células CHO ICOS+ humano y a las células CHO ICOS+ de ratón mediante citometría de flujo. Como control para analizar los anticuerpos panreactivos, se examinó, además, la tinción en células CHO que carecen de la expresión del receptor ICOS. Todos los anticuerpos seleccionados se unieron a células CHO ICOS+ humano y de ratón, y no a células CHO que carecen de la expresión de ICOS.
Para evaluar adicionalmente la especificidad de los anticuerpos anti-ICOS, los anticuerpos se analizaron para determinar la unión a otros miembros de la familia de proteínas CD28 (CD28, BTLA, PD-1 y CTLA-4). No se observó reactividad cruzada de los anticuerpos seleccionados para CD28, BTLA, PD-1 o CTLÁ4 humanos o de ratón. Específicamente, se evaluó la unión a las fusiones Fc (de CD28, BTLA, PD-1 y CTLA-4) en formas diméricas, y no se observó unión. Para un subconjunto de los miembros de la familia CD28, la proteína humana o de ratón se sobreexpresó en la superficie de las células HEK293 y CHOK1. No se observó ninguna unión del anticuerpo por encima del fondo con respecto a las células originales no transducidas.
Se encontró además que los anticuerpos no se unían a proteínas de suero abundantes ni a plaquetas o glóbulos rojos.
La unión al epítopo se llevó a cabo mediante el uso de BLI. Además, los anticuerpos se agruparon contra una fusión ICOSL-Fc (forma homodimérica, bivalente). Se encontró que todos los anticuerpos seleccionados estaban en el mismo grupo de epítopos y todos bloquearon la unión de ICOS al ligando de ICOS.
Humanización
Los anticuerpos seleccionados se humanizaron mediante la realización de estudios de homología entre las regiones del marco variable de anticuerpo de origen humano y de hámster. Se proporcionó un panel de diseños primarios para el análisis y después se produjeron anticuerpos para la comparación con el anticuerpo natural (forma de hámster o quimera). Una vez que se produjeron los paneles humanizados, los cables se caracterizaron y se clasificaron sobre la base de la afinidad y la actividad in vitro. Se ejecutaron otros diseños de humanización para reducir las vulnerabilidades de las secuencias y sitios inmunogénicos de puntuación baja que resulten en menores variaciones de secuencias. Las vulnerabilidades de las secuencias consideradas incluyeron la presencia de cisteínas libres y posibles sitios para la degradación química (desamidación de asparagina, isomerización de aspartato, metionina/triptófano y glicación de lisina no enzimática).
La afinidad de un anticuerpo humanizado que tiene las regiones variables de 37A10S713 para las formas monoméricas de ICOS de humanos, mono cangrejero y rata se midió mediante tecnología de Interferometría de biocapa (BLI) (ForteBio Octet® RED96) y las Kd se muestran en la Tabla 5. Las Kd se consideraron comparables entre las especies ya que estaban dentro de las 2 a 5 veces. La funcionalidad de la unión se confirmó evaluando la potencia de inducción de la proliferación de células T CD4+ primarias aisladas de cada especie.
Tabla 5. Afinidad de unión monovalente de 37A10S713 a ICOS humano, de mono cangrejero y rata.
Ejemplo 5: Caracterización funcional in vitro de anticuerpos anti-ICOS
Un número de ensayos in vitro basados en células se usaron para evaluar la actividad de los AcM anti-ICOS. El propósito principal fue tamizar los anticuerpos con propiedades agonistas/coestimuladoras. Dado que el sistema celular (célula primaria versus la línea celular transfectada) y el método de presentación de anticuerpos (soluble versus unido a la placa o reticulado) pueden influir en la actividad agonista, se emplearon varios formatos de ensayos diferentes. Además, también se usó un ensayo para detectar actividad superagonista no deseada (ver Suntharalingam y otros, 2006, N. Eng. J. Med., 355: 1018-28).
Los ensayos diseñados para determinar la actividad agonista/coestimuladora de los AcM anti-ICOS se realizaron en los tipos de células descritos a continuación, donde la primera señal para las células T (señal 1) fue proporcionada mediante el uso ya sea de concentraciones subóptimas de anti-CD3 o PMA o en el ensayo con PBMC con estimulación con superantígeno (SEB):
1. Ensayo de células T CD4+ humanas primarias
a. Formato de anticuerpos reticulados/unidos a la placa coestimulados con anti-CD3
b. Formato de anticuerpos solubles coestimulados con PMA
2. Ensayo de Jurkat (línea celular indicadora con construcciones de ICOS humano o de ratón transfectadas)
a. Formato unido a la placa/reticulado con anti-CD3 o PMA
b. Formato de anticuerpos solubles coestimulados con PMA
3. Ensayo con PBMC humanas
a. Formato de anticuerpos solubles con superantígeno (SEB)
Los paneles de AcM anti-ICOS de hámster se tamizaron en los ensayos anteriores para identificar anticuerpos con propiedades agonistas. Un ejemplo de la actividad agonista observada en un ensayo con el uso de células T CD4+ humanas primarias coestimuladas con anti-CD3 subóptimo con la adición del anticuerpo anti-ICOS unido a la placa se muestra en la Figura 10A para una selección de anticuerpos anti-ICOS de hámster. En este ensayo, la célula T CD4+ humana aislada de PBMC se activa con anti-CD3 unido a la placa subóptimo en presencia de anticuerpos anti-ICOS de hámster unidos a la placa (7F12, 37A10 y 16G10) en cuatro concentraciones (mg/ml). El hICOSL unido a la placa y el anti-CD28 soluble en presencia de anti-CD3 se usan como controles positivos. Se grafica el % de células divididas. En este ensayo múltiples anticuerpos anti-ICOS muestran actividad. La Figura 10B muestra los resultados del ensayo que usó anticuerpos solubles y coestimulación con PMA subóptimo. Las células T CD4+ humanas se aislaron de
PBMC mediante selección negativa y se marcaron con CFSE. Las células se activaron en placas de 96 pocilios con PMA subóptimo (0,25 ng/ml) solo o en presencia de diferentes versiones de Fc (de hámster, mG1, mG2a, mGIAgly o hG1) del anticuerpo anti-ICOS 37A10 en las concentraciones indicadas (mg/ml). Se usó un anticuerpo anti-CD28 soluble como control. La proliferación se analizó en el día 3 mediante citometría de flujo. Las versiones de IgG1 de ratón e IgG1-agly de ratón mostraron actividad en el ensayo, junto con el anticuerpo original completamente de hámster. Al menos el anticuerpo 37A10 mostró actividad agonista en ambos formatos soluble y unido a la placa. Ver las Figuras 10A y 10B.
La Figura 10C muestra la actividad agonista de los resultados del anticuerpo 37A10S713 con una IgG1 humana en el ensayo de células T CD4+ humanas primarias. Las células T CD4+ se aislaron a partir de PBMC de 4 donantes saludables, se marcaron con colorante CFSE y después se incubaron en placas recubiertas con una concentración subóptima de anti-CD3 y diversas concentraciones de 37A10S713-hIgG1 o un anticuerpo IgG1 humano control negativo (contra el virus sincitial respiratorio (RSV)). Después de 3 días, el porcentaje de células divididas se determinó mediante el uso de citometría de flujo. Los valores de EC50 variaron de 4,27 a 49,75 nM para los 4 donantes analizados. La proliferación se grafica como el porcentaje de células divididas (medido por dilución de CFSE mediante el uso de citometría de flujo) y son la media de duplicados. La Figura 10C muestra los datos de un donante representativo.
Otro ejemplo de un ensayo en el que los anticuerpos de hámster demostraron actividad agonista es el ensayo indicador de Jurkat. Se desarrolló un ensayo indicador de Jurkat en el laboratorio mediante la transducción de una construcción de expresión quimérica de hICOS-hCD28 en una línea celular reportera de NFkB Jurkat. Las células reporteras JurkathICOS-hCD28 se activaron durante 5 horas con PMA y un anticuerpo anti-ICOS soluble 37A10 de hámster eon diferentes extremos Fc en 11 concentraciones (mg/ml). Se usan anti-CD28 soluble e hICOSL-Fc como controles. Se graficó el % de células GFP+. En la Figura 11 se muestran datos representativos del ensayo indicador de Jurkat con el uso de anticuerpos anti-ICOS de hámster. La Figura 11A muestra los resultados para diferentes versiones de Fc de anticuerpo anti-ICOS 37A10 (hG1, mG1, mG2a, mG1Agly) en las concentraciones indicadas (mg/ml). Todas las versiones de Fc del anticuerpo anti-ICOS, que incluyen la versión mG1-agly (es decir, mínima función efectora de Fc) muestran actividad en este ensayo. La Figura 11B muestra los resultados para los anticuerpos humanizados 37A10S713, 37A10S715, 37A10S716 y 37A10S718 en las concentraciones indicadas (mg/ml). Los cuatro anticuerpos humanizados analizados mostraron actividad agonista en el ensayo.
Otro formato de ensayo que muestra actividad agonista es el ensayo con PBMC con la enterotoxina B estafilocóccica (SEB) como superantígeno mediante el uso de la producción de citocinas (por ejemplo, IFNg) como lectura. Este ensayo típicamente tiene una pequeña ventana de un efecto de 1,5 - 3 veces sobre la liberación de citocinas. De acuerdo con los datos de anti-PD-1 publicados, la inducción de citocinas con los anticuerpos anti-ICOS es de ~2 veces pero se observa de manera reproducible en este ensayo a través de múltiples donantes. Véase, por ejemplo, Korman y otros, 2014, Cancer Res., 2: 846-856. Un ensayo representativo se muestra en la Figura 12. Las PBMC congeladas de donantes saludables se estimularon con SEB y el anticuerpo anti-ICOS 37A10 soluble (con mG1, mG1-agly o hG1 Fc) en las concentraciones indicadas (mg/ml) durante 3 días. Los sobrenadantes se recogieron y los niveles de IFNg se midieron mediante la matriz de perlas de citocinas con el uso de citometría de flujo. Los anticuerpos anti-CD28 y de isotipo IgG1 de ratón se usaron como controles. IFNg es inducido por las PBMC después de la estimulación con SEB en presencia de anticuerpos anti-ICOS solubles. En este formato de ensayo, una versión de mG1-agly de 37A10 mostró una actividad reducida.
Para analizar cualquier posible superagonista, se empleó un ensayo en el que las células T CD4+ humanas primarias se incubaron con anticuerpos anti-ICOS solubles o unidos a la placa en ausencia de una señal 1 mediante el uso de un anticuerpo superagonista anti-CD28 conocido como control positivo. Las células T CD4 humanas activadas en ausencia de anti-CD3 proliferarán solo cuando se tratan con un anticuerpo superagonista anti-CD28 (clon CD28.2/5D10) en forma soluble, pero no cuando se tratan con ICOSL-Fc o anticuerpo anti-ICOS 37A10 (versiones Fc de hámster y hG1) o un anticuerpo anti-CD28 no superagonista (CD28.2). Ninguno de los AcM anti-ICOS analizados mostró actividad superagonista en este ensayo. En la Figura 13 se representan datos representativos.
Se establece bien que ICOS puede señalizar a través de la vía de señalización de AKT (revisado en Simpson y otros, 2010, Curr. Opin. Immunol., 22: 326-332). La capacidad del anticuerpo anti-ICOS para inducir la señalización a través de AKT se evaluó como un medio adicional para demostrar la actividad agonista del anticuerpo.
Las células T CD4 aisladas a partir de PBMC humanas se estimularon durante 24 horas con anti-CD3/anti-CD28, y después se dejaron en reposo durante 24 horas en medio de cultivo. Las células se incubaron después con anti-ICOS 37A10-mG2a, hICOSL-mG2a Fc o PBS durante 2, 5, 15 o 30 minutos con o sin anticuerpo reticulado anti-IgG de ratón. Después de la incubación, las células se fijaron, permeabilizaron y después se tiñeron con un anticuerpo anti-fosfo-AKT. El porcentaje de células positivas a pAKT se analizó por citometría de flujo.
Como se muestra en las Figuras 14A-B, pAKT se indujo en células T CD4 después del tratamiento con 37A10-mG2a con una cinética similar en comparación con el tratamiento con hICOSL-mG2a. La inducción de la señalización de pAKT se observó solamente en presencia del agente de reticulación secundario.
Ejemplo 6: Caracterización funcional in vivo de anticuerpos anti-ICOS
Los anticuerpos seleccionados de los ensayos de tamizaje descritos anteriormente se evaluaron in vivo mediante el uso de modelos tumorales singénicos.
El modelo de fibrosarcoma Sa1N (Ostrand-Rosenberg, 2001, Curr. Protoc. Immunol., Capítulo 20) puede usarse para evaluar anticuerpos anti-ICOS in vivo. El perfil inmunitario del modelo de ratón Sa1/N muestra que está altamente infiltrado con células T CD4, y que las células T CD4 expresan altos niveles de ICOS. Este perfil inmunitario es similar a los perfiles inmunitarios de las muestras de pacientes con NSCLC en los que observamos altos niveles de infiltración de c D4, con la expresión de ICOS restringida en gran medida al compartimento CD4.
Un segundo modelo usado para evaluar la eficacia de los anticuerpos anti-ICOS es el modelo de carcinoma de colon CT26 (Wang y otros, 1995, J. Immunol., 9: 4685-4692). El perfil inmunitario del modelo de ratón CT26 mostró altos niveles de infiltración de CD8. Se observó la expresión de ICOS en el subconjunto de células T CD8 en este modelo. Una pequeña proporción de muestras de NSCLC humanas demuestra de manera similar la expresión de ICOS en las células T CD8.
Formatos de anticuerpos para la evaluación in vivo
La IgG1 humana (hIgG1) puede unirse a múltiples receptores de Fc, que incluyen una fuerte unión a los receptores de Fc activadores que son capaces de unirse de forma cruzada con el receptor y mediar ADCC y CDC. Dada la capacidad de unirse a los receptores de Fc activadores, hIgG1 es típicamente capaz de agotar las células que expresan un alto nivel del objetivo. El equivalente de ratón más cercano a hIgG1 es IgG2a de ratón (mIgG2a). Por lo tanto, como un ejemplo, los experimentos in vivo para evaluar un anticuerpo agonista de ICOS con capacidad de agotamiento utilizarían mIgG2a para imitar las propiedades de hIgG1.
La IgG4 humana (hIgG4) se utiliza en situaciones terapéuticas donde no se desea el agotamiento, aunque hIgG4 es capaz de cierto nivel de agotamiento. Se encuentra aproximadamente, aunque no perfectamente, alineada con la IgG1 de ratón (mIgG1), que casi se une exclusivamente a los receptores FcyRII inhibidores, y por lo tanto es capaz de unirse de forma cruzada, aunque no es particularmente competente, en el agotamiento.
Con respecto a los anticuerpos anti-ICOS, los anticuerpos de hámster se evaluaron inicialmente in vivo como Ac de hámster completos. Los Ac de hámster tienen una IgG1 de hámster, que tiene características de unión a FcR similar a mIgG1. Los anticuerpos de hámster de interés se clonaron como quimeras con regiones Fc de ratón, ya sea como mIgG2a o mIgG1.
Modelo de fibrosarcoma Sa1N
El modelo in vivo principal usado para seleccionar candidatos a anticuerpos anti-ICOS para determinar su eficacia es el modelo de fibrosarcoma Sa1N. Por lo tanto, los anticuerpos seleccionados de los ensayos de tamizaje descritos anteriormente se evaluaron en el modelo Sa1N. En estudios iniciales, varios anticuerpos de hámster (clones 7F12, 36E10, 37A10, 16G10 y 35A9) demostraron una actividad antitumoral robusta cuando se administraron como agentes únicos a una dosis de 8 mg/kg en el modelo Sa1N. Ver la Figura 15. Se inyectaron células de fibrosarcoma Sa1N (1x106) por vía s.c. en el flanco derecho de ratones A/J vírgenes (6-8 semanas de edad, hembras). Cuando los volúmenes tumorales alcanzaron 50-100 mm3 en el día 7, los ratones se aleatorizaron. Los ratones recibieron dosis de anticuerpo anti-ICOS de hámster (7F12, 36E10, 37A10, 16G10 y 35A9) o anticuerpo de isotipo IgG de hámster por vía i.p. en los días 7, 10, 14 y 17. El crecimiento tumoral se monitoreó dos veces por semana. N=10.
Un rasgo potencialmente beneficioso de un agente inmunoterapéutico contra el cáncer es la capacidad de levantar una respuesta inmunitaria sostenida y duradera contra el tumor. Se determinó la capacidad de un ratón tratado previamente con un anticuerpo anti-ICOS para rechazar posteriormente un tumor. Los ratones se trataron a 8 mg/kg de anticuerpo en los días 7, 10, 14 y 17. Posteriormente, los ratones que estaban libres de tumor se volvieron a implantar con un tumor el día 60. Todos los ratones tratados previamente con el anticuerpo anti-ICOS 7F12 (n=7) rechazaron el tumor recién implantado, en contraste con los ratones vírgenes (n=10) en los que los tumores crecieron en todos los ratones. Ver la Figura 16.
Los anticuerpos de hámster se clonaron como anticuerpos quiméricos con regiones Fc de ratón (mG1 o mG2a) para permitir la evaluación de la contribución de la función efectora de Fc a la actividad in vivo. Los ratones recibieron un total de 4 dosis bisemanalmente de 4 mg/kg de anticuerpo a partir del día 11. El anticuerpo anti-CTLA-4 se incluyó como un control positivo. El experimento de tamizaje inicial se realizó a una dosis de 4 mg/kg y se observó eficacia con ambos formatos mG1 y mG2a. En la Figura 17 se muestran datos representativos para uno de los anticuerpos de hámster, 37A10.
Modelo tumoral singénico CT26 de colon
El modelo tumoral singénico CT26 de colon se usó para evaluar la actividad de un solo agente, así como también la
terapia de combinación con anticuerpo anti-PD-1.
En el modelo CT26, varios de los anticuerpos de hámster anti-ICOS (por ejemplo, 7F12, 35A9, 36E10, 37A10) exhibieron actividad como agente único. Ver la Figura 18. El modelo CT26 también se usó para evaluar la posible actividad de combinación con anti- PD-1. Cuando los anticuerpos anti-ICOS se combinaron con un anticuerpo anti-PD-1, la eficacia antitumoral mejoró notablemente. Los ratones portadores de tumores CT26 se trataron bisemanalmente (4 dosis a partir del día 3) con anticuerpos anti-ICOS de hámster (8 mg/kg) solos o en combinación con anticuerpo anti-PD-1 (8 mg/kg). Debemos señalar que la combinación de anticuerpo anti-PD-1 con anti-ICOS 37A10 dio como resultado 9/10 ratones sin tumor. Ver la Figura 18.
Ejemplo 7: El agotamiento selectivo de las células Tregs contribuye a la eficacia de los anticuerpos anti-ICOS
Se realizaron estudios ex vivo para caracterizar el estado de los infiltrados de células inmunitarias después de la dosificación con anticuerpos anti-ICOS. Los estudios en el modelo Sa1N mostraron una disminución en la población de células Tregs después del tratamiento con 7F12. Los ratones recibieron dos dosis de anti-ICOS de hámster 7F12, 7F12-mG1 o 7F12-mG2a a 8 mg/kg en los días 7 y 10. Los tumores y los bazos se extrajeron y analizaron en el día 12. Hubo una reducción marcada en las células Tregs pero no en las células Tef, pero poco impacto en las poblaciones de células T en los órganos linfoides como el bazo o ganglios linfáticos. Ver la Figura 19.
También se han observado resultados similares con otros anticuerpos anti-ICOS, como 37A10. Ver la Figura 20. Los ratones recibieron dos dosis de anticuerpos anti-ICOS a 8 mg/kg en los días 7 y 10. Los tumores se extrajeron y analizaron en el día 12. Una reducción similar en la población de células Tregs también se ha observado en el modelo CT26 después de la dosificación con el anticuerpo anti- ICOS.
En conjunto, los estudios de TIL (linfocitos infiltrantes de tumores) respaldan la hipótesis de que las células Treg se agotan selectivamente por los anticuerpos anti-ICOS descritos en la presente descripción, sin el agotamiento correspondiente de las poblaciones de células Tef, y específicamente en los tumores pero no en otros órganos o en la periferia.
Para demostrar formalmente la contribución del sistema inmunitario a la eficacia del anticuerpo anti-ICOS, se realizaron experimentos de agotamiento celular en el contexto del modelo tumoral, Sa1N. Específicamente, los ratones se agotaron de células T CD8, células T CD4 o una combinación de células T CD4 CD8. En los días 6 y 13 después de la implantación del tumor, los ratones se trataron con anti-CD8, anti-CD4, anti-CD4 anti-CD8 o anticuerpo Ig control (n=10 por grupo). El anticuerpo anti-ICOS 7F12 se administró a 8 mg/kg de anticuerpo en los días 7, 10, 14 y 17. El crecimiento tumoral se monitoreó dos veces por semana.
Se observó una marcada reducción en la eficacia antitumoral de 7F12 cuando los ratones se agotaron de células T CD4, CD8 o CD4 CD8. Ver la Figura 21.
Ejemplo 8: Reducción selectiva de células Treg por un anticuerpo anti-ICOS humanizado
Se incubaron PBMC humanas a 37 °C con IL-2 humana recombinante a 100 ng/ml durante 48 horas en una incubadora humidificada con 5 % de CO2. Después de 48 horas, se agregó el anticuerpo 37A10S713 a las concentraciones indicadas. El anticuerpo se preparó como diluciones en serie de 10 veces en medios de cultivo que contenían IL-2. La mezcla de anticuerpo/célula se dejó incubar unas 72 horas adicionales. Después de la incubación, las células se tiñeron para CD3, CD4, CD8, CD25 y FoxP3 mediante métodos estándar y se analizaron por citometría de flujo. La cuantificación de células Treg (CD4+ CD25+ FoxP3+) y Tef (CD4+ CD25- FoxP3-) se llevó a cabo para cada concentración y tratamiento. Los datos se normalizaron en por ciento de cada subconjunto en un grupo tratado con trastuzumab para cada concentración.
Los resultados de ese experimento se muestran en la Figura 22A. El anticuerpo 37A10S713 provocó una reducción de las células Treg dependiente de la dosis. Como se muestra en la Figura 22B, las células Tef y Treg expresaban niveles similares de ICOS después de cinco días de tratamiento con IL-2.
Ejemplo 9: Nuevo reto tumoral después del tratam iento con anticuerpo anti-ICOS
Los ratones A/J hembras de seis a ocho semanas de edad se inocularon por vía subcutánea en el flanco derecho con 1x106 células Sa1/N en 100 ml de PBS con jeringas de tuberculina con agujas calibre 27. El crecimiento tumoral se monitoreó y el día 7, los animales se redistribuyeron en nuevas cajas después de normalizar el volumen tumoral promedio a 100-150 mm3 para cada grupo de tratamiento. Se incluyeron diez ratones en cada grupo de tratamiento. Los animales se trataron con anticuerpos mediante inyecciones intraperitoneales de 0,25 mg/kg de anticuerpo anti ICOS (VH y VL de 37A10S713 (SEQ ID NO: 60 y 61) con una IgG2a de ratón) o un control de isotipo. La dosificación se realizó en el día 7 para una dosis única o los días 7 y 14 para 2 dosis. El crecimiento tumoral y los pesos corporales de los animales se controlaron dos veces por semana. Los ratones se sacrificaron cuando los volúmenes tumorales alcanzaron -2000 mm3 o si existiera signos de malestar clínico como ulceraciones graves conforme al protocolo del IACUC.
La Figura 23, panel izquierdo, muestra el volumen tumoral en ratones administrados con una única dosis de anticuerpo anti-ICOS (n=10) o dos dosis de anticuerpo anti-ICOS (n=10).
Se realizó un experimento de un nuevo reto tumoral para evaluar la durabilidad de la respuesta. Los 7 ratones retados previamente con células Sa1/N cuyos tumores se erradicaron con una dosis única o dos dosis de 0,25 mg/kg de anticuerpo 37A10S713-mIgG2a se volvieron a retar en el flanco contralateral con células Sa1/N 10 semanas después del reto tumoral inicial. Como un control, los ratones vírgenes también se retaron con células Sa1/N (N=10). Los animales se evaluaron para determinar el crecimiento tumoral bisemanalmente.
Como se muestra en la Figura 23, panel derecho, ninguno de los ratones que habían tenido previamente tumores erradicados con tratamiento con anticuerpo anti-ICOS mostró crecimiento tumoral en el experimento de nuevo reto.
Ejemplo 10: Expresión de ligando de ICOS (ICOSL) en ratones portadores de tumores Sa1/N y monos cangrejeros administrados con anticuerpo anti-ICOS
Se inocularon ratones A/J hembras de ocho semanas de edad con células tumorales Sa1/N en el día cero. En el día 7, cuando los tumores alcanzaron ~100 mm3, los ratones se administraron con una dosis única de 5 o 100 mg i.p. de anticuerpo 37A10 con una IgG1 o IgG2a de ratón o un anticuerpo control de isotipo. Los ratones se administraron con una dosis posterior de anticuerpo en el día 10, y se extrajeron los tejidos (sangre, bazo y tumor) en el día 12. Después del procesamiento de los tejidos, las células se incubaron con un bloqueador de Fc al 5 % (5 % de suero normal reconstituido de rata, humano y ratón, 5 % de suero fetal bovino, 0,1 mg/ml de Ac bloqueador de Fc 2.4G2, 0,01 % de azida sódica) durante 15 min en hielo en tampón de tinción de flujo (FSB: 5 % de FBS, 0,01 % de azida sódica en PBS 1x). Después del bloqueo de Fc, las células se tiñeron con un cóctel de tinción extracelular (anti-CD45-BV510, anti-CD19-BV605, anti-ICOSLPE, colorante de viabilidad fijable eFluor 780) en FSB durante 1 hora en hielo. Las células se lavaron dos veces con FSB. Las células se fijaron con solución de fijación/permeabilización durante 30 min en hielo. Las células se lavaron dos veces con tampón de permeabilización 1x, después se tiñeron con un cóctel de tinción intracelular (anti-CD3-BUV496, en tampón de permeabilización durante 1 hora en hielo. Las células se lavaron dos veces con tampón de permeabilización, después se resuspendieron en solución de FSB PFA al 1,5 %. Las células se examinaron en BD Fortessa y los datos se analizaron mediante el uso del programa informático FlowJo.
Las muestras se analizaron en un citómetro de flujo BD Fortessa. Para el análisis de la expresión de ICOSL, se analizó la tinción de ICOSL en células B CD45+ CD3- CD19+ viables. Se informan las intensidades de fluorescencia (MFI) promedio de ICOSL.
El anticuerpo 37A10S713 con una IgG1 humana se administró a través de una infusión intravenosa durante 1 hora a tres monos cangrejeros por grupo de dosis (0,5 mg/kg, 5 mg/kg, 75 mg/kg y vehículo solo). Se obtuvo sangre previo a la primera dosis (día 1), 48 horas después de la primera dosis (día 3), 7 días después de la primera dosis (previo a la segunda dosis; día 8) y 48 horas después de la segunda dosis (día 10). En las muestras de 95 pl de sangre total primero se bloqueó el Fc con TruStain humano 5mE durante 15 min en hielo. Después del bloqueo de Fc, se agregó 100 pl de una mezcla de anticuerpos que contenía anti-CD3 FITC, anti-CD20 PE, anti-CD14 PE/Cy7, colorante de viabilidad e780 y cinoICOSFc DyLight 650. La mezcla de sangre y anticuerpos se incubó en hielo durante 60 min. Después de la incubación, las muestras se centrifugaron a 500 xg durante 5 min. El sobrenadante se decantó y las muestras se resuspendieron en 200 pl de tampón de tinción de FACS. Las etapas de lavado se repitieron tres veces, con resuspensión final en 200 ml de tampón de tinción paraformaldehído al 0,1 %.
Las muestras se analizaron en un citómetro de flujo BD Fortessa. Para el análisis de la expresión de ICOSL, se analizó la tinción de ICOSL por cinoICOS-Fc marcado con DyLight 650 en células B CD3- CD20+ viables. Las MFI de ICOSL se normalizaron con respecto al vehículo en cada punto de tiempo.
Los resultados de esos experimentos se muestran en las Figuras 24A y 24B. Se observó un aumento dependiente de la dosis en la expresión de ICOS-L en todos los tratamientos con anticuerpos y en las dosis y en todos los tejidos con relación a los ratones tratados con control de isotipo. Ver la Figura 24a . Similarmente, se observó un aumento dependiente de la dosis en la expresión de ICOS-L en todos los puntos de tiempo para los grupos de dosis de 0,5 y 5 mg/kg en monos cangrejeros con relación a las muestras del vehículo y previas al estudio. Ver la Figura 24B. También se observó inducción de ICOSL en el grupo de 75mg/kg, pero la expresión observada puede ser una subrepresentación debido a una posible interferencia del fármaco ya que el anticuerpo anti-ICOS es capaz de unirse al reactivo de tinción (cinoICOS-Fc).
El acoplamiento de ICOS y el objetivo puede evaluarse, además, según se mide mediante el ensayo de disponibilidad de receptores, por ejemplo, de la siguiente manera. Los ratones vírgenes se inyectaron por vía i.p. con el control de isotipo mIgG2a a 2,5 mg/kg o 37A10S713 con una IgG2a de ratón a 2,5 mg/kg. En varios puntos de tiempo posteriores a la inyección, se extrajo sangre en microtubos recubiertos con EDTA a mediante una extracción submandibular.
En la sangre completa se bloqueó el Fc mediante el uso de TruStain de ratón (BioLegend) durante 5 minutos en hielo. Después de la incubación, se agregaron 100 pl de la mezcla de anticuerpos de tinción extracelular concentrada 2x a
cada muestra durante 30 minutos a 4 °C. Las muestras se centrifugaron y se fijaron y permeabilizaron en tampón de tinción de Foxp3 (eBioSciences) durante 30 minutos a 4 °C. Después las muestras se centrifugaron y se resuspendieron en tinción de anticuerpo intracelular durante 30 minutos a 4 °C. Las muestras se centrifugaron y se resuspendieron en PFA al 0,1 %. Las muestras se analizaron en un BD LSRII Fortessa. Las células Tregs se identificaron como CD45+ CD3+ CD4+ Foxp3+ vivas. Las células Tef se identificaron como CD45+ CD3+ CD4+ Foxp3-vivas. Las células CD8+ se identificaron como CD45+ CD3+ CD8+ vivas. Se usó 37A10S713-mG2a marcado de manera fluorescente (conjugado con DyLight 650) como el reactivo de tinción para ICOS. La disponibilidad del receptor en cada punto de tiempo se determinó mediante el uso de la siguiente fórmula:
% de Receptor disponible en el tiempo t =((MFI de 37A10S713-mG2aDy650 en el tiempo t-MFI de isotipo de Dy650 en el tiempo t))/((MFI de 37A10S713-mG2aDy650preestudio-MFI de isotipoDy650preestudio)) x 100
Los resultados mostraron que después de la administración de anticuerpos anti-ICOS, los niveles de receptor libre son indetectables, lo que sugiere que los anticuerpos saturan todas las moléculas de ICOS objetivo disponibles.
Ejemplo 11: Inducción de quim iocinas y citocinas de Th-1 después del tratamiento con anticuerpo anti-ICOS
Los tumores frescos de pulmón de pacientes se obtuvieron 24 horas después de la cirugía. El tejido blando se eliminó manualmente del tumor y el tumor sólido restante se embebió en agar de baja fusión al 4 % en un molde contenedor y se permitió solidificar en hielo. El tumor incorporado en gel se cortó con vibrátomo (Leica) (velocidad: 2, frecuencia: 9) para generar cortes con un grosor de 300 mm. Si el tumor era demasiado blando y no podía cortarse por el vibrátomo, el tejido se cortó manualmente con una cuchilla.
Los cortes tumorales se colocaron en un filtro transwell de 40 mm (Millipore) (~1 corte/pocillo) y la unidad se movió a los pocillos de una placa de 6 pocillos, que contenía 1,5 ml de medio de cultivo tisular (RPMI completo 1640/AIM-V). El tratamiento adecuado se añadió después al medio del pocillo correspondiente. Los tratamientos incluyeron 10 mg/ml de anti-RSV hIgG1 (Lake Pharma, lote núm. 3086-849598) como un control de isotipo, anticuerpo 37A10S713 a 10 mg/ml con una IgG1 humana (SEQ ID NO: 188 y 189) o anticuerpo anti-PD-1 (IgG4) a 10 mg/ml. Las placas de replicación se prepararon para varios puntos de tiempo, en el intervalo de 6-72 horas. Las placas se colocaron en una incubadora a 37 °C, 5 % de CO2.
En los puntos de tiempo deseados, los cortes tumorales se recogieron y sumergieron en RNALater (Ambion). El ARN se extrajo con un Mini kit RNeasy (Qiagen, cat#74106) de acuerdo con las instrucciones del fabricante. Después de la extracción de ARN, se usó 1 mg de ARN para la transcripción inversa mediante el uso de un kit de síntesis de ADNc Bio-Rad iScript (cat#170-8891). El producto de RT se diluyó 1 a 7 y se usaron 3 ml para cada reacción de qPCR. La qPCR se llevó a cabo mediante el uso de la mezcla TaqMan Gene Expression Master Mix de Thermo Fisher Scientific (cat#4369016) con el uso de un sistema en tiempo real de Bio-Rad. Los ensayos TaqMan usados se enumeran en la Tabla 6.
La expresión se normalizó con respecto a CD45, donde el cambio en veces se calculó como:
Tabla 6. Ensayos TaqMan de quimiocinas y citocinas
(continuación)
Los resultados de ese experimento se muestran en la Figura 25. En el punto de tiempo de 6 horas con el tumor pulmonar 1, el anticuerpo anti-ICOS dio como resultado un aumento de la expresión de GZMa, GZMb, CSF2, IL2, CXCL9, CXCL10, CXCL11 y CXCL13. El anticuerpo anti-PD-1 también aumentó la expresión de GZMa, GZMb, CSF2, CXCL9 y CXCL10, aunque en menor medida, y mostró un aumento similar en CXCL11. Para el tumor pulmonar 2 en el punto de tiempo de 24 horas, el tratamiento con anticuerpo anti-ICOS mostró un aumento sostenido en CXCL11, y alguna continua elevación de IL2, CXCL9 y CXCL10. El anticuerpo anti-PD-1 sólo mostró una ligera elevación en CXCL11 a las 24 horas.
Ejemplo 12: Inducción del ligando de NKp46 en células Treg después del tratamiento con anticuerpo anti-ICOS agonista
Las células mononucleares de sangre periférica se aislaron a partir de donantes humanos sanos (Research Blood Components) mediante el uso de centrifugación con Ficoll (GE Life Sciences), se congelaron en BamBanker (Wako-Chem) y se almacenaron a -150 °C hasta su uso. Las PBMC se incubaron con anticuerpo anti-ICOS soluble y anti-CD3 humano unido a la placa (1 mg/ml de recubrimiento, Biolegend, OKT3) a 37 °C en RPMI (Gibco) suplementado con suero fetal bovino al 10 % (Sigma-Aldrich) y 1 % de penicilina/estreptomicina (Gibco). En el ensayo se analizaron tres anticuerpos: el anticuerpo agonista fuerte 37A10S713, un anticuerpo agonista débil, y un anticuerpo antagonista débil. Después de cuatro días, las PBMC se rasparon suavemente de las placas y se lavaron con DPBS (Gibco) que contenía suero fetal bovino al 1 %, azida sódica al 0,05 % (Ricco) y EDTA 2 mM (Ambion). Las células se bloquearon después con FcX TruStain Humano (Biolegend). Para detectar el ligando de NKp46, las células se incubaron con 2 mg/ml de NKp46-hIgG1 Fc (R&D Systems, 1850-NK). Se detectó NKp46-hIgG1 Fc unido a las células mediante el uso de un anti-IgG humano conjugado con PE (Biolegend, policlonal). Las células se bloquearon de nuevo con FcX TruStain humano y después se tiñeron con anti-CD56 humano (Biolgend, Violeta brillante 711, HCD56), anti-CD16 humano (Biolegend, Violeta brillante 785, 3G8), anti-CD4 humano (Biolegend, Violeta brillante 510, OKT4), anti-CD8 humano (BD Biosciences, BUV395, RPA-T8), anti-CD25 humano (Biolegend, Violeta brillante 605, BC96), y colorante de viabilidad fijable (eBioscience, eFluor 780). Después de la tinción, las células se fijaron y permeabilizaron con Conjunto de Foxp3 / tampón de tinción para factor de transcripción (eBioscience). Después de la permeabilización, las células se tiñeron intracelularmente con anti-CD3 humano (BD Biosciences, PE-CF594, UCHT1) y anti-Foxp3 humano (eBioscience, APC, PCH101). Las células se fijaron después en paraformaldehído (Alfa Aesar). Los datos se adquirieron en un BD LSRII Fortessa y se analizaron en el programa informático FlowJo v10.1.
Los resultados de ese experimento se muestran en la Figura 26 y la Figura 27. El tratamiento con el anticuerpo agonista anti-ICOS 37A10S713 resultó en una inducción fuerte del ligando de NKp46 en células Treg de tres donantes diferentes. Ver las Figuras 26A-F (las Figuras 26A-B muestran datos del donante 1, las Figuras 26C-D muestran datos del donante 2 y las Figuras 26E-F muestran datos del donante 3). La inducción del ligando de NKp46 en las células Tef no fue tan fuerte como en las células Treg. Ver las Figuras 26A-F. Además, el tratamiento con el anticuerpo agonista anti-ICOS 37A10S713 conduce a la pérdida de CD16 (reducción de CD16) en células NK, lo que sugiere la activación de las células NK. Ver la Figura 27.
Sin pretender estar sujetos a ninguna teoría en particular, se postula que el anticuerpo agonista anti-ICOS 37A10S713 aumenta significativamente los niveles del ligando de NKp46 en las células Treg y también activa las células NK, lo que conduce a un agotamiento selectivo de las células Tregs
LISTADO DE SECUENCIAS
<110> JOUNCE THERAPEUTICS, INC.
<120> ANTICUERPOS CONTRA ICOS
<130> 01140-0001-00PCT
<140> PCT/US16/23524
<141> 22-03-2016
<150> US 62/137.034
<151> 23-03-2015
<150> US 62/147.484 <151> 14-04-2015 <150> US 62/156.588 <151> 04-05-2015 <150> US 62/242.489 <151> 16-10-2015 <150> US 62/255.635 <151> 16-11-2015 <160> 191
<170> Patent In versión 3.5 <210> 1
<211> 199
<212> PRT
<213> Homo sapiens <400> 1
Met Lys Ser Gly Leu Trp Tyr Phe Phe Leu Phe Cys Leu Arg lie Lys
1 5 10 15
Val Leu Thr Gly Glu lie Asn Gly Ser Ala Asn Tyr Glu Met Phe lie
20 25 30
Phe His Asn Gly Gly Val Gln lie Leu Cys Lys Tyr Pro Asp lie Val
35 40 45
Gln Gln Phe Lys Met Gln Leu Leu Lys Gly Gly Gln lie Leu Cys Asp 50 55 60
Leu Thr Lys Thr Lys Gly Ser Gly Asn Thr Val Ser lie Lys Ser Leu
65 70 75 80
Lys Phe Cys His Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu 85 90 95
Tyr Asn Leu Asp His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser
100 105 110
lie Phe Asp Pro Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu
115 120 125
His lie Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro 130 135 140
lie Gly Cys Ala Ala Phe Val Val Val Cys lie Leu Gly Cys lie Leu
145 150 155 160
lie Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro 165 170 175
Asn Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser
180 185 190
Arg Leu Thr Asp Val Thr Leu
195
<210>2
<211 > 179
<212> PRT
<213> Homo sapiens
<400>2
Glu lie Asn Gly Ser Ala Asn Tyr Glu Met Phe lie Phe His Asn Gly
1 5 10 15
Gly Val Gln lie Leu Cys Lys Tyr Pro Asp lie Val Gln Gln Phe Lys
20 25 30
Met Gln Leu Leu Lys Gly Gly Gln lie Leu Cys Asp Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Thr Val Ser lie Lys Ser Leu Lys Phe Cys His 50 55 60
Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Tyr Asn Leu Asp
65 70 75 80
His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser lie Phe Asp Pro 85 90 95
Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu His lie Tyr Glu
100 105 110
Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro lie Gly Cys Ala
115 120 125
Ala Phe Val Val Val Cys lie Leu Gly Cys lie Leu lie Cys Trp Leu 130 135 140
Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro Asn Gly Glu Tyr
145 150 155 160
Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser Arg Leu Thr Asp 165 170 175
Val Thr Leu
<210>3
<211> 200
<212> PRT
<213> Mus musculus
<400>3
Met Lys Pro Tyr Phe Cys Arg Val Phe Val Phe Cys Phe Leu lie Arg
1 5 10 15
Leu Leu Thr Gly Glu lie Asn Gly Ser Ala Asp His Arg Met Phe Ser
20 25 30
Phe His Asn Gly Gly Val Gln lie Ser Cys Lys Tyr Pro Glu Thr Val
35 40 45
Gln Gln Leu Lys Met Arg Leu Phe Arg Glu Arg Glu Val Leu Cys Glu 50 55 60
Leu Thr Lys Thr Lys Gly Ser Gly Asn Ala Val Ser lie Lys Asn Pro
65 70 75 80
Met Leu Cys Leu Tyr His Leu Ser Asn Asn Ser Val Ser Phe Phe Leu 85 90 95
Asn Asn Pro Asp Ser Ser Gln Gly Ser Tyr Tyr Phe Cys Ser Leu Ser
100 105 110
lie Phe Asp Pro Pro Pro Phe Gln Glu Arg Asn Leu Ser Gly Gly Tyr
115 120 125
Leu His lie Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Leu Trp Leu 130 135 140
Pro Val Gly Cys Ala Ala Phe Val Val Val Leu Leu Phe Gly Cys lie
145 150 155 160
Leu H e H e Trp Phe Ser Lys Lys Lys Tyr Gly Ser Ser Val His Asp 165 170 175
Pro Asn Ser Glu Tyr Met Phe Met Ala Ala Val Asn Thr Asn Lys Lys
180 185 190
Ser Arg Leu Ala Gly Val Thr Ser
195 200
<210>4
<211> 180
<212> PRT
<213> Mus musculus
<400> 4
Glu lie Asn Gly Ser Ala Asp His Arg Met Phe Ser Phe His Asn Gly
1 5 10 15
Gly Val Gln lie Ser Cys Lys Tyr Pro Glu Thr Val Gln Gln Leu Lys
20 25 30
Met Arg Leu Phe Arg Glu Arg Glu Val Leu Cys Glu Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Ala Val Ser lie Lys Asn Pro Met Leu Cys Leu 50 55 60
Tyr His Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Asn Asn Pro Asp
65 70 75 80
Ser Ser Gln Gly Ser Tyr Tyr Phe Cys Ser Leu Ser lie Phe Asp Pro 85 90 95
Pro Pro Phe Gln Glu Arg Asn Leu Ser Gly Gly Tyr Leu His lie Tyr
100 105 110
Glu Ser Gln Leu Cys Cys Gln Leu Lys Leu Trp Leu Pro Val Gly Cys
115 120 125
Ala Ala Phe Val Val Val Leu Leu Phe Gly Cys lie Leu lie lie Trp 130 135 140
Phe Ser Lys Lys Lys Tyr Gly Ser Ser Val His Asp Pro Asn Ser Glu
145 150 155 160
Tyr Met Phe Met Ala Ala Val Asn Thr Asn Lys Lys Ser Arg Leu Ala 165 170 175
Gly Val Thr Ser
180
<210>5
<211 > 199
<212> PRT
<213> Macaca fascicularis
<400>5
Met Lys Ser Gly Leu Trp Tyr Phe Phe Leu Phe Cys Leu His Met Lys
1 5 10 15
Val Leu Thr Gly Glu lie Asn Gly Ser Ala Asn Tyr Glu Met Phe lie
20 25 30
Phe His Asn Gly Gly Val Gln lie Leu Cys Lys Tyr Pro Asp lie Val
35 40 45
Gln Gln Phe Lys Met Gln Leu Leu Lys Gly Gly Gln lie Leu Cys Asp 50 55 60
Leu Thr Lys Thr Lys Gly Ser Gly Asn Lys Val Ser lie Lys Ser Leu
65 70 75 80
Lys Phe Cys His Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu 85 90 95
Tyr Asn Leu Asp Arg Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser
100 105 110
lie Phe Asp Pro Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu
115 120 125
His lie Tyr Glu Ser Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro 130 135 140
lie Gly Cys Ala Thr Phe Val Val Val Cys lie Phe Gly Cys lie Leu
145 150 155 160
lie Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Thr Val His Asp Pro 165 170 175
Asn Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser
180 185 190
Arg Leu Thr Gly Thr Thr Pro
195
<210>6
<211 > 179
<212> PRT
<213> Macaca fascicularis
<400>6
Claims (18)
1. Un anticuerpo monoclonal aislado que se une a ICOS, en donde el anticuerpo comprende (a) HCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 62; (b) HCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 63; (c) HCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 64; (d) LCDR1 que comprende la secuencia de aminoácidos de la SEQ ID NO: 65; € LCDR2 que comprende la secuencia de aminoácidos de la SEQ ID NO: 66; y (f) LCDR3 que comprende la secuencia de aminoácidos de la SEQ ID NO: 67, y en donde el anticuerpo comprende una región constante de cadena pesada de IgG1 humana o una región constante de cadena pesada de IgG3 humana.
2. El anticuerpo de conformidad con la reivindicación 1, que comprende una región variable de cadena pesada (Vh) y una región variable de cadena ligera (Vl), en donde
la Vh es al menos 90 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 60 y la Vl es al menos 90 % idéntica a la secuencia de aminoácidos de la SEQ ID NO: 61.
3. El anticuerpo de conformidad con la reivindicación 1, que comprende una región variable de cadena pesada (Vh) y una región variable de cadena ligera (Vl), en donde la Vh comprende la secuencia de aminoácidos de la SEQ ID NO: 60 y la Vl comprende la secuencia de aminoácidos de la SEQ ID NO: 61.
4. El anticuerpo de conformidad con cualquiera de las reivindicaciones anteriores, en donde el anticuerpo comprende una región constante de cadena pesada de IgG1 humana.
5. El anticuerpo de conformidad con cualquiera de las reivindicaciones anteriores, en donde el anticuerpo es un anticuerpo quimérico o un anticuerpo humanizado.
6. El anticuerpo de conformidad con la reivindicación 1, en donde la cadena pesada comprende la secuencia de aminoácidos de la SEQ ID NO: 188 o una variante biológicamente activa de esta en donde uno o más residuos de aminoácidos se añaden o eliminan en el extremo N o C terminal del polipéptido; y la cadena ligera comprende la secuencia de aminoácidos de la SEQ ID NO: 189 o una variante biológicamente activa de esta en donde uno o más residuos de aminoácidos se añaden o eliminan en el N o C terminal del polipéptido.
7. El anticuerpo de conformidad con la reivindicación 1, en donde la cadena pesada comprende la secuencia de aminoácidos de la SEQ ID NO: 188 y la cadena ligera comprende la secuencia de aminoácidos de la SEQ ID NO: 189.
8. El anticuerpo de conformidad con cualquiera de las reivindicaciones anteriores, en donde la administración del anticuerpo a un mamífero da como resultado: (a) un aumento en las células T efectoras (Tef) en el mamífero, y/o (b) la activación de las células T efectoras (Tef) en el mamífero, y/o (c) una disminución en las células T reguladoras (Treg) en el mamífero; en donde las células Tef son opcionalmente células T CD4+ FoxP3- o células T CD4+ FoxP3-y células T CD8+ o células T CD8+; y en donde las células Treg son opcionalmente células T CD4+ FoxP3+.
9. Un ácido nucleico aislado que codifica el anticuerpo de conformidad con cualquiera de las reivindicaciones 1 a 8, en donde el ácido nucleico está comprendido opcionalmente en un vector.
10. Una célula huésped que comprende el vector de conformidad con la reivindicación 9, o que produce el anticuerpo de conformidad con cualquiera de las reivindicaciones 1 a 8.
11. Un método para producir un anticuerpo anti-ICOS, que comprende cultivar la célula huésped de conformidad con la reivindicación 10 en condiciones adecuadas para la expresión del anticuerpo, y recuperar el anticuerpo producido por la célula huésped.
12. Un anticuerpo anti-ICOS que se puede producir por el método de conformidad con la reivindicación 11.
13. El anticuerpo anti-ICOS de conformidad con cualquiera de las reivindicaciones 1 a 8 y 12 para usar en un método para tratar un cáncer en un mamífero.
14. El anticuerpo anti-ICOS para usar de conformidad con la reivindicación 13, en donde el cáncer se selecciona de melanoma, cáncer de pulmón de células no pequeñas (NSCLC), carcinoma de células renales (RCC), cáncer gástrico, cáncer de vejiga, linfoma difuso de células B grandes (DLBCL), linfoma de Hodgkin, cáncer de ovario, cáncer de células escamosas de cabeza y cuello (HNSCC), y cáncer de mama triple negativo (TNBC).
15. El anticuerpo anti-ICOS para usar de conformidad con la reivindicación 13 o la reivindicación 14, que es para usar con al menos un agente terapéutico adicional, en donde el agente terapéutico adicional se selecciona opcionalmente de un anticuerpo anti-PD-1 y un anticuerpo anti-PD-L1, o es opcionalmente una vacuna contra el cáncer, y en donde la vacuna contra el cáncer se selecciona opcionalmente de una vacuna de ADN, una vacuna de virus modificado genéticamente, una vacuna de células tumorales modificadas genéticamente, y una vacuna contra el cáncer desarrollada mediante el uso de neoantígenos.
16. El anticuerpo anti-ICOS para usar de conformidad con cualquiera de las reivindicaciones 13 a 15, en donde el mamífero es un ser humano.
17. El anticuerpo anti-ICOS para usar de conformidad con cualquiera de las reivindicaciones 13 a 16, en donde se ha determinado que una muestra del cáncer expresa ICOS, en donde la muestra opcionalmente muestra una tinción de ICOS de 1+, 2+ o 3+ mediante inmunohistoquímica (IHC).
18. El anticuerpo anti-ICOS para usar de conformidad con cualquiera de las reivindicaciones 13 a 17, en donde se ha determinado que la muestra tiene un nivel bajo de PD-L1, o es negativa para PD-L1, o tiene un nivel elevado de PD-L1, en donde los niveles de PD-L1 se determinan opcionalmente mediante el uso de IHC.
ı32
¿2
£ d * a d
seupojiu inb ep e iu j¡ j
Fuerte (+3) Moderado (+2) Débil (+1 } Negativo (0)
FÍG. 4
( sLUUJ/SB|n|80) S O O I 9 P pep!SU8Q
F/G. 8A
Carcinoma
de células escamosas Adenocarcinoma (5 % área de corte) (5 % área de corte)
M L 1207044 M L 1208251
0 104 105 0 1 0 4 1G5 Comp-BV 711-A=PD-1 Comp-BV ( ! 1 -A= PD-1
PD1 FIG. 9A (Parte 2)
ı43
ı44
í P 7 #\x /82»s i íÚ C
PBS 37A1Q-mG2a hlCGSL-mG2a Fe
F/G. 14B
hits. Ib
151
ı52
( £ w i u ) l e j o L u n i u a w n | O A
IBJOiunj ueiun|OA
O Bazo
FíG. 26 B
161
FIO. 26C
NKp48~L en Treg
FÍG. 26D
162
r K j . 2 b r
/ " / O , £f
Applications Claiming Priority (6)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US201562137034P | 2015-03-23 | 2015-03-23 | |
| US201562147484P | 2015-04-14 | 2015-04-14 | |
| US201562156588P | 2015-05-04 | 2015-05-04 | |
| US201562242489P | 2015-10-16 | 2015-10-16 | |
| US201562255635P | 2015-11-16 | 2015-11-16 | |
| PCT/US2016/023524 WO2016154177A2 (en) | 2015-03-23 | 2016-03-22 | Antibodies to icos |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| ES2811345T3 true ES2811345T3 (es) | 2021-03-11 |
Family
ID=56978549
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| ES16769535T Active ES2811345T3 (es) | 2015-03-23 | 2016-03-22 | Anticuerpos contra ICOS |
Country Status (35)
| Country | Link |
|---|---|
| US (3) | US10023635B2 (es) |
| EP (2) | EP3273992B1 (es) |
| JP (2) | JP6944924B2 (es) |
| KR (2) | KR20170129902A (es) |
| CN (2) | CN107530428B (es) |
| AU (2) | AU2016235362B2 (es) |
| BR (1) | BR112017020054A2 (es) |
| CA (1) | CA2978185A1 (es) |
| CL (1) | CL2017002401A1 (es) |
| CO (1) | CO2017010618A2 (es) |
| CY (1) | CY1123409T1 (es) |
| DK (1) | DK3273992T3 (es) |
| EA (1) | EA037621B1 (es) |
| EC (1) | ECSP17070399A (es) |
| ES (1) | ES2811345T3 (es) |
| HK (1) | HK1248127A1 (es) |
| HR (1) | HRP20201090T1 (es) |
| HU (1) | HUE049938T2 (es) |
| IL (2) | IL254338B (es) |
| LT (1) | LT3273992T (es) |
| MA (1) | MA53297A (es) |
| MD (1) | MD3273992T2 (es) |
| ME (1) | ME03819B (es) |
| MX (2) | MX2017012129A (es) |
| PH (2) | PH12017501748B1 (es) |
| PL (1) | PL3273992T3 (es) |
| PT (1) | PT3273992T (es) |
| RS (1) | RS60614B1 (es) |
| RU (1) | RU2742241C2 (es) |
| SG (2) | SG10202001019TA (es) |
| SI (1) | SI3273992T1 (es) |
| SM (1) | SMT202000444T1 (es) |
| TW (1) | TWI719970B (es) |
| WO (1) | WO2016154177A2 (es) |
| ZA (1) | ZA201705920B (es) |
Families Citing this family (90)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US8709417B2 (en) | 2009-09-30 | 2014-04-29 | Memorial Sloan-Kettering Cancer Center | Combination immunotherapy for the treatment of cancer |
| SG193428A1 (en) | 2011-03-31 | 2013-10-30 | Inst Nat Sante Rech Med | Antibodies directed against icos and uses thereof |
| GB201403775D0 (en) | 2014-03-04 | 2014-04-16 | Kymab Ltd | Antibodies, uses & methods |
| JP6647315B2 (ja) | 2015-01-09 | 2020-02-14 | イーチュービクス コーポレイション | 組み合わせ免疫療法のための方法および組成物 |
| CA2978185A1 (en) * | 2015-03-23 | 2016-09-29 | Stephen Sazinsky | Antibodies to icos |
| EP3286213B1 (en) * | 2015-04-20 | 2021-08-04 | Etubics Corporation | Methods and compositions for combination immunotherapy |
| GB201516047D0 (en) | 2015-09-10 | 2015-10-28 | Cancer Rec Tech Ltd | Method |
| EP3365372A1 (en) | 2015-10-22 | 2018-08-29 | Jounce Therapeutics, Inc. | Gene signatures for determining icos expression |
| CN109563081A (zh) | 2016-04-07 | 2019-04-02 | 葛兰素史克知识产权开发有限公司 | 可用作蛋白调节剂的杂环酰胺类 |
| MY189100A (en) | 2016-04-07 | 2022-01-25 | Glaxosmithkline Ip Dev Ltd | Heterocyclic amides useful as protein modulators |
| BR112018076281A2 (pt) | 2016-06-20 | 2019-03-26 | Kymab Limited | imunocitocina, uso de uma imunocitocina, método, composição farmacêutica, método para tratar uma doença proliferativa em um animal, ácido nucleico, vetor, hospedeiro e anticorpo ou fragmento do mesmo |
| US9567399B1 (en) | 2016-06-20 | 2017-02-14 | Kymab Limited | Antibodies and immunocytokines |
| WO2018029474A2 (en) | 2016-08-09 | 2018-02-15 | Kymab Limited | Anti-icos antibodies |
| CA3032897A1 (en) * | 2016-08-09 | 2018-02-15 | Kymab Limited | Anti-icos antibodies |
| EP3534947A1 (en) | 2016-11-03 | 2019-09-11 | Kymab Limited | Antibodies, combinations comprising antibodies, biomarkers, uses & methods |
| WO2018115859A1 (en) * | 2016-12-20 | 2018-06-28 | Kymab Limited | Multispecific antibody with combination therapy for immuno-oncology |
| JP7290568B2 (ja) * | 2016-12-20 | 2023-06-13 | カイマブ・リミテッド | 癌免疫療法のための併用療法での多重特異性抗体 |
| EP3585782A1 (en) | 2017-02-27 | 2020-01-01 | GlaxoSmithKline Intellectual Property Development Limited | Heterocyclic amides as kinase inhibitors |
| US20200024351A1 (en) * | 2017-04-03 | 2020-01-23 | Jounce Therapeutics, Inc. | Compositions and Methods for the Treatment of Cancer |
| TWI788340B (zh) | 2017-04-07 | 2023-01-01 | 美商必治妥美雅史谷比公司 | 抗icos促效劑抗體及其用途 |
| CN110831971A (zh) | 2017-06-09 | 2020-02-21 | 葛兰素史克知识产权开发有限公司 | 用icos激动剂和ox40激动剂治疗癌症的组合疗法 |
| CA3066048A1 (en) | 2017-06-09 | 2018-12-13 | Glaxosmithkline Intellectual Property Development Limited | Combination therapy |
| CA3066007A1 (en) | 2017-06-09 | 2018-12-13 | Glaxosmithkline Intellectual Property Development Limited | Combination therapy with icos agonist and ox40 agonist to treat cancer |
| CA3065553A1 (en) | 2017-06-18 | 2018-12-27 | Kindred Biosciences, Inc. | Il17a antibodies and antagonists for veterinary use |
| GB201709808D0 (en) | 2017-06-20 | 2017-08-02 | Kymab Ltd | Antibodies |
| US20200129482A1 (en) * | 2017-06-26 | 2020-04-30 | Abbvie Inc. | Treatment of non-small cell lung cancer |
| GB201712032D0 (en) | 2017-07-26 | 2017-09-06 | Bioinvent Int Ab | Antibodies and uses thereof |
| KR102713874B1 (ko) * | 2017-08-11 | 2024-10-10 | 제넨테크, 인크. | 항-cd8 항체 및 이의 용도 |
| EP3692066A2 (en) | 2017-09-14 | 2020-08-12 | GlaxoSmithKline Intellectual Property Development Limited | Combination treatment for cancer |
| WO2019060771A2 (en) | 2017-09-22 | 2019-03-28 | University Of Washington | IN SITU COMBINATORY MARKING OF CELLULAR MOLECULES |
| BR112020006780A2 (pt) | 2017-10-05 | 2020-10-06 | Glaxosmithkline Intellectual Property Development Limited | moduladores do estimulador de genes do interferon (sting) |
| US20210238172A1 (en) | 2017-10-05 | 2021-08-05 | Glaxosmithkline Intellectual Property Development Limited | Heterocyclic amides useful as protein modulators and methods of using the same |
| GB201721338D0 (en) | 2017-12-19 | 2018-01-31 | Kymab Ltd | Anti-icos Antibodies |
| EP3728314A1 (en) | 2017-12-19 | 2020-10-28 | Kymab Limited | Bispecific antibody for icos and pd-l1 |
| TWI833724B (zh) | 2017-12-22 | 2024-03-01 | 美商永斯醫療股份有限公司 | 抗lilrb2抗體 |
| TW202000891A (zh) | 2018-03-07 | 2020-01-01 | 英商葛蘭素史克智慧財產發展有限公司 | 純化抗體之方法 |
| CA3093407A1 (en) | 2018-03-23 | 2019-09-26 | Bristol-Myers Squibb Company | Antibodies against mica and/or micb and uses thereof |
| EP3793598A1 (en) | 2018-05-14 | 2021-03-24 | Jounce Therapeutics, Inc. | Methods of treating cancer |
| GB201807924D0 (en) | 2018-05-16 | 2018-06-27 | Ctxt Pty Ltd | Compounds |
| WO2019229614A1 (en) | 2018-05-31 | 2019-12-05 | Glaxosmithkline Intellectual Property Development Limited | Combination of a type ii protein arginine methyltransferase inhibitor and an icos binding protein to treat cancer |
| EP3801615A1 (en) | 2018-05-31 | 2021-04-14 | GlaxoSmithKline Intellectual Property Development Ltd | Combined therapy with icos binding proteins and argininemethyltransferase inhibitors |
| GB201811410D0 (en) | 2018-07-12 | 2018-08-29 | F Star Beta Ltd | OX40 Binding molecules |
| WO2020031087A1 (en) | 2018-08-06 | 2020-02-13 | Glaxosmithkline Intellectual Property Development Limited | Combination therapy |
| WO2020081905A1 (en) | 2018-10-18 | 2020-04-23 | Jounce Therapeutics, Inc | Methods for treating cancer |
| WO2020086476A1 (en) | 2018-10-22 | 2020-04-30 | Glaxosmithkline Intellectual Property Development Limited | Dosing |
| EP3870610A1 (en) | 2018-10-24 | 2021-09-01 | Jounce Therapeutics, Inc. | Methods and compositions for the treatment of cancer and infectious diseases |
| US20210355222A1 (en) | 2018-10-26 | 2021-11-18 | Jounce Therapeutics, Inc. | Methods of Treating Cancer |
| WO2020185739A1 (en) | 2019-03-11 | 2020-09-17 | Jounce Therapeutics, Inc. | Anti-icos antibodies for the treatment of cancer |
| US20220251079A1 (en) | 2019-05-16 | 2022-08-11 | Stingthera, Inc. | Benzo[b][1,8]naphthyridine acetic acid derivatives and methods of use |
| EP3969438A1 (en) | 2019-05-16 | 2022-03-23 | Stingthera, Inc. | Oxoacridinyl acetic acid derivatives and methods of use |
| US20220363760A1 (en) | 2019-05-30 | 2022-11-17 | Bristol-Myers Squibb Company | Multi-tumor gene signature for suitability to immuno-oncology therapy |
| EP3976832A1 (en) | 2019-05-30 | 2022-04-06 | Bristol-Myers Squibb Company | Methods of identifying a subject suitable for an immuno-oncology (i-o) therapy |
| CN114174537A (zh) | 2019-05-30 | 2022-03-11 | 百时美施贵宝公司 | 细胞定位特征和组合疗法 |
| GB201910304D0 (en) | 2019-07-18 | 2019-09-04 | Ctxt Pty Ltd | Compounds |
| GB201910305D0 (en) | 2019-07-18 | 2019-09-04 | Ctxt Pty Ltd | Compounds |
| WO2021018941A1 (en) | 2019-07-31 | 2021-02-04 | Glaxosmithkline Intellectual Property Development Limited | Methods of treating cancer |
| WO2021046289A1 (en) | 2019-09-06 | 2021-03-11 | Glaxosmithkline Intellectual Property Development Limited | Dosing regimen for the treatment of cancer with an anti icos agonistic antibody and ipilimumab |
| WO2021043961A1 (en) | 2019-09-06 | 2021-03-11 | Glaxosmithkline Intellectual Property Development Limited | Dosing regimen for the treatment of cancer with an anti icos agonistic antibody and chemotherapy |
| US11878009B2 (en) | 2019-09-10 | 2024-01-23 | Great Novel Therapeutics Biotech & Medicals Corporation | Anticancer combination of chidamide and celecoxib salts |
| WO2021056025A2 (en) * | 2019-09-20 | 2021-03-25 | Board Of Regents, The University Of Texas System | Anti-epha10 antibodies and methods of use thereof |
| CN114787188A (zh) | 2019-11-05 | 2022-07-22 | 震动疗法股份有限公司 | 用抗pd-1抗体治疗癌症的方法 |
| CN114981265B (zh) | 2019-12-18 | 2025-01-03 | Ctxt私人有限公司 | 化合物 |
| EP4087922A4 (en) | 2020-01-07 | 2024-01-10 | Board of Regents, The University of Texas System | Improved human methylthioadenosine/adenosine depleting enzyme variants for cancer therapy |
| CN115516111A (zh) | 2020-01-13 | 2022-12-23 | 震动疗法公司 | 治疗癌症的方法 |
| US20230149543A1 (en) | 2020-04-14 | 2023-05-18 | Glaxosmithkline Intellectual Property Development Limited | Combination treatment for cancer based upon an icos antbody and a pd-l1 antibody tgf-bets-receptor fusion protein |
| WO2021209356A1 (en) | 2020-04-14 | 2021-10-21 | Glaxosmithkline Intellectual Property Development Limited | Combination treatment for cancer |
| WO2021209357A1 (en) | 2020-04-14 | 2021-10-21 | Glaxosmithkline Intellectual Property Development Limited | Combination treatment for cancer involving anti-icos and anti-pd1 antibodies, optionally further involving anti-tim3 antibodies |
| WO2021216417A1 (en) | 2020-04-20 | 2021-10-28 | Jounce Therapeutics, Inc. | Compositions and methods for vaccination and the treatment of infectious diseases |
| GB202007099D0 (en) | 2020-05-14 | 2020-07-01 | Kymab Ltd | Tumour biomarkers for immunotherapy |
| EP4204453A1 (en) | 2020-08-31 | 2023-07-05 | Bristol-Myers Squibb Company | Cell localization signature and immunotherapy |
| US20230398150A1 (en) * | 2020-10-16 | 2023-12-14 | Fred Hutchinson Cancer Center | Specific targeting of tumor-infiltrating regulatory t cells (tregs) using icos and il-1r |
| JP2024508207A (ja) | 2020-12-02 | 2024-02-26 | ブイアイビー ブイゼットダブリュ | がんに対する組み合わせ治療におけるltbrアゴニスト |
| WO2022120179A1 (en) | 2020-12-03 | 2022-06-09 | Bristol-Myers Squibb Company | Multi-tumor gene signatures and uses thereof |
| AU2021411486A1 (en) | 2020-12-28 | 2023-06-29 | Bristol-Myers Squibb Company | Antibody compositions and methods of use thereof |
| CA3196999A1 (en) | 2020-12-28 | 2022-07-07 | Masano HUANG | Methods of treating tumors |
| CA3209479A1 (en) | 2021-02-03 | 2022-08-11 | Mozart Therapeutics, Inc. | Binding agents and methods of using the same |
| US20240376224A1 (en) | 2021-04-02 | 2024-11-14 | The Regents Of The University Of California | Antibodies against cleaved cdcp1 and uses thereof |
| TW202313682A (zh) | 2021-05-18 | 2023-04-01 | 英商凱麥博有限公司 | 抗icos抗體之用途 |
| GB202107994D0 (en) | 2021-06-04 | 2021-07-21 | Kymab Ltd | Treatment of cancer |
| WO2023076876A1 (en) | 2021-10-26 | 2023-05-04 | Mozart Therapeutics, Inc. | Modulation of immune responses to viral vectors |
| IL314346A (en) | 2022-03-15 | 2024-09-01 | Compugen Ltd | Antibodies antagonistic to IL-18BP and their use in monotherapy and combination therapy in cancer treatment |
| CN118871451A (zh) | 2022-03-18 | 2024-10-29 | 百时美施贵宝公司 | 分离多肽的方法 |
| EP4525991A1 (en) | 2022-05-18 | 2025-03-26 | Kymab Limited | Uses of anti-icos antibodies |
| WO2023235699A1 (en) | 2022-05-31 | 2023-12-07 | Jounce Therapeutics, Inc. | Antibodies to lilrb4 and uses thereof |
| WO2023235847A1 (en) | 2022-06-02 | 2023-12-07 | Bristol-Myers Squibb Company | Antibody compositions and methods of use thereof |
| WO2024040195A1 (en) | 2022-08-17 | 2024-02-22 | Capstan Therapeutics, Inc. | Conditioning for in vivo immune cell engineering |
| WO2024054992A1 (en) | 2022-09-09 | 2024-03-14 | Bristol-Myers Squibb Company | Methods of separating chelator |
| WO2024196952A1 (en) | 2023-03-20 | 2024-09-26 | Bristol-Myers Squibb Company | Tumor subtype assessment for cancer therapy |
| US20240425607A1 (en) | 2023-06-26 | 2024-12-26 | Compugen Ltd. | Il-18bp antagonist antibodies and their use in monotherapy and combination therapy in the treatment of cancer |
| WO2025038763A1 (en) | 2023-08-15 | 2025-02-20 | Bristol-Myers Squibb Company | Ceramic hydroxyapatite chromatography flow through method |
Family Cites Families (93)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US4816567A (en) | 1983-04-08 | 1989-03-28 | Genentech, Inc. | Recombinant immunoglobin preparations |
| GB8601597D0 (en) | 1986-01-23 | 1986-02-26 | Wilson R H | Nucleotide sequences |
| US6548640B1 (en) | 1986-03-27 | 2003-04-15 | Btg International Limited | Altered antibodies |
| IL85035A0 (en) | 1987-01-08 | 1988-06-30 | Int Genetic Eng | Polynucleotide molecule,a chimeric antibody with specificity for human b cell surface antigen,a process for the preparation and methods utilizing the same |
| AU7247191A (en) | 1990-01-11 | 1991-08-05 | Molecular Affinities Corporation | Production of antibodies using gene libraries |
| US6150584A (en) | 1990-01-12 | 2000-11-21 | Abgenix, Inc. | Human antibodies derived from immunized xenomice |
| US6075181A (en) | 1990-01-12 | 2000-06-13 | Abgenix, Inc. | Human antibodies derived from immunized xenomice |
| US5770429A (en) | 1990-08-29 | 1998-06-23 | Genpharm International, Inc. | Transgenic non-human animals capable of producing heterologous antibodies |
| ATE164395T1 (de) | 1990-12-03 | 1998-04-15 | Genentech Inc | Verfahren zur anreicherung von proteinvarianten mit geänderten bindungseigenschaften |
| EP1400536A1 (en) | 1991-06-14 | 2004-03-24 | Genentech Inc. | Method for making humanized antibodies |
| US6051227A (en) | 1995-07-25 | 2000-04-18 | The Regents Of The University Of California, Office Of Technology Transfer | Blockade of T lymphocyte down-regulation associated with CTLA-4 signaling |
| GB9603256D0 (en) | 1996-02-16 | 1996-04-17 | Wellcome Found | Antibodies |
| PT994903E (pt) | 1997-06-24 | 2005-10-31 | Genentech Inc | Metodos e composicoes para glicoproteinas galactosiladas |
| US6342220B1 (en) | 1997-08-25 | 2002-01-29 | Genentech, Inc. | Agonist antibodies |
| DE19821060A1 (de) * | 1997-09-23 | 1999-04-15 | Bundesrepublik Deutschland Let | Ko-stimulierendes Polypeptid von T-Zellen, monoklonale Antikörper sowie die Herstellung und deren Verwendung |
| WO1999015553A2 (de) | 1997-09-23 | 1999-04-01 | Bundesrepublik Deutschland Letztvertreten Durch Den Direktor Des Robert-Koch-Instituts | Ko-stimulierendes polypeptid von t-zellen, monoklonale antikörper sowie die herstellung und deren verwendung |
| JP2001521909A (ja) | 1997-10-31 | 2001-11-13 | ジェネンテク・インコーポレイテッド | 糖タンパク質グリコフォームを含む方法及び組成物 |
| US6610833B1 (en) | 1997-11-24 | 2003-08-26 | The Institute For Human Genetics And Biochemistry | Monoclonal human natural antibodies |
| WO1999029888A1 (en) | 1997-12-05 | 1999-06-17 | The Scripps Research Institute | Humanization of murine antibody |
| US6194551B1 (en) | 1998-04-02 | 2001-02-27 | Genentech, Inc. | Polypeptide variants |
| JP2002510481A (ja) | 1998-04-02 | 2002-04-09 | ジェネンテック・インコーポレーテッド | 抗体変異体及びその断片 |
| EP2180007B2 (en) | 1998-04-20 | 2017-08-30 | Roche Glycart AG | Glycosylation engineering of antibodies for improving antibody-dependent cellular cytotoxicity |
| CA2352783C (en) | 1998-12-03 | 2012-04-10 | The Regents Of The University Of California | Stimulation of t cells against self antigens using ctla-4 blocking agents |
| US6737056B1 (en) | 1999-01-15 | 2004-05-18 | Genentech, Inc. | Polypeptide variants with altered effector function |
| EP2796465A1 (en) | 1999-02-03 | 2014-10-29 | Amgen, Inc | Polypeptides involved in immune response |
| CA2369292C (en) | 1999-04-09 | 2010-09-21 | Kyowa Hakko Kogyo Co. Ltd. | Method of modulating the activity of functional immune molecules |
| BR0013581A (pt) | 1999-08-23 | 2002-07-02 | Dana Faber Cancer Inst Inc | Pd-1, um receptor para b7-4, e usos para isto |
| CA2589418A1 (en) | 1999-08-24 | 2001-03-01 | Medarex, Inc. | Human ctla-4 antibodies and their uses |
| US7605238B2 (en) | 1999-08-24 | 2009-10-20 | Medarex, Inc. | Human CTLA-4 antibodies and their uses |
| EP1218504B1 (en) | 1999-09-21 | 2007-07-11 | Genetics Institute, LLC | Gl50 molecules and uses therefor |
| AU7950400A (en) | 1999-10-19 | 2001-04-30 | Kyowa Hakko Kogyo Co. Ltd. | Process for producing polypeptide |
| US20030180714A1 (en) | 1999-12-15 | 2003-09-25 | Genentech, Inc. | Shotgun scanning |
| JP3597140B2 (ja) * | 2000-05-18 | 2004-12-02 | 日本たばこ産業株式会社 | 副刺激伝達分子ailimに対するヒトモノクローナル抗体及びその医薬用途 |
| JP2004501624A (ja) | 2000-06-28 | 2004-01-22 | ジェネティックス・インスチチュート・リミテッド・ライアビリティ・カンパニー | Pd−l2分子:新規pd−1リガンドおよびその使用 |
| MXPA03002974A (es) | 2000-10-06 | 2004-05-05 | Kyowa Hakko Kogyo Kk | Celulas que producen composiciones de anticuerpo. |
| US7064191B2 (en) | 2000-10-06 | 2006-06-20 | Kyowa Hakko Kogyo Co., Ltd. | Process for purifying antibody |
| US6946292B2 (en) | 2000-10-06 | 2005-09-20 | Kyowa Hakko Kogyo Co., Ltd. | Cells producing antibody compositions with increased antibody dependent cytotoxic activity |
| US6596541B2 (en) | 2000-10-31 | 2003-07-22 | Regeneron Pharmaceuticals, Inc. | Methods of modifying eukaryotic cells |
| PT1354034E (pt) | 2000-11-30 | 2008-02-28 | Medarex Inc | Roedores transgénicos transcromossómicos para produção de anticorpos humanos |
| US20030124149A1 (en) | 2001-07-06 | 2003-07-03 | Shalaby Shalaby W. | Bioactive absorbable microparticles as therapeutic vaccines |
| NZ571596A (en) | 2001-08-03 | 2010-11-26 | Glycart Biotechnology Ag | Antibody glycosylation variants having increased antibody-dependent cellular cytotoxicity |
| ES2326964T3 (es) | 2001-10-25 | 2009-10-22 | Genentech, Inc. | Composiciones de glicoproteina. |
| CA2466530A1 (en) | 2001-11-09 | 2003-05-15 | Medigene Aktiengesellschaft | Allogenic vaccine that contains a costimulatory polypeptide-expressing tumor cell |
| US20040093621A1 (en) | 2001-12-25 | 2004-05-13 | Kyowa Hakko Kogyo Co., Ltd | Antibody composition which specifically binds to CD20 |
| CA2481837A1 (en) | 2002-04-09 | 2003-10-16 | Kyowa Hakko Kogyo Co., Ltd. | Production process for antibody composition |
| US7691568B2 (en) | 2002-04-09 | 2010-04-06 | Kyowa Hakko Kirin Co., Ltd | Antibody composition-containing medicament |
| US20040259150A1 (en) | 2002-04-09 | 2004-12-23 | Kyowa Hakko Kogyo Co., Ltd. | Method of enhancing of binding activity of antibody composition to Fcgamma receptor IIIa |
| AU2003236022A1 (en) | 2002-04-09 | 2003-10-20 | Kyowa Hakko Kogyo Co., Ltd. | Cells with modified genome |
| CA2481925A1 (en) | 2002-04-09 | 2003-10-16 | Kyowa Hakko Kogyo Co., Ltd. | Therapeutic agent for patients having human fc.gamma.riiia |
| JP4628679B2 (ja) | 2002-04-09 | 2011-02-09 | 協和発酵キリン株式会社 | Gdp−フコースの輸送に関与する蛋白質の活性が低下または欠失した細胞 |
| NZ556507A (en) | 2002-06-03 | 2010-03-26 | Genentech Inc | Synthetic antibody phage libraries |
| PT1537878E (pt) | 2002-07-03 | 2010-11-18 | Ono Pharmaceutical Co | Composições de imunopotenciação |
| US7217797B2 (en) | 2002-10-15 | 2007-05-15 | Pdl Biopharma, Inc. | Alteration of FcRn binding affinities or serum half-lives of antibodies by mutagenesis |
| RU2326127C2 (ru) | 2002-12-16 | 2008-06-10 | Джинентех, Инк. | Варианты иммуноглобулинов и их применение |
| AU2003288675B2 (en) | 2002-12-23 | 2010-07-22 | Medimmune Limited | Antibodies against PD-1 and uses therefor |
| EP1585767A2 (en) | 2003-01-16 | 2005-10-19 | Genentech, Inc. | Synthetic antibody phage libraries |
| CA2542046A1 (en) | 2003-10-08 | 2005-04-21 | Kyowa Hakko Kogyo Co., Ltd. | Fused protein composition |
| JPWO2005035778A1 (ja) | 2003-10-09 | 2006-12-21 | 協和醗酵工業株式会社 | α1,6−フコシルトランスフェラーゼの機能を抑制するRNAを用いた抗体組成物の製造法 |
| CN101979650B (zh) | 2003-10-22 | 2015-09-16 | 凯克研究生院 | 使用单倍体交配策略在酵母中合成异聚多亚基多肽的方法 |
| ES2550311T3 (es) | 2003-11-05 | 2015-11-06 | Roche Glycart Ag | Moléculas de unión a antígeno con afinidad de unión a receptores Fc y función efectora incrementadas |
| WO2005053742A1 (ja) | 2003-12-04 | 2005-06-16 | Kyowa Hakko Kogyo Co., Ltd. | 抗体組成物を含有する医薬 |
| KR101245983B1 (ko) | 2004-03-31 | 2013-06-28 | 제넨테크, 인크. | 인간화 항-tgf-베타 항체 |
| US7785903B2 (en) | 2004-04-09 | 2010-08-31 | Genentech, Inc. | Variable domain library and uses |
| US8318905B2 (en) * | 2004-04-23 | 2012-11-27 | Richard Kroczek | Antibodies for depletion of ICOS-positive cells in vivo |
| AU2005259958A1 (en) | 2004-06-29 | 2006-01-12 | The Johns Hopkins University | Amelioration of drug-induced toxicity |
| DE602006011574D1 (de) | 2005-01-27 | 2010-02-25 | Five Prime Therapeutics Inc | Leitsequenzen zur anleitung von polypeptidsekretion und verfahren zur herstellung |
| JP2006265155A (ja) | 2005-03-23 | 2006-10-05 | Link Genomics Kk | 癌の免疫療法 |
| ES2397355T5 (es) | 2005-06-08 | 2020-11-10 | Dana Farber Cancer Inst Inc | Métodos y composiciones para el tratamiento de infecciones persistentes y cáncer por inhibición de la vía de muerte celular programada (PD-1) |
| US7923538B2 (en) | 2005-07-22 | 2011-04-12 | Kyowa Hakko Kirin Co., Ltd | Recombinant antibody composition |
| EP2465870A1 (en) | 2005-11-07 | 2012-06-20 | Genentech, Inc. | Binding polypeptides with diversified and consensus VH/VL hypervariable sequences |
| EP1973951A2 (en) | 2005-12-02 | 2008-10-01 | Genentech, Inc. | Binding polypeptides with restricted diversity sequences |
| WO2007113648A2 (en) | 2006-04-05 | 2007-10-11 | Pfizer Products Inc. | Ctla4 antibody combination therapy |
| EP2016101A2 (en) | 2006-05-09 | 2009-01-21 | Genentech, Inc. | Binding polypeptides with optimized scaffolds |
| US20080226635A1 (en) | 2006-12-22 | 2008-09-18 | Hans Koll | Antibodies against insulin-like growth factor I receptor and uses thereof |
| ES2538990T3 (es) | 2007-01-24 | 2015-06-25 | Kyowa Hakko Kirin Co., Ltd. | Composición de anticuerpo genéticamente recombinante que tiene una actividad efectora mejorada |
| RU2549701C2 (ru) * | 2007-05-07 | 2015-04-27 | Медиммун, Ллк | Анти-icos антитела и их применение в лечении онкологических, связанных с трансплантацией и аутоиммунных заболеваний |
| CN100592373C (zh) | 2007-05-25 | 2010-02-24 | 群康科技(深圳)有限公司 | 液晶显示面板驱动装置及其驱动方法 |
| CA2769822C (en) * | 2009-08-13 | 2019-02-19 | The Johns Hopkins University | Methods of modulating immune function |
| US8709417B2 (en) | 2009-09-30 | 2014-04-29 | Memorial Sloan-Kettering Cancer Center | Combination immunotherapy for the treatment of cancer |
| AU2010328347B2 (en) | 2009-12-07 | 2015-12-17 | The Board Of Trustees Of The Leland Stanford Junior University | Methods for enhancing anti-tumor antibody therapy |
| CA2791975C (en) * | 2010-02-04 | 2021-02-09 | The Trustees Of The University Of Pennsylvania | Icos critically regulates the expansion and function of inflammatory human th17 cells |
| US9410205B2 (en) | 2010-02-18 | 2016-08-09 | New York University | Methods for predicting survival in metastatic melanoma patients |
| CN103476258B (zh) * | 2011-02-25 | 2017-04-26 | 默沙东公司 | 用作抗糖尿病药剂的新的环状氮杂苯并咪唑衍生物 |
| SG193428A1 (en) * | 2011-03-31 | 2013-10-30 | Inst Nat Sante Rech Med | Antibodies directed against icos and uses thereof |
| NO2872646T3 (es) | 2012-07-12 | 2018-01-27 | ||
| EP2911669B1 (en) | 2012-10-26 | 2024-04-10 | The University of Chicago | Synergistic combination of immunologic inhibitors for the treatment of cancer |
| SG11201507333XA (en) | 2013-03-15 | 2015-10-29 | Genentech Inc | Biomarkers and methods of treating pd-1 and pd-l1 related conditions |
| BR112015022462A8 (pt) | 2013-03-15 | 2019-11-26 | Bristol Myers Squibb Co | inibidores de ido, composição farmacêutica, método de inibição de atividade de indolamina 2,3-dioxigenase e uso dos referidos inibidores |
| MA41414A (fr) * | 2015-01-28 | 2017-12-05 | Centre Nat Rech Scient | Protéines de liaison agonistes d' icos |
| CA2978185A1 (en) * | 2015-03-23 | 2016-09-29 | Stephen Sazinsky | Antibodies to icos |
| EP3365372A1 (en) * | 2015-10-22 | 2018-08-29 | Jounce Therapeutics, Inc. | Gene signatures for determining icos expression |
| CA3026563C (en) * | 2016-06-10 | 2023-11-28 | Io Therapeutics, Inc. | Receptor selective retinoid and rexinoid compounds and immune modulators for cancer immunotherapy |
| US9567399B1 (en) * | 2016-06-20 | 2017-02-14 | Kymab Limited | Antibodies and immunocytokines |
-
2016
- 2016-03-22 CA CA2978185A patent/CA2978185A1/en active Pending
- 2016-03-22 RU RU2017137020A patent/RU2742241C2/ru active
- 2016-03-22 MA MA053297A patent/MA53297A/fr unknown
- 2016-03-22 DK DK16769535.2T patent/DK3273992T3/da active
- 2016-03-22 CN CN201680026223.2A patent/CN107530428B/zh not_active Expired - Fee Related
- 2016-03-22 PT PT167695352T patent/PT3273992T/pt unknown
- 2016-03-22 KR KR1020177030281A patent/KR20170129902A/ko active IP Right Grant
- 2016-03-22 SG SG10202001019TA patent/SG10202001019TA/en unknown
- 2016-03-22 AU AU2016235362A patent/AU2016235362B2/en not_active Ceased
- 2016-03-22 SM SM20200444T patent/SMT202000444T1/it unknown
- 2016-03-22 SI SI201630870T patent/SI3273992T1/sl unknown
- 2016-03-22 KR KR1020237040603A patent/KR20230164243A/ko active Pending
- 2016-03-22 CN CN202210469652.4A patent/CN114907481A/zh active Pending
- 2016-03-22 US US15/076,867 patent/US10023635B2/en active Active
- 2016-03-22 EP EP16769535.2A patent/EP3273992B1/en active Active
- 2016-03-22 PL PL16769535T patent/PL3273992T3/pl unknown
- 2016-03-22 JP JP2018501142A patent/JP6944924B2/ja active Active
- 2016-03-22 ME MEP-2020-166A patent/ME03819B/me unknown
- 2016-03-22 RS RS20200941A patent/RS60614B1/sr unknown
- 2016-03-22 EA EA201792078A patent/EA037621B1/ru unknown
- 2016-03-22 SG SG11201707769VA patent/SG11201707769VA/en unknown
- 2016-03-22 BR BR112017020054A patent/BR112017020054A2/pt not_active Application Discontinuation
- 2016-03-22 ES ES16769535T patent/ES2811345T3/es active Active
- 2016-03-22 HU HUE16769535A patent/HUE049938T2/hu unknown
- 2016-03-22 EP EP20167918.0A patent/EP3735986A1/en not_active Withdrawn
- 2016-03-22 LT LTEP16769535.2T patent/LT3273992T/lt unknown
- 2016-03-22 PH PH1/2017/501748A patent/PH12017501748B1/en unknown
- 2016-03-22 PH PH1/2021/552158A patent/PH12021552158A1/en unknown
- 2016-03-22 WO PCT/US2016/023524 patent/WO2016154177A2/en active Application Filing
- 2016-03-22 MD MDE20180112T patent/MD3273992T2/ro unknown
- 2016-03-22 MX MX2017012129A patent/MX2017012129A/es unknown
- 2016-03-22 TW TW105108884A patent/TWI719970B/zh active
-
2017
- 2017-08-31 ZA ZA201705920A patent/ZA201705920B/en unknown
- 2017-09-05 IL IL254338A patent/IL254338B/en unknown
- 2017-09-22 CL CL2017002401A patent/CL2017002401A1/es unknown
- 2017-09-25 MX MX2021011667A patent/MX2021011667A/es unknown
- 2017-10-18 CO CONC2017/0010618A patent/CO2017010618A2/es unknown
- 2017-10-23 EC ECIEPI201770399A patent/ECSP17070399A/es unknown
-
2018
- 2018-06-14 HK HK18107710.4A patent/HK1248127A1/zh unknown
- 2018-06-20 US US16/012,867 patent/US10570203B2/en active Active
-
2020
- 2020-07-13 HR HRP20201090TT patent/HRP20201090T1/hr unknown
- 2020-08-12 CY CY20201100755T patent/CY1123409T1/el unknown
- 2020-09-16 US US17/022,856 patent/US20210171635A1/en not_active Abandoned
-
2021
- 2021-09-13 JP JP2021148509A patent/JP2022002520A/ja active Pending
- 2021-09-29 IL IL286807A patent/IL286807A/en unknown
-
2022
- 2022-03-01 AU AU2022201408A patent/AU2022201408A1/en not_active Abandoned
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| ES2811345T3 (es) | Anticuerpos contra ICOS | |
| ES2898025T3 (es) | Anticuerpos contra PD-1 y sus usos | |
| US20210340256A1 (en) | Gene Signatures for Determining ICOS Expression | |
| US20200024351A1 (en) | Compositions and Methods for the Treatment of Cancer | |
| US20220135682A1 (en) | Anti-ICOS Antibodies for the Treatment of Cancer | |
| WO2023235699A1 (en) | Antibodies to lilrb4 and uses thereof | |
| EP4055052A1 (en) | Methods of treating cancer with anti-pd-1 antibodies |