JP5905187B2 - 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 - Google Patents
甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 Download PDFInfo
- Publication number
- JP5905187B2 JP5905187B2 JP2009274976A JP2009274976A JP5905187B2 JP 5905187 B2 JP5905187 B2 JP 5905187B2 JP 2009274976 A JP2009274976 A JP 2009274976A JP 2009274976 A JP2009274976 A JP 2009274976A JP 5905187 B2 JP5905187 B2 JP 5905187B2
- Authority
- JP
- Japan
- Prior art keywords
- sequence
- sweet
- hg16gust44
- expression construct
- cells
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Active
Links
- 235000009508 confectionery Nutrition 0.000 title claims description 136
- 102000005962 receptors Human genes 0.000 title claims description 106
- 108020003175 receptors Proteins 0.000 title claims description 106
- 230000014509 gene expression Effects 0.000 title claims description 89
- 210000005056 cell body Anatomy 0.000 title description 4
- 239000002299 complementary DNA Substances 0.000 claims description 136
- 108090000623 proteins and genes Proteins 0.000 claims description 70
- 239000000126 substance Substances 0.000 claims description 62
- 101000659774 Homo sapiens Taste receptor type 1 member 3 Proteins 0.000 claims description 52
- 102100035942 Taste receptor type 1 member 3 Human genes 0.000 claims description 52
- 101000659765 Homo sapiens Taste receptor type 1 member 2 Proteins 0.000 claims description 48
- 102100035948 Taste receptor type 1 member 2 Human genes 0.000 claims description 48
- 230000006461 physiological response Effects 0.000 claims description 41
- 108091006099 G alpha subunit Proteins 0.000 claims description 32
- 102000034353 G alpha subunit Human genes 0.000 claims description 32
- 235000019605 sweet taste sensations Nutrition 0.000 claims description 31
- 108091005708 gustatory receptors Proteins 0.000 claims description 26
- 102000010292 Peptide Elongation Factor 1 Human genes 0.000 claims description 17
- 108010077524 Peptide Elongation Factor 1 Proteins 0.000 claims description 17
- 239000013612 plasmid Substances 0.000 claims description 14
- 238000013518 transcription Methods 0.000 claims description 11
- 230000035897 transcription Effects 0.000 claims description 11
- 101001010097 Shigella phage SfV Bactoprenol-linked glucose translocase Proteins 0.000 claims description 3
- 230000002708 enhancing effect Effects 0.000 claims description 2
- 238000012546 transfer Methods 0.000 claims description 2
- 210000004027 cell Anatomy 0.000 description 166
- 210000004748 cultured cell Anatomy 0.000 description 74
- 239000013598 vector Substances 0.000 description 61
- 239000012634 fragment Substances 0.000 description 56
- 230000000692 anti-sense effect Effects 0.000 description 49
- 230000000638 stimulation Effects 0.000 description 49
- 238000003752 polymerase chain reaction Methods 0.000 description 42
- 238000006243 chemical reaction Methods 0.000 description 40
- 239000011575 calcium Substances 0.000 description 38
- JKMHFZQWWAIEOD-UHFFFAOYSA-N 2-[4-(2-hydroxyethyl)piperazin-1-yl]ethanesulfonic acid Chemical compound OCC[NH+]1CCN(CCS([O-])(=O)=O)CC1 JKMHFZQWWAIEOD-UHFFFAOYSA-N 0.000 description 34
- 239000007995 HEPES buffer Substances 0.000 description 33
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 30
- 229910052791 calcium Inorganic materials 0.000 description 30
- 235000019640 taste Nutrition 0.000 description 29
- 239000002609 medium Substances 0.000 description 26
- 229930006000 Sucrose Natural products 0.000 description 25
- 230000004044 response Effects 0.000 description 25
- 239000005720 sucrose Substances 0.000 description 25
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 24
- 108020004414 DNA Proteins 0.000 description 23
- 238000000799 fluorescence microscopy Methods 0.000 description 22
- 238000000034 method Methods 0.000 description 21
- 108010011485 Aspartame Proteins 0.000 description 20
- 239000000605 aspartame Substances 0.000 description 20
- 235000010357 aspartame Nutrition 0.000 description 20
- IAOZJIPTCAWIRG-QWRGUYRKSA-N aspartame Chemical compound OC(=O)C[C@H](N)C(=O)N[C@H](C(=O)OC)CC1=CC=CC=C1 IAOZJIPTCAWIRG-QWRGUYRKSA-N 0.000 description 20
- 229960003438 aspartame Drugs 0.000 description 20
- 108091008146 restriction endonucleases Proteins 0.000 description 20
- 108091006027 G proteins Proteins 0.000 description 18
- 102000030782 GTP binding Human genes 0.000 description 18
- 108091000058 GTP-Binding Proteins 0.000 description 18
- 108091028043 Nucleic acid sequence Proteins 0.000 description 18
- 230000005284 excitation Effects 0.000 description 15
- 239000000047 product Substances 0.000 description 15
- 108091026890 Coding region Proteins 0.000 description 14
- 229920000936 Agarose Polymers 0.000 description 13
- 238000001962 electrophoresis Methods 0.000 description 13
- 238000002360 preparation method Methods 0.000 description 13
- 239000006144 Dulbecco’s modified Eagle's medium Substances 0.000 description 12
- 241000544066 Stevia Species 0.000 description 12
- 230000008859 change Effects 0.000 description 12
- 229940109275 cyclamate Drugs 0.000 description 12
- HCAJEUSONLESMK-UHFFFAOYSA-N cyclohexylsulfamic acid Chemical group OS(=O)(=O)NC1CCCCC1 HCAJEUSONLESMK-UHFFFAOYSA-N 0.000 description 12
- HELXLJCILKEWJH-NCGAPWICSA-N rebaudioside A Chemical group O([C@H]1[C@H](O)[C@@H](CO)O[C@H]([C@@H]1O[C@H]1[C@@H]([C@@H](O)[C@H](O)[C@@H](CO)O1)O)O[C@]12C(=C)C[C@@]3(C1)CC[C@@H]1[C@@](C)(CCC[C@]1([C@@H]3CC2)C)C(=O)O[C@H]1[C@@H]([C@@H](O)[C@H](O)[C@@H](CO)O1)O)[C@@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O HELXLJCILKEWJH-NCGAPWICSA-N 0.000 description 12
- 235000019204 saccharin Nutrition 0.000 description 12
- CVHZOJJKTDOEJC-UHFFFAOYSA-N saccharin Chemical group C1=CC=C2C(=O)NS(=O)(=O)C2=C1 CVHZOJJKTDOEJC-UHFFFAOYSA-N 0.000 description 12
- 229940081974 saccharin Drugs 0.000 description 12
- 239000000901 saccharin and its Na,K and Ca salt Substances 0.000 description 12
- 229930182832 D-phenylalanine Natural products 0.000 description 11
- ITVGXXMINPYUHD-CUVHLRMHSA-N neohesperidin dihydrochalcone Chemical group C1=C(O)C(OC)=CC=C1CCC(=O)C(C(=C1)O)=C(O)C=C1O[C@H]1[C@H](O[C@H]2[C@@H]([C@H](O)[C@@H](O)[C@H](C)O2)O)[C@@H](O)[C@H](O)[C@@H](CO)O1 ITVGXXMINPYUHD-CUVHLRMHSA-N 0.000 description 11
- COLNVLDHVKWLRT-MRVPVSSYSA-N D-phenylalanine Chemical compound OC(=O)[C@H](N)CC1=CC=CC=C1 COLNVLDHVKWLRT-MRVPVSSYSA-N 0.000 description 10
- 238000012258 culturing Methods 0.000 description 10
- 238000011156 evaluation Methods 0.000 description 10
- 241000701022 Cytomegalovirus Species 0.000 description 9
- 108010046276 FLP recombinase Proteins 0.000 description 9
- GRRNUXAQVGOGFE-UHFFFAOYSA-N Hygromycin-B Natural products OC1C(NC)CC(N)C(O)C1OC1C2OC3(C(C(O)C(O)C(C(N)CO)O3)O)OC2C(O)C(CO)O1 GRRNUXAQVGOGFE-UHFFFAOYSA-N 0.000 description 9
- QOMNQGZXFYNBNG-UHFFFAOYSA-N acetyloxymethyl 2-[2-[2-[5-[3-(acetyloxymethoxy)-2,7-difluoro-6-oxoxanthen-9-yl]-2-[bis[2-(acetyloxymethoxy)-2-oxoethyl]amino]phenoxy]ethoxy]-n-[2-(acetyloxymethoxy)-2-oxoethyl]-4-methylanilino]acetate Chemical compound CC(=O)OCOC(=O)CN(CC(=O)OCOC(C)=O)C1=CC=C(C)C=C1OCCOC1=CC(C2=C3C=C(F)C(=O)C=C3OC3=CC(OCOC(C)=O)=C(F)C=C32)=CC=C1N(CC(=O)OCOC(C)=O)CC(=O)OCOC(C)=O QOMNQGZXFYNBNG-UHFFFAOYSA-N 0.000 description 9
- 230000036755 cellular response Effects 0.000 description 9
- 238000010367 cloning Methods 0.000 description 9
- 238000002073 fluorescence micrograph Methods 0.000 description 9
- VPSRLGDRGCKUTK-UHFFFAOYSA-N fura-2-acetoxymethyl ester Chemical compound CC(=O)OCOC(=O)CN(CC(=O)OCOC(C)=O)C1=CC=C(C)C=C1OCCOC(C(=C1)N(CC(=O)OCOC(C)=O)CC(=O)OCOC(C)=O)=CC2=C1OC(C=1OC(=CN=1)C(=O)OCOC(C)=O)=C2 VPSRLGDRGCKUTK-UHFFFAOYSA-N 0.000 description 9
- GRRNUXAQVGOGFE-NZSRVPFOSA-N hygromycin B Chemical compound O[C@@H]1[C@@H](NC)C[C@@H](N)[C@H](O)[C@H]1O[C@H]1[C@H]2O[C@@]3([C@@H]([C@@H](O)[C@@H](O)[C@@H](C(N)CO)O3)O)O[C@H]2[C@@H](O)[C@@H](CO)O1 GRRNUXAQVGOGFE-NZSRVPFOSA-N 0.000 description 9
- 229940097277 hygromycin b Drugs 0.000 description 9
- 230000003834 intracellular effect Effects 0.000 description 9
- 239000001329 FEMA 3811 Substances 0.000 description 8
- 238000003149 assay kit Methods 0.000 description 8
- 229940089953 neohesperidin dihydrochalcone Drugs 0.000 description 8
- 235000010434 neohesperidine DC Nutrition 0.000 description 8
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 7
- 108010084455 Zeocin Proteins 0.000 description 7
- 231100000673 dose–response relationship Toxicity 0.000 description 7
- 239000008103 glucose Substances 0.000 description 7
- CWCMIVBLVUHDHK-ZSNHEYEWSA-N phleomycin D1 Chemical compound N([C@H](C(=O)N[C@H](C)[C@@H](O)[C@H](C)C(=O)N[C@@H]([C@H](O)C)C(=O)NCCC=1SC[C@@H](N=1)C=1SC=C(N=1)C(=O)NCCCCNC(N)=N)[C@@H](O[C@H]1[C@H]([C@@H](O)[C@H](O)[C@H](CO)O1)O[C@@H]1[C@H]([C@@H](OC(N)=O)[C@H](O)[C@@H](CO)O1)O)C=1N=CNC=1)C(=O)C1=NC([C@H](CC(N)=O)NC[C@H](N)C(N)=O)=NC(N)=C1C CWCMIVBLVUHDHK-ZSNHEYEWSA-N 0.000 description 7
- 235000018102 proteins Nutrition 0.000 description 7
- 102000004169 proteins and genes Human genes 0.000 description 7
- 230000001953 sensory effect Effects 0.000 description 7
- 108091081024 Start codon Proteins 0.000 description 6
- 238000003556 assay Methods 0.000 description 6
- 230000008901 benefit Effects 0.000 description 6
- 108010006025 bovine growth hormone Proteins 0.000 description 6
- 238000013461 design Methods 0.000 description 6
- 239000003269 fluorescent indicator Substances 0.000 description 6
- 235000013305 food Nutrition 0.000 description 6
- 238000003384 imaging method Methods 0.000 description 6
- 235000003599 food sweetener Nutrition 0.000 description 5
- 235000000346 sugar Nutrition 0.000 description 5
- 239000003765 sweetening agent Substances 0.000 description 5
- 235000019583 umami taste Nutrition 0.000 description 5
- 102000004190 Enzymes Human genes 0.000 description 4
- 108090000790 Enzymes Proteins 0.000 description 4
- 241000282412 Homo Species 0.000 description 4
- 235000019658 bitter taste Nutrition 0.000 description 4
- 239000013604 expression vector Substances 0.000 description 4
- 230000006870 function Effects 0.000 description 4
- 239000003550 marker Substances 0.000 description 4
- 230000007246 mechanism Effects 0.000 description 4
- 108020004999 messenger RNA Proteins 0.000 description 4
- 230000010473 stable expression Effects 0.000 description 4
- BHPQYMZQTOCNFJ-UHFFFAOYSA-N Calcium cation Chemical compound [Ca+2] BHPQYMZQTOCNFJ-UHFFFAOYSA-N 0.000 description 3
- 238000001712 DNA sequencing Methods 0.000 description 3
- OUVXYXNWSVIOSJ-UHFFFAOYSA-N Fluo-4 Chemical compound CC1=CC=C(N(CC(O)=O)CC(O)=O)C(OCCOC=2C(=CC=C(C=2)C2=C3C=C(F)C(=O)C=C3OC3=CC(O)=C(F)C=C32)N(CC(O)=O)CC(O)=O)=C1 OUVXYXNWSVIOSJ-UHFFFAOYSA-N 0.000 description 3
- 102000003688 G-Protein-Coupled Receptors Human genes 0.000 description 3
- 108090000045 G-Protein-Coupled Receptors Proteins 0.000 description 3
- 230000005540 biological transmission Effects 0.000 description 3
- 229910001424 calcium ion Inorganic materials 0.000 description 3
- 238000001976 enzyme digestion Methods 0.000 description 3
- 238000005259 measurement Methods 0.000 description 3
- 150000007523 nucleic acids Chemical group 0.000 description 3
- 230000008488 polyadenylation Effects 0.000 description 3
- 230000035945 sensitivity Effects 0.000 description 3
- 239000000243 solution Substances 0.000 description 3
- 239000006188 syrup Substances 0.000 description 3
- 235000020357 syrup Nutrition 0.000 description 3
- 210000001779 taste bud Anatomy 0.000 description 3
- 238000001890 transfection Methods 0.000 description 3
- 235000019607 umami taste sensations Nutrition 0.000 description 3
- 238000011144 upstream manufacturing Methods 0.000 description 3
- MIEKOFWWHVOKQX-UHFFFAOYSA-N (S)-2-(4-Methoxyphenoxy)propanoic acid Chemical compound COC1=CC=C(OC(C)C(O)=O)C=C1 MIEKOFWWHVOKQX-UHFFFAOYSA-N 0.000 description 2
- 241000251468 Actinopterygii Species 0.000 description 2
- 108091003079 Bovine Serum Albumin Proteins 0.000 description 2
- UXVMQQNJUSDDNG-UHFFFAOYSA-L Calcium chloride Chemical compound [Cl-].[Cl-].[Ca+2] UXVMQQNJUSDDNG-UHFFFAOYSA-L 0.000 description 2
- 108020004635 Complementary DNA Proteins 0.000 description 2
- 241000938605 Crocodylia Species 0.000 description 2
- SRBFZHDQGSBBOR-IOVATXLUSA-N D-xylopyranose Chemical compound O[C@@H]1COC(O)[C@H](O)[C@H]1O SRBFZHDQGSBBOR-IOVATXLUSA-N 0.000 description 2
- QGWNDRXFNXRZMB-UUOKFMHZSA-K GDP(3-) Chemical compound C1=NC=2C(=O)NC(N)=NC=2N1[C@@H]1O[C@H](COP([O-])(=O)OP([O-])([O-])=O)[C@@H](O)[C@H]1O QGWNDRXFNXRZMB-UUOKFMHZSA-K 0.000 description 2
- 108090000862 Ion Channels Proteins 0.000 description 2
- 102000004310 Ion Channels Human genes 0.000 description 2
- ZDXPYRJPNDTMRX-VKHMYHEASA-N L-glutamine Chemical compound OC(=O)[C@@H](N)CCC(N)=O ZDXPYRJPNDTMRX-VKHMYHEASA-N 0.000 description 2
- 229930182816 L-glutamine Natural products 0.000 description 2
- 241000124008 Mammalia Species 0.000 description 2
- 241001465754 Metazoa Species 0.000 description 2
- 241000699670 Mus sp. Species 0.000 description 2
- 241000700159 Rattus Species 0.000 description 2
- 229920002472 Starch Polymers 0.000 description 2
- 230000004913 activation Effects 0.000 description 2
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 2
- 102000005936 beta-Galactosidase Human genes 0.000 description 2
- 108010005774 beta-Galactosidase Proteins 0.000 description 2
- 239000000872 buffer Substances 0.000 description 2
- 210000004899 c-terminal region Anatomy 0.000 description 2
- 239000001110 calcium chloride Substances 0.000 description 2
- 229910001628 calcium chloride Inorganic materials 0.000 description 2
- 210000000170 cell membrane Anatomy 0.000 description 2
- 238000001816 cooling Methods 0.000 description 2
- 230000001419 dependent effect Effects 0.000 description 2
- -1 dultin Chemical compound 0.000 description 2
- 239000012091 fetal bovine serum Substances 0.000 description 2
- YFHXZQPUBCBNIP-UHFFFAOYSA-N fura-2 Chemical compound CC1=CC=C(N(CC(O)=O)CC(O)=O)C(OCCOC=2C(=CC=3OC(=CC=3C=2)C=2OC(=CN=2)C(O)=O)N(CC(O)=O)CC(O)=O)=C1 YFHXZQPUBCBNIP-UHFFFAOYSA-N 0.000 description 2
- 230000012010 growth Effects 0.000 description 2
- QGWNDRXFNXRZMB-UHFFFAOYSA-N guanidine diphosphate Natural products C1=2NC(N)=NC(=O)C=2N=CN1C1OC(COP(O)(=O)OP(O)(O)=O)C(O)C1O QGWNDRXFNXRZMB-UHFFFAOYSA-N 0.000 description 2
- 239000000833 heterodimer Substances 0.000 description 2
- 238000001727 in vivo Methods 0.000 description 2
- 230000001965 increasing effect Effects 0.000 description 2
- 230000031146 intracellular signal transduction Effects 0.000 description 2
- HPIGCVXMBGOWTF-UHFFFAOYSA-N isomaltol Chemical compound CC(=O)C=1OC=CC=1O HPIGCVXMBGOWTF-UHFFFAOYSA-N 0.000 description 2
- 101150066555 lacZ gene Proteins 0.000 description 2
- 238000001638 lipofection Methods 0.000 description 2
- 238000012423 maintenance Methods 0.000 description 2
- 238000004519 manufacturing process Methods 0.000 description 2
- 229920001542 oligosaccharide Polymers 0.000 description 2
- 150000002482 oligosaccharides Chemical class 0.000 description 2
- 230000008447 perception Effects 0.000 description 2
- 230000008569 process Effects 0.000 description 2
- RXWNCPJZOCPEPQ-NVWDDTSBSA-N puromycin Chemical compound C1=CC(OC)=CC=C1C[C@H](N)C(=O)N[C@H]1[C@@H](O)[C@H](N2C3=NC=NC(=C3N=C2)N(C)C)O[C@@H]1CO RXWNCPJZOCPEPQ-NVWDDTSBSA-N 0.000 description 2
- 230000006798 recombination Effects 0.000 description 2
- 238000005215 recombination Methods 0.000 description 2
- 230000004043 responsiveness Effects 0.000 description 2
- 230000019491 signal transduction Effects 0.000 description 2
- 239000008107 starch Substances 0.000 description 2
- 235000019698 starch Nutrition 0.000 description 2
- HDTRYLNUVZCQOY-UHFFFAOYSA-N α-D-glucopyranosyl-α-D-glucopyranoside Natural products OC1C(O)C(O)C(CO)OC1OC1C(O)C(O)C(O)C(CO)O1 HDTRYLNUVZCQOY-UHFFFAOYSA-N 0.000 description 1
- NUFKRGBSZPCGQB-FLBSXDLDSA-N (3s)-3-amino-4-oxo-4-[[(2r)-1-oxo-1-[(2,2,4,4-tetramethylthietan-3-yl)amino]propan-2-yl]amino]butanoic acid;pentahydrate Chemical compound O.O.O.O.O.OC(=O)C[C@H](N)C(=O)N[C@H](C)C(=O)NC1C(C)(C)SC1(C)C.OC(=O)C[C@H](N)C(=O)N[C@H](C)C(=O)NC1C(C)(C)SC1(C)C NUFKRGBSZPCGQB-FLBSXDLDSA-N 0.000 description 1
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 1
- OWEGMIWEEQEYGQ-UHFFFAOYSA-N 100676-05-9 Natural products OC1C(O)C(O)C(CO)OC1OCC1C(O)C(O)C(O)C(OC2C(OC(O)C(O)C2O)CO)O1 OWEGMIWEEQEYGQ-UHFFFAOYSA-N 0.000 description 1
- PVXPPJIGRGXGCY-DJHAAKORSA-N 6-O-alpha-D-glucopyranosyl-alpha-D-fructofuranose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1OC[C@@H]1[C@@H](O)[C@H](O)[C@](O)(CO)O1 PVXPPJIGRGXGCY-DJHAAKORSA-N 0.000 description 1
- 241000208140 Acer Species 0.000 description 1
- 239000004377 Alitame Substances 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 241000282472 Canis lupus familiaris Species 0.000 description 1
- 108091005462 Cation channels Proteins 0.000 description 1
- 108091006146 Channels Proteins 0.000 description 1
- 108020004705 Codon Proteins 0.000 description 1
- FBPFZTCFMRRESA-FSIIMWSLSA-N D-Glucitol Natural products OC[C@H](O)[C@H](O)[C@@H](O)[C@H](O)CO FBPFZTCFMRRESA-FSIIMWSLSA-N 0.000 description 1
- FBPFZTCFMRRESA-KVTDHHQDSA-N D-Mannitol Chemical compound OC[C@@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-KVTDHHQDSA-N 0.000 description 1
- FBPFZTCFMRRESA-JGWLITMVSA-N D-glucitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)[C@H](O)CO FBPFZTCFMRRESA-JGWLITMVSA-N 0.000 description 1
- 125000001711 D-phenylalanine group Chemical group [H]N([H])[C@@]([H])(C(=O)[*])C([H])([H])C1=C([H])C([H])=C([H])C([H])=C1[H] 0.000 description 1
- 102000012410 DNA Ligases Human genes 0.000 description 1
- 108010061982 DNA Ligases Proteins 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 241000208713 Dionaea Species 0.000 description 1
- YQYJSBFKSSDGFO-UHFFFAOYSA-N Epihygromycin Natural products OC1C(O)C(C(=O)C)OC1OC(C(=C1)O)=CC=C1C=C(C)C(=O)NC1C(O)C(O)C2OCOC2C1O YQYJSBFKSSDGFO-UHFFFAOYSA-N 0.000 description 1
- 239000004386 Erythritol Substances 0.000 description 1
- UNXHWFMMPAWVPI-UHFFFAOYSA-N Erythritol Natural products OCC(O)C(O)CO UNXHWFMMPAWVPI-UHFFFAOYSA-N 0.000 description 1
- 108090000371 Esterases Proteins 0.000 description 1
- 229930091371 Fructose Natural products 0.000 description 1
- 239000005715 Fructose Substances 0.000 description 1
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 1
- 244000068988 Glycine max Species 0.000 description 1
- 235000010469 Glycine max Nutrition 0.000 description 1
- 101000605625 Homo sapiens Polycystic kidney disease 2-like 1 protein Proteins 0.000 description 1
- 101000730587 Homo sapiens Polycystic kidney disease protein 1-like 3 Proteins 0.000 description 1
- 101000659767 Homo sapiens Taste receptor type 1 member 1 Proteins 0.000 description 1
- 101000844521 Homo sapiens Transient receptor potential cation channel subfamily M member 5 Proteins 0.000 description 1
- 102100034343 Integrase Human genes 0.000 description 1
- 108010061833 Integrases Proteins 0.000 description 1
- 108020004684 Internal Ribosome Entry Sites Proteins 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 239000012097 Lipofectamine 2000 Substances 0.000 description 1
- GUBGYTABKSRVRQ-PICCSMPSSA-N Maltose Natural products O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@@H](CO)OC(O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-PICCSMPSSA-N 0.000 description 1
- 229930195725 Mannitol Natural products 0.000 description 1
- 108010052285 Membrane Proteins Proteins 0.000 description 1
- 102000018697 Membrane Proteins Human genes 0.000 description 1
- 108050004114 Monellin Proteins 0.000 description 1
- 229930193140 Neomycin Natural products 0.000 description 1
- 239000004384 Neotame Substances 0.000 description 1
- 208000008589 Obesity Diseases 0.000 description 1
- XCOJIVIDDFTHGB-UEUZTHOGSA-N Perillartine Chemical compound CC(=C)[C@H]1CCC(\C=N\O)=CC1 XCOJIVIDDFTHGB-UEUZTHOGSA-N 0.000 description 1
- 102100038330 Polycystic kidney disease 2-like 1 protein Human genes 0.000 description 1
- 102100032598 Polycystic kidney disease protein 1-like 3 Human genes 0.000 description 1
- MUPFEKGTMRGPLJ-RMMQSMQOSA-N Raffinose Natural products O(C[C@H]1[C@@H](O)[C@H](O)[C@@H](O)[C@@H](O[C@@]2(CO)[C@H](O)[C@@H](O)[C@@H](CO)O2)O1)[C@@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 MUPFEKGTMRGPLJ-RMMQSMQOSA-N 0.000 description 1
- 102000018120 Recombinases Human genes 0.000 description 1
- 108010091086 Recombinases Proteins 0.000 description 1
- 240000004808 Saccharomyces cerevisiae Species 0.000 description 1
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 1
- 238000002105 Southern blotting Methods 0.000 description 1
- 101100318695 Staphylococcus aureus (strain Mu50 / ATCC 700699) SAV2486 gene Proteins 0.000 description 1
- UEDUENGHJMELGK-HYDKPPNVSA-N Stevioside Chemical compound O([C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1O[C@]12C(=C)C[C@@]3(C1)CC[C@@H]1[C@@](C)(CCC[C@]1([C@@H]3CC2)C)C(=O)O[C@H]1[C@@H]([C@@H](O)[C@H](O)[C@@H](CO)O1)O)[C@@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O UEDUENGHJMELGK-HYDKPPNVSA-N 0.000 description 1
- 239000004376 Sucralose Substances 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 102000003609 TRPM5 Human genes 0.000 description 1
- 102100035941 Taste receptor type 1 member 1 Human genes 0.000 description 1
- HDTRYLNUVZCQOY-WSWWMNSNSA-N Trehalose Natural products O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-WSWWMNSNSA-N 0.000 description 1
- MUPFEKGTMRGPLJ-UHFFFAOYSA-N UNPD196149 Natural products OC1C(O)C(CO)OC1(CO)OC1C(O)C(O)C(O)C(COC2C(C(O)C(O)C(CO)O2)O)O1 MUPFEKGTMRGPLJ-UHFFFAOYSA-N 0.000 description 1
- TVXBFESIOXBWNM-UHFFFAOYSA-N Xylitol Natural products OCCC(O)C(O)C(O)CCO TVXBFESIOXBWNM-UHFFFAOYSA-N 0.000 description 1
- MMWCIQZXVOZEGG-HOZKJCLWSA-N [(1S,2R,3S,4S,5R,6S)-2,3,5-trihydroxy-4,6-diphosphonooxycyclohexyl] dihydrogen phosphate Chemical compound O[C@H]1[C@@H](O)[C@H](OP(O)(O)=O)[C@@H](OP(O)(O)=O)[C@H](O)[C@H]1OP(O)(O)=O MMWCIQZXVOZEGG-HOZKJCLWSA-N 0.000 description 1
- YGCFIWIQZPHFLU-UHFFFAOYSA-N acesulfame Chemical compound CC1=CC(=O)NS(=O)(=O)O1 YGCFIWIQZPHFLU-UHFFFAOYSA-N 0.000 description 1
- 229960005164 acesulfame Drugs 0.000 description 1
- 230000036982 action potential Effects 0.000 description 1
- 238000007792 addition Methods 0.000 description 1
- 235000019409 alitame Nutrition 0.000 description 1
- 108010009985 alitame Proteins 0.000 description 1
- HDTRYLNUVZCQOY-LIZSDCNHSA-N alpha,alpha-trehalose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-LIZSDCNHSA-N 0.000 description 1
- WQZGKKKJIJFFOK-PHYPRBDBSA-N alpha-D-galactose Chemical compound OC[C@H]1O[C@H](O)[C@H](O)[C@@H](O)[C@H]1O WQZGKKKJIJFFOK-PHYPRBDBSA-N 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 230000003321 amplification Effects 0.000 description 1
- PYMYPHUHKUWMLA-UHFFFAOYSA-N arabinose Natural products OCC(O)C(O)C(O)C=O PYMYPHUHKUWMLA-UHFFFAOYSA-N 0.000 description 1
- 239000005441 aurora Substances 0.000 description 1
- 230000009286 beneficial effect Effects 0.000 description 1
- SRBFZHDQGSBBOR-UHFFFAOYSA-N beta-D-Pyranose-Lyxose Natural products OC1COC(O)C(O)C1O SRBFZHDQGSBBOR-UHFFFAOYSA-N 0.000 description 1
- GUBGYTABKSRVRQ-QUYVBRFLSA-N beta-maltose Chemical compound OC[C@H]1O[C@H](O[C@H]2[C@H](O)[C@@H](O)[C@H](O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@@H]1O GUBGYTABKSRVRQ-QUYVBRFLSA-N 0.000 description 1
- 235000013361 beverage Nutrition 0.000 description 1
- 230000015572 biosynthetic process Effects 0.000 description 1
- 210000004369 blood Anatomy 0.000 description 1
- 239000008280 blood Substances 0.000 description 1
- 210000004556 brain Anatomy 0.000 description 1
- 238000010804 cDNA synthesis Methods 0.000 description 1
- 235000019577 caloric intake Nutrition 0.000 description 1
- 150000001720 carbohydrates Chemical class 0.000 description 1
- 150000001768 cations Chemical class 0.000 description 1
- 230000021164 cell adhesion Effects 0.000 description 1
- 230000010261 cell growth Effects 0.000 description 1
- 239000007795 chemical reaction product Substances 0.000 description 1
- 210000004978 chinese hamster ovary cell Anatomy 0.000 description 1
- 229960005091 chloramphenicol Drugs 0.000 description 1
- WIIZWVCIJKGZOK-RKDXNWHRSA-N chloramphenicol Chemical compound ClC(Cl)C(=O)N[C@H](CO)[C@H](O)C1=CC=C([N+]([O-])=O)C=C1 WIIZWVCIJKGZOK-RKDXNWHRSA-N 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 230000000052 comparative effect Effects 0.000 description 1
- 230000009918 complex formation Effects 0.000 description 1
- 150000001875 compounds Chemical class 0.000 description 1
- 108010010165 curculin Proteins 0.000 description 1
- XUJNEKJLAYXESH-UHFFFAOYSA-N cysteine Natural products SCC(N)C(O)=O XUJNEKJLAYXESH-UHFFFAOYSA-N 0.000 description 1
- 235000018417 cysteine Nutrition 0.000 description 1
- 210000000805 cytoplasm Anatomy 0.000 description 1
- HEBKCHPVOIAQTA-NGQZWQHPSA-N d-xylitol Chemical compound OC[C@H](O)C(O)[C@H](O)CO HEBKCHPVOIAQTA-NGQZWQHPSA-N 0.000 description 1
- 235000013365 dairy product Nutrition 0.000 description 1
- 230000007423 decrease Effects 0.000 description 1
- 238000012217 deletion Methods 0.000 description 1
- 230000037430 deletion Effects 0.000 description 1
- 238000000586 desensitisation Methods 0.000 description 1
- 239000003814 drug Substances 0.000 description 1
- 239000012636 effector Substances 0.000 description 1
- 230000000694 effects Effects 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 230000008451 emotion Effects 0.000 description 1
- 108010048367 enhanced green fluorescent protein Proteins 0.000 description 1
- 235000019414 erythritol Nutrition 0.000 description 1
- UNXHWFMMPAWVPI-ZXZARUISSA-N erythritol Chemical compound OC[C@H](O)[C@H](O)CO UNXHWFMMPAWVPI-ZXZARUISSA-N 0.000 description 1
- 229940009714 erythritol Drugs 0.000 description 1
- 210000003527 eukaryotic cell Anatomy 0.000 description 1
- 230000001747 exhibiting effect Effects 0.000 description 1
- 238000002474 experimental method Methods 0.000 description 1
- 239000000796 flavoring agent Substances 0.000 description 1
- 235000019634 flavors Nutrition 0.000 description 1
- FTSSQIKWUOOEGC-RULYVFMPSA-N fructooligosaccharide Chemical compound OC[C@H]1O[C@@](CO)(OC[C@@]2(OC[C@@]3(OC[C@@]4(OC[C@@]5(OC[C@@]6(OC[C@@]7(OC[C@@]8(OC[C@@]9(OC[C@@]%10(OC[C@@]%11(O[C@H]%12O[C@H](CO)[C@@H](O)[C@H](O)[C@H]%12O)O[C@H](CO)[C@@H](O)[C@@H]%11O)O[C@H](CO)[C@@H](O)[C@@H]%10O)O[C@H](CO)[C@@H](O)[C@@H]9O)O[C@H](CO)[C@@H](O)[C@@H]8O)O[C@H](CO)[C@@H](O)[C@@H]7O)O[C@H](CO)[C@@H](O)[C@@H]6O)O[C@H](CO)[C@@H](O)[C@@H]5O)O[C@H](CO)[C@@H](O)[C@@H]4O)O[C@H](CO)[C@@H](O)[C@@H]3O)O[C@H](CO)[C@@H](O)[C@@H]2O)[C@@H](O)[C@@H]1O FTSSQIKWUOOEGC-RULYVFMPSA-N 0.000 description 1
- 229940107187 fructooligosaccharide Drugs 0.000 description 1
- 230000004927 fusion Effects 0.000 description 1
- 235000021255 galacto-oligosaccharides Nutrition 0.000 description 1
- 150000003271 galactooligosaccharides Chemical class 0.000 description 1
- 229930182830 galactose Natural products 0.000 description 1
- 229960001031 glucose Drugs 0.000 description 1
- 235000001727 glucose Nutrition 0.000 description 1
- 125000003630 glycyl group Chemical group [H]N([H])C([H])([H])C(*)=O 0.000 description 1
- 238000012203 high throughput assay Methods 0.000 description 1
- 238000012188 high-throughput screening assay Methods 0.000 description 1
- 235000012907 honey Nutrition 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 230000010354 integration Effects 0.000 description 1
- 229960000318 kanamycin Drugs 0.000 description 1
- 229930027917 kanamycin Natural products 0.000 description 1
- SBUJHOSQTJFQJX-NOAMYHISSA-N kanamycin Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CN)O[C@@H]1O[C@H]1[C@H](O)[C@@H](O[C@@H]2[C@@H]([C@@H](N)[C@H](O)[C@@H](CO)O2)O)[C@H](N)C[C@@H]1N SBUJHOSQTJFQJX-NOAMYHISSA-N 0.000 description 1
- 229930182823 kanamycin A Natural products 0.000 description 1
- 239000000832 lactitol Substances 0.000 description 1
- 235000010448 lactitol Nutrition 0.000 description 1
- VQHSOMBJVWLPSR-JVCRWLNRSA-N lactitol Chemical compound OC[C@H](O)[C@@H](O)[C@@H]([C@H](O)CO)O[C@@H]1O[C@H](CO)[C@H](O)[C@H](O)[C@H]1O VQHSOMBJVWLPSR-JVCRWLNRSA-N 0.000 description 1
- 229960003451 lactitol Drugs 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 229940069445 licorice extract Drugs 0.000 description 1
- 239000002502 liposome Substances 0.000 description 1
- 239000000845 maltitol Substances 0.000 description 1
- VQHSOMBJVWLPSR-WUJBLJFYSA-N maltitol Chemical compound OC[C@H](O)[C@@H](O)[C@@H]([C@H](O)CO)O[C@H]1O[C@H](CO)[C@@H](O)[C@H](O)[C@H]1O VQHSOMBJVWLPSR-WUJBLJFYSA-N 0.000 description 1
- 235000010449 maltitol Nutrition 0.000 description 1
- 229940035436 maltitol Drugs 0.000 description 1
- 239000000594 mannitol Substances 0.000 description 1
- 235000010355 mannitol Nutrition 0.000 description 1
- 229960001855 mannitol Drugs 0.000 description 1
- 230000028161 membrane depolarization Effects 0.000 description 1
- HEBKCHPVOIAQTA-UHFFFAOYSA-N meso ribitol Natural products OCC(O)C(O)C(O)CO HEBKCHPVOIAQTA-UHFFFAOYSA-N 0.000 description 1
- DZNKOAWEHDKBEP-UHFFFAOYSA-N methyl 2-[6-[bis(2-methoxy-2-oxoethyl)amino]-5-[2-[2-[bis(2-methoxy-2-oxoethyl)amino]-5-methylphenoxy]ethoxy]-1-benzofuran-2-yl]-1,3-oxazole-5-carboxylate Chemical compound COC(=O)CN(CC(=O)OC)C1=CC=C(C)C=C1OCCOC(C(=C1)N(CC(=O)OC)CC(=O)OC)=CC2=C1OC(C=1OC(=CN=1)C(=O)OC)=C2 DZNKOAWEHDKBEP-UHFFFAOYSA-N 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 235000013379 molasses Nutrition 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 229960004927 neomycin Drugs 0.000 description 1
- 235000019412 neotame Nutrition 0.000 description 1
- HLIAVLHNDJUHFG-HOTGVXAUSA-N neotame Chemical compound CC(C)(C)CCN[C@@H](CC(O)=O)C(=O)N[C@H](C(=O)OC)CC1=CC=CC=C1 HLIAVLHNDJUHFG-HOTGVXAUSA-N 0.000 description 1
- 108010070257 neotame Proteins 0.000 description 1
- 210000005036 nerve Anatomy 0.000 description 1
- 210000002569 neuron Anatomy 0.000 description 1
- 239000002858 neurotransmitter agent Substances 0.000 description 1
- 238000003199 nucleic acid amplification method Methods 0.000 description 1
- 108020004707 nucleic acids Proteins 0.000 description 1
- 102000039446 nucleic acids Human genes 0.000 description 1
- 239000002773 nucleotide Substances 0.000 description 1
- 125000003729 nucleotide group Chemical group 0.000 description 1
- 235000015097 nutrients Nutrition 0.000 description 1
- 235000020824 obesity Nutrition 0.000 description 1
- 230000003204 osmotic effect Effects 0.000 description 1
- 235000019629 palatability Nutrition 0.000 description 1
- 239000012466 permeate Substances 0.000 description 1
- 238000001556 precipitation Methods 0.000 description 1
- 229950010131 puromycin Drugs 0.000 description 1
- MUPFEKGTMRGPLJ-ZQSKZDJDSA-N raffinose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO[C@@H]2[C@@H]([C@@H](O)[C@@H](O)[C@@H](CO)O2)O)O1 MUPFEKGTMRGPLJ-ZQSKZDJDSA-N 0.000 description 1
- 210000003370 receptor cell Anatomy 0.000 description 1
- 230000000717 retained effect Effects 0.000 description 1
- 238000010839 reverse transcription Methods 0.000 description 1
- 238000003757 reverse transcription PCR Methods 0.000 description 1
- 229910052701 rubidium Inorganic materials 0.000 description 1
- IGLNJRXAVVLDKE-UHFFFAOYSA-N rubidium atom Chemical compound [Rb] IGLNJRXAVVLDKE-UHFFFAOYSA-N 0.000 description 1
- 235000019600 saltiness Nutrition 0.000 description 1
- 235000019643 salty taste Nutrition 0.000 description 1
- 238000012216 screening Methods 0.000 description 1
- 239000006152 selective media Substances 0.000 description 1
- 230000035807 sensation Effects 0.000 description 1
- 235000019615 sensations Nutrition 0.000 description 1
- 230000008786 sensory perception of smell Effects 0.000 description 1
- 230000008054 signal transmission Effects 0.000 description 1
- 230000011664 signaling Effects 0.000 description 1
- SKORRGYRKQDXRS-UHFFFAOYSA-M sodium;2-(4-methoxyphenoxy)propanoate Chemical compound [Na+].COC1=CC=C(OC(C)C([O-])=O)C=C1 SKORRGYRKQDXRS-UHFFFAOYSA-M 0.000 description 1
- 210000001082 somatic cell Anatomy 0.000 description 1
- 239000000600 sorbitol Substances 0.000 description 1
- 229960002920 sorbitol Drugs 0.000 description 1
- 235000010356 sorbitol Nutrition 0.000 description 1
- 235000019614 sour taste Nutrition 0.000 description 1
- 229940013618 stevioside Drugs 0.000 description 1
- OHHNJQXIOPOJSC-UHFFFAOYSA-N stevioside Natural products CC1(CCCC2(C)C3(C)CCC4(CC3(CCC12C)CC4=C)OC5OC(CO)C(O)C(O)C5OC6OC(CO)C(O)C(O)C6O)C(=O)OC7OC(CO)C(O)C(O)C7O OHHNJQXIOPOJSC-UHFFFAOYSA-N 0.000 description 1
- 235000019202 steviosides Nutrition 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 235000019408 sucralose Nutrition 0.000 description 1
- BAQAVOSOZGMPRM-QBMZZYIRSA-N sucralose Chemical compound O[C@@H]1[C@@H](O)[C@@H](Cl)[C@@H](CO)O[C@@H]1O[C@@]1(CCl)[C@@H](O)[C@H](O)[C@@H](CCl)O1 BAQAVOSOZGMPRM-QBMZZYIRSA-N 0.000 description 1
- 125000000185 sucrose group Chemical group 0.000 description 1
- 210000000225 synapse Anatomy 0.000 description 1
- 238000003786 synthesis reaction Methods 0.000 description 1
- 238000012360 testing method Methods 0.000 description 1
- 230000005026 transcription initiation Effects 0.000 description 1
- 229940074410 trehalose Drugs 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 239000000811 xylitol Substances 0.000 description 1
- 235000010447 xylitol Nutrition 0.000 description 1
- HEBKCHPVOIAQTA-SCDXWVJYSA-N xylitol Chemical compound OC[C@H](O)[C@@H](O)[C@H](O)CO HEBKCHPVOIAQTA-SCDXWVJYSA-N 0.000 description 1
- 229960002675 xylitol Drugs 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C07—ORGANIC CHEMISTRY
- C07K—PEPTIDES
- C07K14/00—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof
- C07K14/435—Peptides having more than 20 amino acids; Gastrins; Somatostatins; Melanotropins; Derivatives thereof from animals; from humans
- C07K14/705—Receptors; Cell surface antigens; Cell surface determinants
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N33/00—Investigating or analysing materials by specific methods not covered by groups G01N1/00 - G01N31/00
- G01N33/48—Biological material, e.g. blood, urine; Haemocytometers
- G01N33/50—Chemical analysis of biological material, e.g. blood, urine; Testing involving biospecific ligand binding methods; Immunological testing
- G01N33/53—Immunoassay; Biospecific binding assay; Materials therefor
- G01N33/5308—Immunoassay; Biospecific binding assay; Materials therefor for analytes not provided for elsewhere, e.g. nucleic acids, uric acid, worms, mites
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2333/00—Assays involving biological materials from specific organisms or of a specific nature
- G01N2333/435—Assays involving biological materials from specific organisms or of a specific nature from animals; from humans
- G01N2333/705—Assays involving receptors, cell surface antigens or cell surface determinants
-
- G—PHYSICS
- G01—MEASURING; TESTING
- G01N—INVESTIGATING OR ANALYSING MATERIALS BY DETERMINING THEIR CHEMICAL OR PHYSICAL PROPERTIES
- G01N2400/00—Assays, e.g. immunoassays or enzyme assays, involving carbohydrates
Landscapes
- Health & Medical Sciences (AREA)
- Life Sciences & Earth Sciences (AREA)
- Chemical & Material Sciences (AREA)
- Molecular Biology (AREA)
- Immunology (AREA)
- Engineering & Computer Science (AREA)
- Biomedical Technology (AREA)
- Medicinal Chemistry (AREA)
- Cell Biology (AREA)
- Hematology (AREA)
- General Health & Medical Sciences (AREA)
- Urology & Nephrology (AREA)
- Organic Chemistry (AREA)
- Biochemistry (AREA)
- Biotechnology (AREA)
- Gastroenterology & Hepatology (AREA)
- Analytical Chemistry (AREA)
- Food Science & Technology (AREA)
- Microbiology (AREA)
- General Physics & Mathematics (AREA)
- Pathology (AREA)
- Toxicology (AREA)
- Zoology (AREA)
- Physics & Mathematics (AREA)
- Biophysics (AREA)
- Genetics & Genomics (AREA)
- Proteomics, Peptides & Aminoacids (AREA)
- Tropical Medicine & Parasitology (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Peptides Or Proteins (AREA)
- Measuring Or Testing Involving Enzymes Or Micro-Organisms (AREA)
Description
具体的には、例えば、特許文献1の実施例11において、Gα15発現細胞株(Aurora BioscienceのHEK-293細胞株)に、hT1R2の発現コンストラクト(プラスミドSAV2486)を含む線状化したpEAK10由来(Edge Biosystems)ベクターと、hT1R3の発現コンストラクト(プラスミドSXV550)を含むpCDNA3.1/ZEO由来(Invitrogen)ベクターとを、トランスフェクトすることにより、hT1R2/hT1R3を共発現させて生成した細胞株が報告されている。
そのため、得られた細胞系には、以下の問題点があった。
(1)従来の細胞系の中には、T1R2及びT1R3のいずれか一方又は両方が発現されておらず、中には、細胞内の一連のシグナル伝達機構を実質的に備えていないものがあった。すなわち、従来の細胞系は、甘味の受容・伝達機構を備えた機能的に優れたモデルであるとは言えなかった。
(2)Gタンパク質は細胞間で一定量が発現されるものの、T1R2、T1R3の発現量及び発現比率が細胞間で異なり、バラツキが大きかった。そのため、甘味に関する評価結果について、細胞間での直接比較、特に各種点変異体コンストラクトを用いて発現させた細胞間での直接比較は困難であった。
(3)細胞の継代に伴い、導入されたT1R2、T1R3をコードする遺伝子が排除されることが高頻度で起こり、したがって、連続して継代的に甘味受容体を安定的に発現できる細胞系ではなかった。
(4)細胞系のGタンパク質を変更する場合には、所望するGタンパク質を発現する細胞を新たに作製、入手しておく必要があった。
(5)スクロース等の閾値が高い甘味物質に対しては、細胞の応答性が実用上必ずしも十分とは言えなかった。
現コンストラクトを発現させた安定発現細胞体を提供することにある。
(1)本発明の甘味受容体発現コンストラクトは、甘味受容体刺激による細胞内シグナル伝達が生じるように、T1R2、T1R3、及びそれらに共役するGタンパク質を機能的に発現することができる。したがって、上記コンストラクトを発現させた細胞株は、実際の甘味受容によって生じる甘味の知覚を、in vivoで客観的に評価することができる最適なモデルとなり、また、甘味物質、甘味調節物質などを同定、選択する味覚センサーとして使用することができ、特に高スループットスクリーニングアッセイにおいて極めて有用である。なお、甘味調節物質とは、甘味受容体(T1R2+T1R3)に作用することによって、甘味物質単独で得られる甘味受容体からの生理的応答、すなわち、甘味の強度を改変させる物質を意味する。
(2)本発明の甘味受容体発現コンストラクトは、T1R2、T1R3、及びそれらに共役するGタンパク質を同等の比率で機能的に発現することができる。したがって、本発明の細胞株によれば、異なる点変異体を導入した甘味受容体、あるいは種々のGタンパク質を発現させた複数の安定発現株について、直接比較による比較実験を行うことができる。
(3)本発明の細胞株は、長期にわたってT1R2、T1R3、及びそれらに共役するGタンパク質を発現することができ、安定性が高い。継代数にかかわらず、T1R2、T1R3、及びそれらに共役するGタンパク質の発現量の変化が小さいため、長期間継代して利用することができる。
(4)本発明の細胞株を作製する場合、従来のように、所望するGタンパク質を発現する細胞を作製、入手しておく必要がない。
(5)本発明の細胞株は、甘味物質に対して高い応答性を示すため、甘味の強度が閾値以下にある物質であっても、甘味の強度を評価することができる。
本発明において、発現コンストラクトとは、所望のコード配列及び該コード配列を発現させるために必要なプロモーター、ターミネーター、マーカー遺伝子、FRT部位をコードする遺伝子などの適切な核酸配列を連結したDNA断片を細胞に移動させる核酸分子の意味であり、発現ベクターと同様の意味である。
また、甘味受容体サブユニットT1R2及びT1R3には、各サブユニットの全ての部分、すなわち、7つの膜貫通領域及び対応する細胞質及び細胞外のループからなる膜貫通ドメイン、ビーナス・フライ・トラップドメイン、高システインドメイン及びC末端ドメインの全ての領域が含まれる。
また、甘味受容体サブユニットT1R2及びT1R3には、T1R2及びT1R3の点変異体、キメラも含む。
ところで、本発明においては、特に断りがない限り、核酸の塩基配列には、本明細書に記載される特定の配列に加えて、適宜置換、欠失、挿入又は付加変異を導入したものも含み、例えば、縮重コドンを有する相同的配列なども含む。
Flp-Inシステム(Invitrogen)を用いて、安定的にT1R2及びT1R3並びにGタンパク質αサブユニットを発現する培養細胞株を迅速かつ効果的に得ることができる。図1に、pcDNA5/FRT(Invitrogen)の構造を示す。
それに対し、本発明の甘味受容体発現コンストラクトは、上記特徴を有することにより、甘味受容体サブユニットT1R2及びT1R3並びにGタンパク質αサブユニットをコードする各遺伝子を迅速かつ同時に発現することができる。これら3つの遺伝子は、1つもしくは2つのmRNAに転写された後、3つのタンパク質、すなわち、T1R2及びT1R3並びにGタンパク質αサブユニットに翻訳されて、各遺伝子を同時に発現させるため、上記のような問題点は生じない。
(a) pcDNA5/FRT(Invitrogen)のマルチクローニングサイト(塩基番号895−1010)以外の場所に6塩基の置換を行い、制限酵素EcoRVの認識配列(5’-GATATC-3’)を新たに作製する(図3参照)。
この工程(a)の好適な態様の1つとして、pcDNA5/FRT(Invitrogen)の制限酵素Bgl IIの認識配列(塩基番号12―17)の直下(塩基番号18―23)にEcoRVの認識配列を導入する態様が挙げられる。この態様は、例えば、以下のサブ工程(a1)〜(a5)に従うことにより、実施することができる。
(a1) 5’末端にBgl IIの認識配列(5’-AGATCT-3’)、その直下にEcoRVの認識配列を持つセンスプライマーを設計、作製する。一方、BGH pA配列中の配列を持つアンチセンスプライマーを設計、作製する。
(a2)(a1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、pcDNA5/FRT(Invitrogen)をテンプレートとして、ポリメラーゼ連鎖反応(PCR)を行い、Bgl IIとEcoRVの各認識配列が連結した配列を含むDNA断片を増幅する。なお、PCRとは、センスプライマー及びアンチセンスプライマーの間のDNA配列を増幅する技術であり、増幅とは、遺伝子配列のコピー数を増加させることをいう。
PCRについては、適宜最適化された条件で行えばよいが、具体例として、98℃で30秒間×1サイクル、(98℃で30秒間、55℃で30秒間、72℃で55秒間)×30サイクル、72℃で10分間×1サイクル、その後、4℃に冷却する条件が例示される。
(a3) (a2)で増幅したDNA断片を、Bgl II及びNot Iで消化する。
(a4) pcDNA5/FRT(Invitrogen)を、Bgl II及びNot Iで消化する。
(a5) (a3)で得られたDNA断片と、(a4)で得られたpcDNA5/FRT(Invitrogen)とを、ライゲーション反応によって結合させることにより、pcDNA5/FRT(Invitrogen)のBgl IIの認識配列の直下にEcoRVの認識配列を持つベクターを作製する。
ライゲーション反応は、通常のT4 DNAリガーゼを用いて行えばよいが、迅速かつ簡便に処理を行うために、1液タイプのDNAライゲーション試薬であるLigation high Ver.2(TOYOBO)を用いることが好ましい。
この工程(b)は、例えば、以下のサブ工程(b1)〜(b8)に従うことにより、実施することができる。
(b1) hT1R3のコード領域の直前及び直後に、それぞれAsc Iの認識配列(5’-GGCGCGCC-3’)及びNot Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製する。
(b2) (b1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、hT1R3をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R3をコードするcDNAを増幅する。
(b3) (b2)で得られたhT1R3をコードするcDNA断片を、制限酵素Asc I及びNot Iで消化する。
(b4) pEAK10(Edge Biosystems)を、制限酵素Asc I及びNot Iで消化する。
(b5) (b3)で得られたhT1R3をコードするcDNA断片と、(b4)で得られたpEAK10(Edge Biosystems)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R3をコードするcDNAを挿入する。
(b6) (b5)で得られたpEAK10(Edge Biosystems)を、制限酵素Hind III及びNot Iで消化し、アガロース電気泳動により、DNA断片を分離して、hT1R3のcDNA断片を精製する。
(b7) (a5)で作製したベクターを、Hind III及びNot Iで消化する。
(b8) (b6)で得られたhT1R3をコードするcDNA断片と、(b7)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させることにより、(a5)で作製したベクターのマルチクローニングサイトに、hT1R3をコードするcDNAを挿入する。
この工程(c)は、例えば、以下のサブ工程(c1)〜(c11)に従うことにより、実施することができる。
(c1) IRES配列の直前にEco52Iの認識配列(5’-CGGCCG-3’)を持つようなセンスプライマーを設計、作製する。一方、IRES配列の終了する部分に相当するアンチセンスプライマーを設計、作製する。
(c2) (c1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅する。なお、EGFPは、Enhansed Green Fluorescence Proteinの略である。
(c3) hG16gust44のコード領域の直前及び直後に、Not Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製し、hG16gust44をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hG16gust44をコードするcDNAを増幅する。増幅断片を、Not Iで消化する。
(c4) pEAK10(Edge Biosystems)を、Not Iで消化する。
(c5) (c3)で得られたhG16gust44をコードするcDNA断片と、(c4)で得られたpEAK10(Edge Biosystems)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hG16gust44をコードするcDNAを挿入したベクター(hG16gust44/pEAK10)を作製する。
(c6) hG16gust44の開始コドンを含む18塩基のセンスプライマーを設計、作製する。一方、hGH pA配列中の配列を持つアンチセンスプライマーを設計、作製する。 なお、pA配列(ポリアデニル化配列)は、組換え転写産物を安定化するために、RNA転写産物の終結及びポリアデニル化を指示するDNA配列である。pA配列以外にも、種々の終結配列が公知であり、本発明の甘味受容体発現コンストラクトに使用しうる。pA配列は、プラスミドに存在する内在性のものでもよい。通常使用されるpA配列は、SV40のポリA配列であり、このポリA配列は237bpの制限酵素BamHI/BcII断片に含まれる。また、通常使用されるpA配列は、ウシ成長ホルモン(BGH:Bovine Growth Hormone)遺伝子に由来する。
(c7) (c5)で得られたhG16gust44/pEAK10をテンプレートとして、(c6)で作製したセンスプライマー及びアンチセンスプライマーを用いてPCRを行い、hG16gust44をコードするcDNAを増幅する。
(c8) (c2)で得られたIRES配列を、Eco52Iで消化する。
(c9) (c7)で得られたhG16gust44をコードするcDNAを、Not Iで消化する。
(c10) pBluescript II SK(-)を、Not Iで消化する。
(c11) (c8)で得られたIRES配列と、(c9)で得られたhG16gust44をコードするcDNA断片と、(c10)で得られたpBluescript II SK(-)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、IRES2-hG16gust44/pBluescript II SK(-)を作製する。
この工程(d)は、例えば、以下のサブ工程(d1)〜(d3)に従うことにより、実施することができる。
(d1) (c11)で作製したIRES2-hG16gust44/pBluescript II SK(-)を、Eco52Iで消化し、アガロース電気泳動により、cDNA断片を分離して、IRES2-hG16gust44配列を精製する。
(d2) (b8)で作製したベクターのhT1R3をコードするDNA配列の直後に存在する部位をNot Iで消化する。
(d3) (d1)で得られたIRES2-hG16gust44配列と、(d2)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、(b8)で作製したベクターのhT1R3をコードするDNA配列の直後に、IRES2-hG16gust44配列を挿入する。この結果、hT1R3-IRES2-hG16gust44配列を含むpcDNA5/FRTが得られる。
この工程は、例えば、以下のサブ工程(e1)〜(e5)に従うことにより、実施することができる。
(e1) hT1R2のコード領域の直前及び直後に、それぞれAsc Iの認識配列(5’-GGCGCGCC-3’)及びNot Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製する。
(e2) (e1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、hT1R2をコードするDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R2をコードするcDNAを増幅する。
(e3) (e2)で得られたhT1R2をコードするcDNAを、Asc I及びNot Iで消化する。
(e4) pEAK10(Edge Biosystems)を、Asc I及びNot Iで消化する。
(e5) (e3)で得られたhT1R2をコードするcDNA断片と、(e4)で得られたpEAK10(Edge Biosystems)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R2をコードするcDNAを挿入する。
この工程は、例えば、以下のサブ工程(f1)〜(f3)に従うことにより、実施することができる。
(f1) (c11)で作製したIRES2-hG16gust44/pBluescript II SK(-)を、Eco52Iで消化し、アガロース電気泳動により、cDNA断片を分離して、IRES2-hG16gust44配列を精製する。
(f2) (e5)で得られたベクターのhT1R2をコードするcDNAの直後に存在する部位をNot Iで消化する。
(f3) (f1)で得られたIRES2-hG16gust44配列と、(f2)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、(e5)で得られたベクターのhT1R2をコードするcDNAの直後に、IRES2-hG16gust44配列を挿入する。この結果、hT1R2-IRES2-hG16gust44配列を含むpEAK10(Edge Biosystems)が得られる。
この工程(g)は、例えば、以下のサブ工程(g1)〜(g3)に従うことにより、実施することができる。
(g1) In-Fusion反応を行うためのプライマーを設計、作製し、これを用いて、(f3)で作製したベクターをテンプレートとして、EF-1αプロモーター-hT1R2-IRES2-hG16gust44-hGH pAの領域をPCRによって増幅する。上記プライマーは、(d3)で得られたベクターを制限酵素で線状化したものの末端と相同な約15塩基を、上記領域のDNA断片に付加するようにして設計する。
(g2) (d3)で得られたベクターを、EcoRVで消化する。
(g3) (g1)で得られたEF-1αプロモーター-hT1R2-IRES2-hG16gust44-hGH pA配列断片と、(g2)で得られたベクターとを、In-Fusion Advantage PCR Cloning Kit(Clontech)を用いて結合させて、(d3)で得られたベクターのhT1R3-IRES2-hG16gust44配列の上流に、(f3)で作製したベクターのhT1R2-IRES2-hG16gust44配列を挿入した第一の具体的な態様の甘味受容体発現コンストラクトを作製する。In-Fusion Advantage PCR Cloning Kit(Clontech)による結合は、(g1)で得られたEF-1αプロモーター-hT1R2-IRES2-hG16gust44-hGH pA配列断片と、(g2)で得られたベクターを混合し、In-Fusion酵素と所定のバッファーを加えて、通常、37℃で15分、次いで、50℃で15分反応を行う。In-Fusion Advantage PCR Cloning Kit(Clontech)によれば、制限酵素サイトの制約を受けることなく、また、長鎖DNA断片でも、目的DNA断片のクローニングが可能である。
まず、第一の好適な具体的態様の発現コンストラクトの作製で述べた工程(a)〜(e)を同様にして行う。その後、In-Fusion反応を行うためのプライマーを設計、作製し、これを用いて、工程(e)で作製したベクターをテンプレートとして、EF-1αプロモーター-hT1R2-hGH pAの領域をPCRによって増幅する。そして、得られたEF-1αプロモーター-hT1R2-hGH pA配列断片と、 EcoRVで消化した工程(d)で得られたベクターとを、In-Fusion Advantage PCR Cloning Kit(Clontech)を用いて結合させることにより、工程(d)で得られたベクターのhT1R3-IRES2-hG16gust44配列の上流に、上記PCR産物であるEF-1αプロモーター-hT1R2-hGH pAの配列を挿入して、上述した第二の好適な具体的態様の甘味受容体発現コンストラクトを作製する。
(a’) pcDNA5/FRT(Invitrogen)のマルチクローニングサイト(塩基番号895−1010)に、hT1R3をコードするcDNAを挿入する。
この工程(a’)は、例えば、以下のサブ工程(a’1)〜(a’8)に従うことにより、実施することができる。
(a’1) hT1R3のコード領域の直前及び直後に、それぞれAsc Iの認識配列(5’-GGCGCGCC-3’)及びNot Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製する。
(a’2) (a’1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、hT1R3をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R3をコードするcDNAを増幅する。
(a’3) (a’2)で得られたhT1R3をコードするcDNA断片を、制限酵素Asc I及びNot Iで消化する。
(a’4) pEAK10(Edge Biosystems)を、制限酵素Asc I及びNot Iで消化する。
(a’5) (a’3)で得られたhT1R3をコードするcDNA断片と、(a’4)で得られたpEAK10(Edge Biosystems)とを、Ligation
high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R3をコードするcDNAを挿入する。
(a’6) (a’5)で得られたpEAK10(Edge Biosystems)を、制限酵素Hind III及びNot Iで消化し、アガロース電気泳動により、DNA断片を分離して、hT1R3のcDNA断片を精製する。
(a’7) pcDNA5/FRT(Invitrogen)を、Hind III及びNot Iで消化する。
(a’8) (a’6)で得られたhT1R3をコードするcDNA断片と、(a’7)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させることにより、pcDNA5/FRT(Invitrogen)のマルチクローニングサイトに、hT1R3をコードするcDNAを挿入する。
この工程(b’)は、例えば、以下のサブ工程(b’1)〜(b’16)に従うことにより、実施することができる。
(b’1) IRES配列の直前にEco52Iの認識配列(5’-CGGCCG-3’)を持つようなセンスプライマーを設計、作製する。一方、IRES配列の終了する部分に相当するアンチセンスプライマーを設計、作製する。
(b’2) (b’1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅する。
(b’3) hT1R2のコード領域の直前及び直後に、それぞれAsc Iの認識配列(5’-GGCGCGCC-3’)及びNot Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製する。
(b’4) これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R2をコードするDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R2をコードするcDNAを増幅する。
(b’5) (b’4)で得られたhT1R2をコードするcDNAを、Asc I及びNot Iで消化する。
(b’6) pEAK10(Edge Biosystems)を、Asc I及びNot Iで消化する。
(b’7) (b’5)で得られたhT1R2をコードするcDNA断片と、(b’6)で得られたpEAK10(Edge Biosystems)とを、Ligation
high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R2をコードするcDNAを挿入する。
(b’8) hT1R2の開始コドンを含む18塩基のセンスプライマーを設計、作製する。一方、hGH pA配列中の配列を持つアンチセンスプライマーを設計、作製する。(b’9) (b’7)で得られたhT1R2/pEAK10をテンプレートとして、(b’8)で作製したセンスプライマー及びアンチセンスプライマーを用いてPCRを行い、hT1R2をコードするcDNAを増幅する。
(b’10) (b’2)で得られたIRES配列を、Eco52Iで消化する。
(b’11) (b’9)で得られたhT1R2をコードするcDNAを、Not Iで消化する。
(b’12) pBluescript II SK(-)を、Not Iで消化する。
(b’13) (b’10)で得られたIRES配列と、(b’11)で得られたhT1R2をコードするcDNA断片と、(b’12)で得られたpBluescript II SK(-)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、IRES2-hT1R2/pBluescript II SK(-)を作製する。
この工程(c’)は、例えば、以下のサブ工程(c’1)〜(c’3)に従うことにより、実施することができる。
(c’1) (b’13)で作製したIRES2-hT1R2/pBluescript II SK(-)を、Eco52Iで消化し、アガロース電気泳動により、cDNA断片を分離して、IRES2-hT1R2配列を精製する。
(c’2) (a’8)で作製したベクターのhT1R3をコードするDNA配列の直後に存在する部位をNot Iで消化する。
(c’3) (c’1)で得られたIRES2-hT1R2配列と、(c’2)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、(a’8)で作製したベクターのhT1R3をコードするDNA配列の直後に、IRES2-hT1R2配列を挿入する。この結果、hT1R3-IRES2-hT1R2配列を含むpcDNA5/FRTが得られる。
この工程(d’)は、例えば、以下のサブ工程(d’1)〜(d’11)に従うことにより、実施することができる。
(d’1) IRES配列の直前にEco52Iの認識配列(5’-CGGCCG-3’)を持つようなセンスプライマーを設計、作製する。一方、IRES配列の終了する部分に相当するアンチセンスプライマーを設計、作製する。
(d’2) (d’1)で作製したセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅する。
(d’3) hG16gust44のコード領域の直前及び直後に、Not Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー、アンチセンスプライマーを設計、作製し、hG16gust44をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hG16gust44をコードするcDNAを増幅する。増幅断片を、Not Iで消化する。
(d’4) pEAK10(Edge Biosystems)を、Not Iで消化する。
(d’5) (d’3)で得られたhG16gust44をコードするcDNA断片と、(d’4)で得られたpEAK10(Edge Biosystems)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hG16gust44をコードするcDNAを挿入したベクター(hG16gust44/pEAK10)を作製する。
(d’6) hG16gust44の開始コドンを含む18塩基のセンスプライマーを設計、作製する。一方、hGH pA配列中の配列を持つアンチセンスプライマーを設計、作製する。
(d’7) (d’5)で得られたhG16gust44/pEAK10をテンプレートとして、(d’6)で作製したセンスプライマー及びアンチセンスプライマーを用いてPCRを行い、hG16gust44をコードするcDNAを増幅する。
(d’8) (d’2)で得られたIRES配列を、Eco52Iで消化する。
(d’9) (d’7)で得られたhG16gust44をコードするcDNAを、Not Iで消化する。
(d’10) pBluescript II SK(-)を、Not Iで消化する。
(d’11) (d’8)で得られたIRES配列と、(d’9)で得られたhG16gust44をコードするcDNA断片と、(d’10)で得られたpBluescript II SK(-)とを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、IRES2-hG16gust44/pBluescript II SK(-)を作製する。
この工程は、例えば、以下のサブ工程(e’1)〜(e’3)に従うことにより、実施することができる。
(e’1) (d’11)で作製したIRES2-hG16gust44/pBluescript II SK(-)を、Eco52Iで消化し、アガロース電気泳動により、cDNA断片を分離して、IRES2-hG16gust44配列を精製する。
(e’2) (c’3)で得られたベクターのhT1R2をコードするcDNAの直後に存在する部位をNot Iで消化する。
(e’3) (e’1)で得られたIRES2-hG16gust44配列と、(e’2)で得られたベクターとを、Ligation high Ver.2(TOYOBO)などによるライゲーション反応によって結合させて、(c’3)で得られたベクターのhT1R2をコードするcDNAの直後に、IRES2-hG16gust44配列を挿入する。これにより、上述した第三の好適な具体的態様の甘味受容体発現コンストラクトが得られる。
該培養細胞株の作製は、Flp-Inシステム(Invitrogen)を用いて、ゲノムDNA中にFRT部位が1か所組み込まれた293細胞に、上記甘味受容体発現コンストラクトと、Flpリコンビナーゼ発現ベクターであるpOG44をコトランスフェクションすることによって行われる。Flp-Inシステム(Invitrogen)によれば、トランジエントに発現したFlpリコンビナーゼにより、293細胞のゲノムDNA中に保持されたFRT部位が開裂して、その部分に外来遺伝子が導入され、該遺伝子が発現する。すなわち、T1R2及びT1R3並びにGタンパク質αサブユニットをコードする各遺伝子は、サッカロマイセス・セレビシエ(Saccharomyces cerevisiae)由来のFlpリコンビナーゼと、Flpリコンビナーゼの標的部位であるFRT部位を利用した部位特異的組換えにより、293細胞の染色体のFRT部位に挿入され、その結果、安定的にT1R2及びT1R3並びにGタンパク質αサブユニットを発現する培養細胞株が迅速かつ効果的に得られる。この培養細胞株は、T1R2及びT1R3並びにGタンパク質αサブユニットをコードする各遺伝子は、1つあるいは2つのmRNAに転写された後、3つのタンパク質、すなわち、T1R2及びT1R3並びにGタンパク質αサブユニットに翻訳される。このように、本発明の甘味受容体発現コンストラクトを宿主細胞にトランスフェクションする場合、部位特異的にDNAを切断かつ再結合する酵素を使用することが好ましく、Flpリコンビナーゼのほか、λインテグラーゼ、Kwリコンビナーゼなどが挙げられる。
まず、宿主細胞である293細胞に、FRT部位を導入するためのベクターであるpFRT/lacZeoを遺伝子導入し、導入された細胞をゼオシン(Zeocin、登録商標)で選択する。pFRT/lacZeoは、lacZとゼオシン耐性遺伝子の融合遺伝子を持つため、pFRT/lacZeoがゲノムに導入された細胞は、β-ガラクトシダーゼを発現し、ゼオシン耐性となる。ゼオシン感受性の程度をβ-ガラクトシダーゼアッセイにより確認することができる。
次いで、lacZ遺伝子に対してのサザンブロット法を行う。これにより、ゲノム中の転写されうる位置の1か所だけにFRT部位が組み込まれていることが確認される。ゲノムDNA中にFRT部位が1か所組み込まれた293細胞として、市販されているFlp-In-293細胞(Invitrogen)を用いるのが簡便である。
まず、前述したように、上記甘味受容体発現コンストラクトを発現させた培養細胞株を取得し、次いで、該培養細胞の所定の数を多数のウェル(24穴、48穴、96穴、384穴など)を有するマイクロプレートの各ウェルに撒き(例えば、1万〜50万個/ウェル)、所定の培地(例えば、DMEM培地)中で培養する。
その後、特定の甘味物質を添加した場合において、上記安定培養細胞株に発生した生理的応答を測定し、その測定結果に基づいて甘味物質の甘味を評価する。
まず、ハイグロマイシンBを除いた低グルコース(1,000mg/ml)DMEM培地で細胞を懸濁し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、7〜8万個ずつ撒く。
甘味物質又は甘味物質と試験する物質の添加直後から60〜120秒後にかけての蛍光反応(485nm励起で525nm蛍光)を測定することにより、甘味刺激に対する甘味受容体発現細胞の応答を定量することができる。
まず、ハイグロマイシンBを除いた低グルコース(1,000mg/ml)DMEM培地で細胞を懸濁し、96ウェルプレート(Greiner、Lumox)の各ウェル上に4〜8万個ずつ撒く。
以上のようにして、本発明では、生理的応答を測定する際に、カルシウムイメージング法において、蛍光特性の異なる2種類の上記蛍光指示薬を併用することにより、観察系によらない普遍的な現象を観察することができる。
甘味受容体発現コンストラクト(A)の作製
EF-1αプロモーターの下流に、hT1R2をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有し、かつ、この配列の下流にあるCMVプロモーターの下流に、hT1R3をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有する甘味受容体発現コンストラクト(A)(図2(A)参照)を、以下の手順に従って作製した。
TCGAGG)を設計、作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、pcDNA5/FRT(Invitrogen)をテンプレートとして、ポリメラーゼ連鎖反応(PCR)を行い、Bgl IIとEcoRVの各認識配列が連結した配列を含むDNA断片を増幅した。PCRは、後述する実施例も含め、全て、98℃で30秒間×1サイクル、(98℃で30秒間、55℃で30秒間、72℃で55秒間)×30サイクル、72℃で10分間×1サイクル、その後、4℃に冷却する条件で行った。
次いで、この増幅したDNA断片を、Bgl II及びNot Iで消化し、また、pcDNA5/FRT(Invitrogen)を、Bgl II及びNot Iで消化した。これらの制限酵素消化産物をLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させることにより、pcDNA5/FRT(Invitrogen)のBgl IIの認識配列の直下にEcoRVの認識配列を持つベクターを作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R3をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R3をコードするcDNAを増幅した。
増幅したT1R3をコードするcDNA断片を、Asc I及びNot Iで消化し、また、pEAK10(Edge Biosystems)を、Asc I及びNot Iで消化した。そして、これらの制限酵素消化産物をLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R3をコードするcDNAを挿入した。次いで、これをHind III及びNot Iで消化し、アガロース電気泳動により、DNA断片を分離して、hT1R3のcDNA断片を精製した。
このhT1R3のcDNA断片と、Hind III及びNot Iで消化した、pcDNA5/FRT(Invitrogen)のBgl IIの認識配列の直下にEcoRVの認識配列を持つベクターとを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させることにより、該ベクターのマルチクローニングサイトに、hT1R3をコードするcDNAを挿入した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅した。
hG16gust44のコード領域の直前及び直後に、Not Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー(配列番号7:GATCGCGGCCGCATGGCCCGCTCGCTGACC)、アンチセンスプライマー(配列番号8:GATCGCGGCCGCGAATTCACTAGTGATTTA)を設計、作製した。hG16gust44をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hG16gust44をコードするcDNAを増幅した。増幅断片をNot Iで消化し、pEAK10(Edge Biosystems)をNot Iで消化して、これらをLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hG16gust44をコードするcDNAを挿入したベクター(hG16gust44/pEAK10)を作製した。
次いで、hG16gust44の開始コドンを含む18塩基のセンスプライマー(配列番号9:ATGGCCCGCTCGCTGACC)及びhGH pA配列中の配列を持つアンチセンスプライマー(配列番号10:CTGGATGCAGGCTACTCTA)を設計、作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hG16gust44/pEAK10をテンプレートとして、PCRを行い、hG16gust44をコードするcDNAを増幅した。
次いで、Eco52Iで消化した上記IRES配列と、Not Iで消化した上記hG16gust44をコードするcDNAと、Not Iで消化したpBluescript II SK(-)とを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、IRES2-hG16gust44/pBluescript II SK(-)を作製した。
そして、マルチクローニングサイトにhT1R3をコードするcDNAを挿入した上記pcDNA5/FRT(Invitrogen)をNot Iで消化し、該ベクター中のhT1R3をコードするDNA配列の直後に存在する部分を切断した。このベクターと上記のIRES2-hG16gust44配列とを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、hT1R3をコードするDNA配列の直後に、IRES2-hG16gust44配列を挿入してなる、hT1R3-IRES2-hG16gust44配列を含むpcDNA5/FRTを得た。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R2をコードするDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R2をコードするcDNAを増幅した。得られたhT1R2をコードするcDNAを、Asc I及びNot Iで消化し、pEAK10(Edge Biosystems)を、Asc I及びNot Iで消化して、これらをLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R2をコードするcDNAを挿入した。
そして、hT1R2をコードするcDNAを挿入したpEAK10(Edge Biosystems)をNot Iで消化し、該ベクター中のhT1R2をコードするcDNAの直後に存在する部分を切断した。このベクターと上記IRES2-hG16gust44配列とを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、hT1R2をコードするcDNAの直後に、IRES2-hG16gust44配列を挿入し、hT1R2-IRES2-hG16gust44配列を含むpEAK10(Edge Biosystems)を得た。
そして、このEF-1αプロモーター-hT1R2-IRES2-hG16gust44-hGH pA配列断片と、EcoRVで消化したhT1R3-IRES2-hG16gust44配列を含むpcDNA5/FRTとを、In-Fusion Advantage PCR Cloning Kit(Clontech)を用いて結合させて、甘味受容体発現コンストラクト(A)を作製した。得られた甘味受容体発現コンストラクト(A)は、DNAシーケンシングにより、塩基配列に誤りがないことを確認した。
甘味受容体発現コンストラクト(B)の作製
EF-1αプロモーターの下流に、hT1R2をコードするcDNAの配列を有し、かつ、この配列の下流にあるCMVプロモーターの下流に、hT1R3をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有する甘味受容体発現コンストラクト(B)(図2(B)参照)を、以下の手順に従って作製した。
まず、実施例1の甘味受容体発現コンストラクト(A)の作製で述べた工程に従って、pcDNA5/FRT(Invitrogen)のBgl IIの認識配列の直下にEcoRVの認識配列を持つベクターの作製から、pEAK10(Edge Biosystems)に、hT1R2をコードするcDNAを挿入するまでの工程を実施した。
その後、In-Fusion反応を行うためのプライマー(配列番号15:ATCGGGAGATCTGATGCATAACTAGTGAGGCTC及び配列番号16:GCACCATAGGGGGATAGCGGATCCAGACATGAT)を設計、作製し、これを用いて、hT1R2をコードするcDNAを挿入したpEAK10(Edge Biosystems)をテンプレートとして、EF-1αプロモーター-hT1R2-hGH pAの領域をPCRによって増幅した。そして、得られたEF-1αプロモーター-hT1R2-hGH pA配列断片と、 EcoRVで消化したhT1R3-IRES2-hG16gust44配列を含むpcDNA5/FRT(Invitrogen)とを、In-Fusion Advantage PCR Cloning Kit(Clontech)を用いて結合させて、甘味受容体発現コンストラクト(B)を作製した。得られた甘味受容体発現コンストラクト(B)は、DNAシーケンシングにより、塩基配列に誤りがないことを確認した。
甘味受容体発現コンストラクト(C)の作製
CMVプロモーターの下流に、hT1R3をコードするcDNAとhT1R2をコードするcDNAを、IRES配列を挿んで連結してなり、かつ、該hT1R2をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有する甘味受容体発現コンストラクト(C)(図2(C)参照)を、以下の手順に従って作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R3をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R3をコードするcDNAを増幅した。
次いで、得られたhT1R3をコードするcDNA断片を、制限酵素Asc I及びNot Iで消化し、また、pEAK10(Edge Biosystems)を、制限酵素Asc I及びNot Iで消化した。これらの制限酵素消化産物を、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、hT1R3をコードするcDNAを持つpEAK10(Edge Biosystems)を得た。
そして、このpEAK10(Edge Biosystems)を、制限酵素Hind III及びNot Iで消化し、アガロース電気泳動により、DNA断片を分離して、hT1R3のcDNA断片を精製した。
次いで、このhT1R3をコードするcDNA断片と、Hind III及びNot Iで消化したpcDNA5/FRT(Invitrogen)とを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させることにより、マルチクローニングサイトに、hT1R3をコードするcDNAを持つpcDNA5/FRT(Invitrogen)を作製した。
TC)を設計、作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅した。
次いで、hT1R2のコード領域の直前及び直後に、それぞれAsc Iの認識配列(5’-GGCGCGCC-3’)及びNot Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー(配列番号21:GATCGGCGCGCCGCCATGGGGCCCAGGGCAAAG)及びアンチセンスプライマー(配列番号22:GATCGCGGCCGCCTAGTCCCTCCTCATGGT)を設計、作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R2をコードするDNA配列を含む配列をテンプレートとしてPCRを行い、hT1R2をコードするcDNAを増幅した。得られたhT1R2をコードするcDNAを、Asc I及びNot Iで消化し、pEAK10(Edge Biosystems)を、Asc I及びNot Iで消化して、これらをLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hT1R2をコードするcDNAを挿入した。
次いで、hT1R2の開始コドンを含む18塩基のセンスプライマー(配列番号23: ATGGGGCCCAGGGCAAAG)及びhGH pA配列中の配列を持つアンチセンスプライマー(配列番号24:CTGGATGCAGGCTACTCTA)を設計、作製した。
そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、hT1R2/pEAK10をテンプレートとして、PCRを行い、hT1R2をコードするcDNAを増幅した。
次いで、前述したIRES配列を、Eco52Iで消化し、また、hT1R2をコードするcDNAを、Not Iで消化し、また、pBluescript II SK(-)を、Not Iで消化して、これらの制限酵素消化産物をLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、IRES2-hT1R2/pBluescript II SK(-)を作製した。
そして、IRES2-hT1R2配列と、Not Iで消化したhT1R3をコードするcDNAを持つpcDNA5/FRT(Invitrogen)とを、Ligation
high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、hT1R3-IRES2-hT1R2配列を含むpcDNA5/FRTを得た。
TC)を設計、作製した。そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、pIRES2-EGFP(Clontech)をテンプレートとしてPCRを行い、IRES配列を増幅した。
次いで、hG16gust44のコード領域の直前及び直後に、Not Iの認識配列(5’-GCGGCCGC-3’)を持つようなセンスプライマー(配列番号27:GATCGCGGCCGCATGGCCCGCTCGCTGACC)、アンチセンスプライマー(配列番号28:GATCGCGGCCGCGAATTCACTAGTGATTTA)を設計、作製した。hG16gust44をコードするcDNA配列を含む配列をテンプレートとしてPCRを行い、hG16gust44をコードするcDNAを増幅した。増幅断片をNot Iで消化し、pEAK10(Edge Biosystems)をNot Iで消化して、これらをLigation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、pEAK10(Edge Biosystems)に、hG16gust44をコードするcDNAを挿入したベクター(hG16gust44/pEAK10)を作製した。
次いで、hG16gust44の開始コドンを含む18塩基のセンスプライマー(配列番号29:ATGGCCCGCTCGCTGACC)及びhGH pA配列中の配列を持つアンチセンスプライマー(配列番号30:CTGGATGCAGGCTACTCTA)を設計、作製した。そして、これらのセンスプライマー及びアンチセンスプライマーを用いて、上記hG16gust44/pEAK10をテンプレートとして、PCRを行い、hG16gust44をコードするcDNAを増幅した。
次いで、作製したIRES配列を、Eco52Iで消化し、また、hG16gust44をコードするcDNAを、Not Iで消化し、また、pBluescript II SK(-)を、Not Iで消化し、これらの制限酵素消化産物を、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、IRES2-hG16gust44配列を有するベクター(IRES2-hG16gust44/pBluescript II SK(-))を作製した。
そして、hT1R3-IRES2-hT1R2配列を含むpcDNA5/FRTのhT1R2をコードするcDNAの直後に存在する部位をNot Iで消化した。この制限酵素消化産物とIRES2-hG16gust44配列とを、Ligation high Ver.2(TOYOBO)を用いて、ライゲーション反応によって結合させて、hT1R3-IRES2-hT1R2配列を含むpcDNA5/FRTのhT1R2をコードするcDNAの直後に、IRES2-hG16gust44配列を挿入することにより、甘味受容体発現コンストラクト(C)を作製した。得られた甘味受容体発現コンストラクト(C)は、DNAシーケンシングにより、塩基配列に誤りがないことを確認した。
ヒト甘味受容体(hT1R2+hT1R3)とhG16gust44を発現させた培養細胞株(A)〜(C)の作製
実施例1で作製した甘味受容体発現コンストラクト(A)0.8μgと、pOG44 7.2μgとをリポフェクション法で、200万個のFlp-In-293細胞(Invitrogen)にトランスフェクションして48時間経過後、ハイグロマイシンB添加(100μg/ml)培地でスクリーニングを行い、安定発現株である培養細胞株(A)を取得した。
次いで、ハイグロマイシンBに対して生存した細胞を増殖させ、その一部をゼオシン添加(100μg/ml)培地で培養して、ゼオシン(100μg/ml)によって細胞が死滅していたことから、培養細胞株(A)には、ヒト甘味受容体(hT1R2+hT1R3)及びhG16gust44の遺伝子がFRT部位に導入されていることを確認した。
その後、これらの培養細胞株(A)〜(C)を10%HI-FBS(Heat Inactivated Fetal Bovine Serum)、100μg/mlのハイグロマイシンB(Invitrogen)、L-グルタミン4mMを含む低グルコース(1,000mg/ml)ダルベッコ改変イーグル(DMEM)培地で、37℃で増殖、維持した。
アスパルテーム刺激に対する、実施例4で作製した培養細胞株(A)〜(C)の生理的応答の比較
実施例4で作製した培養細胞株(A)〜(C)のそれぞれに、蛍光カルシウム指示薬Fura-2 AMを負荷して、細胞に取り込ませた。そして、Fura-2 AMを負荷した細胞を340nmと380nmで励起し、510nmにおいて観察される蛍光画像を蛍光顕微鏡ならびにCCDカメラを用いて取得した。画像取得中に細胞に終濃度10mMのアスパルテームを投与し、その後、継続して画像の取得を行った。取得した画像の視野中から細胞100個をランダムに選択し、応答細胞数を計測した。
また、比較として、CMVプロモーターの下流に、hT1R3をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結してなる配列を有するpcDNA5/FRTからなる発現コンストラクト(図10)を用いて、実施例4と同様に培養細胞株(D)を作製し、上記と同様に応答細胞数を計測に供した。
上記計測の結果を図11に示す。この結果からわかるように、培養細胞株(A)については、アスパルテーム投与により、非常に強い細胞応答が観察され、また、培養細胞株(B)、(C)については、培養細胞株(A)に比べて、弱い細胞応答が観察されたのに対し、培養細胞株(D)では、細胞応答が観察されなかった。したがって、培養細胞株(A)〜(C)は、ヒト甘味受容体が機能的に発現していることが確認された。
アスパルテーム刺激に対する、培養細胞株(A)の生理的応答
実施例4で作製した培養細胞株(A)に、蛍光カルシウム指示薬Fura-2 AMを負荷して、細胞に取り込ませた。そして、Fura-2 AMを負荷した細胞を340nmと380nmで励起し、510nmにおいて観察される蛍光画像を蛍光顕微鏡ならびにCCDカメラを用いて取得した。画像取得中に細胞に終濃度0.1mM、0.5mM、1mM、2mM、5mMの各濃度のアスパルテームを投与し、その後、継続して画像の取得を行った。各濃度において最も強い応答が観察された時点の画像を図12に示した。結果からわかるように、投与する甘味料としてアスパルテームを用いた場合、0.1mMから5mMの範囲において濃度依存的な甘味応答が観察された。
スクロース刺激に対する、培養細胞株(A)の生理的応答
実施例4で作製した培養細胞株(A)に、蛍光カルシウム指示薬Fura-2 AMを負荷して、細胞に取り込ませた。そして、Fura-2 AMを負荷した細胞を340nmと380nmで励起し、510nmにおいて観察される蛍光画像を蛍光顕微鏡ならびにCCDカメラを用いて取得した。画像取得中に細胞に終濃度20mM、50mM、100mM、200mM、500mM、1Mの各濃度のスクロースを投与し、その後、継続して画像の取得を行った。各濃度において最も強い応答が観察された時点の画像を図13に示した。
結果からわかるように、投与する甘味料としてスクロースを用いた場合、20mMから1Mの範囲において濃度依存的な甘味応答が観察された。
スクロース刺激に対する、培養細胞株(A)の生理的応答
実施例4で作製した培養細胞株(A)に、蛍光カルシウム指示薬Fura-2 AMを負荷して、細胞に取り込ませた。そして、Fura-2 AMを負荷した細胞を340nmと380nmで励起し、510nmにおいて観察される蛍光画像を蛍光顕微鏡ならびにCCDカメラを用いて取得した。画像取得中に細胞に終濃度500mMのスクロースを投与し、その後、継続して画像の取得を行った。最も強い応答が観察された時点の画像を図14に示した。
また、終濃度が500mMのスクロースと1.25mMのラクチゾールを同時に細胞に投与し、上記と同様にして蛍光画像を取得し、最も強い応答が観察された時点の画像を図14に示した。
結果からわかるように、T1R3インヒビターとして知られるラクチゾールの添加により、細胞の応答が抑制されたことが確認できた。この結果より、スクロースに対する細胞応答は、T1R3サブユニットを介した応答であると推測された。
スクロース刺激に対する、培養細胞株(A)の生理的応答
スクロース刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、スクロースの終濃度が表1に記載された濃度となるように、スクロースを含むHEPESバッファーを添加して、27℃でスクロース刺激を行った。
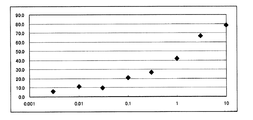
そして、スクロースを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、スクロース刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表1及び図15に示す。図15の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のスクロース濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、スクロースの甘味を評価できることが確認できた。スクロースは甘味の程度が弱いため、50mM程度の濃度で添加しないと甘味受容体細胞の応答は観察されない。しかし、逆に200mMを超える濃度のスクロース溶液を添加すると、細胞が浸透圧の影響を受けるため、正常な細胞応答を測定することができなくなる。したがって、通常、細胞を用いた甘味アッセイ系でスクロースの甘味度を数値化できるのは約50から200mMという狭い範囲に限定されるが、上記細胞系を利用すれば、この限界値の下限を更に低くすることが可能になる。
D-フェニルアラニン刺激に対する、培養細胞株(A)の生理的応答
D-フェニルアラニン刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、D-フェニルアラニンの終濃度が表2に記載された濃度となるように、D-フェニルアラニンを含むHEPESバッファーを添加して、27℃でD-フェニルアラニン刺激を行った。
そして、D-フェニルアラニンを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、D-フェニルアラニン刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表2及び図16に示す。図16の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のD-フェニルアラニン濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、D-フェニルアラニンの甘味を評価できることが確認できた。
アスパルテーム刺激に対する、培養細胞株(A)の生理的応答
アスパルテーム刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、アスパルテームの終濃度が表3に記載された濃度となるように、アスパルテームを含むHEPESバッファーを添加して、27℃でアスパルテーム刺激を行った。
そして、アスパルテームを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、アスパルテーム刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表3及び図17に示す。図17の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のアスパルテーム濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、アスパルテームの甘味を評価できることが確認できた。
サッカリン刺激に対する、培養細胞株(A)の生理的応答
サッカリン刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、サッカリンの終濃度が表4に記載された濃度となるように、サッカリンを含むHEPESバッファーを添加して、27℃でサッカリン刺激を行った。
そして、サッカリンを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、サッカリン刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表4及び図18に示す。図18の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のサッカリン濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、サッカリンの甘味を評価できることが確認できた。
ステビア刺激に対する、培養細胞株(A)の生理的応答
ステビア刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、ステビアの終濃度が表5に記載された濃度となるように、ステビアを含むHEPESバッファーを添加して、27℃でステビア刺激を行った。
そして、ステビアを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、ステビア刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表5及び図19に示す。図19の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のステビア濃度(mg/ml)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、ステビアの甘味を評価できることが確認できた。
ネオヘスペリジンジヒドロカルコン(NHDC)刺激に対する、培養細胞株(A)の生理的応答
ネオヘスペリジンジヒドロカルコン刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、ネオヘスペリジンジヒドロカルコンの終濃度が表6に記載された濃度となるように、ネオヘスペリジンジヒドロカルコンを含むHEPESバッファーを添加して、27℃でネオヘスペリジンジヒドロカルコン刺激を行った。
そして、ネオヘスペリジンジヒドロカルコンを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、ネオヘスペリジンジヒドロカルコン刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表6及び図20に示す。図20の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のネオヘスペリジンジヒドロカルコン濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、ネオヘスペリジンジヒドロカルコンの甘味を評価できることが確認できた。
シクラメート刺激に対する、培養細胞株(A)の生理的応答
シクラメート刺激に対して、培養細胞株(A)の生理的応答を、自動化蛍光イメージングにより解析した。
実施例4で作製した培養細胞株(A)をトリプシナイズし、該DMEM培地に懸濁して細胞密度を計測し、96ウェルプレート(Corning、CellBIND Surface)の各ウェル上に、約8万個ずつ撒いた。
そして、37℃で24時間培養した後、培地を除去し、50μlのHEPESバッファーで置換し、蛍光カルシウム指示薬(Molecular Devices、FLIPR Ca 4 Assay Kitに付属のFluo-4 AM)を含むHEPESバッファーをさらに50μl添加した。次いで、27℃で45分間インキュベートして、自動化蛍光イメージングに供する細胞を調製した。
次いで、この細胞に、シクラメートの終濃度が表7に記載された濃度となるように、シクラメートを含むHEPESバッファーを添加して、27℃でシクラメート刺激を行った。
そして、シクラメートを含むHEPESバッファーの添加直後から100秒後にかけての蛍光反応(485nm励起で525nm蛍光)を、FlexStation 3(Molecular Devices)を用いて測定し、シクラメート刺激に対する甘味受容体発現細胞の応答を自動化蛍光イメージングにより定量した。結果を表7及び図21に示す。図21の縦軸は、刺激直後と刺激後100秒後の間における蛍光強度の変化量の最大値(ΔF)、すなわち、細胞の応答強度であり、横軸は、対数表示のシクラメート濃度(mM)を示す。
得られた結果から、本発明の培養細胞株(A)を用いて、シクラメートの甘味を評価できることが確認できた。
Claims (8)
- 甘味受容体サブユニットT1R2及びT1R3並びにGタンパク質αサブユニットをコードする各遺伝子を同一のプラスミドに挿入してなる甘味受容体発現コンストラクトであって、
EF-1αプロモーターの下流に位置する、甘味受容体サブユニットT1R2をコードする遺伝子の直後に、IRES配列を介して、Gタンパク質αサブユニットをコードする遺伝子を連結し、さらに、その下流に存在するCMVプロモーターの下流に位置する、甘味受容体サブユニットT1R3をコードする遺伝子の直後に、IRES配列を介して、Gタンパク質αサブユニットをコードする遺伝子を連結してなる、甘味受容体発現コンストラクト。 - 甘味受容体サブユニットT1R2及びT1R3並びにGタンパク質αサブユニットをコードする各遺伝子を転写方向が同じ向きになるように、同一のプラスミドに挿入してなる、請求項1に記載の甘味受容体発現コンストラクト。
- プラスミドが、pcDNA5/FRT(Invitrogen)である請求項1又は2に記載の甘味受容体発現コンストラクト。
- Gタンパク質αサブユニットが、hG16gust44である請求項1〜3のいずれか1項に記載の甘味受容体発現コンストラクト。
- EF-1αプロモーターの下流に、hT1R2をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有し、かつ、この配列の下流にあるCMVプロモーターの下流に、hT1R3をコードするcDNAとhG16gust44をコードするcDNAを、IRES配列を挿んで連結した配列を有する甘味受容体発現コンストラクト。
- ゲノム中にFRT(Flippase Recognition Target)配列を1か所組み込まれた293細胞に、請求項1〜5のいずれか1項に記載の甘味受容体発現コンストラクトを遺伝子導入して甘味受容体サブユニットT1R2及びT1R3並びにGタンパク質αサブユニットを同時に発現させた細胞株。
- 甘味物質に対する生理的応答を測定するための請求項6に記載の細胞株の使用。
- 請求項6に記載の細胞株を使用した、甘味物質に対する生理的応答の測定により同定された、特定の甘味物質に対する甘味増強物質を、該甘味物質の生理的応答を測定する際に添加することにより、該甘味物質の甘味の閾値以下の濃度で該甘味物質の生理的応答を測定する方法。
Priority Applications (5)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2009274976A JP5905187B2 (ja) | 2009-12-02 | 2009-12-02 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
| US13/512,683 US8658383B2 (en) | 2009-12-02 | 2010-09-08 | Sweet taste receptor-expressing construct, cell body expressing the same, and utilization thereof |
| EP10834423.5A EP2520647B1 (en) | 2009-12-02 | 2010-09-08 | Sweet taste receptor expression construct, cell body expressing same, and uses therefor |
| CA2781655A CA2781655C (en) | 2009-12-02 | 2010-09-08 | Sweet taste receptor-expressing construct, cell body expressing the same, and utilization thereof |
| PCT/JP2010/065366 WO2011067970A1 (ja) | 2009-12-02 | 2010-09-08 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
Applications Claiming Priority (1)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| JP2009274976A JP5905187B2 (ja) | 2009-12-02 | 2009-12-02 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| JP2011115088A JP2011115088A (ja) | 2011-06-16 |
| JP5905187B2 true JP5905187B2 (ja) | 2016-04-20 |
Family
ID=44114829
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| JP2009274976A Active JP5905187B2 (ja) | 2009-12-02 | 2009-12-02 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
Country Status (5)
| Country | Link |
|---|---|
| US (1) | US8658383B2 (ja) |
| EP (1) | EP2520647B1 (ja) |
| JP (1) | JP5905187B2 (ja) |
| CA (1) | CA2781655C (ja) |
| WO (1) | WO2011067970A1 (ja) |
Families Citing this family (5)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| JP5905187B2 (ja) | 2009-12-02 | 2016-04-20 | 国立大学法人 東京大学 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
| JP6755640B2 (ja) | 2014-03-28 | 2020-09-16 | 味の素株式会社 | 甘味受容体キメラタンパク質及びその利用 |
| JP7173980B2 (ja) * | 2017-10-23 | 2022-11-17 | 日本たばこ産業株式会社 | Gタンパク質共役受容体若しくはそのサブユニットを発現するためのコンストラクト及びその利用 |
| CR20210617A (es) | 2019-05-09 | 2022-05-03 | Gyrus Acmi Inc D/B/A Olympus Surgical Tech America | Sistema y métodos electroquirúrgicos |
| CN116554350B (zh) * | 2023-04-26 | 2024-01-09 | 之江实验室 | 基于人类甜味受体蛋白的生物传感器及其应用 |
Family Cites Families (15)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US20050085625A1 (en) * | 2000-10-30 | 2005-04-21 | Senomyx, Inc. | Chimeric Galpha 15 variants and their use in the analysis and discovery of modulators of G-protein coupled receptors |
| US7368285B2 (en) | 2001-03-07 | 2008-05-06 | Senomyx, Inc. | Heteromeric umami T1R1/T1R3 taste receptors and isolated cells that express same |
| US6955887B2 (en) | 2001-03-30 | 2005-10-18 | Senomyx, Inc. | Use of T1R hetero-oligomeric taste receptor to screen for compounds that modulate taste signaling |
| DE10143237A1 (de) * | 2001-09-04 | 2003-03-20 | Icon Genetics Ag | Herstellung künstlicher interner ribosomaler Eingangsstellenelemente (Ires-Elemente) |
| US7297536B2 (en) * | 2003-01-23 | 2007-11-20 | Wisconsin Alumni Research Foundation | Inducible protein expression system |
| WO2005015158A2 (en) | 2003-08-06 | 2005-02-17 | Senomyx Inc. | T1r hetero-oligomeric taste receptors, cell lines that express said receptors, and taste compounds |
| US7906627B2 (en) | 2003-08-06 | 2011-03-15 | Senomyx, Inc. | Chimeric human sweet-umami and umami-sweet taste receptors |
| US7531523B2 (en) * | 2005-02-17 | 2009-05-12 | Vertex Pharmaceuticals Incorporated | Sodium channel protein type III alpha-subunit splice variant |
| CA2952660C (en) * | 2005-10-20 | 2018-09-11 | Senomyx, Inc. | Chimeric human sweet-umami and umami-sweet taste receptors |
| WO2007121604A2 (en) * | 2006-04-20 | 2007-11-01 | Givaudan Sa | Method relating to sweetness enhancement |
| EP1865316B1 (en) | 2006-06-07 | 2010-02-24 | Nutrinova Nutrition Specialties & Food Ingredients GmbH | Screening methods for compounds that modulate the activity of G-protein coupled receptors |
| EP2166871A4 (en) | 2007-05-14 | 2012-01-25 | TASTE EXHAUSTING COMPOSITIONS IN ORAL DELIVERY SYSTEMS | |
| WO2009026389A2 (en) * | 2007-08-20 | 2009-02-26 | Mount Sinai School Of Medicine | Regulating glp-1 and sglt-1 in gastrointestinal cells |
| US20120245213A1 (en) | 2009-10-01 | 2012-09-27 | Bedrich Mosinger | Human type i taste receptor subunit 3 modulators and methods of using same |
| JP5905187B2 (ja) | 2009-12-02 | 2016-04-20 | 国立大学法人 東京大学 | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 |
-
2009
- 2009-12-02 JP JP2009274976A patent/JP5905187B2/ja active Active
-
2010
- 2010-09-08 US US13/512,683 patent/US8658383B2/en not_active Expired - Fee Related
- 2010-09-08 EP EP10834423.5A patent/EP2520647B1/en not_active Not-in-force
- 2010-09-08 CA CA2781655A patent/CA2781655C/en not_active Expired - Fee Related
- 2010-09-08 WO PCT/JP2010/065366 patent/WO2011067970A1/ja active Application Filing
Also Published As
| Publication number | Publication date |
|---|---|
| JP2011115088A (ja) | 2011-06-16 |
| EP2520647A1 (en) | 2012-11-07 |
| US20120315652A1 (en) | 2012-12-13 |
| EP2520647A4 (en) | 2013-07-10 |
| EP2520647B1 (en) | 2018-07-04 |
| WO2011067970A1 (ja) | 2011-06-09 |
| US8658383B2 (en) | 2014-02-25 |
| CA2781655C (en) | 2019-01-08 |
| CA2781655A1 (en) | 2011-06-09 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| JP7018000B2 (ja) | 甘味増強に関する方法 | |
| JP5225986B2 (ja) | 核酸、ポリペプチドおよびその利用 | |
| JP2007512227A5 (ja) | ||
| JP2007512227A (ja) | T1rヘテロオリゴマー味覚受容体、この受容体を発現する細胞株、および味覚化合物 | |
| JP5905187B2 (ja) | 甘味受容体発現コンストラクト、これを発現させた細胞体、及びその利用 | |
| US20200247859A1 (en) | Construct for expressing g protein-coupled receptor or subunit thereof, and use of said construct | |
| US20110097741A1 (en) | Partial t1r2 nucleic acid sequence, receptor protein and its use in screening assays | |
| WO2011067971A1 (ja) | 甘味物質に対するヒト甘味受容体作用性甘味調節物質 | |
| WO2006066064A2 (en) | A voltage-gated, ph-sensitive anion channel and its novel splice variant involved in taste sensation |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A621 | Written request for application examination |
Free format text: JAPANESE INTERMEDIATE CODE: A621 Effective date: 20120413 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20120413 |
|
| A131 | Notification of reasons for refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A131 Effective date: 20131206 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20140129 |
|
| A02 | Decision of refusal |
Free format text: JAPANESE INTERMEDIATE CODE: A02 Effective date: 20140804 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20141028 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A821 Effective date: 20141028 |
|
| A521 | Request for written amendment filed |
Free format text: JAPANESE INTERMEDIATE CODE: A523 Effective date: 20160127 |
|
| A61 | First payment of annual fees (during grant procedure) |
Free format text: JAPANESE INTERMEDIATE CODE: A61 Effective date: 20160316 |
|
| R150 | Certificate of patent or registration of utility model |
Ref document number: 5905187 Country of ref document: JP Free format text: JAPANESE INTERMEDIATE CODE: R150 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |
|
| R250 | Receipt of annual fees |
Free format text: JAPANESE INTERMEDIATE CODE: R250 |