ESCRT
ESCRT(endosomal sorting complexes required for transport)は細胞質のタンパク質複合体で、ESCRT-0、ESCRT-I、ESCRT-II、ESCRT-IIIという種類が存在する。これらのESCRT複合体は、他の補助タンパク質とともに独特な形式で細胞膜のリモデリングを行い、膜を屈曲させたり切り離したりする[1][2]。酵母やヒトを含む多くの生物で、ESCRTの構成要素の単離と研究が行われている[3]。
ESCRTは、多胞体(multivesicular body)の生合成、細胞質分裂、ウイルスの出芽など、多数の細胞過程に重要な役割を果たす。多胞体の生合成とは、ユビキチンでタグ付けされたタンパク質が小胞形成を介してエンドソームと呼ばれる細胞小器官へ入る過程である。この過程は、誤ったフォールディングをしたタンパク質や損傷したタンパク質を破壊するために必須の過程である[4]。ESCRTがなければこれらのタンパク質は蓄積し、神経変性疾患が引き起こされる。例えば、ESCRT-IIIの構成要素の異常は遺伝性痙性対麻痺などの神経疾患を引き起こす可能性がある[5]。細胞のabscission(切断、分離)は膜が連結されている2つの娘細胞を切り離す過程であり、これもESCRTによって媒介される。ESCRTがなければ娘細胞は分離することができず、2倍量のDNAを含む異常な細胞が形成される。このような細胞はアポトーシスによって破壊される。最後に、ウイルスの出芽は特定のタイプのウイルスが細胞を出る過程であり、ESCRTがなければ起こらない。その結果、ウイルスは細胞から細胞へ拡散することができなくなる。
多胞体の生合成と積み荷のシャトリング
[編集]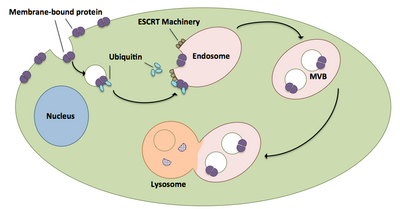
多胞体は、ユビキチン化されたタンパク質と受容体のリソソームへの輸送に大きな役割を果たす[6]。ESCRT複合体は、ユビキチン化された積み荷をエンドソーム区画内に直接出芽する小胞へ運搬し、多胞体を形成する[6]。最終的に多胞体はリソソームと融合し、積み荷の分解を引き起こす[7]。より詳細な過程は次のようになる。
- ESCRT-0複合体の構成要素であるVps27とHse1がユビキチン化された積み荷に結合する[1][6]。
- Vps27がエンドソームの脂質ホスファチジルイノシトール-3-リン酸に結合し、複合体全体がエンドソームへリクルートされる[1][6]。
- Vps27がESCRT-IのVps23サブユニットに結合し、ESCRT-Iがエンドソームへもたらされる。ESCRT-Iはユビキチン化されたタンパク質にも結合する[1][6]。
- Vps36がESCRT-IのサブユニットVps28に結合し、ESCRT-II複合体がリクルートされる[1]。
- ESCRT-IIのVps25サブユニットがESCRT-III複合体のVps20に結合し、活性化する[1][6][7]。
- Vps20がSnf7多量体鎖の形成核となり、その後Vps24によってキャップされる[7]。
- Vps24がVps2をリクルートし、Vps4が複合体へもたらされる[7]。
- Vps4は、Vta1の結合に伴って2つの六量体リングからなるポアを形成する[1]。Vps4-Vta1複合体は、ESCRT-IIIの解体を開始し、多胞体形成の終結の印となる[2]。
ESCRT複合体と補助タンパク質
[編集]
各ESCRT複合体と補助タンパク質は、異なる生化学的機能を可能にする独特の構造を持っている。ESCRTを構成する各タンパク質には、酵母と後生動物の双方で多数のシノニムが存在する。これらのタンパク質をすべてまとめた表が右に示されている。
酵母では、次に挙げる複合体または補助タンパク質が存在している。
ESCRT-0
[編集]ESCRT-0複合体は、ユビキチン化されたタンパク質や細胞表面の受容体に結合して密集することで、多胞体の形成に重要な役割を果たす。その後複合体はエンドソームの膜脂質への結合を担い、これらタグ付けされたタンパク質をエンドソームへリクルートする[8]。適切な局在化が行われると、これらのタンパク質は小胞を介してエンドソームへ取り込まれて多胞体を形成し、最終的にリソソームへ運ばれて分解される。この経路は、ゴルジ体を通過した傷害タンパク質の分解の主要な経路である[4]。
ESCRT-0複合体は、Vps27とHse1が1:1で結合したヘテロ二量体である[1][5]。Vps27とHse1は逆平行コイルドコイルを形成するGATドメインを介して二量体化する[1]。Vps27とHse1の双方がN末端にVHSドメインを持っている[9]。これらのVHSドメインは、細胞が分解しようとするタンパク質に付加されたユビキチンに結合する。また、ユビキチンはHse1に1つ、またはVps27の両側面にそれぞれ存在するユビキチン相互作用モチーフにも結合する。Vps27には、VHSドメインとユビキチン相互作用モチーフに挟まれてFYVEドメインが存在する[5][10]。エンドソームの一般的な脂質であるホスファチジルイノシトール-3-リン酸は、このFYVEドメインに結合してESCRT-0をエンドソームへリクルートする[5]。
ESCRT-I
[編集]ESCRT-I複合体の役割は、ユビキチン化されたタンパク質に密集し、ESCRT-0複合体とESCRT-II複合体のブリッジとして機能することで多胞体の形成を助けることである[11]。またESCRT-Iは膜のabscissionの際、分裂している細胞の中央体(midbody)の両側にリングを形成し、膜の認識とリモデリングに機能を果たす。ESCRT-Iは、細胞が分裂する直前に狭窄帯を形成するESCRT-IIIのリクルートも担う[12]。さらに、ESCRT-Iは特定のウイルスタンパク質と相互作用してウイルスの出芽にも関与し、ウイルス放出部位に他のESCRT複合体をリクルートする[13]。
ESCRT-I複合体は、Vps23、Vps28、Vps37、Mvb12が1:1:1:1で結合したヘテロ四量体である[3]。組み立てられたヘテロ四量体は、Vps23、Vps37、Mvb12からなる棒状のストークに、Vps23、Vps28、Vps37のヘリックスからなる扇型のキャップが結合したような形状をしている[3][5]。Vps23はユビキチンE2バリアントドメインを持っており、ユビキチン、ESCRT-0複合体、そして、ウイルスのgagタンパク質のPTAPモチーフとの結合を担う[3][5]。このユビキチンE2バリアントドメインの直後にはプロリンリッチモチーフ(GPPX3Y)が存在しており、膜のabscissionの際にESCRT-Iを中央体へ差し向ける[5]。Mvb12もC末端を介してユビキチンに結合することができる。Vps28はESCRT-IとESCRT-II間の相互作用を担い、C末端の4ヘリックスバンドルドメインを介してESCRT-IIのVps36のGLUEドメインへ結合する[1]。
ESCRT-II
[編集]ESCRT-II複合体は主に、多胞体の生合成とユビキチン化されたタンパク質のエンドソームへの輸送の際に機能する。ユビキチンタグの付いたタンパク質はESCRT-0からESCRT-Iへ、そしてその後ESCRT-IIへと受け渡される。ESCRT-IIは、積み荷を含む小胞をくびり切る複合体ESCRT-IIIと結合する[5]。
ESCRT-IIは、2つのVps25サブユニット、1つのVps22サブユニット、そして1つのVps36サブユニットからなるヘテロ四量体である[3]。Vps25のPPXYモチーフはVps22とVps26のwinged-helix(WH)モチーフと結合し、Vps22とVps36が基部、2つのVps25分子がアームとなるY字型の複合体を形成する[3][5]。Vps25もWHモチーフを含んでおり、ESCRT-IIとESCRT-IIIの間の相互作用を担う。Vps36はGLUEドメインを含んでおり、ホスファチジルイノシトール-3-リン酸とESCRT-IのVps28に結合する[3][5]。酵母のVps36では、GLUEドメイン内のループには2つのジンクフィンガードメインが存在している。これらのジンクフィンガードメインの一方はVps28のC末端ドメインを、もう一方はユビキチンを結合する[5]。
ESCRT-III
[編集]ESCRT-III複合体はESCRTが媒介する全ての過程に関与し、おそらく最も重要である[14]。膜のabscissionとウイルスの出芽において、膜の切断の直前にESCRT-IIIは膜の狭窄部位に巻き付く長いフィラメントを形成する[12][15]。膜のabscissionの媒介は、セントラルスピンドリン複合体との相互作用によって行われる[16]。ESCRT-IIIのフィラメント構造は多胞体の形成時にも存在し、積み荷タンパク質が細胞質へ流出するのを防ぐために出芽小胞に栓をするリング状の柵として機能する[12]。
ESCRT-III複合体は一過的にしか存在しないという点で他のESCRT複合体をは異なっており、必須サブユニットと非必須サブユニットを含んでいる[1][12]。複合体が機能するためには、必須サブユニットは適切な順序(Vps20、Snf7、Vps24、その後にVps2)で集合しなければならない[5]。非必須サブユニットにはVps60、Did2、Ist1が含まれる[12]。Vps20は、Snf7の多量体化の核として機能することでESCRT-IIIの組み立てを開始する。その後Vps24がSnf7に結合し、複合体のキャップとなってVps2をリクルートする[1][3]。Vps2はその後Vps4を複合体へリクルートする[7]。細胞質の遊離型の各サブユニット単量体は「閉じた」構造であると考えられている。すなわち、各サブユニットのC末端部分が自身の上に折り畳まれ、自己阻害的に単量体サブユニットを安定化している[1][3]。ESCRT-IIIのサブユニットのC末端は、必須サブユニットも非必須サブユニットも、MIMモチーフを含んでいる[17]。このモチーフはVps4やAAA-ATPアーゼのスパスチンとの結合を担う[3]。
Vps4-Vta1
[編集]Vps4-Vta1タンパク質複合体は、特定の過程の完了後に膜からESCRT構成要素(通常はESCRT-III)を除去する際に必要とされる。Vps4がESCRT-III複合体を切断するのか、それとも構成要素が特定の時期に切断されるよう複合体をリモデリングするのかについては議論がある[13]。Vta1はVps4の活性化因子として機能し、組み立てを補助してAAA-ATPアーゼ活性を向上させると考えられている[14][18]。
Vps4サブユニットは2つの機能的ドメイン、N末端のMITドメインと中心部のAAA-ATPアーゼドメインを持っている[3]。MITドメインはVps2のMIMモチーフとの相互作用を担う[1]。AAA-ATPアーゼドメインは、ATPを加水分解してESCRT-III複合体の解体の動力となる[12]。ESCRT-IIIは除去されて、全ての結合サブユニットは再利用される[12][13]。Vta1は二量体タンパク質で、VSLドメインがVps4との結合を担い、MITドメインがESCRT-IIIのサブユニットVps60に結合する。Vta1は必須ではないものの、Vps4のリング状構造の組み立てを助け、ATPアーゼ活性を促進し、ESCRT-IIIの解体を促進することが示されている[5]。
Bro1
[編集]Bro1の主要な機能は、脱ユビキチン化酵素をESCRT-III複合体へリクルートすることである[19]。その結果、リソソームでの分解標的となったタンパク質からユビキチンタグが多胞体の形成の直前に除去される。Bro1は積み荷タンパク質からユビキチンが除去される間、ESCRT-IIIの安定化を助けていると想定されている[19]。
Bro1のN末端ドメインはESCRT-IIIのSnf7に結合する[20]。この結合によってBro1は膜のabscission部位にリクルートされる。Bro1は脱ユビキチン化酵素Doa4の触媒ドメインにも結合し、Doa4を膜のabscission部位へリクルートする。Doa4はリソソームへ送られる積み荷タンパク質からユビキチンを除去する[20]。
膜のabscission
[編集]
細胞質分裂における膜のabscissionは、細胞分裂の際に2つの娘細胞を連結している膜が切断される過程である。この過程は真核生物に近い一部の古細菌でも保存されており、ESCRTの最古の役割であると考えられている[5]。2分岐説に従えば、プロテオ古細菌の祖先がESCRTを獲得し、アスガルド古細菌段階でESCRTシリーズをほぼ全て備えた。
この過程は、中心体タンパク質Cep55が、微小管と結合する有糸分裂キネシン様タンパク質MKLP1との結合によって、分裂中の細胞の中央体へリクルートされることで開始される[5][22]。その後、Cep55はESCRT-IのVps23サブユニットと補助タンパク質ALIXをリクルートし、中央体の両側にリングを形成する[5][12][13]。ESCRT-IとALIXはSnf7サブユニットを介してESCRT-IIIをリクルートする[5]。ESCRT-IIIのサブユニットVps20、Snf7、Vps24、Vps2、Did2は、Vps23によって形成されたリングに近接してらせん状の繊維を形成する[1][13][19]。このらせん状構造の形成によって膜が変形し、中央体に形成された微小管を切断するためにAAA-ATPアーゼのスパスチンがDid2とIst1によってリクルートされる[13][19]。その後、Vps4がESCRT-III複合体の解体を触媒し、新たに分離した2つの娘細胞となる[19]。膜のabscissionの過程は後生動物でより詳細に研究されているため、後生動物のタンパク質名で記している。
ウイルスの出芽
[編集]
ウイルスの出芽としても知られるウイルス粒子放出過程は、宿主のESCRTを乗っ取って細胞内から遊離ビリオンが放出される過程である。HIV-1やヒトTリンパ好性ウイルスなどのレトロウイルスや、エボラウイルスなどエンベロープを持つウイルスの多くは、宿主細胞から出るためにESCRTを必要とする[1]。この過程は、レトロウイルスの被覆の主要な構造タンパク質であるGagタンパク質によって開始され、ESCRT-I複合体のTSG101、補助タンパク質ALIXと相互作用する[13][14]。ESCRT-IIIのサブユニット(CHMP4とCHMP2のみが必須である[11])がウイルスの出芽部位にリクルートされ、細胞質分裂中の膜のabscissionと似た過程で、出芽部分のネックを狭窄し切断する[1][5][13]。その後Vps4がESCRT-III構成要素を細胞質へリサイクルし、ウイルスは細胞から放出される[5]。ウイルスの出芽の過程は後生動物でより詳細に研究されているため、後生動物のタンパク質名で記している。
出典
[編集]- ^ a b c d e f g h i j k l m n o p q r “The ESCRT machinery”. Curr. Biol. 22 (4): R116–20. (February 2012). doi:10.1016/j.cub.2012.01.028. PMC 3314914. PMID 22361144.
- ^ a b Babst M (August 2011). “MVB vesicle formation: ESCRT-dependent, ESCRT-independent and everything in between”. Curr. Opin. Cell Biol. 23 (4): 452–7. doi:10.1016/j.ceb.2011.04.008. PMC 3148405. PMID 21570275.
- ^ a b c d e f g h i j k l “Membrane budding and scission by the ESCRT machinery: it's all in the neck”. Nat. Rev. Mol. Cell Biol. 11 (8): 556–66. (August 2010). doi:10.1038/nrm2937. PMC 2922035. PMID 20588296.
- ^ a b “Biogenesis and function of multivesicular bodies”. Annu. Rev. Cell Dev. Biol. 23: 519–47. (2007). doi:10.1146/annurev.cellbio.23.090506.123319. PMC 2911632. PMID 17506697.
- ^ a b c d e f g h i j k l m n o p q r s t Hurley JH (December 2010). “The ESCRT complexes”. Crit. Rev. Biochem. Mol. Biol. 45 (6): 463–87. doi:10.3109/10409238.2010.502516. PMC 2988974. PMID 20653365.
- ^ a b c d e f “The ESCRT complexes: structure and mechanism of a membrane-trafficking network”. Annu Rev Biophys Biomol Struct 35: 277–98. (2006). doi:10.1146/annurev.biophys.35.040405.102126. PMC 1648078. PMID 16689637.
- ^ a b c d e “ESCRT-II coordinates the assembly of ESCRT-III filaments for cargo sorting and multivesicular body vesicle formation”. EMBO J. 29 (5): 871–83. (March 2010). doi:10.1038/emboj.2009.408. PMC 2837172. PMID 20134403.
- ^ “Molecular mechanism of multivesicular body biogenesis by ESCRT complexes”. Nature 464 (7290): 864–9. (April 2010). Bibcode: 2010Natur.464..864W. doi:10.1038/nature08849. PMC 2851844. PMID 20305637.
- ^ “VHS domains of ESCRT-0 cooperate in high-avidity binding to polyubiquitinated cargo”. EMBO J. 29 (6): 1045–54. (March 2010). doi:10.1038/emboj.2010.6. PMC 2845278. PMID 20150893.
- ^ “Comparative genomics reveals selective distribution and domain organization of FYVE and PX domain proteins across eukaryotic lineages”. BMC Genomics 11: 83. (2010). doi:10.1186/1471-2164-11-83. PMC 2837644. PMID 20122178.
- ^ a b “ESCRT-III protein requirements for HIV-1 budding”. Cell Host Microbe 9 (3): 235–42. (March 2011). doi:10.1016/j.chom.2011.02.004. PMC 3070458. PMID 21396898.
- ^ a b c d e f g h “Assembly and disassembly of the ESCRT-III membrane scission complex”. FEBS Lett. 585 (20): 3191–6. (October 2011). doi:10.1016/j.febslet.2011.09.001. PMC 3192940. PMID 21924267.
- ^ a b c d e f g h “Membrane abscission: first glimpse at dynamic ESCRTs”. Curr. Biol. 22 (15): R603–5. (August 2012). doi:10.1016/j.cub.2012.06.063. PMC 3414845. PMID 22877781.
- ^ a b c “No strings attached: the ESCRT machinery in viral budding and cytokinesis”. J. Cell Sci. 122 (Pt 13): 2167–77. (July 2009). doi:10.1242/jcs.028308. PMC 2723143. PMID 19535732.
- ^ “Dynamics of ESCRT protein recruitment during retroviral assembly”. Nat. Cell Biol. 13 (4): 394–401. (April 2011). doi:10.1038/ncb2207. PMC 3245320. PMID 21394083.
- ^ Glotzer, Michael. "Cytokinesis: Centralspindlin Moonlights as a Membrane Anchor", Current Biology, 18 February 2013
- ^ “Structure and ESCRT-III protein interactions of the MIT domain of human VPS4A”. Proc. Natl. Acad. Sci. U.S.A. 102 (39): 13813–8. (September 2005). Bibcode: 2005PNAS..10213813S. doi:10.1073/pnas.0502165102. PMC 1236530. PMID 16174732.
- ^ “Recycling of ESCRTs by the AAA-ATPase Vps4 is regulated by a conserved VSL region in Vta1”. J. Cell Biol. 172 (5): 705–17. (February 2006). doi:10.1083/jcb.200508166. PMC 2063703. PMID 16505166.
- ^ a b c d e “Regulation of Vps4 during MVB sorting and cytokinesis”. Traffic 12 (10): 1298–305. (October 2011). doi:10.1111/j.1600-0854.2011.01230.x. PMC 3171586. PMID 21658171.
- ^ a b “Bro1 binding to Snf7 regulates ESCRT-III membrane scission activity in yeast”. J. Cell Biol. 192 (2): 295–306. (January 2011). doi:10.1083/jcb.201007018. PMC 3172170. PMID 21263029.
- ^ Carmena M (July 2012). “Abscission checkpoint control: stuck in the middle with Aurora B”. Open Biol 2 (7): 120095. doi:10.1098/rsob.120095. PMC 3411112. PMID 22870391.
- ^ “Recruitment of MKLP1 to the spindle midzone/midbody by INCENP is essential for midbody formation and completion of cytokinesis in human cells”. Biochem. J. 389 (Pt 2): 373–81. (July 2005). doi:10.1042/BJ20050097. PMC 1175114. PMID 15796717.