タンパク質凝集
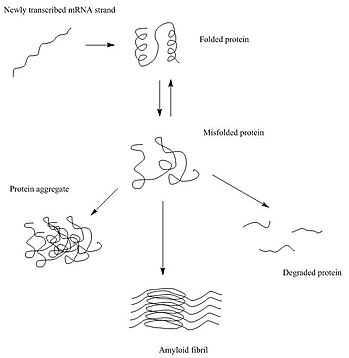
タンパク質の凝集(ぎょうしゅう、英: aggregation)は、天然変性タンパク質やミスフォールドタンパク質(誤ったフォールディングを行ったタンパク質)が細胞内や細胞外に蓄積して塊(凝集体)を形成する生物学的現象である[1][2]。ミスフォールドタンパク質の凝集体は疾患と関係していることが多い。タンパク質の凝集はALS、アルツハイマー病、パーキンソン病、プリオン病を含む、アミロイドーシスとよばれる広範囲の疾患との関係が示唆されている[3][4]。
タンパク質は合成後、熱力学的に最も有利な三次元コンフォメーションへとフォールディングし、その状態はネイティブ状態と呼ばれる[5]。このフォールディング過程は疎水効果によって駆動される。タンパク質の疎水的部分は自身をタンパク質の内部に埋め込むことで細胞の親水的環境から自身を隠す傾向がある。そのため、一般的にはタンパク質の外部は親水的となり、内部は疎水的となる。
タンパク質の構造は非共有結合的相互作用や、2つのシステイン残基の間のジスルフィド結合によって安定化される。非共有結合的相互作用には、イオン性相互作用や弱いファンデルワールス相互作用が含まれる。イオン性相互作用はアニオンとカチオンの間で形成され、タンパク質の安定化を助ける塩橋を形成する。ファンデルワールス相互作用には非極性相互作用(ロンドン分散力など)と極性相互作用(水素結合や双極子間相互作用など)が含まれる。これらはタンパク質の二次構造(αヘリックスやβシートなど)や三次構造の形成に重要な役割を果たしている。タンパク質内のアミノ酸残基間の相互作用はタンパク質の最終的な構造に非常に重要である。
アミノ酸配列の変化などによって非共有結合的相互作用に変化が生じた場合には、タンパク質はミスフォールディングやアンフォールディング(フォールディングの解消)を起こしやすくなる。こうした場合、細胞がタンパク質のリフォールディング(再フォールディング)の補助やアンフォールドタンパク質の分解を行わなければ、アンフォールド/ミスフォールドタンパク質は露出した疎水的部分を介して他のタンパク質の露出した疎水的部分と相互作用して凝集する可能性がある[6][7]。タンパク質凝集体には、アモルファス凝集体(不定形凝集体、amorphous aggregates)、オリゴマー、アミロイド線維という3つの主要なタイプが存在する[8]。
原因
[編集]タンパク質の凝集はさまざまな原因で生じる。ここではその原因を4つに分類して詳細を記載する。
突然変異
[編集]DNA配列に生じた突然変異はタンパク質のアミノ酸配列に影響を与える場合(ミスセンス変異)と与えない場合(サイレント変異)がある。アミノ酸配列が影響を受ける場合、アミノ酸の変化によって側鎖間の相互作用が変化し、タンパク質のフォールディングに影響が生じる可能性がある。その結果タンパク質の疎水性領域が露出し、同じミスフォールド/アンフォールドタンパク質どうし、または異なるタンパク質間での相互作用により、凝集体が形成される。
タンパク質の凝集は、凝集体を形成するタンパク質自体の変異以外にも、リフォールディング経路(分子シャペロン)やユビキチン-プロテアソーム経路(ユビキチンリガーゼ)などの調節経路のタンパク質の変異によって間接的に引き起こされる場合もある[9]。シャペロンはタンパク質がフォールディングを行う安全な環境を提供し、タンパク質のリフォールディングを助ける。ユビキチンリガーゼはタンパク質をユビキチン化修飾することでタンパク質分解の標的とする。
タンパク質合成の問題
[編集]タンパク質の凝集は転写や翻訳時の問題によっても引き起こされる。転写時にDNAはRNAにコピーされ、pre-mRNA鎖はプロセシングを受けてmRNAとなる[10]。翻訳時には、リボソームとtRNAがmRNA配列からアミノ酸配列への翻訳を助ける[10]。これらの段階のいずれかに問題が生じた場合、不適切なmRNAや不適切なアミノ酸配列を有するタンパク質が形成され、ミスフォールディングやタンパク質の凝集が引き起こされる可能性がある。
環境ストレス
[編集]極度の温度やpHの変化、酸化ストレスなどの環境ストレスもタンパク質の凝集をもたらす[11]。こうした疾患の例としてはクリオグロブリン血症が挙げられる。
極度の温度変化はアミノ酸残基間の非共有結合的相互作用を弱めて不安定化する。pHの変化もアミノ酸のプロトン化状態を変化させ、非共有結合的相互作用を強めたり弱めたりする。その結果、相互作用の安定性が低下し、タンパク質のアンフォールディングが引き起こされることがある。
酸化ストレスは活性酸素種などのラジカルによって引き起こされる場合がある。こうした不安定なラジカルはアミノ酸残基を攻撃し、側鎖(芳香族アミノ酸やメチオニンの側鎖)の酸化やペプチド結合の切断を引き起こす[12]。こうした攻撃はタンパク質を適切に保持している非共有結合的相互作用にも影響を与え、タンパク質の不安定化やアンフォールディングが引き起こされる場合がある[11]。
老化
[編集]細胞にはタンパク質凝集体をリフォールディングしたり分解したりする機構が存在する。しかしながら、老化によってこうした制御機構は弱くなり、細胞は凝集体を解消することが難しくなる[11]。
タンパク質の凝集が老化の原因となる過程であるという仮説に関して、現在では老化遅延生物モデルがいくつか開発されており、それらを用いた検証がなされている。タンパク質凝集体の形成が老化とは無関係な過程である場合、老化は経時的なタンパク質毒性に影響を与えないはずである。一方、老化がタンパク質毒性の防止機構の活性低下と関係している場合、老化遅延モデルでは凝集とタンパク質毒性の低下がみられるはずである。この問題に取り組むため、いくつかの毒性アッセイが線虫Caenorhabditis elegansで行われている。こうした研究からは、主要な老化調節経路であるインスリン/IGFシグナル伝達経路の活性の低下によって、神経変性と関係した有毒なタンパク質凝集に対して保護効果がもたらされることが示されている。哺乳類でもアルツハイマー病モデルマウスにおいて、IGF-1シグナル伝達経路の活性の低下によって疾患と関係した行動的・生化学的異常から保護されることが示されており、このアプローチの妥当性は確証されている[13]。
凝集の局在
[編集]タンパク質凝集に対する細胞応答はよく調節され、組織化されたものであることがいくつかの研究から示されている。タンパク質凝集体は細胞内の特定の領域に局在することが知られており、原核生物(大腸菌)や真核生物(酵母、哺乳類細胞)でこうした局在の研究が行われている。
細菌
[編集]細菌では凝集体は非対称的に蓄積し、細胞の"older pole"と呼ばれる極に到達する。細胞分裂後、タンパク質凝集体はolder poleを含む側の娘細胞が受け取り、その生育は凝集体を持たない娘細胞よりも遅くなる。この機構は細菌集団中のタンパク質凝集体を減少させる自然選択機構となっている[14]。
酵母
[編集]
酵母細胞内のタンパク質凝集体の大部分は、分子シャペロンによってリフォールディングされる。しかしながら、酸化損傷したタンパク質や分解標的となったタンパク質など、一部の凝集体はリフォールディングされない。細胞内にはこうした凝集体が到達する2つの区画JUNQとIPODが存在する。酵母細胞内でJUNQ(juxtanuclear quality-control compartment)は核膜に近接して存在し、IPOD(insoluble protein deposit)は液胞に近接して存在する[11]。タンパク質凝集体はユビキチン化され分解標的となった場合にはJUNQに局在する。凝集したタンパク質や不溶性タンパク質はIPODに局在し、より恒久的に蓄積する。この区画のタンパク質はオートファジーによって除去される可能性を示すエビデンスが得られている[15]。これらの2つの経路は協働的にに機能しており、プロテアソーム経路の負荷が高い場合にはタンパク質はIPODに移動する傾向がある[15]。
哺乳類細胞
[編集]哺乳類細胞では、こうしたタンパク質凝集体はアグリソームと呼ばれており、細胞が病的状態となったときに形成される。凝集体は細胞内に異種性タンパク質が存在する場合に形成される傾向があり、こうしたタンパク質は細胞に変異が生じた場合に生じうる。E3ユビキチンリガーゼはミスフォールドタンパク質を認識し、ユビキチン化する。その後、HDAC6がユビキチンとモータータンパク質ダイニンに結合し、ユビキチン標識された凝集体を微小管形成中心(MTOC)へ運ぶ。そこで凝集体はMTOCを囲むように球状にパッキングされる。そしてシャペロンやプロテアソームがそこへ呼び寄せられ、オートファジーが活性化される[16]。
除去
[編集]細胞内にはタンパク質凝集体の除去を担う2つの主要なタンパク質品質管理システムが存在する。ミスフォールドタンパク質はbi-chaperone systemによってリフォールディングされるか、ユビキチンプロテアソームシステムまたはオートファジーによって分解される[17]。
リフォールディング
[編集]Bi-chaperone systemはHsp70システム(大腸菌ではDnaK-DnaJ-GrpE、酵母ではSsa1-Ydj1/Sis1-Sse1/Fes1)とHsp100(大腸菌ではClpB、酵母ではHsp104)シャペロンを利用してタンパク質の脱凝集とリフォールディングを行う[18]。
Hsp70はタンパク質凝集体と相互作用し、Hsp100をリクルートする。Hsp70は活性化されたHsp100を安定化する。Hsp100は芳香族アミノ酸に富むポアループが存在し、スレッディング(糸通し)によってポリペプチドのもつれを解くために利用される。このスレッディング活性はポリペプチドN末端からでもC末端からでも、また中間からでも開始することができる。ポリペプチドは一連の過程によってHsp100の内部を通過し、その各段階でATPが利用される[18]。この過程を経てポリペプチドはアンフォールドされ、その後自発的にまたは他の熱ショックタンパク質の助けのもとでリフォールディングを行う[19]。
分解
[編集]ミスフォールドタンパク質はユビキチンプロテアソームシステムよって除去される場合もある。このシステムはタンパク質をユビキチン化して分解の標識を付加するE1-E2-E3経路から構成される。真核生物では、タンパク質は26Sプロテアソームによって分解される。哺乳類細胞では、E3リガーゼであるCHIPはHsp70に結合したタンパク質を標的とする。酵母では、Doa10とHrd1が小胞体タンパク質に対して類似した機能を果たす[20]。

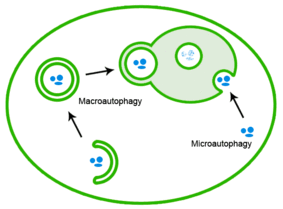
ミスフォールドタンパク質はオートファジーによっても除去される。この過程ではタンパク質凝集体はリソソームへ運ばれる[20]。
毒性
[編集]成熟したタンパク質凝集体はそれ自身が毒性を持つと考えられてきたが、実際に最も毒性が高いのは未成熟のタンパク質凝集体であることを近年のエビデンスは示唆している[21][22]。こうした凝集体の疎水性パッチは細胞の他の構成要素と相互作用し、それらを損傷する。タンパク質凝集体が毒性をもたらす機構としては、細胞の構成要素の隔離、活性酸素種の産生、膜中の特定の受容体への結合、または膜の破壊と関係した機構であるといった仮説が提唱されている[23]。定量的アッセイによって高分子量の分子種が膜の透過化に関与していることが示されており[24]、タンパク質凝集体がin vitroで人工的リン脂質二重膜を不安定化し、膜の透過性をもたらすことが知られている。
出典
[編集]- ^ Aguzzi, A.; O'Connor, T. (March 2010). “Protein aggregation diseases: pathogenicity and therapeutic perspectives.”. Nature Reviews Drug Discovery 9 (3): 237–48. doi:10.1038/nrd3050. PMID 20190788.
- ^ Stefani, M.; Dobson, CM. (November 2003). “Protein aggregation and aggregate toxicity: new insights into protein folding, misfolding diseases and biological evolution.”. J Mol Med (Berl) 81 (11): 678–99. doi:10.1007/s00109-003-0464-5. PMID 12942175.
- ^ De Felice, FG.; Vieira, MN.; Meirelles, MN.; Morozova-Roche, LA.; Dobson, CM.; Ferreira, ST. (July 2004). “Formation of amyloid aggregates from human lysozyme and its disease-associated variants using hydrostatic pressure.”. FASEB J 18 (10): 1099–101. doi:10.1096/fj.03-1072fje. PMID 15155566.
- ^ Tanzi, RE.; Bertram, L. (February 2005). “Twenty years of the Alzheimer's disease amyloid hypothesis: a genetic perspective.”. Cell 120 (4): 545–55. doi:10.1016/j.cell.2005.02.008. PMID 15734686.
- ^ Brüning, Ansgar; Jückstock, Julia (2015-01-01). “Misfolded proteins: from little villains to little helpers in the fight against cancer”. Frontiers in Oncology 5: 47. doi:10.3389/fonc.2015.00047. PMC 4338749. PMID 25759792.
- ^ Gething, MJ.; Sambrook, J. (January 1992). “Protein folding in the cell”. Nature 355 (6355): 33–45. Bibcode: 1992Natur.355...33G. doi:10.1038/355033a0. PMID 1731198.
- ^ Roberts, CJ. (December 2007). “Non-native protein aggregation kinetics”. Biotechnol Bioeng 98 (5): 927–38. doi:10.1002/bit.21627. PMID 17705294.
- ^ Cox, David L.; Nelson, Michael M. (2013). Lehninger Principles of Biochemistry. New York: W.H. Freeman. pp. 143. ISBN 978-1-4292-3414-6
- ^ Berke, Sarah J Shoesmith; Paulson, Henry L (2003-06-01). “Protein aggregation and the ubiquitin proteasome pathway: gaining the UPPer hand on neurodegeneration”. Current Opinion in Genetics & Development 13 (3): 253–261. doi:10.1016/S0959-437X(03)00053-4. PMID 12787787.
- ^ a b Weaver, Robert F. (2012). Molecular Biology. New York: McGraw-Hill. pp. 122–156, 523–600. ISBN 978-0-07-352532-7
- ^ a b c d Tyedmers, Jens; Mogk, Axel; Bukau, Bernd (November 2010). “Cellular strategies for controlling protein aggregation”. Nature Reviews Molecular Cell Biology 11 (11): 777–788. doi:10.1038/nrm2993. PMID 20944667.
- ^ Stadtman, E. R.; Levine, R. L. (2003-07-29). “Free radical-mediated oxidation of free amino acids and amino acid residues in proteins” (英語). Amino Acids 25 (3–4): 207–218. doi:10.1007/s00726-003-0011-2. ISSN 0939-4451. PMID 14661084.
- ^ “The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditiselegans”. PNAS 99 (16): 10417–10422. (2002). Bibcode: 2002PNAS...9910417M. doi:10.1073/pnas.152161099. PMC 124929. PMID 12122205.
- ^ Bednarska, Natalia G.; Schymkowitz, Joost; Rousseau, Frederic; Van Eldere, Johan (2013-01-01). “Protein aggregation in bacteria: the thin boundary between functionality and toxicity”. Microbiology 159 (9): 1795–1806. doi:10.1099/mic.0.069575-0. PMID 23894132.
- ^ a b Takalo, Mari; Salminen, Antero; Soininen, Hilkka; Hiltunen, Mikko; Haapasalo, Annakaisa (2013-03-08). “Protein aggregation and degradation mechanisms in neurodegenerative diseases”. American Journal of Neurodegenerative Disease 2 (1): 1–14. ISSN 2165-591X. PMC 3601466. PMID 23516262.
- ^ Garcia-Mata, Rafael; Gao, Ya-Sheng; Sztul, Elizabeth (2002-06-01). “Hassles with Taking Out the Garbage: Aggravating Aggresomes” (英語). Traffic 3 (6): 388–396. doi:10.1034/j.1600-0854.2002.30602.x. ISSN 1600-0854. PMID 12010457.
- ^ Gregersen, Niels; Bolund, Lars; Bross, Peter (2005-10-01). “Protein misfolding, aggregation, and degradation in disease” (英語). Molecular Biotechnology 31 (2): 141–150. doi:10.1385/MB:31:2:141. ISSN 1073-6085. PMID 16170215.
- ^ a b Mogk, Axel; Kummer, Eva; Bukau, Bernd (2015-01-01). “Cooperation of Hsp70 and Hsp100 chaperone machines in protein disaggregation”. Frontiers in Molecular Biosciences 2: 22. doi:10.3389/fmolb.2015.00022. ISSN 2296-889X. PMC 4436881. PMID 26042222.
- ^ Liberek, Krzysztof; Lewandowska, Agnieszka; Ziętkiewicz, Szymon (2008-01-23). “Chaperones in control of protein disaggregation”. The EMBO Journal 27 (2): 328–335. doi:10.1038/sj.emboj.7601970. ISSN 0261-4189. PMC 2234349. PMID 18216875.
- ^ a b Chen, Bryan; Retzlaff, Marco; Roos, Thomas; Frydman, Judith (2011-08-01). “Cellular Strategies of Protein Quality Control” (英語). Cold Spring Harbor Perspectives in Biology 3 (8): a004374. doi:10.1101/cshperspect.a004374. ISSN 1943-0264. PMC 3140689. PMID 21746797.
- ^ Zhu, YJ.; Lin, H.; Lal, R. (June 2000). “Fresh and nonfibrillar amyloid beta protein(1-40) induces rapid cellular degeneration in aged human fibroblasts: evidence for AbetaP-channel-mediated cellular toxicity”. FASEB J 14 (9): 1244–54. doi:10.1096/fasebj.14.9.1244. PMID 10834946.
- ^ Nilsberth, C.; Westlind-Danielsson, A.; Eckman, CB.; Condron, MM.; Axelman, K.; Forsell, C.; Stenh, C.; Luthman, J. et al. (September 2001). “The 'Arctic' APP mutation (E693G) causes Alzheimer's disease by enhanced Abeta protofibril formation”. Nat Neurosci 4 (9): 887–93. doi:10.1038/nn0901-887. PMID 11528419.
- ^ Soto C (2003). “Unfolding the role of protein misfolding in neurodegenerative diseases”. Nat. Rev. Neurosci 4 (1): 49–60. doi:10.1038/nrn1007. PMID 12511861.
- ^ “Ultrasensitive Measurement of Ca2+Influx into Lipid Vesicles Induced by Protein Aggregates”. Angew. Chem. Int. Ed. Engl. 56 (27): 7750–7754. (2017). doi:10.1002/anie.201700966. PMC 5615231. PMID 28474754.