Entrecruzamento cromosómico



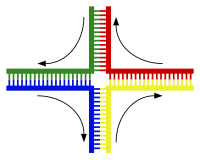
O entrecruzamento cromosómico, tamén chamado sobrecruzamento, crossing over ou crossover, é o intercambio de material xenético entre cromosomas homólogos que orixina cromosomas recombinantes. É unha das fases finais da recombinación xenética, que ten lugar durante o paquiteno da profase I da meiose nun proceso chamado sinapse cromosómica. A sinapse empeza antes de que se forme o complexo sinaptonémico, e non se completa ata case ao final da profase I. O entrecruzamento xeralmente ocorre cando rexións homólogas de cromosomas homólogos rompen e despois reconéctanse pero co outro cromosoma homólogo.
O entrecruzamento foi descrito teoricamente por Thomas Hunt Morgan, que se baseou no descubrimento feito polo profesor belga da Universidade de Lovaina Frans Alfons Janssens que describiu en 1909 un fenómeno cromosómico que chamou "quiasmatipia". O termo quiasma está moi relacioado co entrecruzamento. Morgan decatouse da grande importancia da interpretación citolóxica de Janssens dos quiasmas para explicar os resultados experimentais das súas investigacións sobre a herdanza na mosca Drosophila. A base física do entrecruzamento foi demostrada por Harriet Creighton e Barbara McClintock en 1931.[1]
Bases moleculares
[editar | editar a fonte]A recombinación meiótica iníciase con roturas de dobre cadea no ADN causadas pola proteína Spo11.[2] Unha ou máis exonucleases dixiren despois os extremos 5' xerados polas roturas nas dúas cadeas para formar colas 3' de ADN de cadea simple. A recombinase específica da meiose Dmc1 e a recombinase xeral Rad51 cobre o ADN monocatenario para formar filamentos nucleoproteínicos.[3] As recombinases catalizan a invasión da cromátide oposta polo filamento monocatenario de ADN dun extremo da rotura. Despois, o extremo 3' dos ADN invasores serve de cebador para a síntese de ADN, causando o desprazamento da cadea complementaria, que seguidamente se aparea (anneals) co filamento monocatenario de ADN xerado a partir do outro extremo da rotura bicatenaria inicial. A estrutura que se orixina é un intercambio de filamentos cruzado, tamén chamado unión de Holliday. O contacto entre as dúas cromátides que pouco despois sufrirá o entrecruzamento coñécese como quiasma. A unión de Holliday é unha estrutura tetraédrica que pode ser "empurrada" por outras recombinases, que a moven ao longo da estrutura de catro cadeas.
Consecuencias
[editar | editar a fonte]
Na maioría dos eucariotas, unha célula leva dúas copias de cada xene chamadas alelos. Cada proxenitor pasa un alelo aos seus fillos. Cada gameto herda un complemento haploide completo de alelos nos cromosomas que son seleccionados independentemente de cada par de cromátides que se sitúan na placa metafásica. Sen recombinación, todos os alelos destes xenes que están ligados no mesmo cromosoma herdaríanse sempre xuntos. A recombinación meiótica permite unha selección máis independente entre os dous alelos que ocupan as posicións dun mesmo locus, xa que a recombinación intercambia os alelos entre os cromosomas homólogos.
A recombinación dá lugar a unha nova distribución dos alelos maternos e paternos no mesmo cromosoma. Aínda que aparecen os mesmos xenes na mesma orde, os alelos son diferentes. Deste xeito, é teoricamente posible que se forme calquera combinación de alelos parentais na descendencia, e o feito de que dous alelos aparezan xuntos nun descendente non ten ningunha influencia na probabilidade estatística de que outro descendente teña a mesma combinación. Esta lei da distribución independente dos alelos é un principio fundamental na herdanza xenética.[4]
Porén, a frecuencia de recombinación en realidade non é a mesma para todas as combinacións de xenes. Isto leva á noción de "distancia xenética", que é unha medida da frecuencia de recombinación promediada nunha mostra (grande dabondo) de pedigrís. Grosso modo, pódese dicir que isto se debe a que a recombinación está moi influída pola proximidade entre os xenes. Se dous xenes están situados moi xuntos no cromosoma, a probabilidade de que un evento de recombinación separe estes dous xenes é menor ca no caso de que os xenes estivesen afastados. O ligamento xenético describe a tendencia dos xenes a herdárense xuntos como resultado da súa localización no mesmo cromosoma. O desequilibrio de ligamento describe a situación na cal algunhas combinacións de xenes ou marcadores xenéticos aparecen máis ou menos frecuentemente nunha poboación do que se agardaría da súa distancia de separación. Este concepto aplícase cando se busca un xene no xenoma causante dalgunha determinada doenza. Isto faise comparando a aparición dunha secuencia de ADN específica coa da aparición da doenza. Cando se encontra que hai unha elevada correlación entre ambas as dúas, é probable que a secuencia xénica apropiada estea próxima.[4]
Problemas
[editar | editar a fonte]Aínda que o entrecruzamento ocorre normalmente entre rexións homólogas de cromosomas homólogos, as similitudes na secuencia con outros cromosomas poden dar lugar a apareamentos entre cromosomas de distintos pares cromosómicos. Estes procesos denomínanse recombinación desequilibrada. A recombinación desequilibrada é bastante rara comparada coa normal, pero pode orixinar graves problemas se un gameto que contén recombinantes desequilibrados acaba formando un cigoto. O resultado pode ser unha duplicación xénica nun cromosoma e unha deleción de ditos xenes no outro, ou ben unha translocación cromosómica de parte dun cromosoma noutro diferente, ou unha inversión cromosómica.
Notas
[editar | editar a fonte]- ↑ Creighton H, McClintock B (1931). "A Correlation of Cytological and Genetical Crossing-Over in Zea Mays". Proc Natl Acad Sci USA 17 (8): 492–7. PMC 1076098. PMID 16587654. doi:10.1073/pnas.17.8.492. (Original paper)
- ↑ Keeney, S; Giroux, CN; Kleckner, N (1997). "Meiosis-Specific DNA Double-Strand Breaks Are Catalyzed by Spo11, a Member of a Widely Conserved Protein Family". Cell 88 (3): 375–84. PMID 9039264. doi:10.1016/S0092-8674(00)81876-0.
- ↑ Sauvageau, S; Stasiak, Az; Banville, I; Ploquin, M; Stasiak, A; Masson, Jy (2005). "Fission Yeast Rad51 and Dmc1, Two Efficient DNA Recombinases Forming Helical Nucleoprotein Filaments". Molecular and Cellular Biology 25 (11): 4377–87. ISSN 0270-7306. PMC 1140613. PMID 15899844. doi:10.1128/MCB.25.11.4377-4387.2005. Arquivado dende o orixinal (Free full text) o 28 de maio de 2020. Consultado o 22 de agosto de 2012.
- ↑ 4,0 4,1 http://www.daviddarling.info/encyclopedia/G/genetic_recombination.html